CRISPR-Cas系统在病原核酸检测中的研究进展
2024-03-22杜瑶高宏丹刘家坤刘孝荣邢志浩张涛马东礼
杜瑶,高宏丹,刘家坤,刘孝荣,邢志浩,张涛,马东礼
(1 蚌埠医学院检验医学院,安徽 蚌埠 233030; 2 深圳市儿童医院儿科研究所,广东 深圳 518034; 3 中国科学院深圳先进技术研究所深圳合成生物研究所,广东 深圳 518055; 4 深圳市海微生物科技有限公司,广东 深圳 518057; 5 蚌埠医学院病原生物学教研室,安徽 蚌埠 233030)
病原体(pathogens)指可以侵犯人体,引起感染甚至传染病的微生物,包括病毒、细菌、真菌、寄生虫等,以细菌和病毒的危害性最大[1]。目前,实验室诊断病原体的方法[2]主要有病原体培养、抗原抗体检测、实时荧光定量PCR(quantitative realtime PCR, qPCR)和下一代测序[3](next-generation sequencing, NGS)等,但这些检测技术都存在一定局限性:病原体培养周期长;抗原抗体检测的应用范围有限且灵敏度不高。qPCR和NGS是目前核酸检测最常用和相对理想的方法,但是qPCR对仪器、实验室环境和操作人员的专业技能等的高要求,限制了其在医疗保健环境中的应用, NGS的操作复杂,也使其应用受限。
2019年新型冠状病毒(SARS-CoV-2)肆虐全球[4],造成了全球公共卫生危机,RT-qPCR(quantitative reverse transcription PCR)成为世界卫生组织、中国国家卫生健康委员会等单位推荐的新冠肺炎确诊的标准之一。但是,从大规模的检测结果来看,RT-qPCR具有“假阳性”问题,对于病毒载量较低的样本(如康复期患者),其检出率并不能令人满意[5],因此迫切需要开发灵敏度更高的方法。作为斩获2020年诺贝尔化学奖的CRISPR-Cas系统,不仅可用于基因编辑,还可用于核酸检测,而且基于CRISPR-Cas系统开发的新一代诊断技术,展现出快速、高通量、灵敏度高、特异性好等优势[6-7],而且在资源匮乏地区等特定场景的适用性中,CRISPR-Cas系统具有难以替代的地位。本文主要介绍了CRISPR-Cas系统的生物学机制和分类,侧重总结基于Cas蛋白的反式切割活性建立的不同形式、不同读数的病原核酸检测技术。
1 CRISPR-Cas系统的生物学机制
1987年,CRISPR位点在细菌基因组中被发现[8],2002年CRISPR相关蛋白被发现[9],随后证实CRISPR-Cas系统是一种RNA引导的适应性免疫系统,可以抵御病毒、质粒等入侵遗传元素[10-11],其免疫过程大致可以分为三个阶段:适应、表达和干扰[12-13]。
(1)适应阶段 Cas蛋白识别并捕获外来核酸片段,获取新间隔序列,并整合插入自身CRISPR阵列,形成免疫记忆。
(2)表达阶段 当外来核酸再次入侵时,从CRISPR阵列中相对应的间隔序列,转录出CRISPR RNA(crRNA)前体并加工获得小的、成熟的crRNA,其中包含一个保守的重复序列和一个间隔序列,crRNA进一步与一个或多个Cas蛋白相互作用,形成RNP(ribonucleoprotein)复合体[14]。
(3)干扰阶段 Cas-crRNA复合体识别外来核酸,通过crRNA靶向目标核酸区域,介导Cas蛋白特异性地破坏入侵核酸[15]。
在不同的CRISPR-Cas系统中,参与适应阶段的Cas1和Cas2蛋白,在很大程度上是高度保守的。相比之下,参与表达和干扰阶段的蛋白质,差异很大[16]。
2 CRISPR-Cas系统的分类
CRISPR-Cas系统被划分为2个类别、6个类型、48个亚型[17]。第一个类别可分为3种类型:Ⅰ、Ⅲ和Ⅳ型,利用由多个Cas蛋白和crRNA组成的复合体,切割靶核酸序列[18]。第二个类别也包括三种类型:Ⅱ、Ⅴ和Ⅵ型,具有单一的多功能Cas蛋白和crRNA用于干扰[19],该类系统已被广泛应用于病原体的基因组编辑和核酸检测,特别是CRISPR-Cas9、Cas12和Cas13[20]。
2.1 CRISPR-Cas系统类别Ⅰ
CRISPR-Cas第一类系统的三种类型,具有多种Cas蛋白组成的效应复合物。
CRISPR-CasⅠ型的共享效应模块Cascade(CRISPR-associated complex for antiviral defense),是Cas蛋白与crRNA组成的复合物[21]。Cascade识别Protospacer相邻基序(protospacer adjacent motif,PAM)序列,通过crRNA靶向DNA目标位点,利用Cas3蛋白实现靶位点切割[22]。目前,在已确定的7个亚型(Ⅰ-A到Ⅰ-F和Ⅰ-U)中[23],Ⅰ-E型CRISPR-Cas3也具有反式切割活性[24]。
CRISPR-Cas Ⅲ型的功能基于由crRNA与Ⅲ-A亚型系统中的Csm复合物或Ⅲ-B亚型系统中的Cmr复合物组成的多亚单位复合物,标志性蛋白为Cas10。Ⅲ型整体组成和结构与Ⅰ型效应复合物具有高度相似性[25-26]。Ⅲ型系统的靶位点识别可激活Cas10蛋白的聚合酶活性,然后由Cas10介导产生环状寡核苷酸(cOA),Csm6与cOA的结合可激活Csm6的RNase结构域活性,从而切割靶RNA和其他相邻RNA分子[27]。
CRISPR-Cas Ⅳ型系统被分为Ⅳ-A、Ⅳ-B、Ⅳ-D等亚型,主要存在于质粒中。研究发现Ⅳ型系统对质粒有很强的靶向性,某些质粒可以利用Ⅳ型CRISPR-Cas系统对抗其他质粒对同一宿主菌的竞争,Ⅳ型CRISPR-Cas系统的这一特性可能具有应用于临床耐药基因治疗的潜力[28]。
2.2 CRISPR-Cas系统类别Ⅱ
第二类CRISPR-Cas系统的三种类型,都具有独特的效应蛋白[29]。与第一类系统相比,第二类系统的效应模块仅为一个单一的多结构域、多功能的蛋白。
CRISPR-CasⅡ型Cas9蛋白是一种双RNA引导的DNA内切酶,通过crRNA和反式激活crRNA(tracrRNA)组成的复合物,介导Cas9识别目标DNA上的PAM序列(5′-NGG-3′),然后利用Cas9的HNH和RuvC结构域,切割目标双链DNA,产生平末端双链断裂[19]。最近,Jinek等[30-31]构建的单向导RNA(single-guide RNA, sgRNA),通过将三组分系统(Cas9、crRNA、tracrRNA)减少到两组分(Cas9和sgRNA),简化了CRISPR-Cas基因组编辑[30-31]。
CRISPR-CasⅤ型系统分为Ⅴ-A、Ⅴ-B、Ⅴ-C等亚型,其效应蛋白分别为Cas12a(Cpf1)、Cas12b(C2c1)、Cas12c(C2c3)等[17]。CRISPR-CasⅤ型系统只需要Cas蛋白和crRNA来编辑目标位点,在crRNA识别PAM位点(5′-TTTN-3′)并与目标DNA充分碱基配对后,Cas12a利用RuvC结构域,顺式切割目标序列,产生5′黏性末端的同时,利用反式切割活性,切割任何相邻的单链DNA(singlestranded DNA, ssDNA)[32]。该特性使CRISPRCas12系统成为核酸检测领域研究的热点。最近该系统也被应用于分子标志物检测,如微RNA(microRNA)[33]、心肌肌钙蛋白Ⅰ(cardiac troponinⅠ,cTnⅠ)[34]等,针对该领域的研究对实现精准的体外诊断具有重要意义。此外,Cas12也经常被应用于基因工程[35-36]。Cas14蛋白是CRISPR-CasⅤ型系统中一类结构紧凑的RNA引导的核酸酶(400~700 aa),几乎只出现在DPANN(一种以极小基因组为特征的超门古菌)中[37]。与一些Cas12蛋白相似,Cas14也具有反式切割活性,而且可以在不依赖PAM序列的情况下,靶向切割目标DNA,再反式切割任意相邻单链DNA[38]。与Cas12相比,Cas14对crRNA与靶标模板之间的核苷酸错配的容忍性更低,可实现高保真的单核苷酸多态性(single nucleotide polymorphism, SNP)基因分型[39]。
CRISPR-CasⅥ型系统可分为Ⅵ-A、Ⅵ-B、Ⅵ-C和Ⅵ-D亚型,标志性蛋白是Cas13,具有两个HEPN结构域[40],此系统的独特性在于该蛋白能够识别单链RNA分子靶标。在crRNA的靶向作用下,Cas13-crRNA复合物识别靶标核酸上的Protospacer侧翼位点(Protospacer flanking site,PFS)序列,在切割目标RNA的同时,反式切割单链RNA[41-42]。
综上,CRISPR-Cas系统是多样性的,其关键要素的组成、结构和作用机制各不相同[43]。对CRISPR-Cas系统的深入探索,可能会为开发新的诊断平台提供方向。
3 基于反式切割活性的病原核酸检测
CRISPR-Cas最初作为基因编辑系统[44],在合成生物学领域被广泛研究与应用,根据不同蛋白的生物学功能和特性,开发和设计了大量新的基因编辑元件、工具等[45]。2016年,CRISPR-Cas系统首次被应用于核酸检测[46],在随后的研究与开发中展现出高效、精准的病原核酸检测。而合成生物学作为一门新兴交叉学科,专注于设计生物分子或生物系统[47],这也为分子诊断提供了新思路和新机会[48-49]。
早期基于CRISPR开发的核酸检测技术,主要依赖于Ⅱ型的Cas9蛋白,但部分技术没有明显的优势[50-51]。随着对CRISPR-Cas系统的不断探索,特别是Cas12、Cas13和Cas14蛋白的反式切割活性的发现,极大扩展了CRISPR-Cas系统的应用范围,也进一步革新了核酸检测领域。最近新发现Cas3蛋白也具有反式切割活性[24],其可能会为CRISPR系统的研究打开另一扇门(表1)。
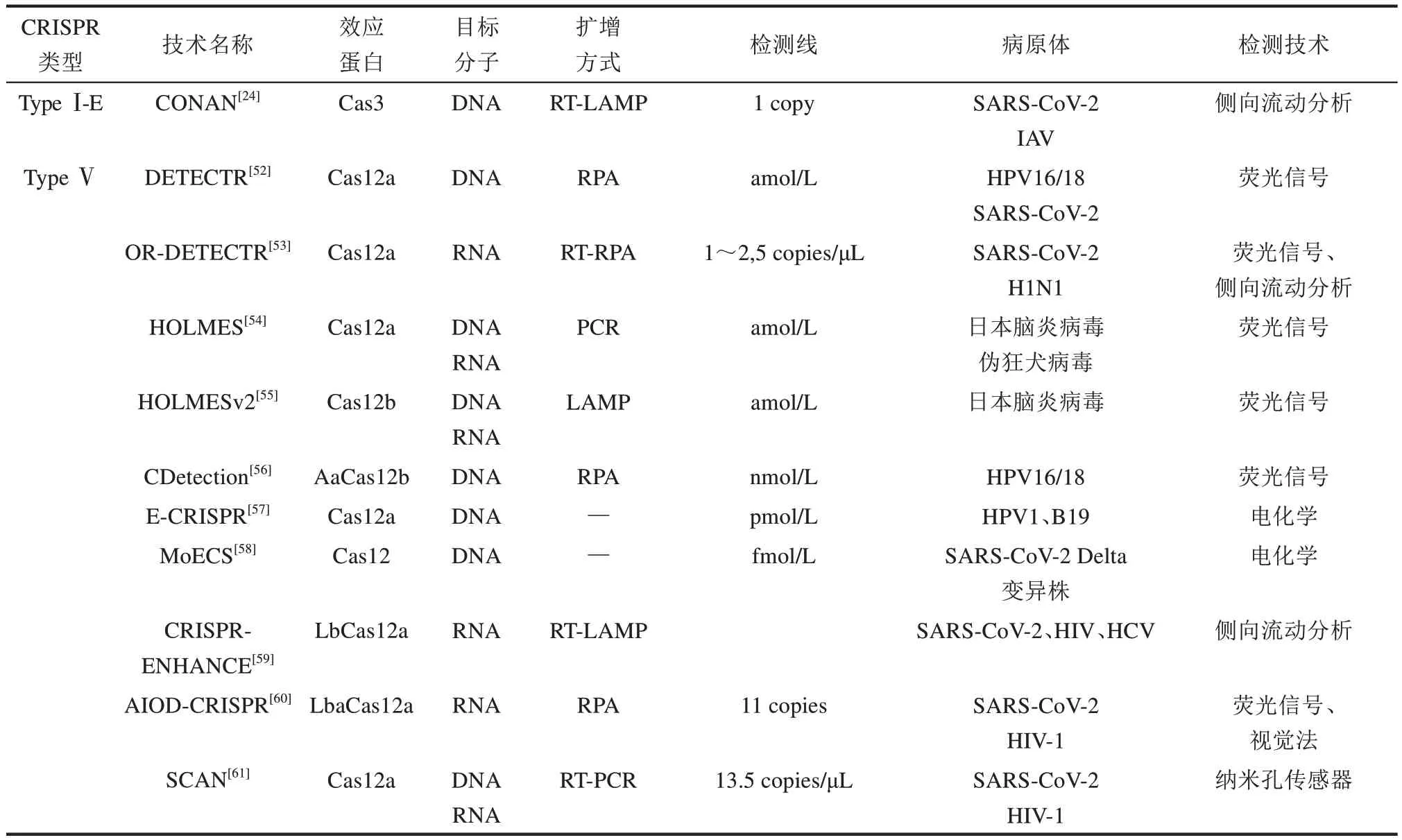
表1 基于CRISPR-Cas系统开发的部分诊断技术Table 1 Technologies for detecting pathogens based on the CRISPR-Cas system
3.1 基于CRISPR-Cas12系统的诊断
Cas12是一种RNA引导的内切酶[44],包含RuvC和NuC结构域。除了常见的Cas12a和Cas12b蛋白外,Ⅴ型CRISPR-Cas系统的其他Cas12蛋白,如Cas12d、Cas12h、Cas12i和CasF(Cas12j)也具有反式切割活性[74],但活性较弱,在核酸检测领域没有竞争力[75]。
在广泛应用于核酸检测的三种Cas12a蛋白LbCas12a、AsCas12a和FnCas12a中,LbCas12a的反式切割活性最强[76]。2017年Jennifer Doudna团队[77]开发的DETECTR(DNA endonuclease-targeted CRISPR trans reporter )检测技术,利用LbCas12a(Lachnospiraceae bacteriumND2006)与crRNA复合物,并在crRNA引导下利用RuvC结构域切割PAM(TTTN)序列下游18~25 nt的靶DNA,在识别并切割靶标dsDNA的同时,激发Cas12a的反式切割活性,切割反应体系中的ssDNA报告分子。ssDNA报告分子标记有荧光基团和猝灭基团,一旦切割,荧光基团和猝灭基团被分离,就会产生稳定而强烈的荧光信号,荧光信号的强度可以指示检测样本中靶标的量。此外,DETECTR还与重组酶聚合酶等温扩增技术结合(recombinase polymerase amplification, RPA),不仅提高了检测分析的灵敏度(amol/L水平),而且避免了对复杂、昂贵设备的需求。该技术现已成功应用于人乳头状瘤病毒(human papilloma virus, HPV)[77]和SARS-CoV-2[78]的检测(图1)。
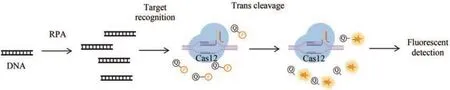
图1 基于CRISPR-Cas12a的DETECTR检测方法原理图(部分绘图素材来自Figdraw)Fig. 1 Schematics of the CRISPR-Cas12a-based CRISPR-diagnostic method DETECTR(Some drawing elements are from Figdraw)
2018年王金、赵国屏团队[54]联合LbCas12a与PCR扩增技术,建立的HOLMES(one-hour lowcost multipurpose highly efficient system)检测技术,可以在1 h内完成对伪狂犬病毒(pseudorabies virus,PRV)和日本脑炎病毒(Japanese encephalitis virus, JEV)的检测,灵敏度可以达到1~10 amol/L,此外还可以准确区分病毒基因型以及人类的单核苷酸多态性[54]。但是,该技术联合PCR扩增提高稳定性的同时也增加了检测时间和设备依赖性。
随着对Cas蛋白的深入研究,Cas12b蛋白被发现。来源于嗜热菌的Cas12b含有单个RuvC结构域[79],脱靶效应低且同样具有反式切割活性。根据这些特性,该团队将AacCas12b(Alicyclobacillus acidoterrestris)蛋白与环介导等温扩增(loopmediated isothermal amplification, LAMP)结合,建立了升级版的HOLMESv2[55],只有含有5′-TTC-3′的dsDNA或切割后具有的5′-TAC-3′的DNA序列才能激活AacCas12b反式切割活性[74]。该技术实现了DNA、RNA特异性检测以及区分SNP和定量DNA甲基化,展现出在分子诊断和表观遗传学方面应用的潜力。
而同样联合AacCas12b蛋白和LAMP建立的“TB-QUICK”,可以在2 h内完成低至1.3 copies/μL的结核分枝杆菌的检测(mycobacterium tuberculosis)[62]。该方法比常规检测方法抗酸杆菌涂片具有更高的灵敏度和特异性。目前,CRISPR-Cas12系统已广泛应用于细菌快速、灵敏的现场检测,如副溶血性弧菌(Vibrio parahaemolyticus, VP)[80]、铜绿假单胞菌(Pseudomonas aeruginosa)[81]、创伤弧菌(Vibrio vulnificus)[82]等。
随着对Cas蛋白的进一步认识,李伟团队[83]发现,AaCas12b(Alicyclobacillus acidiphilus)可以在很宽的温度(31~59 ℃)和pH范围(1.0~8.0)内保持核酸酶活性,而且Ⅴ-B型CRISPRCas12b是双RNA引导的DNA核酸内切酶系统,可以识别和切割靶向dsDNA或ssDNA,也可以非特异性切割ssDNA。当靶向dsDNA时,Cas12b依靠PAM序列(TTC或TAC)完成顺式切割。当靶向ssDNA时,Cas12b可不依赖PAM序列实现切割,而且切割活性高于dsDNA[56]。基于此特性开发的CDetection(Cas12b-mediated DNA detection)结合优化的tgRNA(tuned guide RNA)(在间隔区序列中引入单核苷酸错配),实现了6种ABO血型等位基因以及乳腺癌相关基因BRCA1的SNP分型[56]。另一个利用优化的crRNA设计的CRISPR-ENHANCE(enhanced analysis of nucleic acids with crRNA extensions),通过将具有5′-TATTATT-3′序列的7-mer DNA添加到crRNA的3′末端,在明显提高了SARS-CoV-2核酸检测的灵敏度的同时,保持了特异性[59]。
为了优化检测技术,拓展信号阅读方式。基于电化学的生物传感平台因其快速的信号阅读、经济的传导元件和简单的传感平台而得到广泛的开发和应用,但这种传感平台的准确度不高。而CRISPR-Cas系统在靶向核酸方面具有高准确性,两者的联合成为开发精准、低成本的快速检测技术的强大推力。2019年,Dai等[57]将电化学生物传感平台和CRISPR结合开发的E-CRISPR检测技术,通过CRISPR-Cas系统来影响电信号输出,利用Cas12a的反式切割活性获得高传导信号。该检测技术将修饰有3′-亚甲基蓝(3′-MB)标签的ssDNA报告分子固定于金电极上,在靶标DNA存在的情况下,Cas12a-crRNA反式切割MB-ssDNA分子,亚甲基蓝标签的分离,会使电化学信号降低。在没有靶标DNA存在时,Cas12a的反式切割活性保持沉默,ssDNA分子保留在电极表面,从而导致亚甲基蓝的高电化学电流[57]。该技术率先在HPV-16和细小病毒(PB-19)的检测中证明了可行性[57]。而同样利用电化学CRISPR传感建立的MoECS(methodology of electrochemical CRISPR sensing),在SARS-CoV-2 Delta变异株检测中,展现出高特异性和稳定性,为诊断其他新出现的SARS-CoV-2变体提供了借鉴[58]。目前,电化学传感联合CRISPR-Cas系统逐渐成为医学诊断中强大的分析工具,其还成功应用于鼠伤寒沙门氏菌[84]、单核李斯特氏菌[85]、心肌肌钙蛋白Ⅰ(cardiac troponinⅠ,cTnⅠ)[34]等的检测(图2)。

图2 基于CRISPR-Cas12a的E-CRISPR检测方法原理图(图片绘制来自BioRender)Fig. 2 Schematics of the CRISPR-Cas12a-based diagnostic method E-CRISPR(The picture was created in BioRender)
而Nouri等[86]开发的SCAN(solid-state CRISPRCas12a-assisted nanopores)检测技术,将Cas12a与固态纳米孔传感器结合,利用固态纳米孔传感器独特的单分子灵敏度和无标记电子传感,定量分析被Cas12a切割的环状ssDNA报告分子[86]。其已成功应用于HIV-1和SARS-CoV-2的检测[61]。该检测平台还可以扩展到其他类型纳米孔和靶标核酸检测中,为POCT(point-of-care testing)提供一种快速、低成本的方法。
为了优化检测流程,Ding等[60]开发AIODCRISPR(all-in-one dual CRISPR-Cas12a)检测技术,将RPA扩增和CRISPR检测的所有反应组分混合,在引入两个crRNA的同时,添加高浓度ssDNA报告分子[60],一步即可完成对SARS-CoV-2[87]和HIV-1[88]的检测。最近新发布的sPAMC(suboptimal protospacer adjacent motifs of Cas12a-based)一步法,利用次优PAM基序(非传统PAM),可将反应速度加快2~3倍,20 min即可完成SARS-CoV-2检测。而且在针对新冠病毒载量很低(Ct值>35)的感染初期患者,检出率也较高[88]。相较于一步反应STOPCovid[63](SHER-LOCK Testing in One Pot COVID-19 version 1)和STOPCovid.v2[64],具有良好的优势,但是该技术对反应温度要求比较高,需要37 ℃左右的恒温,无法室温进行。
3.2 基于CRISPR-Cas13系统的诊断
Cas13是RNA引导和RNA靶向的核酸酶,具有两个HEPN结构域[89],特异性作用于RNA靶标,发挥特异性识别和非特异性切割的功能[90]。这一特性使其很快在核酸检测和疾病诊断中显示出独特优势,成为开发快速检测工具的研究热点之一。在一系列Cas13蛋白中,Cas13a是第一个被明确的亚型,也是第一个被应用于核酸检测的效应蛋白。2017年,张峰团队[68]首次利用Cas13的非特异性切割活性,开发了SHERLOCK(specific high-sensitivity enzymatic reporter unlocking )核酸检测技术[68]。该技术与DETECTR原理相同,但依赖于LwaCas13a(Leptotirichia wadei)核酸酶的活性,识别和切割靶标RNA的同时,反式切割ssDNA报告分子产生荧光信号[91]。目前,该技术已成功应用于寨卡病毒(Zika virus, ZIKA)、登革病毒(Dengue virus, DENV)、SARS-CoV-2和恶性疟原虫[92]等的诊断,可以在短时间内完成高灵敏度的检测。而且SHERLOCK反应体系的所有组分可以冻干储存,方便运输的同时不会影响检测的灵敏度和特异性[91](图3)。
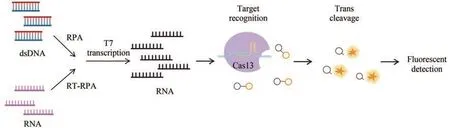
图3 基于CRISPR-Cas13a的SHERLOCK检测方法原理图(部分绘图素材来自Figdraw)Fig. 3 Schematics of the CRISPR-Cas13-based diagnostic method SHERLOCK(Some drawing elements are from Figdraw)
上述的SHERLOCK检测技术有一个缺点就是只能定性,不能定量。而升级的SHERLOCKv2,将4种类型的Cas蛋白(LwaCas13a 、PsmCas13b、CcaCas13b和AsCas12a)组合使用,切割用不同的荧光基团标记的报告分子,在FAM、Cy5、HEX和TEX通道中进行检测,实现了定量同时检测4种核酸序列[73],但是仍然无法实现单管多重检测。此外,通过使用CRISPR相关酶Csm6(Csm6是一种来自Ⅲ型CRISPR-Cas系统的二聚体RNA核酸内切酶,Cas13剪切后的序列是Csm6的靶向序列)进一步放大检测信号,提高了检测灵敏度[70]。同时,SHERLOCKv2也被应用于侧向流动分析。通过金纳米颗粒标记的抗FAM抗体,在试纸条上检测被切割的报告分子,产生视觉比色信号[73]。该检测平台具有SNP检测特异性,可快速、便携和准确进行核酸检测,为资源设备缺乏地区的流行病监测和病原检测提供了技术支持(图4)。
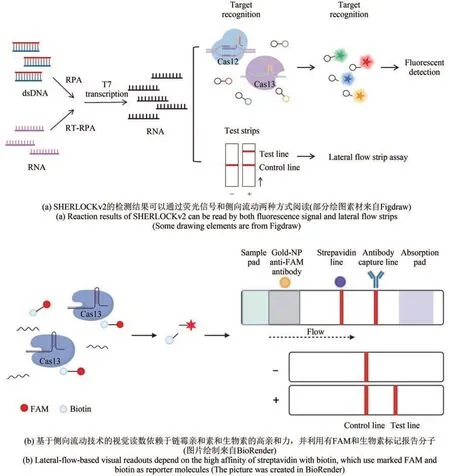
图4 基于CRISPR-Cas13的SHERLOCKv2检测平台Fig. 4 Schematics of the CRISPR-Cas13 diagnostic platform SHERLOCKv2
为了简化工作流程,张峰团队[93]又引入HUDSON(加热未提取样本以消除核酸酶)技术,成功简化了病原检测过程中的核酸提取步骤。HUDSON可有效提取各种临床样本(包括尿液、全血、血浆、血清和唾液等)中的病毒核酸,且不影响检测灵敏度。HUDSON-SHERLOCK已实现对DENV、ZIKA、埃博拉病毒等的快速检测[93-94]。该技术在缺乏基础设施的地区是必需的,非常适合应用于现场检测。
为提高基于CRISPR的检测能力,联合CRISPR诊断和微流控技术开发了CARMEN(combinatorial arrayed reactions for multiplexed evaluation of nucleic acids)检测平台[71]。在微流控技术的众多分类中,基于液滴的微流控系统由于其高比表面积、高通量等优点,在合成生物学实验中展现出非凡的前景[95]。而且用于微流控检测的微流控芯片具有简单、体积小、便携等优势。在第一代检测方法CARMEN v.1中,样本和Cas13-crRNA复合物液滴分别编码和乳化后配对组合,实现针对性的样本检测,该技术可以同时检测8个样本,169种人类相关病毒。CARMEN v.1证明了基于CRISPR多重检测的可行性,但由于需要定制的成像芯片和读出硬件,以及8~10 h的手工操作和低通量样本评估,因此很难应用于临床(图5)。

图5 CARMEN-Cas13检测方法原理图(图片绘制来自BioRender)Fig. 5 Schematics of the CARMEN-Cas13 detection method(The picture was created in BioRender)
为优化检测性能,在CARMEN v.1的基础上开发了mCARMEN(microfluidic combinatorial arrayed reactions for multiplexed evaluation of nucleic acids),将CRISPR诊断、微阵列技术与简化的临床工作流程相结合[96],开发的mCARMEN呼吸道病毒检测面板,可同时检测21种呼吸道病毒,包括SARS-CoV-2及其他冠状病毒、流感病毒等[72]。mCARMEN是唯一一种将监测功能结合到单一技术平台中的诊断方法,能够在一天内检测数百个样本中的多种呼吸道病毒。
微流控技术为POCT的发展提供了强有力的工具。将CRISPR-Cas12a/Cas13a与微流控装置相结合,具有快速、高灵敏度、低成本、自动化和现场检测等优点。但还有许多需要解决的问题,如扩散过程中可能发生交叉反应、芯片外的手动步骤等(核酸提取、扩增等)[97]。
为了更好地实现现场检测,Arizti-Sanz团队[98]将扩增与CRISPR-Cas13系统检测整合为一步诊断平台SHINE(streamlined highlight of infection to navigate pandemic),通过添加RNase H提高核酸检测灵敏度[98],并采用管内荧光和配套的智能手机应用程序,实现了SARS-CoV-2有效的核酸检测[69]。SHINE诊断平台最大限度地减少了对设备的要求,也减少了结果读数偏差。而另一个联合微流控芯片和集成型荧光检测器的检测技术FIND-IT[70](this fast integrated nuclease detection in Tandem)和采用物理隔离方法建立的检测技术OR-SHERLOCK及OR-DETECTR[53],也均在单管SARS-CoV-2的一步核酸检测中证明了可行性。
3.3 基于CRISPR-Cas14系统的诊断
除了Cas12和Cas13系统外,结构紧凑的Cas14蛋白也具有类似的反式切割活性。Cas14是一种靶向ssDNA的CRISPR内切酶,与CRISPRCas12相比,Cas14识别和切割目标核酸序列不受PAM序列的限制,而且对crRNA与靶标模板之间的核苷酸错配的容忍性更低,内部序列对核苷酸错配更敏感,这一特性使Cas14能够实现高保真的区分SNP[38]。基于此特性建立的Cas14a-DETECTR检测平台,已应用于人类HERC2基因(该基因决定眼睛颜色[99])SNP分型。该检测技术也可以与HUDSON结合,实现病原的快速检测,其已成功应用于人类博卡病毒(HBoV-1)的检测[66]。最近,另一个基于Cas14a1蛋白的ACasB(aptamerbased Cas14a1 biosensor)新检测平台,可以在复杂样本中实现100%检出金黄色葡萄球菌(Staphylococcus aureus)[67]。CRISPR-Cas14是病原突变筛查的一种高效且经济的方法,但由于Cas14仅结合和切割ssDNA,因此需要额外的步骤使dsDNA靶标生成ssDNA,这一特殊性将增加应用的复杂性。因此,到目前为止,Cas14蛋白在分子诊断应用领域并没有展现出明显优势。
3.4 基于CRISPR-Cas 3系统的诊断
最近,有研究描述Ⅰ-E型CRISPR-Cas3也具有反式切割活性[24],主要利用Cascade效应模块(由Cas5、Cas6、Cas7、Cas8、和Cas11等多种蛋白组成)、Cas3蛋白和crRNA组成的复合物,在与靶标结合后,完成ssDNA探针的切割[100]。而且有研究表明,Cas3-Cascade/crRNA效应复合物必须与靶标DNA形成完整的R-loop结构,才能利用Cas3蛋白完成切割[101],这一特性可以防止Cas3过早与DNA结合,引起非特异性切割。基于此新开发的CONAN(Cas3-operated nucleic acid detection)检测技术对单碱基区分具有很高的特异性,不仅成功检测出临床样本中SARS-CoV-2,还可以特异性检测甲型流感病毒(influenza A virus)变体中的单碱基对突变。Cas3的反式切割活性被发现不久,未来一定会开发更多检测方法,拓展CRISPR技术研究的方向。
4 总结与展望
CRISPR-Cas系统作为新颖而强大的核酸诊断工具,利用不同Cas蛋白的固有特性,开发了许多检测技术,而基于Cas12和Cas13蛋白开发的检测技术最具潜力。迄今为止,DETECTR[77]、SHERLOCKv2[73]、CARMEN[71]等很多检测技术已经应用于多种病原检测,包括SARS-CoV-2[78,102],这些检测方法通常可以达到fmol/L至amol/L级的灵敏度以及实现单碱基分辨。
为了简化和优化检测的流程,研究人员开发了一些简单的样本预处理,如HUSDON等,可以实现直接扩增和检测病原核酸;而一管式反应技术的开发,降低了样本污染的风险,将靶标核酸扩增与Cas蛋白切割两步反应于闭管中进行;开发不同的结果读数方法(荧光读数、比色读数、电化学读数等)并结合便携低成本的仪器(智能手机等),非常适合应用于现场检测。此外,在一些检测技术中,设计使用优化的crRNA降低了脱靶效应,而利用核苷酸错配耐受性更低的Cas蛋白也解决了潜在的假阳性问题。
虽然CRISPR-Cas系统具有很多优势,但在该技术转化为临床应用之前,仍有很多挑战需要克服。例如,Cas12、Cas13等蛋白对靶标核酸的识别与切割,依赖于PAM或PFS序列,其限制了目标核酸区域的选择,大大缩小了引物设计的选择范围;而且现有的检测技术都无法摆脱核酸预扩增步骤以及实现单管多重检测;同时基于CRISPRCas系统开发的检测技术与PCR技术一样,都无法为未来可能新出现的病毒威胁或大流行做出监测。
CRISPR-Cas系统的发现与探索,为分子生物学开辟了许多新的研究方向。目前,CRISPR-Cas系统已经被应用于生命科学的各个领域,本文主要综述CRISPR-Cas系统在病原核酸检测方向的研究,讨论了具有代表性的诊断平台,而CRISPR的应用范围远远不止核酸检测或临床相关突变的鉴定。针对非核酸检测的其他领域,如蛋白质、氨基酸代谢物、脂质、糖类、离子等,CRISPR-Cas系统也具有强大的潜力。目前,已经开发了几种基于CRISPR的新检测平台,不仅可以识别蛋白质[57],还可以检测小分子物质(有毒物质、药物等)[103],但仍有许多具有重要功能的蛋白质尚未得到全面研究[104]。因此,将CRISPR-Cas系统与蛋白组学联合,可能会为蛋白质功能的研究提供强大的工具。与此同时,基于CRISPR的诊断也可以联合材料学,开发可以检测固体表面、穿戴物品、医疗设备上的核酸,实现实时病原体检测。
随着对CRISPR-Cas系统的优化和进一步认识,基于CRISPR-Cas系统开发的检测技术可能会成为病原核酸检测的主流平台之一。虽然很多检测技术不能很快从实验室走向临床应用,发挥其作用,但在不久的将来,可以为大规模人口筛查、更好更快地控制病原体传播、实现现场快速检测提供良好平台。
