应用深度学习进行基于前列腺癌转移报告和数据系统指南的晚期前列腺癌盆腔外脏器及转移灶分割
2024-03-12刘想曲别雪蕾吴静云吴鹏升张晓东王霄英
刘想,曲别雪蕾,吴静云,吴鹏升,张晓东,王霄英*
1.北京大学第一医院医学影像科,北京 100034;2.武警四川省总队医院医学影像科,四川 乐山 614000;3.北京赛迈特锐医学科技有限公司,北京 100011;
晚期前列腺癌患者常发生远处转移,而不同转移情况的治疗策略也不同,因此,全面、准确地评估前列腺癌患者的远处转移十分重要[1-2]。前列腺癌转移报告和数据系统(metastasis reporting and data system for prostate cancer,MET-RADS-P)是由多学科专家组基于全身MRI 制订的评估晚期转移性前列腺癌全身情况及临床治疗效果的指南[3-4]。该指南将前列腺癌全身转移部位分为4 组14 个区域,包括:①淋巴结3 个区域;②脏器3 个区域;③前列腺局部病灶1 个区域;④骨骼7 个区域[5]。然而在临床工作中,全身多区域转移灶的检出及评估仍存在一定困难,且需要耗费大量时间。基于深度学习的区域及转移灶自动分割方法有望简化评估过程,提高影像医师的读片效率[6-8]。本团队前期已完成了在扩散加权成像(DWI)序列上基于深度学习进行前列腺癌患者盆腔范围内骨质结构、骨转移灶及淋巴结的分割[9-11]。本研究尝试基于深度学习进行盆腔外的区域分割及转移灶分割,以期为后续前列腺癌全身转移疗效评估奠定基础。
1 资料与方法
1.1 研究对象 按照扫描部位(头部、颈部、胸部、腹部)回顾性收集北京大学第一医院2017年1月—2022年1月经病理证实的恶性肿瘤且临床综合诊断存在全身转移灶的479例患者,最终纳入研究的4组数据集包括头部转移68例,颈部转移91例(颈椎转移瘤4例、淋巴结转移77例、多部位转移10例),胸部转移57例(包括肺转移瘤16例、胸椎转移瘤18例、纵隔淋巴结转移21例、多部位转移瘤2例),腹部转移263例(包括肝转移186例、腹膜后淋巴结转移13例、腰椎转移瘤6例、肾上腺转移3例、多发转移瘤55例)。4组数据集用于扫描范围的分类模型及不同区域和转移灶的分割模型训练。
另收集2017年1月—2022年1月经病理证实为前列腺癌,且在此期间行全身MRI的患者144例,排除转移灶切除患者23例;转移灶定位不明确患者9例;扫描不规范(包括扫描范围不足、图像序列缺失等)患者12例;图像质量不佳(包括各类伪影如呼吸伪影、运动伪影及化学位移伪影等,图像信噪比低等)患者10例。最终共90例前列腺癌患者的全身MRI纳入研究并用做分类及分割模型的外部验证。本研究经北京大学第一医院伦理委员会批准(2020-170)。
1.2 扫描参数模型 训练及外部验证数据集中的MRI 图像来源于3 台3.0T MRI(Achieva,Philips Healthcare;Discovery MR750,GE Healthcare;Intera,Philips Healthcare),主要扫描序列包括轴位T1WI、轴位T2WI、DWI及DCE序列,并根据DWI序列自动重建表观扩散系数(ADC)图像。DWI序列为本研究的研究对象,不同扫描部位的DWI扫描参数见表1。

表1 不同3.0T MRI各部位的DWI参数
1.3 扫描部位分类 本研究纳入的外部验证数据集为前列腺癌患者的全身DWI扫描,因此在进行区域分割及转移灶分割前应先进行不同扫描部位的自动分类(图1)。按照临床实践规则,将图像范围分为4个类别,定义如下:头部:从颅顶到颅底;颈部:从颅底到锁骨,肺尖可见,纵隔不可见;胸部:从肺尖到膈面,全肺可见;腹部:从膈面到髂嵴,肝脏可见,膀胱未见。
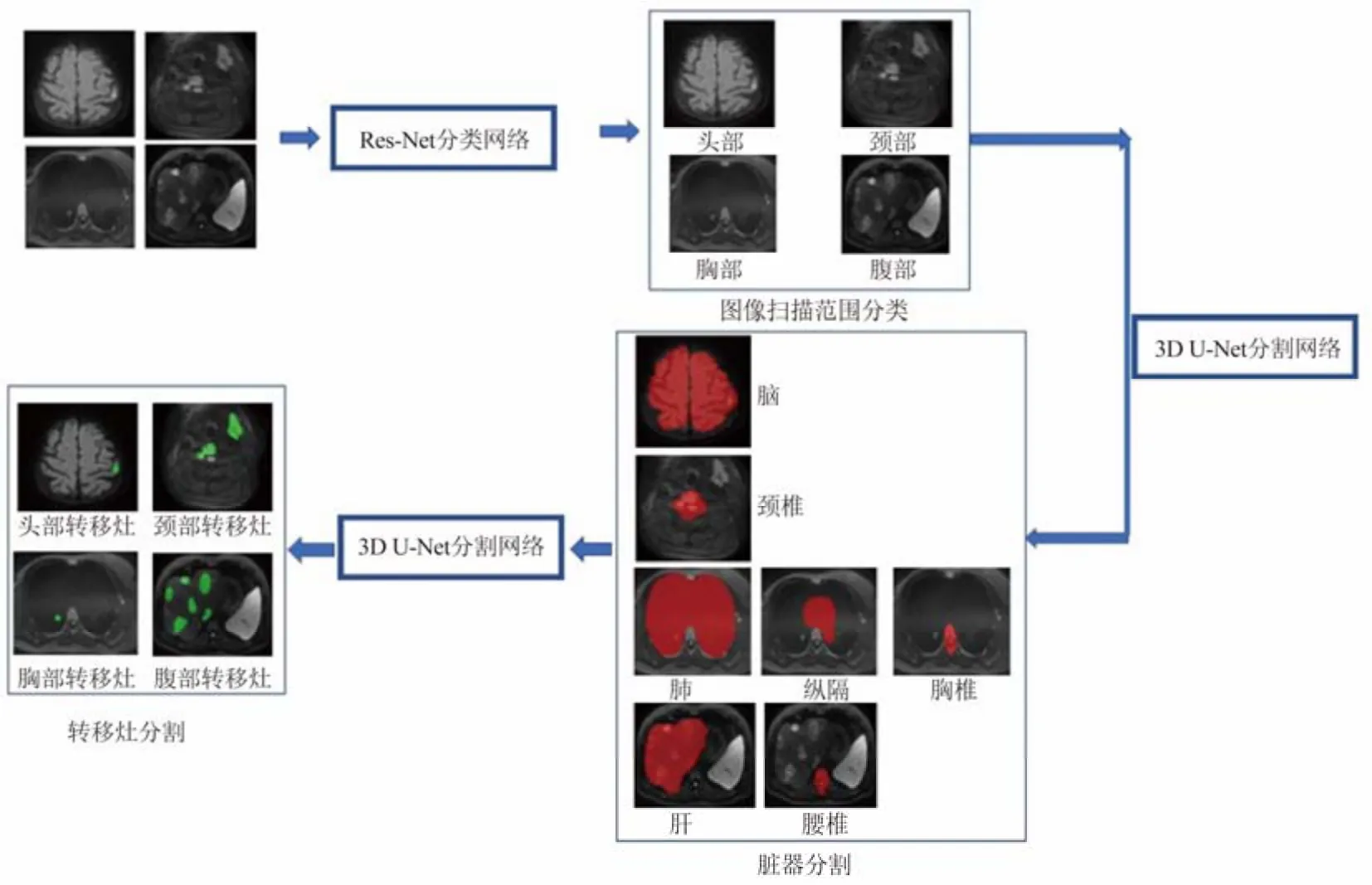
图1 不同扫描部位的自动分类
1.4 模型训练 训练3D-ResNet[12]深度学习分类模型用于全身DWI扫描范围的分类。首先将模型训练数据集中的479例不同部位DWI数据按照8∶1∶1随机分为训练集383例、调优集48例和测试集48例。模型训练过程中,输入图像大小为256×256,输出数据为对模型分类的预测结果。图像扩增方法包括0~10°以内的水平及垂直旋转,上下、左右随机平移的最大幅度为图像大小的10%。使用Adam(https://arxiv. org/abs/1412.6980)作为训练优化器。模型训练时批尺寸设定为4,学习率为0.001,训练迭代次数设置为300个周期。
1.5 数据标注 根据MET-RADS-P指南的分区,对不同部位的解剖结构及转移灶进行标注,主要包括,①头部DWI:脑实质、脑转移灶;②颈部DWI:颈椎、颈部转移灶;③胸部DWI:双肺实质、纵隔、胸椎、胸部转移灶;④腹部DWI:肝、腰椎、腹部转移灶。其中转移灶在DWI图像呈明显高信号且相应ADC图为低信号(图2)。
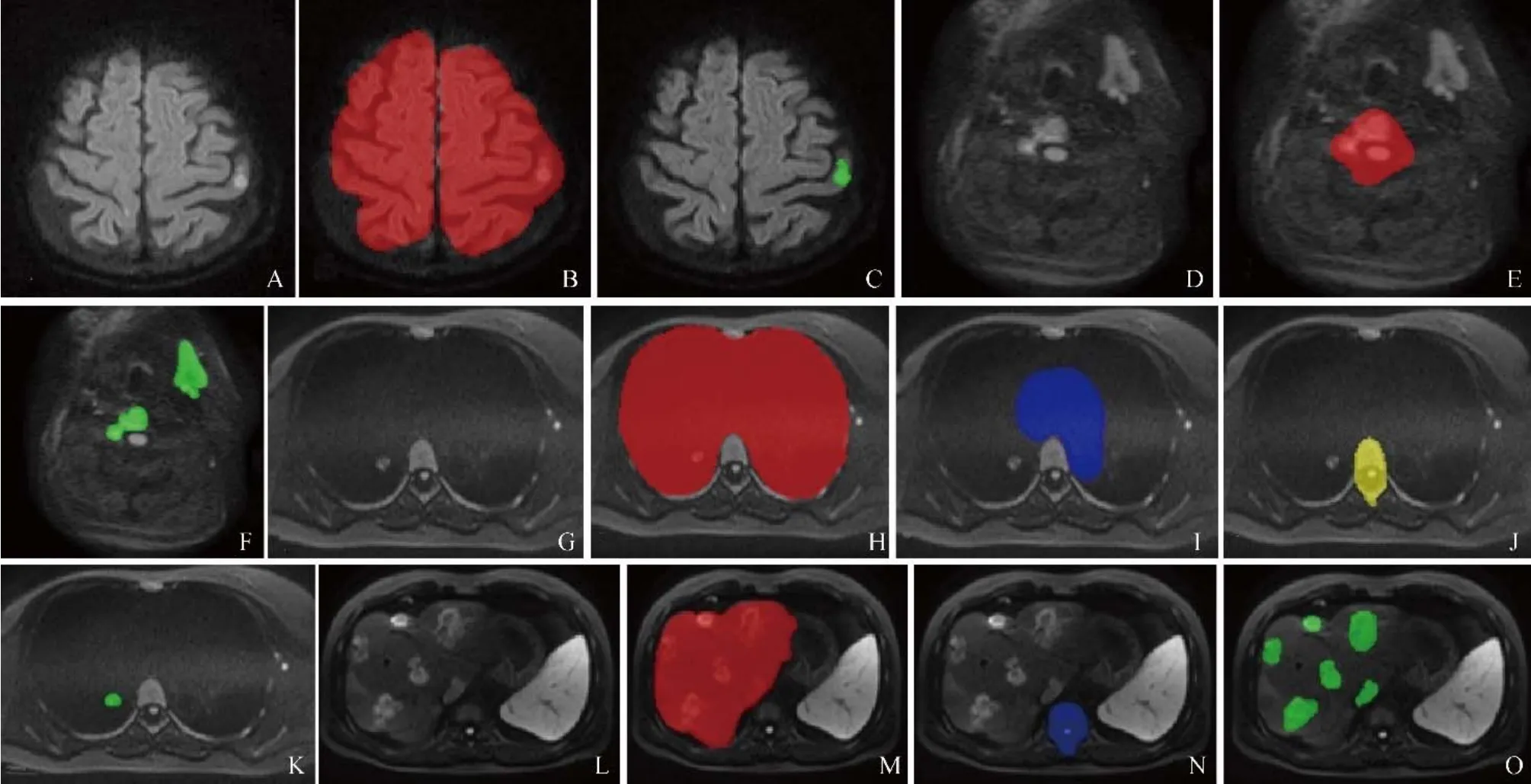
图2 不同部位解剖结构及转移灶标注。A~C:头部标注。A:DWI原图;B:脑实质标注;C:脑转移灶标注;D~F:颈部标注。D:DWI原图;E:颈椎标注;F:颈部转移灶标注;G~K:胸部标注。G:DWI原图;H:肺实质标注;I:纵隔标注;J:胸椎标注;K:肺转移灶标注;L~O:腹部标注。L:DWI原图;M:肝实质标注;N:腰椎标注;O:肝转移灶标注
标注前将所有DICOM格式的DWI图像转换为NIFTI格式。由1名影像科住院医师(读片经验3年)使用ITK-SNAP 3.6.0软件在DWI图像上对脏器及转移灶进行标注;再由1位影像科专家(读片经验≥15年)对标注进行修改确认。以确认后的图像标签作为评估分割模型的“金标准”。
1.6 分割模型训练 采用3D U-Net深度学习网络[13-15]进行各部位解剖结构及转移灶分割。颅脑分割模型包括脑实质和脑转移灶分割;颈部分割模型包括颈椎及颈部转移灶分割;胸部分割模型包括肺、纵隔、胸椎及胸部转移灶分割;腹部分割模型包括肝、腰椎及腹部转移灶分割。所有图像数据经预处理后大小均为64×256×256(z,y,x),自动窗宽、窗位。按照8∶1∶1将不同分割模型数据随机分为训练集、调优集和测试集(颅脑:54∶7∶7;颈部:73∶9∶9;胸部:45∶6∶6;腹部:211∶26∶26)。各模型输入的训练集数据为DWI图像和手工标注的各结构标签,使用Adam(https://arxiv.org/abs/1412.6980)作为训练优化器,模型训练时批尺寸设定为1,学习率为0.000 1,训练迭代次数设置为200个周期。
1.7 模型训练框架 本研究的深度学习框架为PyTorch0.4.1[16-17],Python版本3.6,所有训练过程均在GPUNVIDIA Tesla P100 16G(https://www.nvidia.com/en-us/data-center/tesla-p100/)上完成,运行系统为ubuntu16.04,相关软件包括Opencv 3.4.0.12、Numpy 1.16.2、Simple ITK 2.2.0等。
1.8 统计学分析 采用Prism 9.0软件。符合正态分布的计量资料以±s表示,非正态分布的计量资料以M(Q1,Q3)表示;计数资料以例数或百分比表示;利用测试集及外部验证数据对3D-ResNet的分类结果进行评估。以影像专家的分类结果为参考标准,使用混淆矩阵进行模型分类结果与参考标准比较,并计算模型分类的符合率。
同样利用测试集及外部验证数据对3D U-Net模型的分割性能进行定量评估,评估指标包括基于重合度的指标:Dice相似系数(Dice similarity coefficient,DSC)[18-19];基于体积的指标:体积相似度(volumetric similarity,VS)[20]。三者联合使用可以从整体重叠程度、体积以及边缘匹配程度多方面评估分割性能。
2 结果
2.1 数据特征与分布 本研究纳入的用于模型训练的4组数据集及外部验证数据集的患者临床信息分布见表2。其中,脑转移灶的原发肿瘤多为肺癌(50,73.53%),颈部转移灶的原发肿瘤多为喉癌(35,38.46%),胸部转移灶的原发肿瘤多为肺癌(25,43.86%),腹部转移灶的原发肿瘤多为结肠癌(79,30.04%)。

表2 不同数据集患者临床信息
2.2 扫描范围 分类模型结果以扫描序列为单位统计,为扫描范围分类模型在测试集及外部验证数据集中的预测结果。在测试集中,分类模型在头部、颈部、胸部和腹部的符合率分别为100%(7/7)、100%(9/9)、100%(6/6)和96.15%(25/26)。在外部验证数据集中,分类模型在头部、颈部、胸部和腹部的符合率分别为100%(90/90)、98.89%(89/90)、96.67%(87/90)和94.44%(85/90)。错误分类的情况主要包括:1个颈部DWI序列因包含部分颅底层面而被自动分类为头部;3个胸部DWI序列因包含较多肝脏层面而被自动分类为腹部;5个腹部DWI序列因包含纵隔而被自动分类为胸部。
2.3 脏器分割 脏器分割DSC、VS值见表3,模型在测试集及外部验证集对于不同区域的脏器均具有较好的分割性能,所有脏器分割的DSC及VS值均>0.85。
表3 脏器分割结果(±s)

表3 脏器分割结果(±s)
注:DSC:Dice相似系数;VS:体积相似度
项目脑实质颈椎肺实质纵隔胸椎肝脏腰椎测试集DSC 0.96±0.01 0.85±0.08 0.95±0.03 0.90±0.03 0.91±0.03 0.93±0.04 0.96±0.02 VS 0.99±0.01 0.99±0.01 0.98±0.03 0.96±0.04 0.95±0.02 0.98±0.03 0.99±0.01外部验证集DSC 0.99±0.01 0.97±0.03 0.96±0.05 0.86±0.10 0.96±0.05 0.94±0.06 0.94±0.02 VS 0.99±0.01 0.99±0.01 0.96±0.01 0.89±0.10 0.97±0.04 0.96±0.04 0.94±0.07
2.4 转移灶分割 不同部位转移灶分割DSC、VS见表4。模型在测试集及外部验证集的脏器分割DSC及VS值均>0.65。
表4 不同部位转移灶分割结果(±s)

表4 不同部位转移灶分割结果(±s)
注:DSC:Dice相似系数;VS:体积相似度
项目头部转移灶颈部转移灶胸部转移灶腹部转移灶测试集DSC 0.75±0.07 0.70±0.12 0.68±0.13 0.71±0.11 VS 0.79±0.12 0.75±0.16 0.77±0.13 0.83±0.13外部验证集DSC --0.65±0.07 0.72±0.13 VS --0.74±0.04 0.82±0.13
3 讨论
全身MRI可突破单部位的限制,对晚期转移性前列腺癌患者的全身肿瘤负荷、疗效评估及生化复发做出精准评估[21-22],同时,在检测癌症患者的骨转移及骨外转移方面具有极好的观察者间一致性[23-24]。然而,复杂的评估流程限制了全身MRI在临床上的广泛应用。本研究尝试应用深度学习技术自动分割前列腺癌转移灶,以辅助影像医师进行全身MRI评估。
本研究在进行转移灶分割模型训练前,基于深度学习方法建模,对全身DWI图像做出扫描范围的分类,结果显示,模型对分类任务的准确度高(符合率:94.44%~100.00%),可基本达到临床需求,与本团队前期通过深度学习模型建立的腹盆部CT图像范围分类的研究结果类似[25]。
本研究在扫描范围分类的基础上,进一步进行不同扫描部位的脏器及转移灶分割。结果表明,模型在测试集和外部验证集中对不同扫描部位的脏器均具有很高的分割性能,尽管外部验证集的脑实质及颈椎分割性能优于测试集,但其DSC及VS值均>0.85。相较于脏器分割,不同扫描部位的转移灶分割性能较差,其DSC值范围在测试集为0.68~0.71,外部验证集为0.65~0.72。其中胸部转移灶的分割性能最低,分析其原因可能是在模型训练中所纳入的胸部转移患者数量较少。
本研究中,为了尽可能多地纳入不同区域的转移灶特征,所收集的模型训练数据集包括前列腺癌在内的多种原发病灶的转移瘤;而在外部验证数据集中仅有晚期转移性前列腺癌患者。本研究结果显示,模型在测试集及外部验证数据集中的分割性能相似,表明模型在分割不同原发肿瘤的转移灶间具有较好的泛化性能。
深度学习技术目前已广泛用于脏器及病灶的分割。谢婷婷等[26]应用深度学习技术实现了肝段的自动分割,并基于分割结果进一步为术前评估提供了指导;奈日乐等[18]进行CT图像上脑血肿分割;陈元翀等[27]基于深度学习技术实现了肾上腺结构分割。尽管取得了较为理想的分割结果,但以上研究均为单个器官结构的分割,不同于本研究基于全身MRI所实现的全身多个脏器的分割。
本研究也存在一定局限性。第一,本研究的区域分割及转移灶分割模型并未完全囊括MET-RADS-P指南的14个区域,主要原因是部分区域(如胸廓、颅骨等)在DWI序列的成像存在一定局限性,因此,在后续研究中,计划纳入其他序列如T1WI或T2WI,进行多序列的分割模型训练,以进一步完善前列腺癌的全身转移评估。第二,由于外部验证数据集中纳入的患者数量有限,转移灶的分布存在较大的偏倚,腹部转移患者较多,而无头部及颈部转移患者。第三,模型训练数据集包括但不限于前列腺癌在内的多种原发病灶的转移瘤;而在外部验证数据集中仅有晚期转移性前列腺癌患者,不同原发癌的转移表现和好发部位存在很大差异,将非前列腺癌转移用于训练是该研究的主要缺陷之一。最后,本研究仅进行了不同区域脏器及转移灶的分割模型训练,但其对于影像医师的临床意义还需进一步证实。
总之,基于深度学习的3D U-Net模型可实现晚期前列腺癌患者的盆腔外区域及转移灶分割,有望用于后续的前列腺癌全身转移疗效评估。
