福建省茶树病毒种类鉴定及多重PCR检测技术的建立
2024-03-08陈细红蔡伟虞赟李敏王念武杜振国沈建国高芳銮
陈细红,蔡伟,虞赟,李敏,王念武,杜振国,沈建国,高芳銮
福建省茶树病毒种类鉴定及多重PCR检测技术的建立

1福建农林大学植物病毒研究所,福州 350002;2福州海关技术中心/福建省检验检疫技术研究重点实验室,福州 350001;3榕城海关综合技术服务中心,福建福清 350300
【目的】明确福建省茶树主要病毒种类和分布情况,并建立同时快速检测多种病毒的多重PCR检测技术。【方法】2019—2023年,从福建省福州、南平、宁德、泉州、漳州、厦门、三明、莆田和龙岩9个地市区采集具有褪绿、皱缩和坏死等疑似病毒感染症状的茶树样品1 869份,采用高通量测序技术结合PCR和RT-PCR检测的方法对茶树病毒病的病原进行鉴定,并将PCR扩增获得的特异性目的片段进行克隆和测序,对获得的序列进行系统发育分析;同时,根据GenBank上已报道的病毒序列设计特异性引物,通过退火温度、引物浓度、循环数等反应条件和反应程序的优化,建立同时检测福建茶树主要病毒的多重PCR检测技术,并测定该技术的特异性、灵敏度及实际应用效果。【结果】从所采集的茶树病样上检出3种病毒,按检出率从高到低依次为油茶双生病毒(oil tea associated geminivirus,OTaGV)(48.90%)、茶树坏死环斑病毒(tea plant necrotic ring blotch virus,TPNRBV)(26.75%)和茶树潜隐病毒1(camellia cryptic virus 1,CCV1)(17.98%);在检出病毒的1 258份样品中,有807份为OTaGV、TPNRBV或CCV1单独侵染,检出率分别为37.20%、21.38%和5.56%;其余451份样品为2种或3种病毒复合侵染,复合侵染检出率达35.85%,OTaGV+CCV1、TPNRBV+CCV1、TPNRBV+OTaGV、OTaGV+CCV1+TPNRBV 4种类型复合侵染检出率分别为17.49%、0.40%、14.71%、3.26%。在地理分布上,OTaGV在福州、南平、宁德、泉州、漳州、厦门、三明、莆田和龙岩9个地区均有分布,其中漳州OTaGV检出率最高,为96.77%;CCV1在福州、南平、宁德、泉州、漳州、三明、莆田和龙岩8个地区均有分布,其中三明CCV1检出率最高,为66.00%;TPNRBV在福州、南平、宁德、泉州和漳州5个地区均有分布,其中泉州TPNRBV检出率最高,为79.13%;在福建省9个地区中,厦门地区仅检出OTaGV,三明、莆田和龙岩地区检出OTaGV和CCV1,其他5个地区同时检出OTaGV、TPNRBV和CCV1;福州、南平、宁德、泉州、漳州、三明、莆田7个地区检测到病毒复合侵染,其中漳州地区病毒复合侵染检出率最高(85.00%)、宁德地区病毒复合侵染检出率最低(23.03%)。利用测定的CCV1和TPNRBV部分基因序列构建系统发育树,结果表明本研究获得的CCV1分离物(FW)与已报道的福建分离物FJ-SH104(GenBank登录号:ON807095)亲缘关系最近、TPNRBV分离物(FU)与福建分离物QZHA92(GenBank登录号:OQ948454)亲缘关系最近。经优化建立的多重PCR检测技术特异性强,仅OTaGV、TPNRBV和CCV1能扩增出特异性目的片段,而其他病毒及健康茶树样品上均未扩增出特异性目的片段;多重PCR灵敏度最低可以检测到稀释至10-4倍的OTaGV、TPNRBV和10-3倍的CCV1;利用该多重PCR检测技术对茶园中采集的60份病样进行检测,检测结果与单一PCR检测结果完全相符。【结论】OTaGV、TPNRBV和CCV1是当前福建茶树上主要病毒种类,其中OTaGV为福建地区首次报道,该病毒可侵染茶树也为首次发现;在检出的3种病毒中,以OTaGV发生分布范围最广,其次为CCV1、TPNRBV;福建地区茶树病毒目前仍以单独侵染为主,但同时存在较多的复合侵染,复合侵染类型包括TPNRBV+CCV1、TPNRBV+OTaGV、CCV1+OTaGV和OTaGV+CCV1+TPNRBV;建立的多重PCR检测技术特异性强、灵敏度高,可用于茶园茶树上OTaGV、CCV1和TPNRBV 3种病毒的快速检测。研究结果可为福建茶树病毒病的综合防控提供理论依据和技术支持。
茶树病毒;病原鉴定;多重PCR;分子检测;福建省
0 引言
【研究意义】茶树()是山茶科(Theaceae)山茶属()的一种具有重要经济价值的多年生木本植物[1-2],主要分布于亚洲、非洲和南美洲等温暖湿润的热带和亚热带地区[3]。茶是世界上最受欢迎的饮料之一,其富含矿物元素、糖、氨基酸等营养物质[4-5]。此外,茶叶因富含儿茶素等次级代谢物而具有抗菌、抗氧化、消炎镇静等药用功效,不仅有益人体健康,还能保护作物植株免受生物或非生物因素的损害[3]。我国是世界上最大的茶叶生产国和消费国,国内茶树种植主要分布在南部20多个省[6]。其中,福建是我国重要的产茶大省,2020年福建茶园面积为22.93万公顷,茶产量46.14万吨,综合产值达1 300亿元。随着茶树种植规模的不断扩大,茶树上病虫害的发生和危害也逐渐增多,给茶叶的产量和品质造成了不同程度的影响。据报道,茶树上已知的病虫害主要为真菌性病害,少数为细菌、昆虫和线虫危害[7]。病毒病是近年来茶树上新报道的一种病害[8],为防止病毒病的进一步扩散和蔓延,加强茶树病毒种类、分布及快速检测技术研究具有十分重要的现实意义。【前人研究进展】茶树病毒病症状主要表现为叶片褪绿、斑驳、花叶、卷曲、坏死和环斑等,严重时植株死亡[8-14]。作为茶树上的新病害,国内外关于茶树病毒病的研究报道较少。HAO等[8]以我国浙江省茶园采集的茶树病叶为材料,采用高通量测序方法发现了两种新病毒,即茶树坏死环斑病毒(tea plant necrotic ring blotch virus,TPNRBV)和茶树网斑病毒(tea plant line pattern virus,TPLPV),并测定了全基因组序列,建立了用于两种病毒快速检测的普通RT-PCR技术。JO等[9]通过对茶树转录组数据的分析,发现了一种与油茶()上油茶潜隐病毒1(camellia oleifera cryptic virus 1,CoCV1)具有序列同源性的病毒,将其命名为茶树潜隐病毒1(camellia cryptic virus 1,CCV1)。笔者前期采用RT-PCR方法从福建茶树上检测到CCV1,并测定了该病毒的全基因组序列,分析了其序列特征和分子进化关系[10]。Nazerian等[11]在伊朗首次报道了TPNRBV,随后Maruyama等[12]报道了TPNRBV日本分离物(TPNRBV-J)的全基因组序列。Ren等[13]建立了检测TPNRBV 4个RNA片段的SYBR Green Ⅰ荧光定量PCR检测方法,并利用该方法研究了TPNRBV在茶树不同部位的分布和传播特性。Xie等[14]采用ELISA和RT-PCR技术,从我国山东泰安茶树上检测发现了葡萄卷叶伴随病毒7号(grapevine leafroll-associated virus 7,GLRaV-7),该病毒是国内外茶树上的首次报道。WANG等[15]在安徽茶树上发现了一种新的杆状病毒——茶树杆状病毒1(camellia sinensis badnavirus 1,CSBV1),报道了该病毒的全基因组序列。【本研究切入点】茶树是福建省重要的经济作物,茶产业已被列为福建九大支柱产业之一。然而,由病毒引起的茶树病毒病有进一步扩散和蔓延的趋势,并可能成为制约茶叶安全生产的主要因素之一。为保护福建省茶树生产安全、促进茶产业可持续发展,生产上亟需加强茶树病毒病的预防与控制。然而,针对茶树病毒病,目前福建地区缺乏系统全面的研究,病毒发生的种类、分布、侵染类型等尚不明确,关于茶树病毒分子快速检测鉴定技术研究的报道也较少。【拟解决的关键问题】系统调查福建省茶树病毒病的发生和危害情况,通过高通量测序、PCR和RT-PCR检测各地区采集的疑似病样,明确福建省茶树病毒的主要种类、分布特征及侵染类型,并建立用于茶树病毒快速检测的多重PCR检测技术,以期为福建省茶树病毒病的早期预警与防控提供理论依据和技术支持。
1 材料与方法
试验于2019—2023年在福建农林大学植物病毒研究所和福州海关技术中心完成。
1.1 材料
2019年1月至2023年3月,在福建省福州、南平、宁德、泉州、漳州、厦门、三明、莆田和龙岩9个茶树主要种植区采集具有疑似病毒感染症状(褪绿、斑驳、花叶、卷曲、坏死和环斑等)病株样品1 869份,其中福州414份、南平328份、宁德153份、泉州345份、漳州62份、厦门65份、三明50份、莆田81份、龙岩371份。样品采集后液氮速冻,于-80 ℃保存备用。
Random Hexamer Primer购自Thermo Fisher Scientific(中国)有限公司;RNasin Plus RNase Inhibitor(40 U·μL-1)、M-MLV RT 5×Buffer、M-MLV Reverse Transcriptase(200 U·μL-1)、dNTP Mix(10 mmol·L-1)、Go Taq Green Master Mix(2×)购自美国Promega公司;DL 1000 DNA Marker购自宝生物(大连)有限公司产品;HiScriptⅡOne Step RT-PCR Kit(Dye Plus)购自南京诺唯赞生物科技股份有限公司;胶回收试剂盒EasyPure Quick Gel Extraction Kit、pEASY-T5 Zero载体、Trans-T1感受态细胞载体购自北京全式金生物技术有限公司。
1.2 高通量测序
选取具有褪绿、斑驳、花叶、卷曲、坏死和环斑等典型症状的茶树病株进行高通量测序,测序委托生工生物工程(上海)股份有限公司完成。
1.3 引物设计
根据高通量测序结果及GenBank上已报道的病毒序列设计特异性引物(表1),用于后续PCR/RT-PCR对采集的茶树叶片进行病毒检测和验证,引物由生工生物工程(上海)股份有限公司合成。
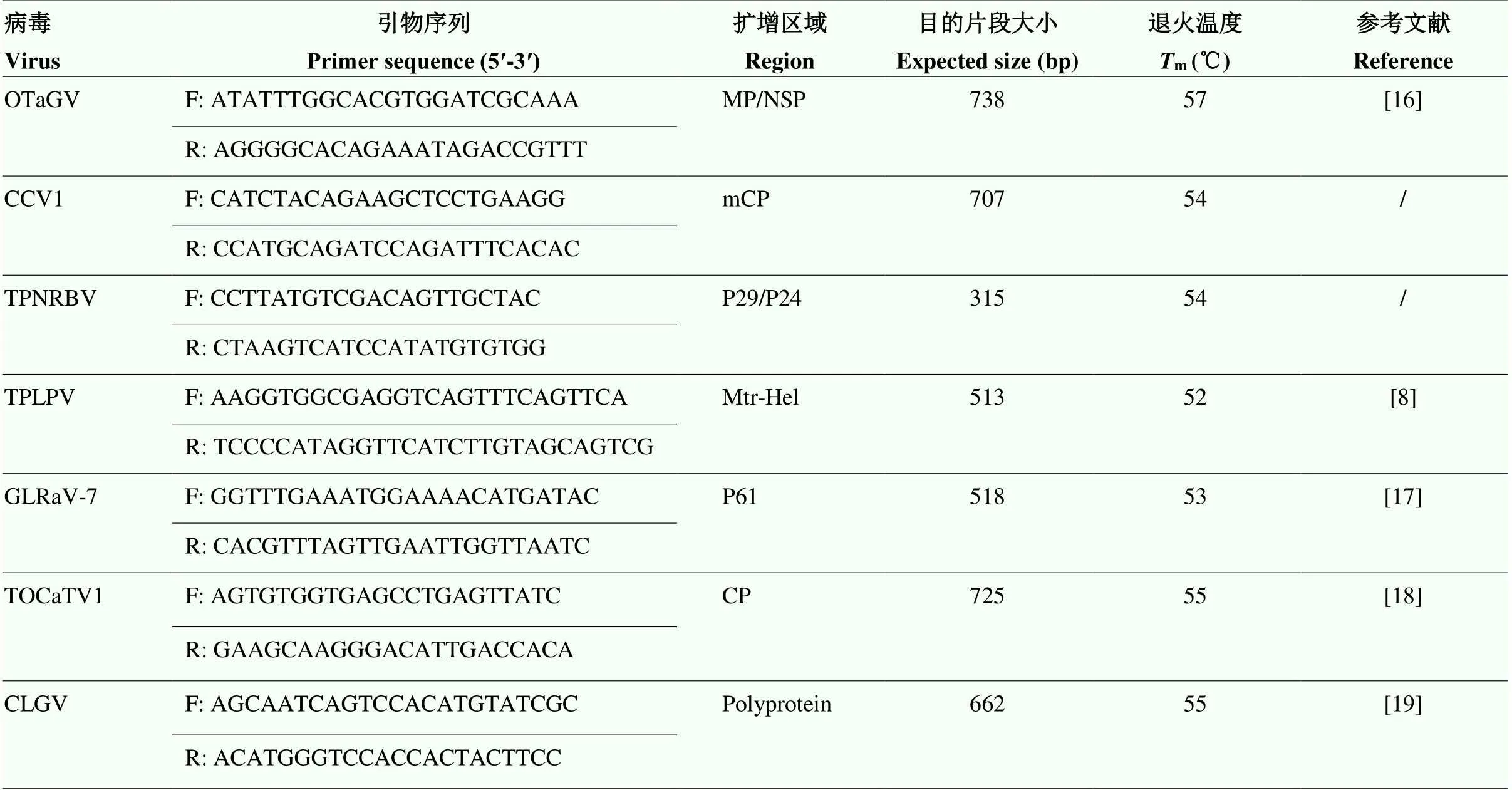
表1 福建茶树病毒检测所用引物
F:上游引物Forward primer;R:下游引物Reverse primer;Mtr-Hel:甲基化转移酶-解旋酶Methyltransferase-helicase;NSP:核穿梭蛋白nuclear shuttle protein
1.4 总核酸提取和反转录
称取茶树叶片100 mg,采用CTAB法[20]提取总核酸,总核酸-80 ℃保存备用。DNA病毒检测直接以总核酸为模板,RNA病毒检测以总核酸反转录合成的cDNA为模板。反转录反应体系(20 μL):在PCR管中加入7 μL ddH2O、1 μL Random Hexamer Primer(0.2 μg·μL-1)、3 μL模板,72 ℃ 10 min,冰上5 min,瞬时离心;再加入以下试剂:5 μL M-MLV RT 5× Buffer、2 μL dNTP Mix(10 mmol·L-1)、1 μL RNasin Plus RNase Inhibitor(40 U·μL-1)、1 μL M-MLV Reverse Transcriptase(200 U·μL-1),42 ℃1 h,72 ℃ 10 min合成cDNA。
1.5 PCR扩增及克隆测序
PCR扩增反应体系(25 μL):病毒上下游引物(10 μmol·L-1)各1 μL,12.5 μL 2×Go Taq Green Master Mix,2 μL模板,8.5 μL ddH2O。PCR扩增反应条件:94 ℃ 5 min;94 ℃ 30 s、X ℃(各病毒具体退火温度见表1)45 s、72 ℃ 1 min,35个循环,最后一轮循环后72 ℃延伸10 min。目的片段经EasyPure Quick Gel Extraction Kit回收后,连接到pEASY-T5 Zero载体,连接产物转化Trans-T1感受态细胞中,然后涂布含氨苄青霉素的LB培养基,筛选阳性克隆,委托生工生物工程(上海)股份有限公司进行测序。
1.6 序列分析及系统发育树重建
序列测定的结果采用NCBI中的BLAST程序进行一致性分析。从GenBank中已报道的各病毒基因序列中,分别选取代表性基因序列作为参考序列,采用MEGAX[21]文献软件MUSCLE算法[22]对核苷酸序列进行多重比对后,通过IQ-tree软件[23]应用最大似然法(maximum likelihood,ML)重建系统发育树。使用ModelFinder[24]分析确定CCV1和TPNRBV数据集最适的核苷酸替代模型分别K80+I和HKY+F,并基于BIC标准设置相关参数,发育树中各分支节点的可靠性通过超快自举法(Ultrafast bootstrap)10 000次重复抽样进行评估。
1.7 多重PCR建立及优化
根据GenBank上已报道的OTaGV序列,设计1对特异性引物OTV-F1/OTV-R1(F1:5′-TAGGAGAC GCATTGTGGCAGAAG-3′,R1:5′-GTTAGTCTTGCC TTCGATGCTGC-3′,预期扩增目的片段大小为446 bp),与1.3中TPNRBV、CCV1的引物对组成多重PCR引物组,引物委托生工生物工程(上海)股份有限公司合成。建立多重PCR检测方法,PCR扩增反应体系(20 μL):病毒上下游引物(10 μmol·L-1)各0.8 μL,10 μL 2×One Step Mix(Dye Plus),1 μL One Step Enzyme Mix,2 μL模板,2.2 μL ddH2O。PCR扩增反应条件:50 ℃ 30 min;94 ℃ 3 min;94 ℃ 30 s,53 ℃ 30 s,72 ℃ 1 min,35个循环,最后一轮循环后72 ℃延伸7 min。在建立的多重PCR检测方法基础上,分别对退火温度、引物浓度、循环数等反应体系和反应程序进行优化。退火温度分别设53、55、57、59和61 ℃ 5个处理,引物浓度分别设0.1、0.2、0.3、0.4和0.5 μmol·L-15个处理,循环数分别设25、30、35和40 4个处理。在不同处理下分别进行多重PCR,确定多重PCR的最佳反应体系和反应程序。
1.8 多重PCR特异性、灵敏度测定
根据建立的多重PCR的反应体系和反应程序,对OTaGV、CCV1、TPNRBV及3种病毒不同复合侵染类型的茶树样品进行多重PCR扩增,以健康茶树样品为阴性对照,测定多重PCR特异性;将CCV1、TPNRBV和OTaGV阳性样品提取的总核酸等量混合后按10倍梯度稀释,分别稀释至10-1、10-2、10-3、10-4、10-5和10-6倍后作为模板进行多重PCR扩增,以健康茶树样品为阴性对照,测定多重PCR灵敏度,并与单一PCR进行比较。
1.9 多重PCR实际应用
以茶园采集的60份病样为检测对象,应用建立的多重PCR检测技术进行病毒检测,同时用单一PCR进行验证,评价该技术的实际检测效果。
2 结果
2.1 福建省茶树病毒病的田间症状
在福建省茶树病毒病的调查中发现,福州、南平、宁德、泉州、漳州、厦门、三明、莆田和龙岩9个地区的茶树田间症状主要表现为叶片褪绿、斑驳、花叶、卷曲、坏死和环斑等多种类型(图1),其中以褪绿、花叶、坏死和环斑较为普遍。在同一棵茶树病株上,有时会出现多种症状。

A:褪绿、斑驳Chlorosis, mottle;B:坏死、环斑Necrosis, ringspots;C:皱缩、泡斑Crinkle, blotch;D:卷曲Curl
图1 茶树病毒病主要症状
Fig. 1 Main symptoms caused by tea plant viruses
2.2 高通量测序结果及PCR/RT-PCR验证
茶树病株高通量测序后共获得75 922 118 raw reads(读长),通过质量控制后进行序列拼接组装,得到592 335个contigs(重叠群)。经BLAST比对和注释分析,发现与植物病毒及类病毒相关的contigs中,与3种已报道能够侵染山茶属植物的病毒(OTaGV、CCV1和TPNRBV)核苷酸序列一致性较高,为94.9%—100%。根据高通量测序结果和GenBank上已报道的3种病毒基因组序列设计特异性引物,进行PCR/RT-PCR验证。验证结果表明,仅OTaGV、CCV1和TPNRBV分别扩增出预期大小的目的片段,而其余病毒均未扩增出目的片段(图2)。将PCR产物进行克隆测序和BLAST比对,结果显示OTaGV、CCV1和TPNRBV目的片段核苷酸序列分别与GenBank已报道的OTaGV江西分离物(GenBank登录号:MN161582)、CCV1福建分离物(GenBank登录号:ON807098)和TPNRBV日本分离物(GenBank登录号:LC566239)核苷酸序列一致性为91.6%、100%和95.5%,进一步证实福建茶树样品中存在OTaGV、CCV1和TPNRBV侵染。
2.3 福建茶树病样的病毒检测
针对高通量测序发现的3种病毒(OTaGV、CCV1、TPNRBV)以及茶树上已报道的TPLPV、GLRaV-7,采用PCR、RT-PCR对采集的1 869份样品进行检测。结果表明,利用OTaGV、CCV1、TPNRBV特异性引物能够扩增到与预期大小相近的738、707和315 bp目的片段(图3),而TPLPV、GLRaV-7特异性引物未扩增到任何特异性目的片段。福建茶树病样的病毒检测结果表明(表2),1 869份样品中检出病毒的有1 258份,病毒检出率为67.31%;检出的病毒种类有3种,分别为OTaGV、CCV1和TPNRBV,检出率分别为48.90%、17.98%和26.75%,其中以OTaGV检出率最高;OTaGV在福州、南平、宁德、泉州、漳州、厦门、三明、莆田和龙岩9个地区均有分布,其中漳州地区OTaGV检出率最高,为96.77%;CCV1在福州、南平、宁德、泉州、漳州、三明、莆田和龙岩8个地区均有分布,其中三明地区CCV1检出率最高,为66.00%;TPNRBV在福州、南平、宁德、泉州和漳州5个地区均有分布,其中泉州地区TPNRBV检出率最高,为79.13%;在福建省9个地区中,厦门地区仅检出OTaGV,三明、莆田和龙岩地区检出OTaGV、CCV1,其他5个地区同时检出OTaGV、CCV1和TPNRBV。

M:DNA分子量标准DNA Marker (DL 1000);1:OTaGV;2:OTaGV检测的阴性对照Negative control for OTaGV;3:CCV1;4:CCV1检测的阴性对照Negative control for CCV1;5:TPNRBV;6:TPNRBV检测的阴性对照Negative control for TPNRBV
图2 3种茶树病毒PCR/RT-PCR扩增结果
Fig. 2 PCR/RT-PCR amplification results of three types of tea plant viruses
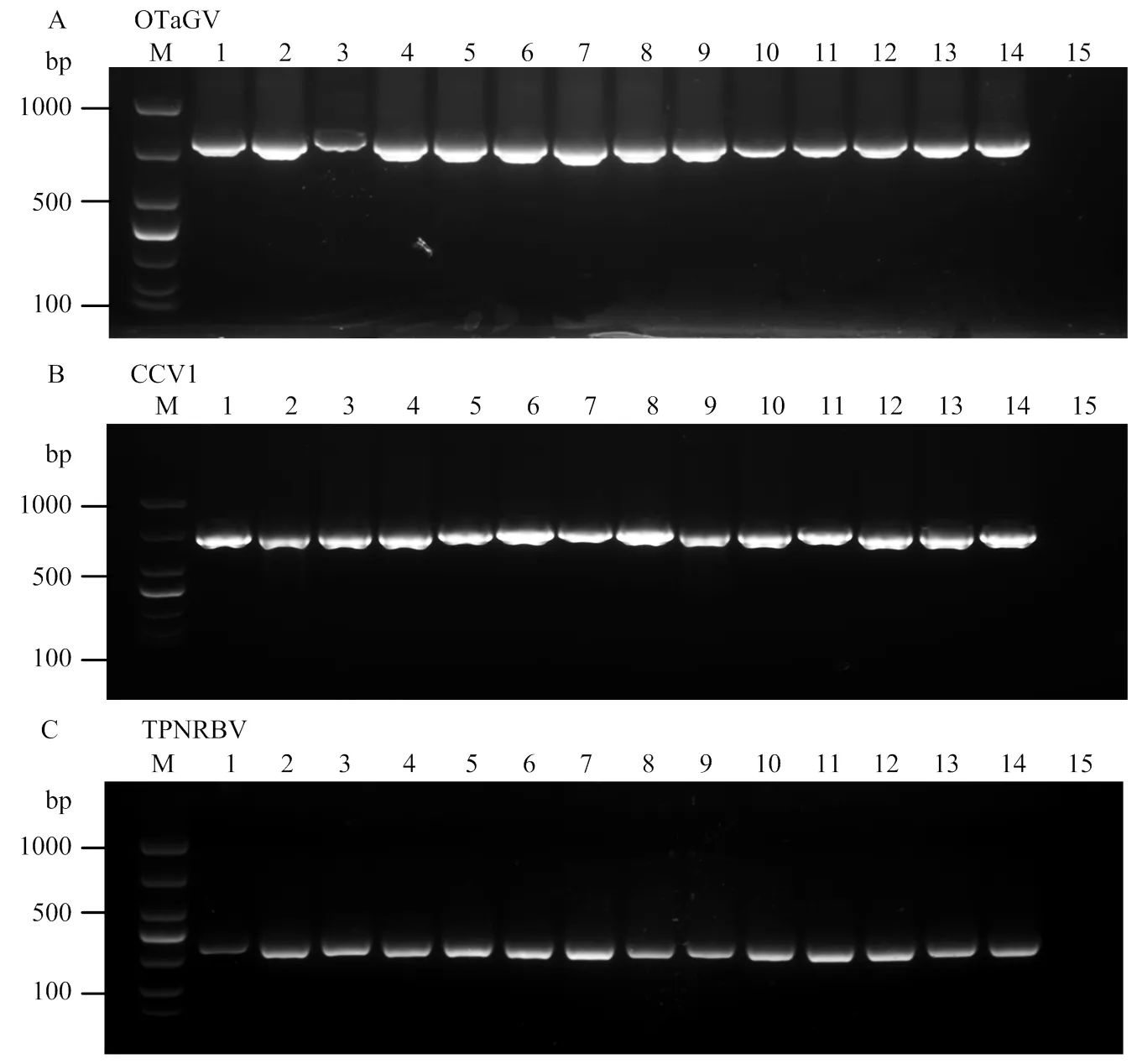
M:DNA分子量标准DNA Marker (DL 1000);1—14:茶树样品Tea plant sample;15:阴性对照Negative control
图3 福建茶树部分病样OTaGV、CCV1和TPNRBV检测结果
Fig. 3 The detection results of OTaGV, CCV1 and TPNRBV in some susceptible samples of tea plant from Fujian Province
病毒侵染情况分析结果显示(表3),在检出病毒阳性的1 258份茶树病样中,有807份样品为单一病毒侵染,TPNRBV、OTaGV、CCV1单独侵染率分别为21.38%、37.20%和5.56%;其余451份样品为2种或3种病毒复合侵染,复合侵染率达35.85%,OTaGV+CCV1、TPNRBV+CCV1、TPNRBV+OTaGV、OTaGV+CCV1+TPNRBV 4种类型复合侵染检出率分别为17.49%、0.40%、14.71%和3.26%。在福建省9个地区中,福州、南平、宁德、泉州、漳州、三明和莆田7个地区检测到病毒复合侵染,其中漳州地区病毒复合侵染检出率最高,为85.00%,宁德地区病毒复合侵染检出率最低,为23.03%。
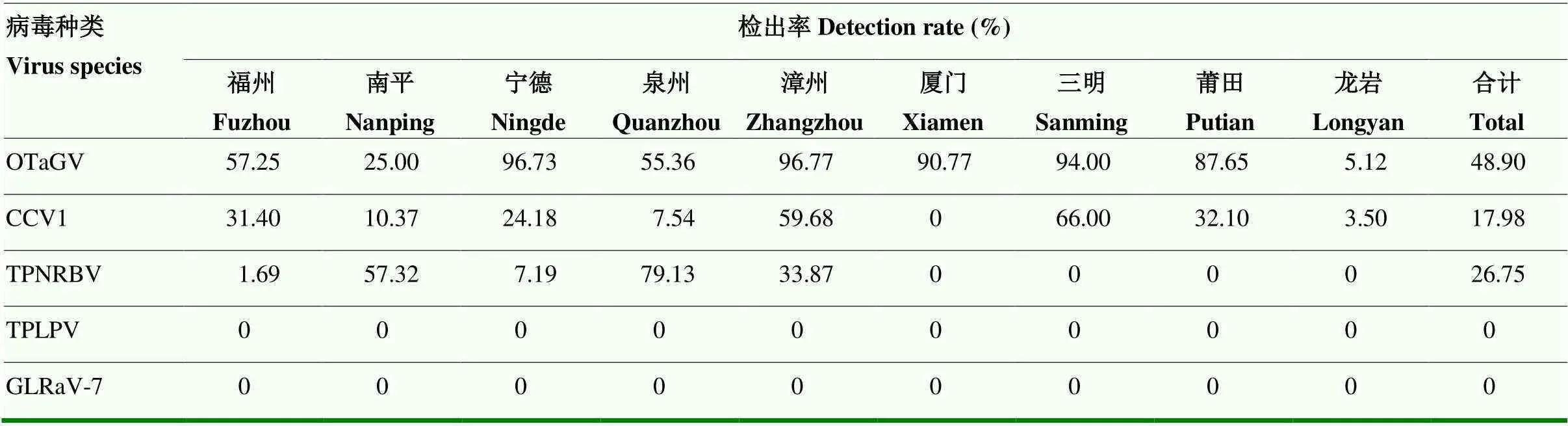
表2 福建各地区茶树病毒检测结果

表3 福建各地区茶树病毒单独侵染和复合侵染情况
2.4 CCV1和TPNRBV分离物的系统发育分析
选取CCV1、TPNRBV单独侵染的茶树样品,RT-PCR检测获得的CCV1、TPNRBV产物分别进行克隆测序,所测目的片段核苷酸序列提交NCBI,GenBank登录号分别为OR282452、OR282451。BLAST比对结果表明,本研究获得的CCV1分离物与CCV1福建分离物FJ_SH90、FJ_SH79、FJ_SH63(GenBank登录号:ON807096—ON807098)以及韩国分离物Won(GenBank登录号:MH898484)RNA3的一致性最高(均为99.72%),与其他分离物RNA3的核苷酸序列一致性为96.46%—99.58%;本研究获得的TPNRBV分离物与GenBank已报道的TPNRBV分离物核苷酸序列一致性为95.24%—96.54%,其中与日本分离物J(GenBank登录号:LC566239)一致性最高(96.5%)。为进一步明确本研究获得的CCV1、TPNRBV分离物与GenBank中已报道其他分离物的进化关系,选取CCV1、TPNRBV其他不同地区代表性分离物,采用ML法构建系统发育树(图4),结果显示本研究获得的CCV1分离物与已报道的福建分离物FJ-SH104(GenBank登录号:ON807095)亲缘关系最近、TPNRBV分离物与福建分离物QZHA92(GenBank登录号:OQ948454)亲缘关系最近。
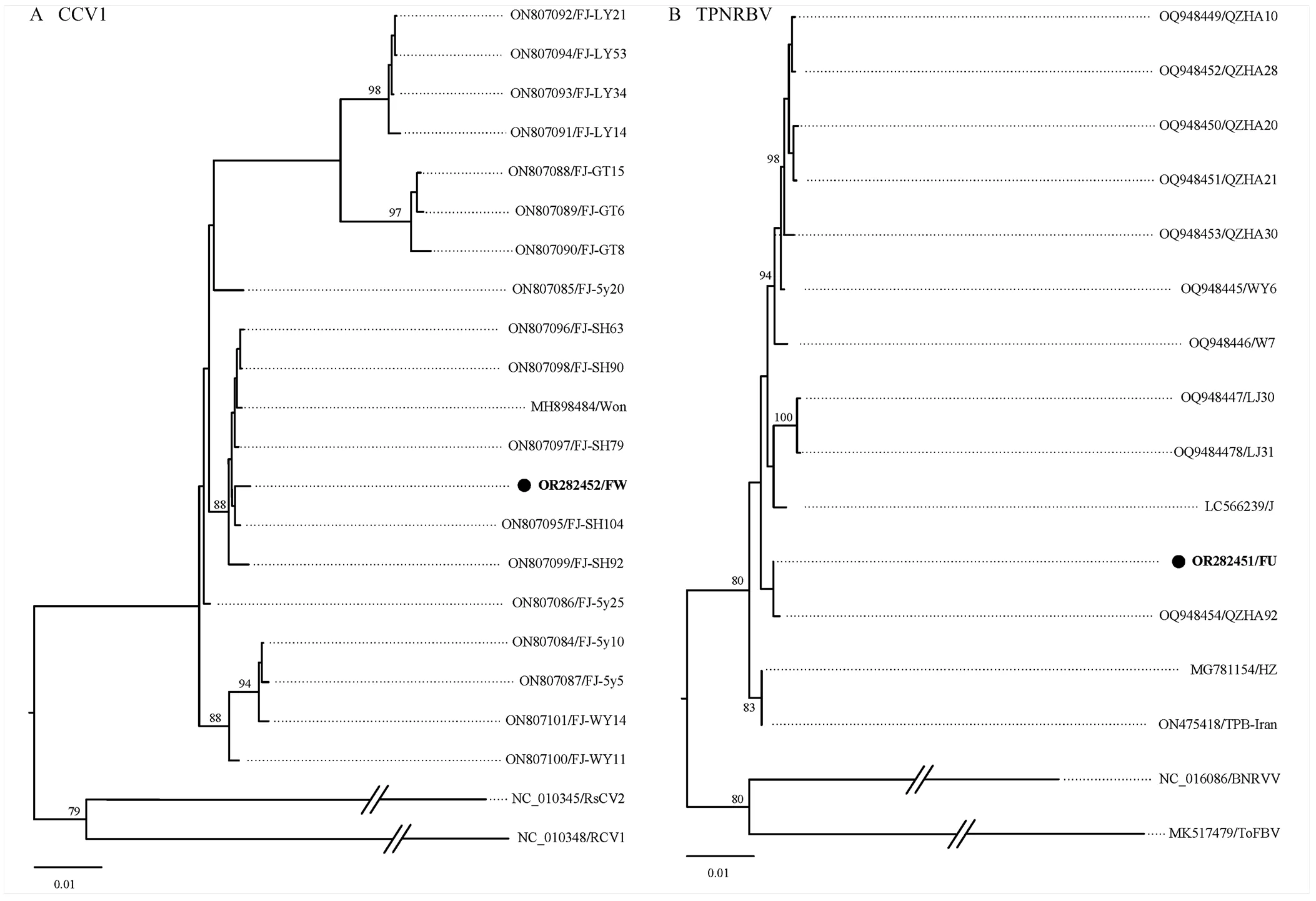
分支节点上的数值为超快自举值(>75%),黑色圆点显示的为本研究获得的病毒分离物Numbers above branches on the left indicated Ultrafast bootstrap values (only shown>75%). The viral isolates from current study were marked with black solid circle
图4 基于最大似然重建的CCV1和TPNRBV系统发育树
Fig. 4 Phylogenetic tree of CCV1 and TPNRBV based on maximum likelihood (ML) algorithm
2.5 多重PCR的优化
在单一PCR基础上,通过固定单一变量,对多重PCR反应条件和程序进行优化,结果表明退火温度为57 ℃时,3个病毒的目的片段条带最为清晰、亮度相近(图5-A);引物浓度各为0.3 μmol·L-1时,3个病毒的目的片段条带最亮,且亮度均一(图5-B);循环数为35以下时,3个病毒的目的片段条带亮度不一致,当循环数达到35、40时,各病毒目的片段条带均较明亮(图5-C)。经综合考虑,确定多重PCR最佳反应体系为总体积20 μL:病毒上下游引物(10 μmol·L-1)各0.6 μL,10 μL 2×One Step Mix(Dye Plus),1 μL One Step Enzyme Mix,1 μL模板,4.4 μL ddH2O。反应程序:50 ℃ 30 min;94 ℃ 3 min;94 ℃ 30 s、57 ℃ 30 s、72 ℃ 1 min,35个循环,最后一轮循环后72 ℃延伸7 min。
2.6 多重PCR的特异性
利用优化建立的多重PCR检测技术对OTaGV、CCV1、TPNRBV 3种病毒不同侵染类型的茶树样品进行检测,仅从OTaGV、CCV1、TPNRBV 3种病毒复合侵染样品上同时扩增到大小约446、707和315 bp特异性目的片段,从OTaGV、TPNRBV复合侵染样品上同时扩增到大小约446和315 bp特异性目的片段,从CCV1、TPNRBV复合侵染样品上同时扩增到大小约707、315 bp特异性目的片段,从OTaGV、CCV1复合侵染样品上同时扩增到大小约446和707 bp特异性目的片段,从OTaGV、CCV1、TPNRBV单独侵染样品上分别仅扩增到大小约446、707和315 bp特异性目的片段,而从健康茶树样品上未扩增到特异性目的片段(图5-D),表明建立的多重PCR具有较强的特异性。

M:DNA分子量标准DNA Marker (DL1000)。A:多重PCR退火温度优化结果Optimization results of annealing temperature for multiplex PCR;1—5:53、55、57、59、61 ℃;6:阴性对照Negative control。B:多重PCR引物浓度优化结果Optimization results of primer concentration for multiplex PCR;1—5:0.1、0.2、0.3、0.4、0.5 μmol·L-1;6:阴性对照Negative control。C:多重PCR循环数优化结果Optimization results of cycle for multiplex PCR;1—4:25、30、35和40个循环25, 30, 35 and 40 cycles, respectively;5:阴性对照Negative control。D:多重PCR特异性Specificity of multiplex PCR;1:OTaGV、CCV1、TPNRBV复合侵染样品Sample co-infected by OTaGV, CCV1 and TPNRBV;2:OTaGV、TPNRBV复合侵染样品Sample co-infected by OTaGV and TPNRBV;3:CCV1、TPNRBV复合侵染样品Sample co-infected by CCV1 and TPNRBV;4:OTaGV、CCV1复合侵染样品Sample co-infected by OTaGV and CCV1;5:TPNRBV单独侵染样品Sample infected by TPNRBV;6:OTaGV单独侵染样品Sample infected by OTaGV;7:CCV1单独侵染样品Sample infected by CCV1;8:阴性对照Negative control
图5 多重PCR优化和特异性结果
Fig. 5 Optimization and specificity results of multiplex PCR
2.7 多重PCR的灵敏度
将OTaGV、CCV1、TPNRBV 3种病毒阳性样品提取的总核酸等量混合后按10倍梯度稀释后分别进行多重PCR和单一PCR检测。结果发现多重PCR最低能同时检测到稀释至10-4倍OTaGV、TPNRBV和10-3倍CCV1(图6-A),单一PCR最低均能检测稀释至10-4倍OTaGV、CCV1和TPNRBV(图6-B—D)。与多重PCR相比,单一PCR灵敏度略高,且在相同稀释倍数模板下各病毒目的片段条带亮度均明显较多重PCR明亮。
2.8 茶园样品的多重PCR检测
利用优化建立的多重PCR检测技术对60份茶园样品进行检测。结果显示OTaGV、CCV1、TPNRBV、OTaGV+CCV1、TPNRBV+CCV1、TPNRBV+OTaGV、OTaGV+CCV1+TPNRBV阳性样品检出率分别为26.7%(16/60)、3.3%(2/60)、1.7%(1/60)、45.0%(27/60)、1.7%(1/60)、13.3%(8/60)和5.0%(3/60)(表4),该检测结果与样品单一PCR检测结果完全相符,符合率为100%,表明建立的多重PCR检测技术能够有效用于茶园样品中OTaGV、CCV1和TPNRBV的检测。

A:多重PCR Multiplex PCR;B:CCV1单一PCR Single PCR of CCV1;C:OTaGV单一PCR Single PCR of OTaGV;D:TPNRBV单一PCR Single PCR of TPNRBV。M:DNA分子量标准DNA Marker (DL1000);1—6:101、102、103、104、105和106倍稀释101, 102, 103, 104, 105, and 106times dilution, respectively;7:阴性对照Negative control
图6 多重PCR灵敏度
Fig. 6 Sensitivity of multiplex PCR

表4 茶园样品多重PCR检测结果
3 讨论
茶树是福建省特色经济作物,茶产业也已成为福建省重要特色优势产业。近年来,随着福建省茶树种植面积和规模化程度不断提高,茶树上病害发生种类和数量也在不断增多,严重威胁了茶叶的产量和品质。针对茶树上新报道的病毒病,福建地区至今尚未开展系统的调查和鉴定。本研究采用高通量测序和PCR、RT-PCR相结合的方法对福建茶树病毒种类进行了检测鉴定,明确了茶树病毒的主要种类、分布特征及侵染类型,并建立了多重快速检测技术。
3.1 福建省茶树病毒主要种类
高通量测序作为检测病毒的一项重要技术手段,目前已被越来越多地用于农作物上病毒的检测鉴定,除检出已知病毒外,还发现了多种新病毒[25-32]。本研究通过高通量测序、PCR和RT-PCR,从采集的1 869份茶树样品中检测出携带病毒的样品1 258份,发现了OTaGV、CCV1和TPNRBV 3种病毒,其中OTaGV为福建地区茶树上首次检出。病毒检出率从高到低依次为OTaGV、TPNRBV和CCV1。OTaGV检出率为48.90%,发生最为普遍,在福建省9个地区均有分布;TPNRBV检出率26.75%,在5个地区有分布;而CCV1检出率为17.98%,除厦门外,在福建省其他8个地区均有分布。从病毒检出率和分布范围推测OTaGV、TPNRBV和CCV1为当前福建省茶树病毒主要种类。我国浙江、山东茶树上先后报道了TPLPV、GLRaV-7的发生,但本研究中未检出这两种病毒。汤亚飞等[33]研究发现,植株体内的某些病毒因含量过低,导致高通量测序可能无法检测到。为防止NGS的漏检,本研究利用TPLPV、GLRaV-7特异性引物对所有样品进行了RT-PCR检测,但均未检出TPLPV和GLRaV-7。分析原因一方面可能是病毒分布存在地理差异,福建茶树上尚无TPLPV和GLRaV-7侵染,另一方面也有可能是未采集到携带有这两种病毒的样品。通过系统发育树分析,发现CCV1分离物与已报道的福建分离物亲缘关系最近、TPNRBV分离物与福建分离物亲缘关系最近。由于GenBank上公布的OTaGV相应序列仅有一条,因此该病毒未构建系统发育树。OTaGV是在油茶上发现的一种新的双生病毒[16],本研究发现OTaGV也可以侵染茶树,该OTaGV茶树分离物的全基因组序列有待进一步测定。
3.2 福建省茶树病毒分布特征及侵染类型
在本研究中,OTaGV、CCV1和TPNRBV 3种病毒在福建不同地区的分布呈现出明显的差异。OTaGV在漳州地区检出率最高(96.77%),龙岩地区检出率最低(5.12%);CCV1在三明地区检出率最高(66.00%),厦门地区未检出;TPNRBV在泉州地区检出率最高(79.13%),厦门、三明、莆田和龙岩4个地区未检出。上述结果表明,福建9个地区茶树的优势病毒存在显著的不同,这可能与各地区气候条件、茶树品种抗性和栽培制度以及病毒传播特性等不同有关。在自然界中,植物病毒复合侵染现象较为普遍[34]。刘勇等[35]报道,我国蔬菜作物中普遍存在多种病毒复合侵染现象,已发现2—6种病毒复合侵染。本研究1 258份病毒检出为阳性的茶树样品中,807份样品为单一病毒侵染,总单独侵染率为64.15%,其中OTaGV、TPNRBV和CCV1单独侵染率分别为37.20%、21.38%和5.56%,451份样品为2种或3种病毒复合侵染,总复合侵染率为35.85%。单一病毒侵染率约为复合侵染率的两倍,说明福建茶树病毒目前仍以单独侵染为主,同时存在较多的复合侵染。茶树病毒复合侵染类型有OTaGV+CCV1、TPNRBV+CCV1、TPNRBV+ OTaGV、OTaGV+CCV1+TPNRBV 4种,检出率分别为17.49%、0.40%、14.71%、3.26%,其中以OTaGV+ CCV1、TPNRBV+OTaGV检出率较高,表明复合侵染是以OTaGV与另一种病毒(CCV1或TPNRBV)的复合侵染为主。在福建省9个地区中,除龙岩、厦门地区未发现复合侵染外,其他7个地区均检测到病毒复合侵染,其中以漳州地区病毒复合侵染检出率最高(85.00%)、宁德地区病毒复合侵染检出率最低(23.03%)。病毒的复合侵染有可能会加重对茶树生产的危害,但目前尚无相关的研究数据,这方面有待进一步研究。
3.3 茶树病毒多重PCR检测
多重PCR检测技术具有快速、准确、高效和检测成本低的优点,在植物病毒常规检测上具有重要应用价值[36]。Xu等[37]根据百合无症病毒(lily symptomless virus)、百合斑驳病毒(lily mottle virus)、黄瓜花叶病毒(cucumber mosaic virus)和车前草花叶病毒(plantago asiatica mosaic virus)的基因组序列保守区域设计特异性引物,建立了同时检测4种百合病毒的多重RT-PCR检测方法。吐逊艾力·艾孜提力等[38]为了快速检测危害无花果的病毒种类,研究建立了一种同时检测无花果斑点相关病毒(fig fleck-associated virus)、无花果叶斑相关病毒(fig leaf mottle-associated virus)和无花果花叶病毒(fig mosaic virus)3种无花果病毒的多重RT-PCR检测体系。本研究针对福建茶树病毒的主要种类和发生特点,建立了可用于OTaGV、CCV1和TPNRBV快速检测的多重PCR技术,适用于目前茶树病毒常见侵染类型的检测。OTaGV为DNA病毒,而TPNRBV、CCV1为RNA病毒,传统的核酸提取方法是将RNA、DNA分开提取,然后将RNA反转录得到的cDNA与DNA混合后作为模板。本研究采用改良的CTAB法[20],提取的总核酸既有RNA,也有DNA,直接作为多重PCR的模板,不仅简化了操作步骤、节约了检测成本,还减少了实验污染。此外,多重PCR采用了一步法RT-PCR扩增,RNA和DNA病毒在同一反应体系中进行检测,进一步提高了检测效率。引物浓度、退火温度、循环数等因素常常影响多重PCR检测技术的建立。本研究在单重PCR反应体系和反应条件基础上,通过引物浓度、退火温度、循环数因素优化,最终建立了同时检测茶树上OTaGV、CCV1和TPNRBV 3种病毒的多重PCR检测技术。特异性和灵敏度是检验多重PCR方法是否成功的关键。本研究建立的多重PCR检测技术,对OTaGV、CCV1、TPNRBV具有良好的特异性和灵敏度。利用建立的多重PCR检测技术对采集的福建茶树疑似病样进行了实际检测,检测结果与单重PCR完全一致,进一步验证了该技术的实用性。
4 结论
对福建省9个地区采集的1 869份样品进行了检测鉴定,明确了当前福建茶树的主要病毒种类为OTaGV、CCV1、TPNRBV,其中OTaGV为福建地区首次报道,并发现该病毒也可以侵染茶树;在检出的3种病毒中,OTaGV分布范围最广,在福建9个地区均有分布,而CCV1、TPNRBV分别在8个、5个地区有分布;福建茶树病毒目前以单独侵染为主,但同时存在较为普遍的复合侵染现象,复合侵染类型包括TPNRBV+CCV1、TPNRBV+OTaGV、CCV1+OTaGV和OTaGV+ CCV1+TPNRBV;建立了快速、准确、灵敏和高效的多重PCR检测技术,可用于茶园茶树上OTaGV、CCV1和TPNRBV的检测鉴定。
[1] Bennetzen J. Culturing better tea research. Nature, 2019, 566(7742): S5.
[2] Wambulwa M C, Meegahakumbura M K, Kamunya S, Wachira F NFrom the wild to the cup: tracking footprints of the tea species in time and space. Frontiers in Nutrition, 2021, 8: 706770.
[3] Drew L. The growth of tea. Nature, 2019, 566(7742): S2-S4.
[4] Brody H. Tea. Nature, 2019, 566(7742): S1.
[5] Das P R, Kim Y, Hong S J, Eun J B. Profiling of volatile and non-phenolic metabolites—Amino acids, organic acids, and sugars of green tea extracts obtained by different extraction techniques. Food chemistry, 2019, 296: 69-77.
[6] 黄晓燕. 图说茶文化. 北京: 华文出版社, 2009.
Huang X Y. Illustration of tea culture. Beijing: Sino-Culture Press, 2009. (in Chinese)
[7] 陈宗懋, 陈雪芬. 世界茶树病原名录. 茶叶科学, 1988, 8(2): 65-76.
Chen Z M, Chen X F. A world list of pathogens reported on tea plant. Journal of Tea Science, 1988, 8(2): 65-76. (in Chinese)
[8] Hao X Y, Zhang W, Zhao F, Liu Y, Qian W, Wang Y, Wang L, Zeng J, Yang Y, Wang XDiscovery of plant viruses from tea plant ((L.) O. Kuntze) by metagenomic sequencing. Frontiers in Microbiology, 2018, 9: 2175.
[9] Jo Y, Cho W K. Identification of viruses belonging to the familyfrom plant transcriptomes. bioRxiv, 2020, doi: 10. 1101/2020.03.11.988063.
[10] 许瑜婷, 丁莹, 沈建国, 陈细红, 章淑玲, 杜振国, 高芳銮. 福建茶树潜隐病毒1的检测及全基因组序列特征. 植物病理学报, 2023, 53(3): 386-394.
Xu Y T, Ding Y, Shen J G, Chen X H, Zhang S L, Du Z G, Gao F L. Detection of camellia cryptic virus 1 in Fujian Province and molecular characterization of its complete genome. Acta Phytopathologica Sinica, 2023, 53(3): 386-394. (in Chinese)
[11] Nazerian E, Bayat H. Occurrence of tea plant necrotic ring blotch virus in Iran. Journal of Plant Protection Research, 2021, 61(2): 200-202.
[12] Maruyama N, Iwabuchi N, Nishikawa M, Nijo T, Yoshida T, Kitazawa Y, Maejima K, Namba S, Yamaji Y. Complete genome sequence of tea plant necrotic ring blotch virus detected from a tea plant in Japan. Microbiology Resource Announcements, 2022, 11(6): e0032322.
[13] Ren H, Chen Y, Zhao F, Ding C, Zhang K, Wang L, Yang Y, Hao X, Wang X. Quantitative distribution and transmission of tea plant necrotic ring blotch virus in. Forests, 2022, 13(8): 1306.
[14] Xie X, Zhu C, Han X. First report on the occurrence of grapevine leafroll-associated virus 7 in tea plants. Journal of Plant Pathology, 2022, 104(1): 455.
[15] Wang F, Zhu J Y, Zhu Y, Yan D K, Dong Q, Jegede O J, Wu Q F. Complete genome sequence of a new badnavirus infecting a tea plant in China. Archives of virology, 2022, 167(12): 2811-2815.
[16] 彭梁. 油茶中两种新病毒的基因克隆及分子特性分析[D].南昌: 南昌大学, 2019.
Peng L. Genome determination and characterization of two novel viruses infectingC. Abel[D]. Nanchang: Nanchang University, 2019. (in Chinese)
[17] LÜ M D, Li X M, Guo R, Li M J, Liu X M, Wang Q, Cheng Y Q. Prevalence and distribution of grapevine leafroll-associated virus 7 in China detected by an improved reverse transcription polymerase chain reaction assay. Plant Pathology, 2014, 63(5): 1168-1176.
[18] Zhang Z Y, Huang H, Han X X, Li R, Wu L P, Wu L.Identification and molecular characterization of tea-oil camellia- associated totivirus 1. Archives of virology, 2021, 166(8): 2347-2351.
[19] Li R H, Zheng L P, Cao M J, Wu L P, Normandy P, Liu H W. First identification and molecular characterization of a new badnavirus infecting camellia. Archives of virology, 2020, 165(9): 2115-2118.
[20] Li R, Mock R, Huang Q, Abad J, Hartung J, Kinard G. A reliable and inexpensive method of nucleic acid extraction for the PCR-based detection of diverse plant pathogens. Journal of Virological Methods, 2008, 154(1/2): 48-55.
[21] Kumar S, Stecher G, Li M, Knyaz C, Tamura K. MEGA X: Molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6): 1547-1549.
[22] Edgar R C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research, 2004, 32(5): 1792-1797.
[23] Nguyen L T, Schmidt H A, von Haeseler A, Minh B Q. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum likelihood phylogenies. Molecular Biology and Evolution, 2015, 32(1): 268-274.
[24] Kalyaanamoorthy S, Minh B Q, Wong T K F, von Haeseler A, Jermiin L S. ModelFinder: fast model selection for accurate phylogenetic estimates. Nature Methods, 2017, 14(6): 587-589.
[25] Adams I P, Glover R H, Monger W A, Mumford R, Jackeviciene E, Navalin-skiene M, Samuitiene M, Boonham N. Next-generation sequencing and metagenomic analysis: A universal diagnostic tool in plant virology. Molecular Plant Pathology, 2009, 10(4): 537-545.
[26] Rott M, Xiang Y, Boyes I, Belton M, Saeed H, Kesanakurti P, Hayes S, Lawrence T, Birch C, Bhagwat B, RasT H. Application of next generation sequencing for diagnostic testing of tree fruit viruses and viroids.Plant Disease, 2017, 101(8): 1489-1499.
[27] Barba M, Czosnek H, Hadidi A. Historical perspective, development and applications of next-generation sequencing in plant virology. Viruses, 2014, 6(1): 106-136.
[28] Roossinck M J, Martin D P, Roumagnac P. Plant virus metagenomics: advances in virus discovery. Phytopathology, 2015, 105(6): 716-727.
[29] Wu Q F, Ding S W, Zhang Y J, Zhu S F. Identification of viruses and viroids by next-generation sequencing and homology-dependent and homology-independent algorithms. Annual Review of Phytopathology, 2015, 53(1): 425-444.
[30] Villamor D E V, Ho T, Al Rwahnih M, Martin R R, Tzanetakis I E. High throughput sequencing for plant virus detection and discovery. Phytopathology, 2019, 109(5): 716-725.
[31] Wu L P, Du Y M, Xiao H, Peng L, Li R. Complete genomic sequence of tea-oil camellia deltapartitivirus 1, a novel virus from. Archives of Virology, 2020, 165(1): 227-231.
[32] Katsarou K, Andronis C, James A, Euthymiou K, Kryovrysanaki N, Pappi P G, Kalantidis K. Complete genome sequence of a carlavirus identified in grapevine (sp.) in Greece. Archives of Virology, 2023, 168(6): 172.
[33] 汤亚飞, 裴凡, 李正刚, 佘小漫, 于琳, 蓝国兵, 邓铭光, 何自福. 基于小RNA深度测序技术鉴定侵染广东辣椒的病毒种类. 中国农业科学, 2019, 52(13): 2256-2267. doi:10.3864/j.issn.0578-1752. 2019.13.006.
Tang Y F, Pei F, Li Z G, She X M, Yu L, Lan G B, Deng M G, He Z F. Identification of viruses infecting peppers in guangdong by small rna deep sequencing. Scientia Agricultura Sinica, 2019, 52(13): 2256-2267. doi:10.3864/j.issn.0578-1752.2019.13.006. (in Chinese)
[34] Syller J. Facilitative and antagonistic interactions between plant viruses in mixed infections. Molecular Plant Pathology, 2012, 13(2): 204-216.
[35] 刘勇, 李凡, 李月月, 张松柏, 高希武, 谢艳, 燕飞, 张安盛, 戴良英, 程兆榜, 等. 侵染我国主要蔬菜作物的病毒种类、分布与发生趋势. 中国农业科学, 2019, 52(2): 239-261. doi:10.3864/j.issn. 0578-1752.2019.02.005.
Liu Y, Li F, Li Y Y, Zhang S B, Gao X W, Xie Y, Yan F, Zhang A S, Dai L Y, Cheng Z B,. Identification, distribution and occurrence of viruses in the main vegetables of China. Scientia Agricultura Sinica, 2019, 52(2): 239-261. doi:10.3864/ j.issn.0578-1752.2019.02.005. (in Chinese)
[36] Pallás V, Sánchez-Navarro J A, James D. Recent advances on the multiplex molecular detection of plant viruses and viroids. Frontiers in Microbiology, 2018, 9: 2087.
[37] Xu L, Ming J. Development of a multiplex RT-PCR assay for simultaneous detection of lily symptomless virus, lily mottle virus, cucumber mosaic virus, and plantago asiatica mosaic virus in lilies. Virology Journal, 2022, 19(1): 219.
[38] 吐逊艾力·艾孜提力, 侯婉莹, 郭庆元,古丽尼沙·卡斯木, 代毅, 李世访, 麦合木提江·米吉提. 三种无花果病毒的多重RT-PCR检测. 分子植物育种, 2021, 19(16): 5414-5420.
Tuxunaili·Aizitili, Hou W Y, Guo Q Y, Gulinisha·Kasimu, Dai Y, Li S F, Maihemutijiang·Mijiti. Multiplex RT-PCR detection of three fig viruses. Molecular Plant Breeding, 2021, 19(16): 5414-5420. (in Chinese)
Identification of Tea Plant Viruses in Fujian Province and Establishment of Multiplex PCR Detection Assay

1Institute of Plant Virology, Fujian Agriculture and Forestry University, Fuzhou 350002;2Technology Center of Fuzhou Custom District/Fujian Key Laboratory for Technology Research of Inspection and Quarantine, Fuzhou 350001;3Comprehensive Technical Service Center of Rongcheng Customs District, Fuqing 350300, Fujian
【Objective】This study endeavors to explore the species diversity and prevalence of viruses within tea plantations in Fujian Province. Furthermore, it aims to devise a multiplex PCR assay capable of swiftly detecting multiple viruses simultaneously.【Method】From 2019 to 2023, a total of 1 869 samples were gathered from tea plants displaying virus-like symptoms, including chlorosis, blotch, and necrosis, across nine regions, encompassing Fuzhou, Nanping, Ningde, Quanzhou, Zhangzhou, Xiamen, Sanming, Putian, and Longyan cities. High-throughput sequencing technology, combined with PCR and RT-PCR detection methods, was used to identify the pathogens causing viral diseases in tea plants. Specific target fragments amplified by PCR were cloned, sequenced, and subjected to phylogenetic analysis. In addition, specific primers were designed based on reported virus sequences in GenBank, and a multiplex PCR assay was established for detecting major tea plant viruses in Fujian Province by optimizing reaction conditions, including annealing temperature, primer concentration, and cycle number. The assay’s specificity, sensitivity, and practical application were subsequently determined.【Result】Three viruses were detected in tea plant samples from Fujian tea plantations, with detection rates ranked as follows: oil tea associated geminivirus (OTaGV) at 48.90%, tea plant necrotic ring blotch virus (TPNRBV) at 26.75%, and camellia cryptic virus 1 (CCV1) at 17.98%. Among the 1 258 samples that tested positive for viruses, 807 samples were infected with only one of the three viruses (OTaGV, TPNRBV, or CCV1), corresponding to detection rates of 37.20%, 21.38%, and 5.56%, respectively. The remaining 451 samples were co-infected with two or three viruses, resulting in a co-infection rate of 35.85%. The most common co-infection types were OTaGV+CCV1 (17.49%), TPNRBV+CCV1 (0.40%), TPNRBV+OTaGV (14.71%), and OTaGV+CCV1+TPNRBV (3.26%). Geographically, OTaGV was distributed in all nine regions, with the highest detection rate in Zhangzhou at 96.77%. CCV1 was present in eight regions (excluding Xiamen), with the highest detection rate in Sanming at 66.00%. TPNRBV was found in five regions (Fuzhou, Nanping, Ningde, Quanzhou and Zhangzhou), with the highest detection rate in Quanzhou at 79.13%. Among the nine regions, only OTaGV was detected in Xiamen, OTaGV and CCV1 were detected in Sanming, Putian, and Longyan, and all three viruses were detected in the other five regions. The viral co-infection rate was highest in Zhangzhou at 85.00% and lowest in Ningde at 23.03%. Phylogenetic tree construction based on CCV1 and TPNRBV gene sequences indicated that the CCV1 isolate FW obtained in this study had the closest resemblance to the reported Fujian isolate FJ_SH104 (GenBank accession number: ON807095), and the TPNRBV isolate FU had the closest resemblance to the Fujian isolate QZHA92 (GenBank accession number: OQ948454). The established multiplex PCR detection assay exhibited strong specificity, as it only amplified specific target fragments of OTaGV, TPNRBV, and CCV1, while no amplification product was observed in other viruses or healthy tea plant samples. The lowest sensitivity of the multiplex PCR was detecting OTaGV and TPNRBV at dilutions of 10-4and CCV1 at a dilution of 10-3. The multiplex PCR assay successfully detected all three viruses in 60 disease samples collected from tea plantations, and the results were consistent with those obtained by single PCR detection.【Conclusion】OTaGV, CCV1, and TPNRBV are the major virus species in tea plants in the Fujian, with OTaGV being reported for the first time in this province. This study also revealed that OTaGV can infect tea plants. Among the detected viruses, OTaGV has the widest distribution, followed by CCV1 and TPNRBV. Presently, tea plants in Fujian are still mainly infected by single virus infections. However, viral co-infections are prevalent, showcasing combinations such as TPNRBV+CCV1, TPNRBV+OTaGV, CCV1+OTaGV, and OTaGV+CCV1+TPNRBV. The established multiplex PCR assay exhibits strong specificity and high sensitivity, making it suitable for the rapid detection of the three viruses (OTaGV, CCV1 and TPNRBV) in tea plantations. Taken together, the findings obtained in this study will provide a theoretical basis and technical support for prevention and control of viral diseases in tea plants in Fujian Province.
tea plant virus; pathogen identification; multiplex PCR; molecular detection; Fujian Province

2023-10-07;
2023-11-05
福建省农业引导性(重点)项目(2020N0024)、福建省检验检疫技术研究重点实验室开放课题(FJKF2019-03)
陈细红,E-mail:kaishilvxing00@163.com。通信作者沈建国,E-mail:shenjg_agri@163.com。通信作者高芳銮,E-mail:raindy@fafu.edu.cn
(责任编辑 岳梅)
