基于FAVOR+和增强损失的蛋白溶解预测
2024-02-22杨子航王顺芳
杨子航,王顺芳
(云南大学 信息学院,云南 昆明 650504)
0 引 言
蛋白质的溶解性往往代表着它的产量,从而在制药与食品等行业中发挥重要作用[1]。迄今为止,在大肠杆菌表达系统中可以成功生产的可溶性重组蛋白实例仍然很少,这导致了难以提高整体生产能力。为了获得可溶性蛋白质,大多数成熟的策略通常涉及一系列试错步骤,却无法保证成功。同时提高产量和最小化生产成本的一种方法是通过使用计算模型来提供准确的溶解性预测[2],在实验工作之前预测出高度可溶解的蛋白质。
目前,已经提出了几种预测方法[3-5],但现有方法存在一定的不足且预测效果上仍然表现不佳。在已有工作的基础上,本文提出了一种多输入的深度学习模型FESOL来应对现有方法的局限性并提高蛋白质溶解性的预测性能。方法受到FAVOR+(fast attention via positive orthogonal random features)[6]这种快速注意力机制的高效性和它与常规Transformer[7]的兼容性的启发,应用它代替传统自注意力使得模型能够在蛋白质长序列的溶解性预测中高效提取全局特征,同时克服由于传统注意力而引起的计算复杂度问题。此外,在交叉熵的基础上,进一步结合余弦相似度,设计了增强的损失函数,文中记名为EhL(enhanced loss)。传统的交叉熵损失只关注样本是否被正确分类[8],EhL一定程度上弥补了这一不足,使得模型在处理多个输入时能够关注到不同输入的差异性。本文提出的方法针对蛋白质长序列和多数据,通过FAVOR+和EhL的相互配合,编码更多特定于不同输入的信息,从而提高溶解性预测的准确性。
1 相关工作
近几年,深度学习领域快速发展,相比于传统机器学习方法不灵活等特点,深度学习能够直接高效地捕获到原始数据中所关心的信息,因此通过深度学习的方法探索蛋白质性质与其序列的内在关系是目前比较热门的一个研究领域,并取得了良好的研究成果[9,10]。在蛋白质溶解性预测中,Khurana S等提出了DeepSol,一种基于深度学习的蛋白质溶解性预测器,框架的主干是一个卷积神经网络,它利用了k-mer 结构以及从蛋白质序列中提取的额外序列和结构特征[3]。Chen J等提出了一种新的结构感知方法GraphSol,通过结合预测的接触图和图神经网络,从序列中预测蛋白质溶解度[11]。Wu X等提出了EPSOL,使用Bi-gram和Tri-gram来增强原始蛋白质序列的表示,通过多维嵌入获得全面的蛋白质特征表示,利用多卷积池进一步整合并最后预测[4]。
利用CNN和多数据融合进行蛋白质溶解性预测已经取得了良好的效果,但仍存在一定的不足,主要体现在两方面:其一,基于卷积神经网络的模型将无法直接高效的捕获到序列的长程依赖信息,这也导致了这类模型不能够最大限度提取有意义的长序列特征进行准确的下游预测;其二,当在多输入下使用传统交叉熵作为模型训练损失时,仅学习各个输入关联到标签的一般特征,而无法充分考虑到不同类型的输入数据可以在高级表示空间呈现出独特的特征,这将导致模型无法有效提取丰富的预测特征。
Transformer通过注意力机制并行处理整个输入,它能够直接有效提取序列全局特征,有效缓解了卷积网络难以捕获长程信息的问题。例如,Thumuluri V提出了NetSolP,一个基于Transformer的深度学习蛋白质语言模型,专注于直接从序列预测溶解性和可用性[12]。但是,Transformer由于其较高计算复杂度而不能很好地扩展到长序列。为了优化Transformer模型的复杂度,Choromanski K等提出了Performer,并在蛋白质序列建模任务上测试了其有效性[6]。其复杂度上的优化主要得益于FAVOR+,它通过核技巧近似传统注意力分布,利用矩阵运算规则将时间复杂度降到了线性,这很好解决了由序列长度引起的计算复杂度问题。因此应用FAVOR+有望改善长序列蛋白质的溶解性预测。
2 FESOL模型
2.1 模型输入
提出的模型接受单个蛋白质的8种不同输入数据,可以概括为4个部分:①原始氨基酸序列、2-mer和3-mer增强表示;②从蛋白质序列预测的二级结构序列;③从蛋白质序列预测的溶剂相对可及性序列;④从蛋白质序列中提取的附加特征,共57个数值形式的特征。表1中总结了这8种输入数据。
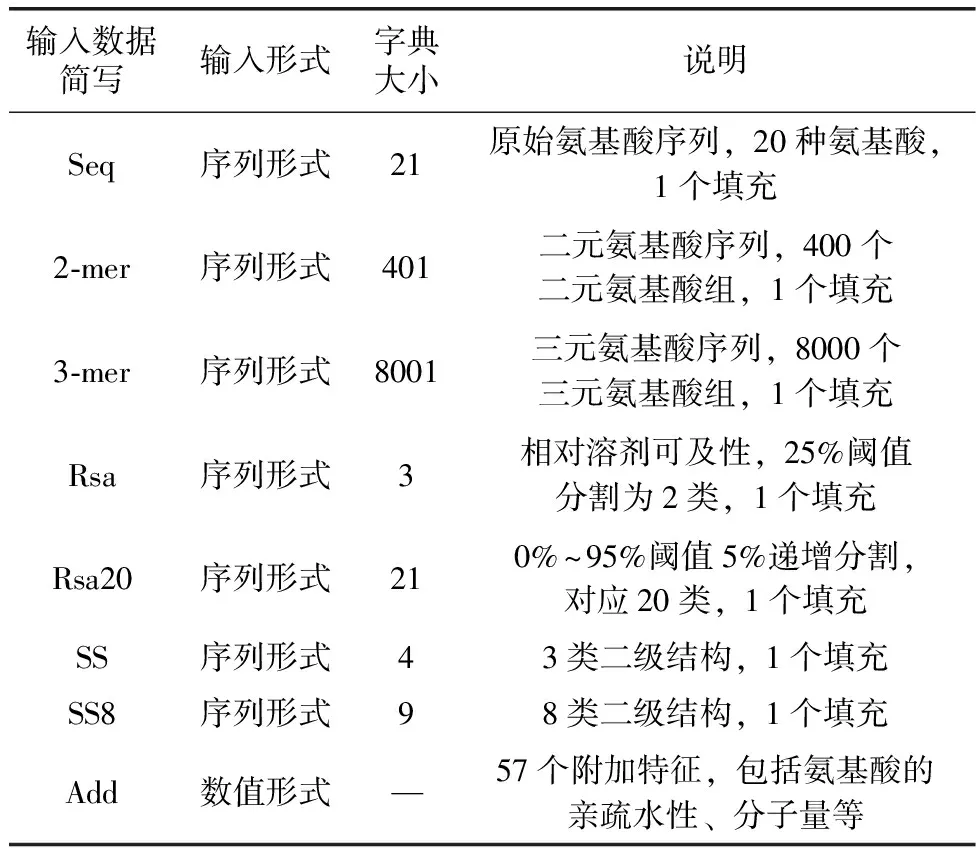
表1 输入数据总结
2.2 模型总体框架
FESOL是一个在多输入数据下完成分类任务的深度学习模型,架构如图1所示。它由7个编码器以及1个分类器组成。并行的7个编码器在结构上都是相同的,从模型的整体工作过程来说,它首先接受一个样本的7种不同序列特征输入,分别是Seq、2-mer、3-mer、Rsa、Rsa20、SS和SS8,它的7个编码器分别为一个样本的不同输入各编码到一个表示向量,产生7个表示向量再与57个附加特征(Add)直接连接为一个全局表示向量,与此同时,7个表示向量计算余弦相似度损失(LCS),以在高级特征空间中约束不同特征向量的相似程度。之后,FESOL的分类器直接将这个全局表示向量映射到预测标签,这个分类器是一个两层的全连接网络。最终,预测标签与实际标签计算交叉熵损失并结合余弦相似度损失训练整个网络。
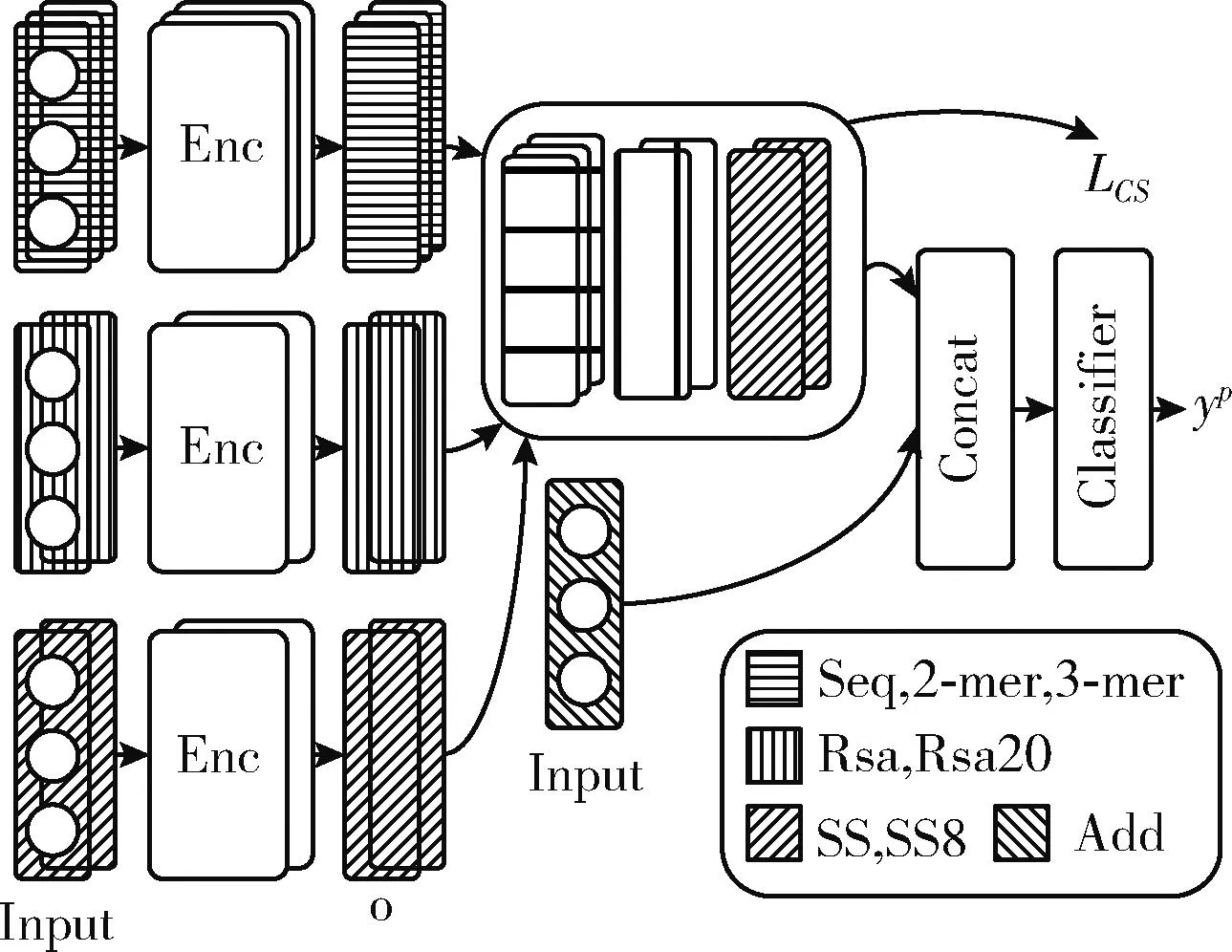
图1 FESOL总体框架
2.3 编码器
单个编码器在Transformer基础上,引入了FAVOR+机制,其结构如图2所示,它由嵌入层(Embedding)、多头注意力层(MulHAttn)和前馈神经网络层(FFN)顺序堆叠组成。由于各个编码器独立工作,且结构相同,本节将以氨基酸序列输入(Seq)为例,介绍其对应的编码器(Enc1)的工作过程。
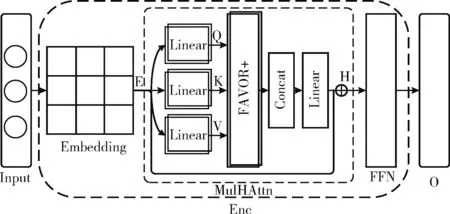
图2 编码器结构
(1)嵌入层
整齐后氨基酸序列是Enc1的输入,它由20种不同的氨基酸和1个填充构成。Enc1从嵌入层开始,将每个氨基酸转换为相应的嵌入向量。经过嵌入层后,得到氨基酸序列嵌入矩阵E∈RL×d, 其中L是氨基酸序列长度,d是嵌入向量的维度大小。
(2)多头注意力层
Enc1的多头注意力与早期工作(Transformer)中的注意力机制[13,14]有所不同,它是基于正交随机特征的快速注意力,通过隐式计算注意力分布,利用矩阵运算规则将时间复杂度降到了线性,使得编码器能够在捕获长序列的上下文信息时更加简洁高效。Enc1的每个子层中,多头注意力层是核心,它帮助编码器将序列的嵌入矩阵转换为潜在特征矩阵。多头注意力的具体计算公式被列出
MH(E)=Concat(head1,head2,…,headn)Wh
(1)
(2)
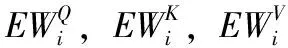
(3)
在FAVOR+的注意力分布矩阵A中,A(i,j)=K(qiT,kiT), 核函数K定义如下[6]
K(x,y)=[φ(x)Tφ(y)]
(4)
这里的φ(u) 是一个随机特征图,qi,ki分别对应到矩阵Q,K的第i行向量。最终高效的注意力机制的计算形式具体表示为
Attn(Q,K,V)=U-1(QP((KP)TV))
(5)
U=diag(QP((KP)T1L))
(6)
对于QP,KP的矩阵行分别由φ(qiT)T和φ(kiT)T给出。 diag(z) 获得以输入向量为对角线的对角矩阵。1L表示长度为L的全1向量。
编码器通过FAVOR+计算注意力分布,能够将FESOL的计算复杂度从O(L2d) 降至了O(Lrd), 有效提高了长序列的计算效率。
除了注意力的有效近似估计外,本层还添加了残差连接。嵌入矩阵E通过多头注意力层后,得到带有残差连接的输出H∈RL×nr
H=LayerNorm(MH(E)+E)
(7)
(3)前馈神经网络层
前馈神经网络层由两个线性变换组成,中间通过一个elu激活函数连接,计算过程描述为
O=elu(HW1+b1)W2+b2
(8)
W1∈Rd×m和W2∈Rm×d是权重矩阵,b1和b2是偏置值。这一层得到的O∈RL×d为氨基酸序列的特征矩阵,取O的第一行o∈Rd作为氨基酸序列的特征表示向量,提供给分类器进行溶解性预测。
2.4 分类器
FESOL的分类器是一个简单的两层全连接神经网络。它接受x作为输入,x由每种输入特定的编码器生成的表示向量和附加的特征连接而成
x=Concat(o1,o2,…,o7,oAdd)
(9)
o1,o2,…,o7分别对应到Seq,2-mer,3-mer,Rsa,Rsa20,SS,SS8这7个不同特征输入的表示向量,oAdd是附加特征组成的特征向量。分类器的输出是预测概率yp
yp=softmax(elu(xW3+b3)W4+b4)
(10)
2.5 EhL损失
在交叉熵损失下学习的表示仅捕获所有输入数据的一般上下文,但可能不是特定于每种输入的信息。这将直接导致预测模型学习到的表示向量中缺乏不同输入之间的差异性,而这些无法学习到的差异性可能是决定蛋白质溶解性的潜在特征,进而影响到溶解性的预测效果。为了体现不同输入的差异性,希望不同输入的表示向量应当互不相同,因此本研究中增强了训练模型的损失函数,将交叉熵和余弦相似度相结合,新增的余弦相似度损失可以在高级特征空间中迫使不同输入的表示互不相同,从而提高FESOL编码器提取丰富特征的能力。
总体上,FESOL的7个编码器分别将7种不同数据的输入编码到7个独立的d维的表示向量,然后,进一步利用每种输入特定的编码器生成的表示向量来构建反映不同输入数据独特性的余弦相似度损失,同时,结合交叉熵损失构建出EhL。这里的交叉熵损失由FESOL分类器给出的预测概率与真实标签计算得到。
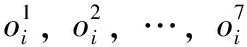
EhL=αLCE+βLCS
(11)
这里的LCE,LCS分别代表交叉熵损失和余弦相似度损失,α,β是权重系数,α约束预测标签与真实标签的接近程度,β控制各个表示向量的差异程度。LCE计算如下,表示二分类交叉熵损失
(12)
LCS先计算单个样本的不同表示间的余弦相似度,然后进行求和
(13)
这里的cs计算两个向量余弦相似度,规范定义在下列等式中给出
(14)
其中,p,q是分别代表一个向量。
3 实验结果与分析
3.1 实验数据集
本文中使用的原始数据集来自于Smialowski等从大肠杆菌中收集的异源表达的蛋白质序列,并由Xiang Wu等[4]进行了预处理的基础上作为训练数据集。数据集由28 972个可溶性和40 448个不溶性蛋白质组成。
研究中使用的独立的测试集由1000个可溶性蛋白质序列和1001个不溶性蛋白质序列组成,它已被广泛用作评估方法性能的基准测试集。为了与现有方法进行公平比较,采用此测试集对几种最先进的基于序列的蛋白质溶解性预测方法进行全面比较。
3.2 实验设置
实验中将预测蛋白质溶解性的任务作为二分类问题,目标是最小化预测的溶解性标签和实验测量的标签之间的差异。Pytorch库被用来实现所提出的模型。模型训练中,使用学习率为0.001和正则率为0.005的AdamW优化器,批次大小为32,训练轮次设置为10。至于输入数据,为了保证方法的可对比性,所有蛋白质的序列数据长度固定为L=1200,这类似于之前的工作[3,4],少于1200个氨基酸用0填充,超过1200个氨基酸被截断为1200。不同的序列形式输入数据的嵌入维数d均设置为64,这些数据依次为Seq、2-mer、3-mer、Rsa、Rsa20、SS和SS8。此外,EhL中的权重系数α,β经过实验探索后均设置为1。
3.3 评价指标
研究中使用到的评价指标包括正确率(Accuracy)、马修斯相关系数(MCC)和Rawi等[5]介绍到的每类选择性(Selectivity)、每类灵敏度(Sensitivity),这些指标已在之前研究中用于评估其它先进方法的性能,MCC的值在-1和1之间,越接近1表示模型预测越好。其它5个指标具体计算为
(15)
(16)
(17)
(18)
(19)
其中,TP和TN分别代表正确分类的可溶和不可溶蛋白质的样本数,FN和FP分别代表被错误分类的可溶和不可溶蛋白质的样本数。
此外,EhL系数影响和消融研究中还使用到Precision、Recall和AUC,Precision、Recall这两个指标被广泛用于评估分类问题的性能[15,16],定义为
(20)
(21)
AUC定义为接受者操作特性曲线(ROC)下的面积,一般来说,提供更大AUC的分类器表明它具有更好的性能。
3.4 增强损失性能分析
为了验证增强损失EhL的有效性,实验中对FESOL依次使用EhL和交叉熵(CE)进行了训练,并在MCC上进行了比较,同时,为了更全面探索EhL在不同序列长度输入下的适用性,设置了从600到1200,以100长度递增的序列截断长度上进行模型训练并比较预测性能,结果如图3所示。可以发现,EhL在所有的不同截断长度下均取得了更高的性能,能够适应于不同长度下的预测。在长度为1200时,EhL最高超过仅使用交叉熵损失时的0.3以上,虽然长度为600和1000时两条折线最为接近,但是也略微胜过了交叉熵。
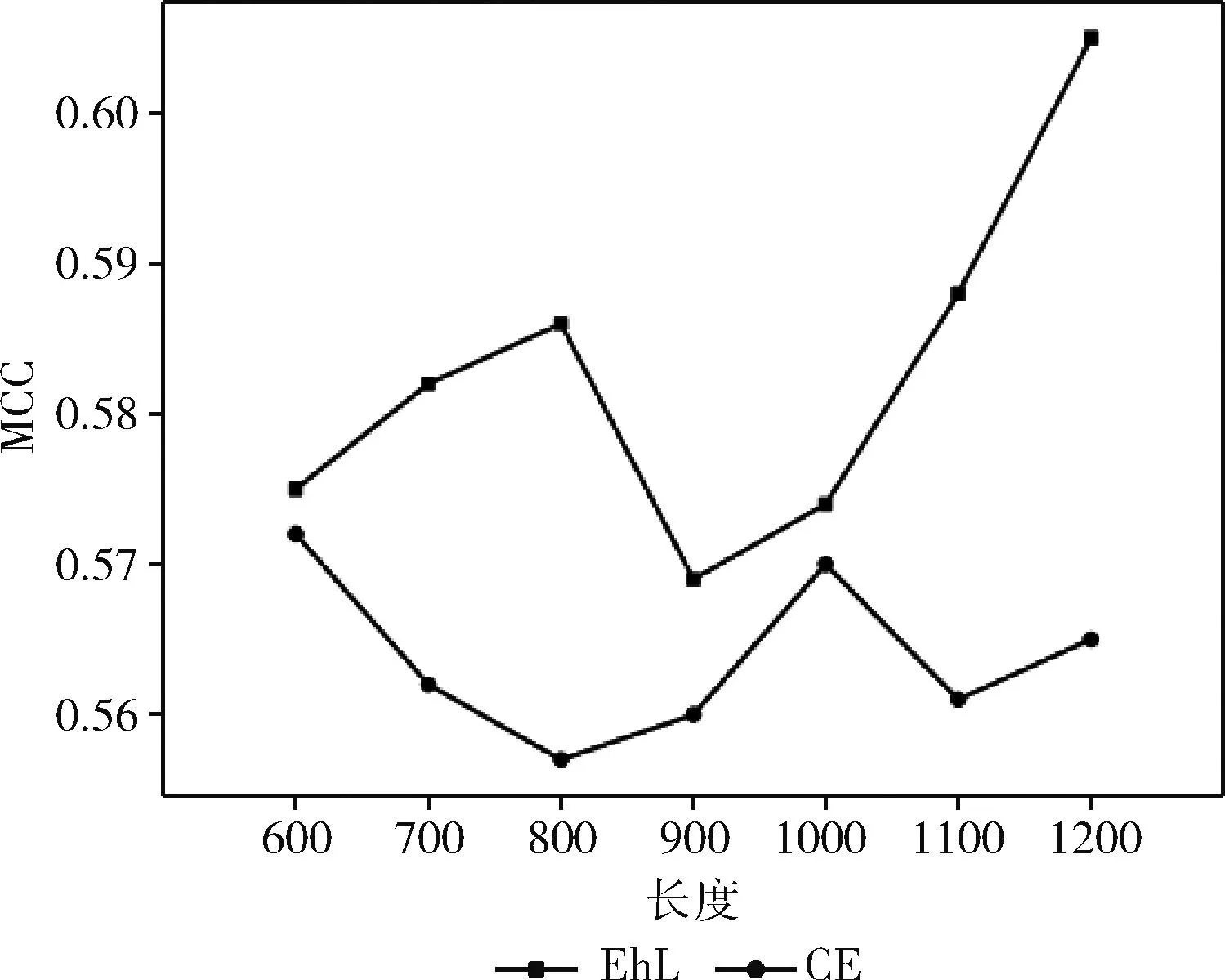
图3 EhL和交叉熵对FESOL性能影响
根据观察到的实验结果,设计的增强损失函数EhL可以提高多输入下的预测性能。总体而言,实验结果表明了损失函数的组合是一个简单且有效的策略,它有助于在多输入模型的学习过程中捕获更丰富的表示并提高分类性能。
3.5 EhL系数分析
为了探究EhL中的权重系数α,β在不同的取值下的对模型预测性能的影响。实验中对0.5、1、2这3个参数上进行了网格搜索,并在Accuracy、Precision、Recall、MCC这4个评价指标上对预测性能进行了比较,具体预测结果见表2。
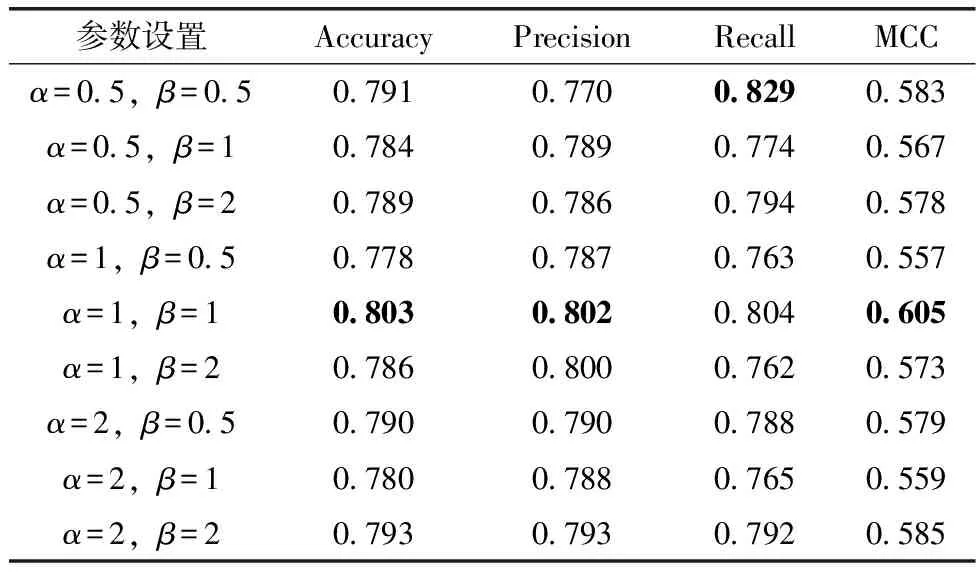
表2 FESOL在不同α,β下的预测性能
实验中主要关注引入不同比重的余弦相似度后对预测效果产生的影响,因此固定α取值,比较β对预测性能的影响。可以发现在比较α=0.5时,β=0.5取的相对最高性能,对于其它两组β=1,2,随着β取到更大值,性能也随之增大;比较α=1时,当β=1时取得全局最高性能,而对于β=0.5,2时,性能也呈现随β增大而增大;比较α=2时,当β=2时取得相对较高性能,但是,对于β=0.5,2时却呈现与之前相反结果,随β增大性能反而下降。根据固定α时的对比可知,在α,β等比例取值时,一致取得了最高性能,也说明了添加余弦相似度的必要性。
3.6 消融研究
FESOL总共有8种不同类型的输入,为了探究不同的输入对FESOL预测性能的贡献程度,实验中通过移除网络中的单个输入组件来进行消融研究。具体来说,将所有输入分为了4组,氨基酸(Seq,2-mer,3-mer)、溶剂可及性(Rsa,Rsa20)、二级结构(SS,SS8)和附加特征(Add),并依次测试了移除氨基酸(Without Seq)、溶剂可及性(Without Rsa)、二级结构(Without SS)和附加特征(Without Add)时模型性能,并与所有输入(All)进行比较。
实验结果见表3,可以发现氨基酸序列对最终的预测性能是最重要的。如果没有氨基酸序列,Accuracy、Precision、Recall和MCC分别从0.803、0.802、0.804和0.605下降到0.696、0.779、0.546和0.410,氨基酸序列的重要程度在之前的研究[4]中也得到了验证。其次,溶剂可及性也是重要的,在没有它们输入时,Accuracy、Precision、Recall和MCC分别下降到0.775、0.796、0.738和0.551。此外,相较于其它输入数据,二级结构和附加特征显得不那么重要,但它们也有利于提高预测性能。
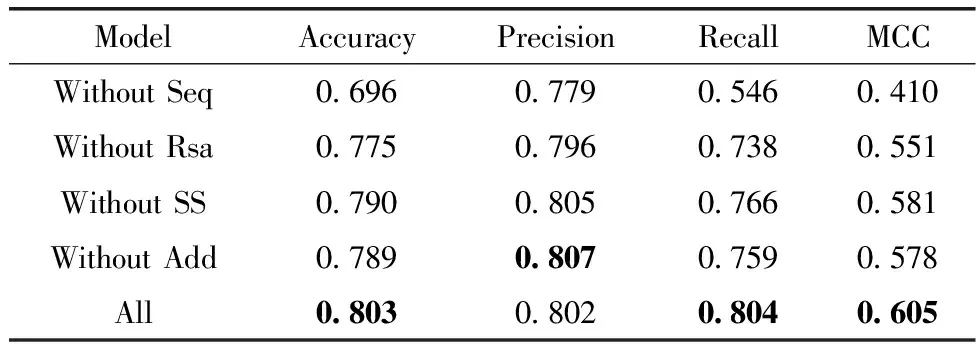
表3 FESOL与移除不同输入的性能
此外,展示了提出的FESOL与移除不同组件后的模型的ROC曲线,如图4所示,所有输入下FESOL的ROC曲线明显高于没有氨基酸输入的模型,但是相较于其它模型则高度并不明显。结果表明,不同类型的输入数据对蛋白质溶解性预测产生的作用也是不同的。最关键的数据是氨基酸输入,其它包括溶剂可及性、二级结构和附加特征仅作为一种改善预测性能的辅助信息。
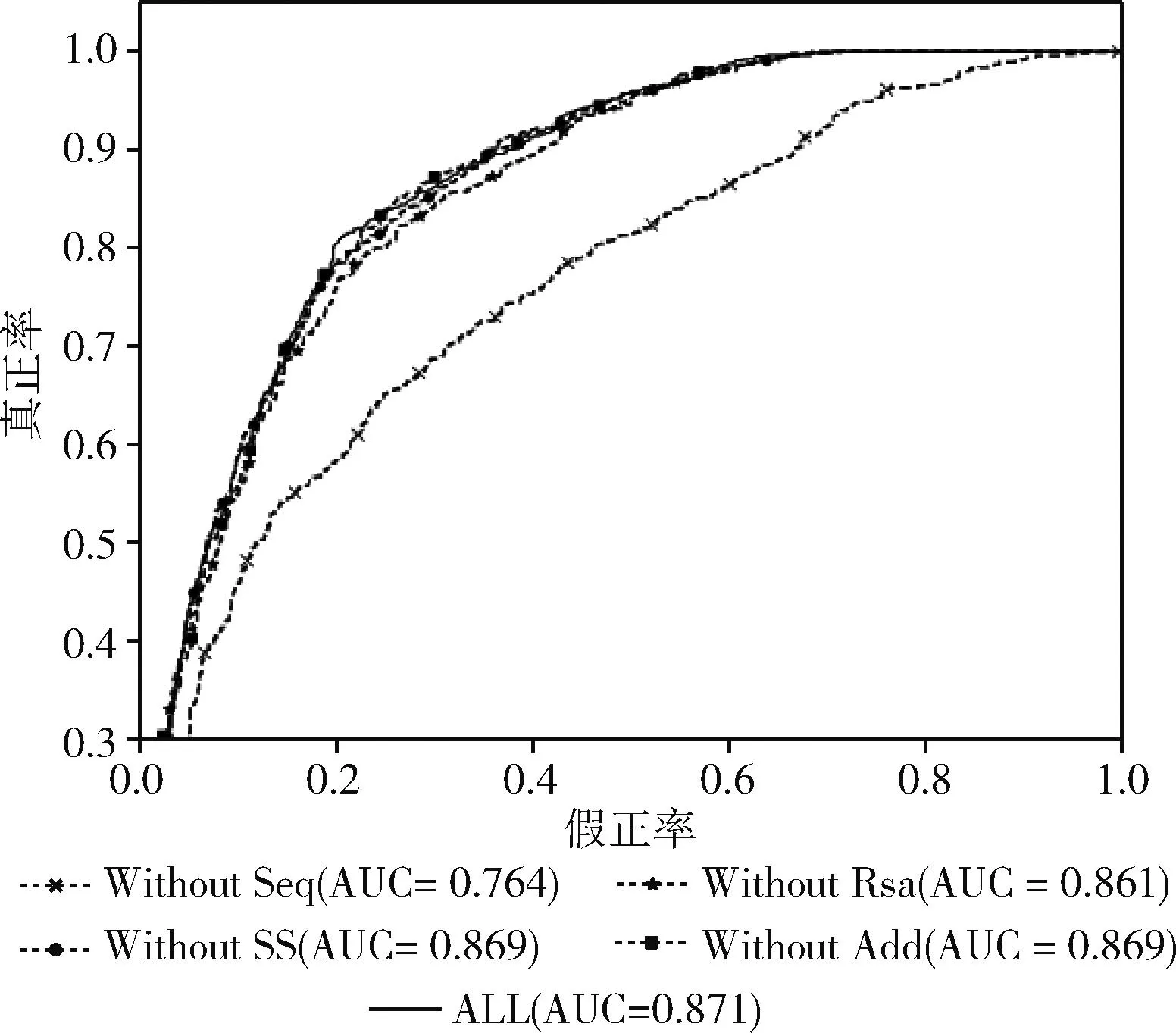
图4 FESOL与移除不同输入的ROC曲线
3.7 FESOL性能分析
为了评估FESOL的预测性能,在6个评价指标上使用独立测试集将FESOL与3种先进的预测方法进行了比较,包括PaRSnIP、DeepSOL和EPSOL。FESOL训练过程中的损失收敛曲线如图5所示,可以发现,训练5轮左右时模型收敛,取验证集上损失达到最低的模型,进而在测试集上进行性能评估。
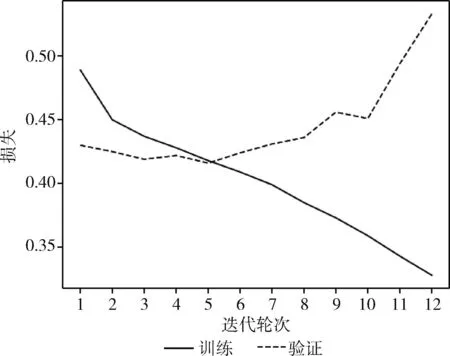
图5 损失收敛曲线
性能预测结果见表4,可以发现FESOL在所有方法中取得了最高的Accuracy=0.80、MCC=0.60、Selectivity(insoluble)=0.80、Sensitivity(soluble)=0.80。唯一例外的是Selectivity(soluble)=0.84和Sensitivity(insoluble)=0.88两个指标,它们由DeepSol S2产生了最高分数。但是, 在使用Selectivity(insoluble)和Sensitivity(soluble)指标评估时,DeepSol S2的表现却明显更低,综合显示,FESOL能够兼顾到正类和类负样本,性能显得更加均衡稳定。
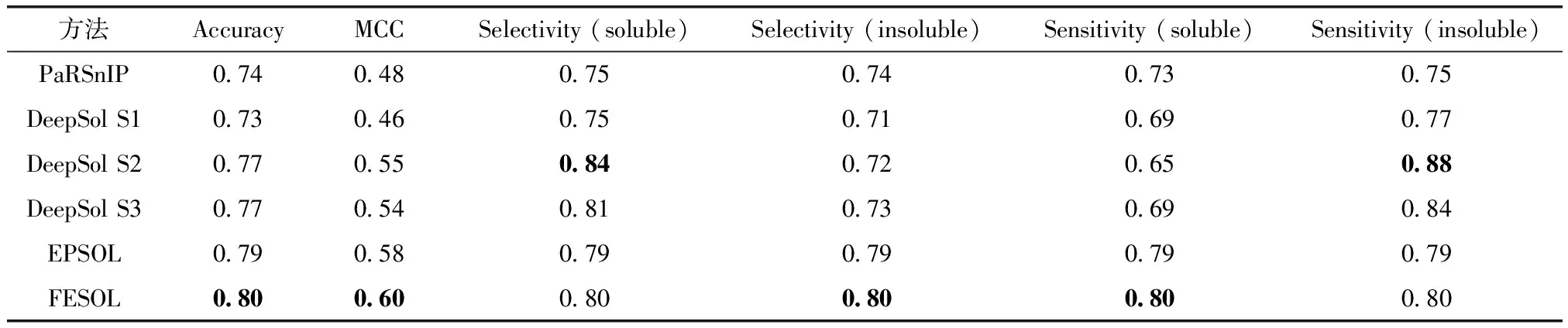
表4 FESOL与其它方法的性能
4 结束语
本文提出了用于蛋白质溶解性预测的多输入深度学习模型FESOL,并在独立测试集上验证了其有效性。该模型巧妙地利用了FAVOR+的线性计算复杂度特点,从而为更长的蛋白质序列预测问题提供有效的解决方案。实验结果表明,余弦相似度与交叉熵相结合的增强损失能够在多输入数据的分类问题下有助于提高性能。未来,主要工作将探索仅基于原始氨基酸序列的高效模型,在进一步提高预测能力的同时,更加注重模型简洁易用。
