基于全基因组重测序的拟鳄龟遗传多样性及遗传结构分析
2024-02-09杨华琳李伟纪利芹朱新平洪孝友






摘要:【目的】从全基因组水平开展拟鳄龟养殖群体遗传多样性及群体遗传结构分析,评估养殖拟鳄龟的种质资源状况,为拟鳄龟种质资源的开发与利用及促进其养殖产业健康发展提供参考依据。【方法】分别从肇庆高要市活道镇荣杰龟鳖专业合作社(RJ)、肇庆市滋源龟鳖养殖专业合作社(ZY)和肇庆市四会肇庆盛科农业发展有限公司(SK)各采集30只拟鳄龟,通过对3个拟鳄龟养殖群体90个样本进行基因组重测序,基于全基因组单核苷酸多态性(SNP)进行系统发育进化树分析和主成分分析,从基因组水平评估养殖拟鳄龟遗传多样性及群体遗传结构。【结果】基于形态性状比例参数的聚类分析结果显示,3个拟鳄龟养殖群体分为两大分支,SK群体与RJ群体先聚类在一起,然后与ZY群体聚合。在3个拟鳄龟养殖群体中共检测出708112个SNPs位点,过滤筛选后获得220950个高质量纯合SNPs位点和440435个高质量杂合SNPs位点。3个拟鳄龟养殖群体的单核苷酸密度均为0.01 SNP/kb,核苷酸多样性(π)为0.00166~0.00171,多态信息含量(PIC)为0.151~0.154,观测杂合度(Ho)为0.171~0.205,近交系数(FHOM)分布在0.0571~0.1110;不同群体间的遗传分化系数(Fs)t在0.0056~0.0138,基因流(Nm)在-0.2486~-0.2466。基于全基因组SNP构建的系统发育进化树显示,3个拟鳄龟养殖群体整体上可分为两大分支,第一分支主要由RJ群体和SK群体组成,第二分支主要由ZY群体组成,与形态聚类分析结果基本一致。主成分分析也发现,3个拟鳄龟养殖群体间的相互距离较近,且存在相互交叉现象。【结论】3个拟鳄龟养殖群体遗传多态性较低,遗传背景不够丰富,群体间遗传分化程度低,推测具有相似的遗传背景,来源于同一引种群体。因此,在今后的育种工作亟需引入新的种质资源,为种质创新提供更丰富的遗传基础。
关键词:拟鳄龟;全基因组重测序;遗传多样性;遗传结构;SNP位点
中图分类号:S917.4;S966.5文献标志码:A 文章编号:2095-1191(2024)11-3392-12
Genetic diversity and structural analysis of Chelydra serpentinabased on whole genome resequencing
YANG Hua-lin1,2, LI Wei2, JI Li-qin2, ZHU Xin-ping2*, HONG Xiao-you2*
(1College of Fisheries, Zhejiang Ocean University, Zhoushan, Zhejiang 316022, China; 2Pearl River Fisheries Re‐search Institute, Chinese Academy of Fishery Sciences/Key Laboratory of Tropical and Subtropical Fishery Resources Application and Cultivation, Ministry of Agriculture and Rural Affairs, Guangzhou, Guangdong 510380, China)
Abstract:【Objective】The genetic diversity and genetic structure of the breeding population of Chelydra serpentina were analyzed from the whole genome level, and the germplasm resources of C. serpentina were evaluated, so as to pro‐vide reference for the development and utilization of germplasm resources of C. serpentina and promote the healthy deve-lopment of the breeding industry of C. serpentina.【 Method】A total of 30 C. serpentina were collected from Rongjie TurtleBreeding Professional Cooperative( RJ) in Huandao Town, Gaoyao City, Zhaoqing City, Ziyuan Turtle Breeding Profes‐sional Cooperative in Zhaoqing City( ZY) and Sihui Zhaoqing Shengke Agricultural Development Co., Ltd. in Zhaoqing City (SK) respectively, and 90 samples from 3 C. serpentina groups were resequenced. Phylogenetic tree analysis and principal component analysis were conducted based on whole genome single nucleotide polymorphisms( SNP) to evalua-te the genetic diversity and population genetic structure of cultured C. serpentina at the genome level. 【Result】The re‐sults of cluster analysis based on morphological trait proportion parameters showed that the 3 groups were divided into 2 branches, SK group and RJ group were first clustered together, and then aggregated with ZY group. A total of 708112 SNPs loci were detected in the 3 groups, and 220950 high quality homozygous SNPs loci and 440435 high quality hetero‐zygous SNPs loci were obtained after filtering. The single nucleotide density, single nucleotide polymorphism( π), poly‐morphism information content( PIC) and observed heterozygosity( Ho) of the 3 groups of C. serpentina were 0.01 SNP /kb, 0.00166-0.00171, 0.151-0.154 and 0.171-0.205. The inbreeding coefficient (FHOM) ranged from 0.0571 to 0.1110. The coefficient of genetic differentiation (Fst) and gene flow (Nm) ranged from 0.0056 to 0.0138 and from -0.2486 to -0.2466. The phylogenetic tree constructed on the basis of the whole genome SNP showed that the 3 groups of C. serpen‐tina could be divided into 2 branches as a whole. The first branch was mainly composed of RJ group and SK group, and the second branch was mainly composed of ZY group, which was basically consistent with the results of morphological cluster analysis. The principal component analysis also found that the distance between the 3 groups of C. serpentina was relatively close, and there was cross-over phenomenon. 【Conclusion】The genetic polymorphism 3 groups of C. serpen‐tina is low, the genetic background is not rich enough, and the degree of genetic differentiation between the groups is low, so it is presumed that they had similar genetic background and come from the same introduced population. There‐fore, it is urgent to introduce new germplasm resources in future breeding work to provide a richer genetic basis for germ‐plasm innovation.
Key words: Chelydra serpentina; whole genome resequencing; genetic diversity; genetic structure; SNP locus
Foundation items: Youth Project of National Natural Science Foundation of China(32102789); Scientific and Tech‐nological Innovation Team Project of Chinese Academy of Fishery Sciences(909231004); Government Purchase Service Project of Ministry of Agriculture and Rural Affairs(191231009); China-ASEAN Maritime Cooperation Fund(CAMC- 2018F)
0 引言
【研究意义】拟鳄龟(Chelydra serpentina)又名蛇鳄龟、小鳄龟、鳄龟等,隶属于爬行纲(Reptilia)龟鳖目(Testudines)鳄龟科(Chelydridae)拟鳄龟属(Chelydra),原产于北美,因其含肉率高(可食用部分高达70%),且肉质细嫩无腥味、营养丰富、生长速度快及饲养技术简单(李振业等,2014),于20世纪90年代引入我国进行人工养殖。2005年,农业部发布第485号公告认定拟鳄龟为适宜推广的引进品种。经过近20年的养殖推广,拟鳄龟逐渐受到养殖户和消费者的青睐,已成为水产养殖的热门品种之一。我国华南地区是拟鳄龟养殖的主产区,经种质资源调查发现以广东肇庆的养殖规模较大,但存在病害频发及生长速度差异等问题。因此,亟待对人工养殖拟鳄龟进行种质资源调查,从基因组水平了解其群体遗传多样性,为养殖拟鳄龟种质资源的保护利用及促进其养殖业健康发展提供参考依据。【前人研究进展】目前,针对野生拟鳄龟的研究主要集中于种质资源调查、分类鉴定及生态学等方面(Phil‐lips et al.,1996;de Solla and Martin,2011;Vervust et al.,2011;Paterson et al.,2012)。作为养殖品种,有关拟鳄龟的研究多集中在营养成分分析(刘翠娥等,2007;叶泰荣等,2007)、生理生化(李思明等,2008;Lawniczak and Teece,2009;Rhen et al.,2009)、养殖技术(沈保平等,2008;Schnars et al.,2011;刘坚红,2012)、病害防治(刘继芳等,2009)等方面,但针对拟鳄龟养殖群体遗传多样性的研究相对较少(刘丽等,2013)。全基因组重测序是通过对已知基因组序列物种的不同个体基因组进行测序,在基因水平上对不同目标个体或群体进行比较分析(王中铎等,2023),在遗传多样性分析和系统发育进化等方面已得到广泛应用(van der Nest et al.,2020;Ma et al.,2021;Zhang et al.,2022)。Spinks等(2014)基于高通量测序获得900个西部池龟(Emys marmorata)个体的89个SNPs位点及1个线粒体基因序列信息,深入分析了西部池龟的进化史。阿旺措吉等(2022)利用全基因组重测序数据对3个昌都黑山羊群体的遗传多样性和群体遗传结构进行分析,发现昌都黑山羊具有丰富的遗传多样性,且3个群体为同一品种,品种内存在一定的杂交现象。王统苗等(2022)基于全基因组重测序对不同地区的鸭种群体进行遗传结构分析,研究结论为获取我国地方鸭遗传资源间的遗传信息和遗传关系提供了重要依据。吴昊天等(2022)利用第二代高通量测序技术对新疆和西藏的2个棕鳟(Salmo trutta)群体进行基因组重测序,从基因组水平分析棕鳟群体的遗传结构,为指导物种保护和育种提供了理论依据。朱皓东(2022)通过Super-GBS简化基因组重测序检测并识别罗氏沼虾(Macrobrachium rosenbergii)全基因组可信度较高的SNP位点,探究3个群体的遗传多样性与遗传变异,并建立了罗氏沼虾快速生长专门化品系。马钧等(2023)利用全基因组重测序,从遗传多样性及亲缘关系等方面对秦川牛进行综合评估,发现保种群体具有丰富的遗传多样性,尚未出现大面积近交,但部分个体存在近交风险。【本研究切入点】目前,基因组重测序已广泛应用于物种遗传多样性研究领域(Saha et al.,2022;黎旺长等,2023;张彦等,2023),但尚无利用重测序数据对拟鳄龟进行遗传多样性及遗传结构分析的研究报道。【拟解决的关键问题】通过对广东肇庆3个具代表性拟鳄龟养殖企业的养殖群体进行基因组重测序,从全基因组水平开展物种遗传多样性及群体遗传结构分析,以评估养殖拟鳄龟的种质资源状况,为拟鳄龟种质资源的开发与利用及促进其养殖产业健康发展提供参考依据。
1 材料与方法
1. 1 试验材料
拟鳄龟样本采自广东省肇庆市,3个采样点分别为肇庆高要市活道镇荣杰龟鳖专业合作社(RJ)、肇庆市滋源龟鳖养殖专业合作社(ZY)和肇庆市四会肇庆盛科农业发展有限公司(SK)。每个采样点采集30只拟鳄龟,共90个样本。动物试验经中国水产科学研究院珠江水产研究所动物伦理委员会批准,批准号LAEC-PRFRI-2022-01-03。
1. 2 试验方法
1. 2. 1 形态测量与聚类分析 利用高精度的游标卡尺(精确到0.1 mm)和电子天平(精确到0.1 g)对3个养殖群体90个样本进行测量,包括背甲长、背甲宽、体高、尾长、腹甲长和腹甲宽等6个形态性状及其体质量,并利用SPSS 27.0中的Analyze-C1assify-Hierarchical-Cluster对3个群体进行聚类分析。
1. 2. 2 DNA提取 按照MGIEasy基因组DNA提取试剂盒说明提取拟鳄龟肌肉基因组DNA,获得的肌肉基因组DNA样品浸泡于70%酒精中,-20 ℃冰箱保存备用。
1. 2. 3 DNA文库构建 利用NanoDrop超微量核酸分析仪检测样品DNA浓度,以琼脂糖凝胶电泳检测样品DNA纯度。通过超声波仪打断0.001 mg基因组DNA,选取0.2~0.4 mg大小的基因片段,并在3'端链接DNA接头,采用PCR扩增加入DNA接头的基因片段,提纯回收后对PCR扩增产物进行解链,将未被破坏的DNA分子进行环化,构建DNA文库。
1. 2. 4 测序及参考基因组比对 委托深圳华大基因股份有限公司利用DNBSEQ-T7测序平台对3个拟鳄龟养殖群体样本进行全基因组重测序,获得的原始序列(Raw reads)以Trimmomatic进行质控分析,过滤后得到的有效序列(Clean reads)用于后续分析。通过BWA-MEM算法将过滤得到的Clean reads比对到参考基因组,利用SAMtools中的sort工具将比对生成的sam文件转换成source.bam文件(Li et al.,2009);比对结果文件利用SAMtools、Picard Tools和ReSeqTools进一步排序、去重复及加ID等操作(He et al.,2013),选择Qgt;30的Clean reads用于变异检测分析。
1. 3 数据分析
1. 3. 1 SNP位点分析 通过 GATK进行基因序列变异检测分析(McKenna et al.,2010),将变异检测获得的SNP集融合,进行个体及群体水平的遗传参数多样性评估。以GATK中的HaplotypeCaller完成每个样品的变异检测,使用CombineGVCFs将得到的gvcf文件合并,通过GenotypeGVCFs进行联合基因分型,得到vcf文件。采用GATK推荐的参数进行过滤:变异置信度与深度的比值(QD)≥2;Fisher检验的P≤60;MQRankSum≥-12.5;ReadPosRankSum≥-8.0;Stran‐dOddsRatio(SOR)gt;3.0。此外,由于基因型缺失率较高的SNP位点通常是基因组区域较复杂或测序数据比对的质量较低所致,SNP可信度也较低,因此只保留基因型检出率在60%以上的SNP位点。采用SelectVariants筛选出所有变异序列中的SNP位点,经Variant Filtraition过滤以获得高可信度SNP数据集,再通过SNP数据集将各样品SNP位点与对应参考基因组SNP杂合及纯合比例结果进行统计分析。
1. 3. 2 遗传多样性分析 在PLINK中设定参数滑动窗口大小为50 kb,步长为20 kb,计算3个群体的核苷酸多样性(PI)(Chang et al.,2015);利用R语言脚本与perl语言脚本对试验数据进行处理,最终生成可视化图形。
1. 3. 3 群体遗传结构分析 通过PLINK将包含SNP信息的vcf文件转换成bed和ped文件(Chang et al.,2015),将结果输入Admixture中判断群体的遗传结构,群体亚群数目设为K=2~12,并计算交叉验证误差(Cross-validation error);绘制交叉验证误差图得到最佳分群数(Alexander et al.,2009),利用R语言脚本和ggplot2软件包进行可视化处理(Wick‐ham et al.,2017)。运用PLINK生成ped、map和bed文件,根据编写的perl脚本计算遗传距离,生成的meg文件输入MEGA 10.0以获得可视化的系统发育进化树(Tamura et al.,2021),再通过Interactive Tree of Life网站(https://itol.embl.de/)对系统发育进化树进行可视化(Letunic and Bork,2021)。使用EIGENSOFT中的smartpca程序进行主成分分析(Patterson et al.,2006),并以R语言脚本及ggplot2软件包生成可视化图形(Wickham et al.,2017)。
2 结果与分析
2. 1 拟鳄龟群体形态性状比例聚类分析结果
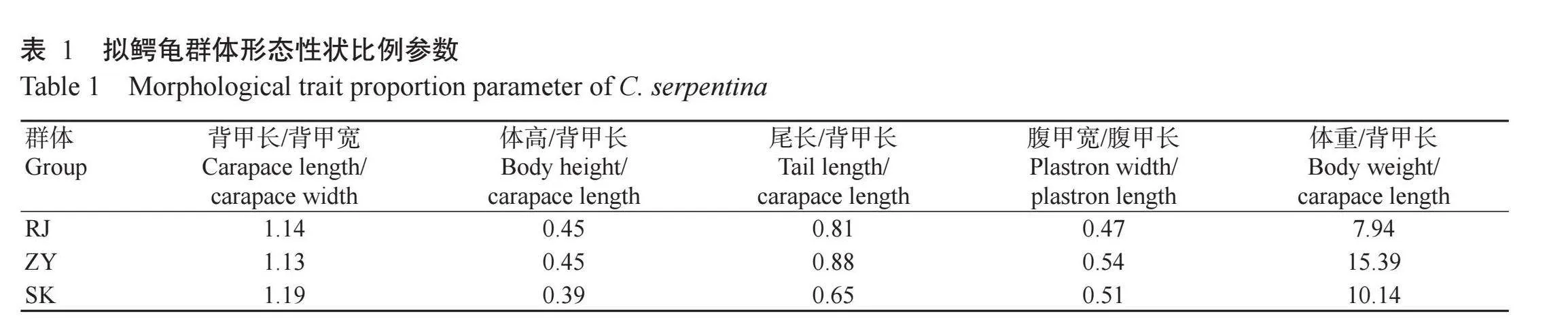
3个拟鳄龟养殖群体90个样本的形态性状比例如表1所示。对拟鳄龟形态性状进行聚类分析,获得不同养殖群体形态性状比例参数的聚类分析结果。由图1可看出,3个养殖群体分为两大分支,其中,SK群体与RJ群体先聚类在一起,然后与ZY群体聚合,表明SK群体与RJ群体的形态更相似。
2. 2 拟鳄龟基因组重测序及参考基因组比对结果
通过DNBSEQ-T7测序平台对3个拟鳄龟养殖群体90个肌肉基因组DNA样品进行全基因组重测序,样品产出的总测序碱基(Total sequencing bases)为1432.14 Gb,平均测序碱基(Average sequencing bases)为15.91 Gb,平均测序深度(Average depth)为6.7X。测序参考基因组为黄喉拟水龟(Mauremys reevesii)基因组,其基因组大小(Genome size)为2.37 Gb,Scaffold N50 为130.47 Mb。测序数据量及过滤结果如表2所示。其中,Q30平均值为92.71%~92.84%,GC含量平均值在44.32%~44.35%,表明样本建库质量良好,符合基因组重测序的标准。
运用BWA-MEM将90个拟鳄龟基因组重测序数据与黄喉拟水龟基因组(NCBI登录号GCA_0161619 35.1)进行比对,结果(表2)显示,质控后得到的平均测序数为100658616~109367305,平均基因组比对率为99.18%~99.31%,平均测序深度为6.10%~6.25%,平均覆盖率在83.74%~83.81%,表明拟鳄龟基因组重测序基本覆盖了黄喉拟水龟参考基因组,可用于后续的SNP位点筛选及遗传结构分析。
2. 3 拟鳄龟全基因组SNP位点鉴定结果
由表3可看出,在3个拟鳄龟养殖群体中共检测出708112个SNPs位点,过滤筛选后获得220950个高质量的纯合SNPs位点和440435个高质量的杂合SNPs位点,这些SNP位点将用于3个拟鳄龟养殖群体的遗传多样性分析。
2. 4 拟鳄龟遗传多样性分析结果
多态信息含量(PIC)是衡量基因变异程度的重要参数,PIC越大表明遗传信息越丰富(贾舒雯,2011)。PICgt;0.50为高度多态位点,0.25lt;PIClt;0.50为中度多态位点,PIClt;0.25属于低度多态位点(Botstein et al.,1980;徐煜等,2021)。由表4可看出,3个拟鳄龟养殖群体的单核苷酸密度均为0.01 SNP/kb;核苷酸多样性(π)为0.00166~0.00171,总群体为0.00169;PIC为0.151~0.154,总群体为0.153;观测杂合度(Ho)为0.171~0.205,总群体为0.186;基于基因组杂合度计算的近交系数(FHOM)分布在0.0571~0.1110,总群体为0.0845。由表5可看出,不同拟鳄龟养殖群体间的遗传分化系数(Fs)t在0.0056~0.0138,平均为0.0097。当Fst小于0.05时,表明遗传分化水平较低(Holsinger and Weir,2009)。不同拟鳄龟养殖群体间的基因流(Nm)在-0.2486~-0.2466,表明不同群体间存在着较小的基因交换概率。综上所述,不同拟鳄龟养殖群体遗传分化程度较低,拟鳄龟种源较单一。
2. 5 拟鳄龟群体遗传结构分析结果
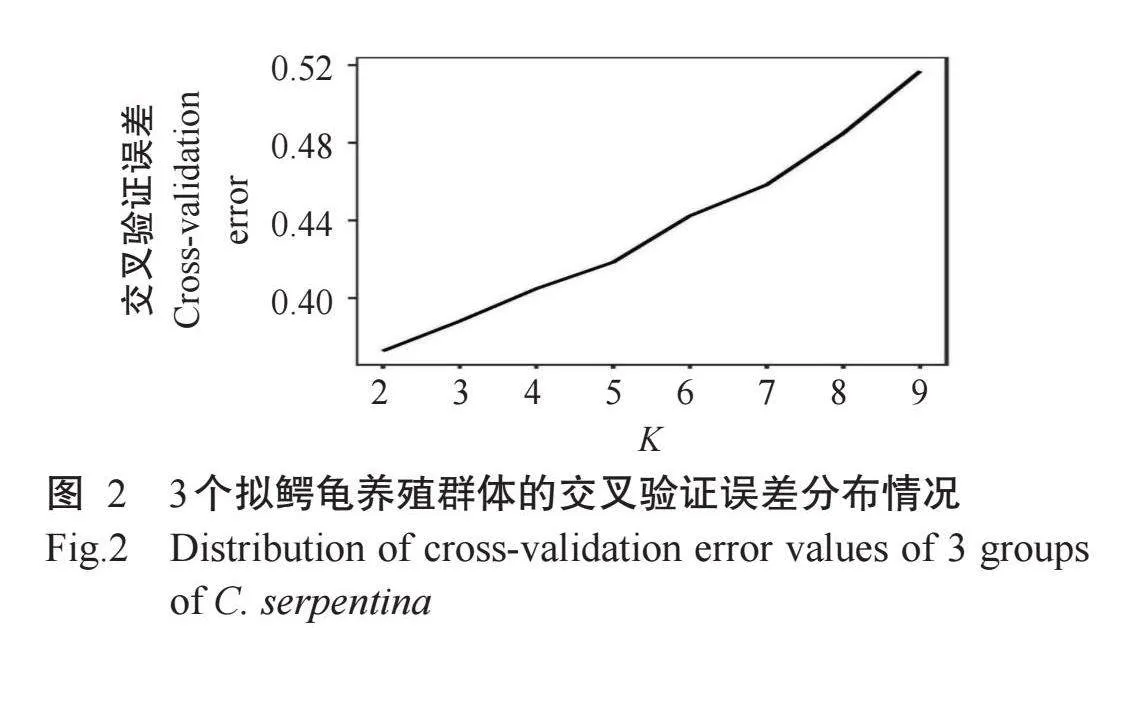
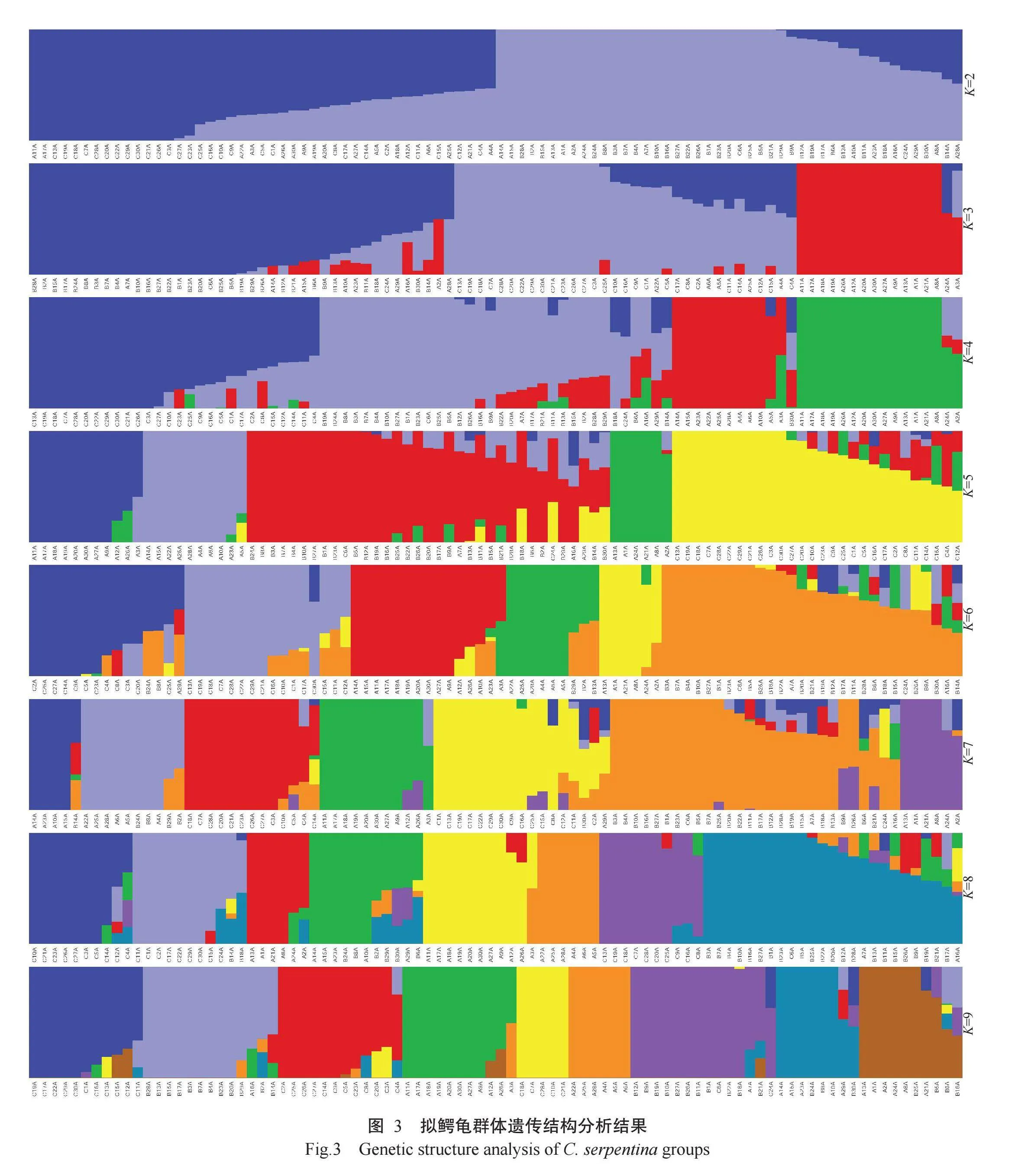
采用Admixture对3个拟鳄龟养殖群体的遗传结构进行分析,对最优亚群进行评估,结果如图2所示。当交叉验证误差最小时,K=2为假定的最佳分群数目。K=2时的拟鳄龟群体遗传结构(图3)显示,3个拟鳄龟养殖群体的遗传结构相似,仅存在较小的杂交特征。
2. 6 拟鳄龟遗传关系及其主成分分析结果
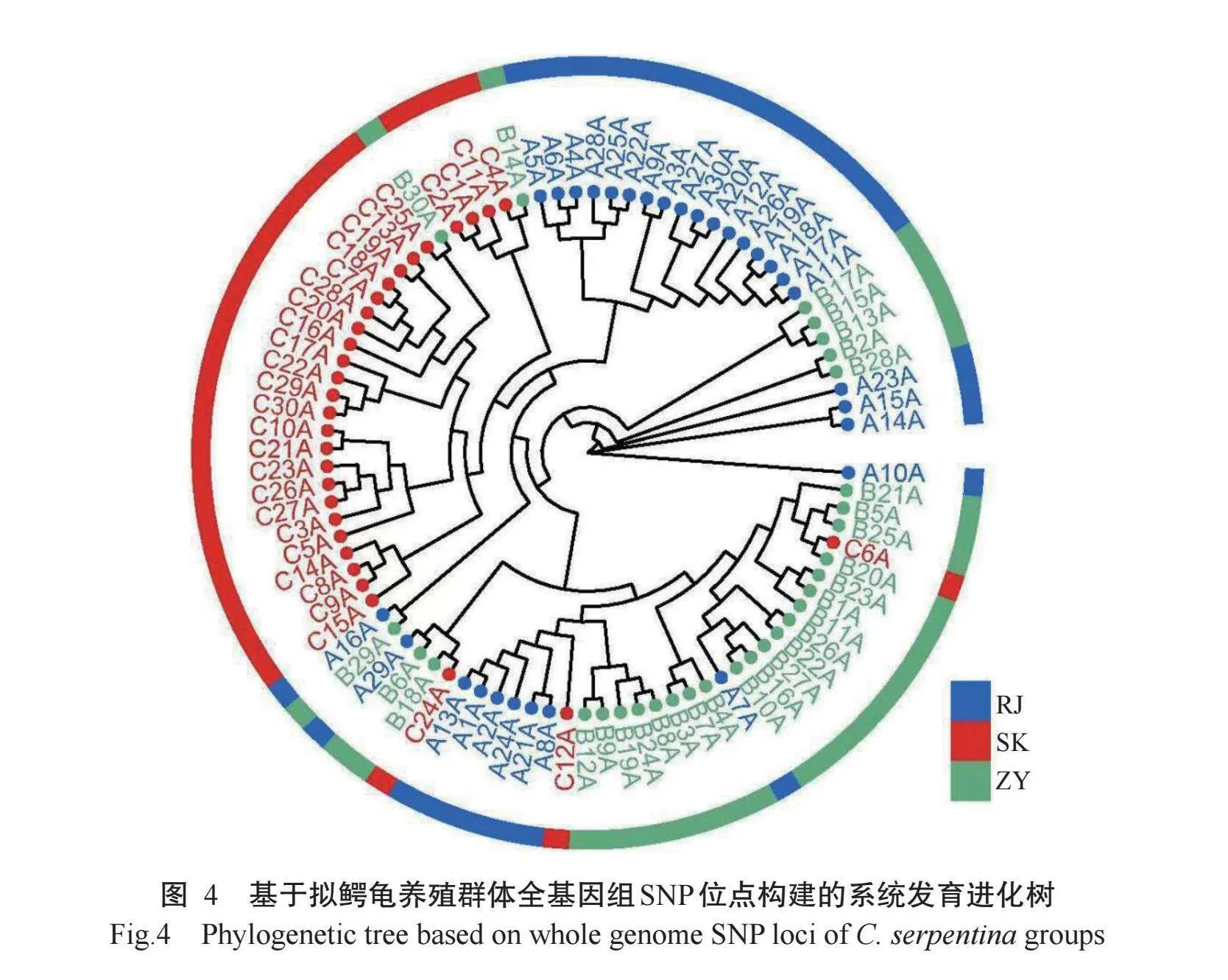
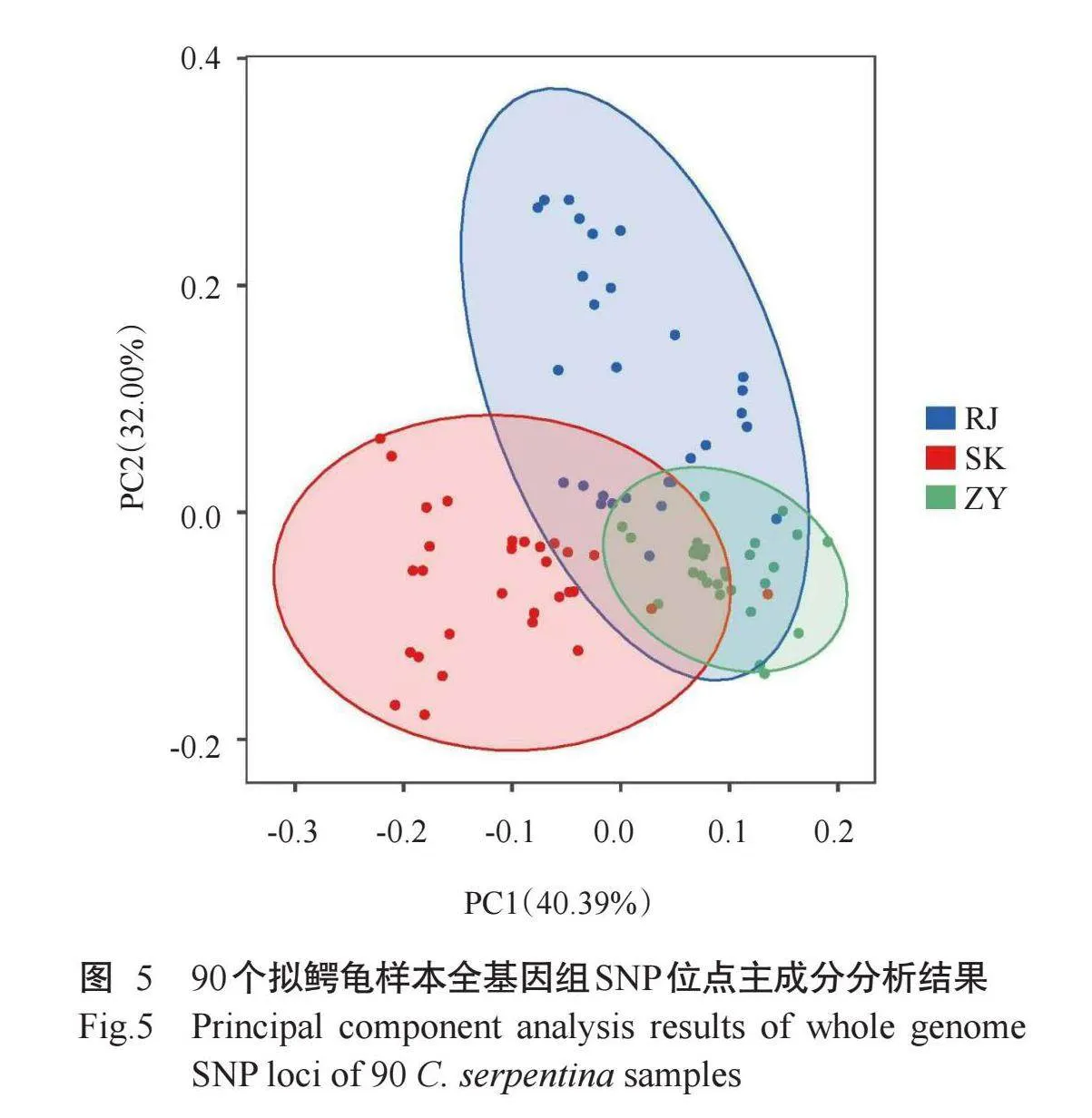
基于90个拟鳄龟样本的全基因组SNP位点,绘制系统发育进化树及进行主成分分析,以评估测序个体的群体遗传结构。结果显示,在构建的系统发育进化树上无法明显区分相邻较近的群体,故推测90个拟鳄龟样本具有相似的遗传背景。由系统发育进化树(图4)的拓扑结构可看出,3个拟鳄龟养殖群体整体上可分为两大分支,第一分支主要由RJ群体和SK群体组成,第二分支主要由ZY群体组成,与形态聚类分析结果基本一致,即群体间的亲缘关系较近。主成分分析结果(图5)表明,3个拟鳄龟养殖群体间的相互距离较近,且存在相互交叉现象。3个拟鳄龟养殖群体中有部分个体紧密聚集在一起,其中,RJ群体较分散,而ZY群体较聚集。系统发育进化树与主成分分析结果基本吻合,说明3个拟鳄龟养殖群体的亲缘关系和遗传距离较近。
3 讨论
Fst是评价群体间遗传分化的重要指标之一(傅洪拓等,2010),当Fst保持在0~0.05时,表明群体间存在很小的遗传分化,基本上不用考虑;当Fst维持在0.05~0.15时,表明群体间呈中等程度的分化;当Fst在0.15~0.25时,表明群体间存在较大的遗传分化;当Fst大于0.25时,表明群体间遗传分化显著(McConnell et al.,1995)。本研究中,3个拟鳄龟养殖群体间的Fst为0.0056~0.0138,且100%的变异来源于群体内部,没有变异来自于群体间,表明人工养殖拟鳄龟群体间的遗传分化程度相对较低。Da是衡量群体间遗传分化程度的重要指标。3个拟鳄龟养殖群体的Dα仅为0.0056~0.0139,说明拟鳄龟选育群体的遗传结构并未发生明显改变。选择育种通常需要连续多代选育才能获得可稳定遗传的优良性状,但随着选育代数的增加,群体遗传多样性不断下降,进而影响选育效果,因此适当拓宽选育群体的遗传基础可为其选育工作的持续进行提供有力保障(刘志刚等,2021)。聚类分析是对不同群体间的差异水平进行初步量化,可反映群体间亲缘关系的远近(李勤生等,1991)。本研究的形态聚类分析和全基因组SNP位点分析均显示,3个拟鳄龟养殖群体可清晰分为两大类,RJ群体与SK群体为一类,ZY群体单独为一类,说明RJ群体与SK群体的亲缘关系较近,二者与ZY群体的亲缘关系相对较远,但由于群体间的Fst较低,因此3个群体无法完全区分开。
遗传多样性是生物多样性的一个层次,是物种在长期进化过程中不断发生变异所产生的特征,能反映物种或种群在进化过程中的适应性与生存能力(Liu et al.,2020;马克岩等,2023)。种质资源的遗传多样性是育种工作中衡量各品种亲缘关系远近、杂交选配和杂种优势强弱的重要指标,因此,育种工作中要格外重视种质资源的遗传背景,明确各品种间的遗传相似系数(刘易科等,2020)。随着测序技术的快速发展,从第一代测序到第三代高通量测序,致使各品种间的遗传多样性研究更透彻深入。与简化基因组测序对比,全基因组重测序理论上覆盖了整个基因组,10.0X以上的测序深度可覆盖90%~99%的基因组,满足群体遗传多样性分析。近十年来,全基因组重测序及其产生的海量SNP位点已广泛应用于动植物和微生物群体进化与重要性状关联分析,为分子育种基因型鉴定系统提供了新的平台(沈颖越等,2020)。滕爽爽等(2021)利用多态性SNP标记分析缢蛏(Sinonovacula constricta)5个群体的遗传多样性,结果显示5个群体的Ho平均值范围为0.265~0.317。吴昊天等(2022)通过全基因组重测序分析新疆和西藏棕鳟(Salmo trutta)群体的遗传多样性,发现西藏棕鳟群体的Ho(0.324)明显高于新疆棕鳟群体(0.316)。本研究中,3个拟鳄龟养殖群体的Ho为0.171~0.205。相对而言,拟鳄龟养殖群体的遗传多样性略低,可能与其为引进种有关。拟鳄龟在国内没有广泛分布,引进基础数量有限,虽然经过多年人工养殖发展,但多个养殖企业的养殖群体可能来源于同一基础群体,群体间的Fst较低。
与微卫星等重复序列多态标记相比,SNP具有遗传稳定性更高的特点,尤其是处于编码区的SNP工作效率更高,在大样本量检测分析时更合适,即SNP标记被视为最具应用前景的分子标记,已广泛应用于水产动物遗传图谱构建、遗传多样性分析及分子标记辅助育种等研究领域(滕爽爽等,2021;雷骆等,2023)。刘丽等(2013)通过开发具有高度多态性的微卫星分子标记分析拟鳄龟养殖群体的遗传多样性,结果显示,拟鳄龟群体的平均期望杂合度(He)为0.600,平均Ho为0.725,故推测拟鳄龟养殖群体存在较高的遗传多样性水平。本研究结果与刘丽等(2013)的研究结果存在差异,究其原因可能是:(1)微卫星分子标记与SNP标记存在差异;(2)拟鳄龟的取样存在差异,与近年来的拟鳄龟养殖规模呈现萎缩,种质资源的保存受到一定影响有关。本研究通过全基因组重测序,从基因组水平解析拟鳄龟养殖群体的种质特性,结果显示3个拟鳄龟养殖群体遗传相似性较高、遗传背景不够丰富、群体间遗传分化程度低,种源较单一。因此,在今后的育种工作亟需引入新的种质资源,为种质创新提供更丰富的遗传基础。
4 结论
3个拟鳄龟养殖群体遗传多态性较低,遗传背景不够丰富,群体间遗传分化程度低,推测具有相似的遗传背景,来源于同一引种群体。因此,在今后的育种工作亟需引入新的种质资源,为种质创新提供更丰富的遗传基础。
参考文献((References)):
阿旺措吉,仁青措姆,黄舒泓,周先辉,德吉,索朗达,王小龙,吴玉江,巴贵. 2022. 基于重测序数据的昌都黑山羊遗传多样性及群体结构分析[J]. 家畜生态学报,43(10):31-35.[ Awangcuoji,Renqingcuomu,Huang S H,Zhou X H,Deji,Solangda,Wang X L,Wu Y J,Bagui. 2022. Genetic diversity and population structure analysis of Changdu black sheep based on resequencing data[J]. Acta Ecologiae Animalis Domastici,43(10):31-35.] doi:10.3969/j.issn. 1673-1182.2022.10.005.
傅洪拓,乔慧,李法君,吴滟,龚永生,蒋速飞,熊贻伟,王宁. 2010. 长江不同江段青虾的遗传多样性[J]. 水产学报,34(2):204-212.[ Fu H T,Qiao H,Li F J,Wu Y,Gong Y S,Jiang S F,Xiong Y W,Wang N. 2010. Genetic diversity of Macrobrachium nipponense on a regional scale in the Yangtze River[J]. Journal of Fisheries of China,34(2):204-212.] doi:10.3724/SP.J.1231.2010.06422.
贾舒雯,刘萍,韩智科,李健,潘鲁青. 2011. 脊尾白虾微卫星富集文库的构建与多态性标记的筛选[J]. 水产学报,35(12):1787-1794.[ Jia S W,Liu P,Han Z K,Li J,Pan L Q. 2011. Construction of microsatellite-enriched library and isolation of microsatellite markers in the ridgetail white prawn (Exopalaemon carinicauda)[J]. Journal of Fishe-ries of China,35(12):1787-1794.] doi:10.3724/SP.J.1231.2011.17612.
雷骆,祝骏贤,陈辰,王亚坤,刘晓莉,洪孝友,于凌云,魏成清,李伟,朱新平. 2023. 基于转录组测序的中华鳖SSR和SNP特征分析及性别标记筛选[J]. 广东海洋大学学报,43(1):25-32.[ Lei L,Zhu J X,Chen C,Wang Y K,Liu X L,Hong X Y,Yu L Y,Wei C Q,Li W,Zhu X P. 2023. Analysis of SSR and SNP characteristics and scree-ning of sex-linked markers in Chinese soft-shelled turtle based on transcriptome sequencing[J]. Journal of Guang‐dong Ocean University,43(1):25-32.] doi:10.3969/j.issn.
1673-9159.2023.01.004.
黎旺长,刘玮玮,龙佳佳,杨小淦. 2023. 基于基因组重测序的广西地方猪种遗传多样性和选择信号分析[J]. 南方农业学报,54(8):2415-2422. [Li W C,Liu W W,Long J J,Yang X G. 2023. Genetic diversity and selection signals analysis of Guangxi local pig breeds based on whole-genome resequencing[J]. Journal of Southern Agriculture,54(8):2415-2422.] doi:10.3969/j.issn.2095-1191.2023.08.023.
李勤生,蔡庆华,华俐,刘建康. 1991. 东湖异养细菌群落的分类结构和聚类分析[J]. 水生生物学报,15(3):242-254. [Li Q S,Cai Q H,Hua L,Liu J K. 1991. The taxonomic structure and cluster analysis of heterotrophic bacterial communities from the Donghu Lake[J]. Acta Hydrobio‐logica Sinica,15(3):242-254.]
李思明,周定刚,欧阳玲花,温安祥,黎德兵. 2008. 鳄龟血液抗菌肽生物活性研究[J]. 经济动物学报,12(1):34-37. [Li S M,Zhou D G,Ouyang L H,Wen A X,Li D B. 2008. Study on biological activity of antimicrobial peptides extracted from blood of Chelydra serpentina[J]. Journal of Economic Animal,12(1):34-37.] doi:10.13326/j.jea. 2008.01.013.
李振业,叶海胜,唐燕高,徐树珍,叶威龙,程家高. 2014. 蛇鳄龟生态产卵与控温孵化技术初步研究[J]. 安徽农业科学,42(17):5509-5510.[ Li Z Y,Ye H S,Tang Y G,Xu S Z,Ye W L,Cheng J G. 2014. Preliminary study of the eco‐logical spawning and temperature control incubation tech‐nology of Chelydraser pentina[J]. Journal of Anhui Agri‐cultural Sciences,42(17):5509-5510.] doi:10.13989/j.cnki. 0517-6611.2014.17.115.
刘翠娥,李若利,王建明,刘继芳,梁启防,叶青生. 2007. 小鳄龟含肉率和肌肉营养成分分析及品质评定[J]. 养殖与饲料,(10):9-13. [Liu C E,Li R L,Wang J M,Liu J F,Liang Q F,Ye Q S. 2007. Analysis and quality assessment of meat content and muscle nutrients of Chelydra serpen‐tina[J]. Animals Breeding and Feed,(10):9-13.] doi:10. 13300/j.cnki.cn42-1648/s.2007.10.034.
刘继芳,蔡嘉庆,李若利,刘翠娥,姚绍云,梁启防. 2009. 鳄龟常见病害及其防治技术[J]. 齐鲁渔业,26(11):43-44. [Liu J F,Cai J Q,Li R L,Liu C E,Yao S Y,Liang Q F. 2009. Common diseases of Chelydra serpentina and their
control techniques[J]. Shandong Fisheries,26(11):43-44.]刘坚红. 2012. 鳄龟稚龟越冬养殖试验[J]. 广西农学报,27(3):55-58. [Liu J H. 2012. Experiment on overwinter breeding of juvenile Chelydra serpentina[J]. Journal of Guangxi Agriculture,27(3):55-58.] doi:10.3969/j.issn. 1003-4374.2012.03.016.
刘丽,刘海情,郭昱嵩,王中铎,刘楚吾. 2013. 蛇鳄龟微卫星标记的开发及一个养殖群体的遗传多样性分析[J]. 水生生物学报,37(4):669-677. [Liu L,Liu H Q,Guo Y S,Wang Z D,Liu C W. 2013. Isolation of microsatellite markers in the common snapping turtle( Chelydra serpen‐tina L.) and its application in genetic diversity analysis in a cultured population[J]. Acta Hydrobiologica Sinica,37(4):669-677.] doi:10.7541/2013.78.
刘易科,朱展望,陈泠,邹娟,佟汉文,朱光,何伟杰,张宇庆,高春保. 2020. 基于SNP标记揭示我国小麦品种(系)的遗传多样性[J]. 作物学报,46(2):307-314.[ Liu Y K,Zhu Z W,Chen L,Zou J,Tong H W,Zhu G,He W J,Zhang Q Y,Gao C B. 2020. Revealing the genetic diversity of wheat varieties( lines) in China based on SNP marker[s J]. Acta Agronomica Sinica,46(2):307-314.] doi:10.3724/SP.J.1006.2020.91039.
刘志刚,曹建萌,高风英,可小丽,王淼,衣萌萌,卢迈新. 2021. 罗非鱼“粤闽1号”母本选育群体世代间遗传差异的微卫星分析[J]. 大连海洋大学学报,36(1):16-22. [Liu Z G,Cao J M,Gao F Y,Ke X L,Wang M,Yi M M,Lu M X. 2021. Genetic differentiation analysis of maternal selective breeding generations of tilapia“ Yuemin No. 1” using microsatellites[J]. Journal of Dalian Ocean Univer‐sity,36(1):16-22.] doi:10.16535/j.cnki.dlhyxb.2020-208.
马钧,樊安平,王武生,张金川,江晓军,马瑞军,贾社强,刘飞,雷初朝,黄永震. 2023. 全基因组重测序解析秦川牛保种群遗传多样性和遗传结构[J]. 遗传,45(7):602-616.[ Ma J,Fan A P,Wang W S,Zhang J C,Jiang X J,Ma R J,Jia S Q,Liu F,Lei C C,Huang Y Z. 2023. Analysis of genetic diversity and genetic structure of Qinchuan cattle conservation population using whole-genome resequencing[J]. Hereditas,45(7):602-616.] doi:10.16288/j.yczz.23-
115.
马克岩,韩金涛,白雅琴,李讨讨,马友记. 2023. 基于简化基因组测序的永登七山羊遗传多样性分析[J]. 畜牧兽医学报,54(5):1939-1950.[ Ma K Y,Han J T,Bai Y Q,Li T T,Ma Y J. 2023. Genetic diversity analysis of Yongdeng Qishan sheep based on specific-locus amplified fragment sequencing[J]. Acta Veterinaria et Zootechnica Sinica,54(5):1939-1950.] doi:10.11843/j.issn.0366-6964.2023.05.016.
沈保平,万全,胡海涛. 2008. 鳄龟的人工繁殖和稚龟培育技术研究[J]. 现代农业科技,(22):227-228.[ Shen B P,Wan Q,Hu H T. 2008. Research on the artificial reproduc‐tion of crocodile turtles and the cultivation techniques of juvenile turtles[J]. Modern Agricultural Science and Tech‐nology,(22):227-228.] doi:10.3969/j.issn.1007-5739.2008.22.159.
沈颖越,金群力,蔡为明,范丽军,冯伟林,宋婷婷. 2020. 基于重测序信息的金针菇菌株资源遗传多样性及群体结构分析[J]. 菌物学报,39(6):1016-1028.[ Shen Y Y,Jin Q L,Cai W M,Fan L J,Feng W L,Song T T. 2020. Analyses of population diversity and structure of Flammulina filifor‐mis strains based on whole genome resequencing data[J]. Mycosystema,39(6):1016-1028.] doi:10.13346/j.myco‐systema.200041.
滕爽爽,胡高宇,范建勋,柴雪良,肖国强. 2021. 缢蛏5个群体遗传多样性和遗传分化的SNP分析[J]. 水生生物学报,45(4):861-870.[ Teng S S,Hu G Y,Fan J X,Chai X L,Xiao G Q. 2021. Genetic diversity and genetic differen‐tiation analysis of Sinonovacula constricta populations revealed by SNP markers[J]. Acta Hydrobiologica Sinica,45(4):861-870.] doi:10.7541/2021.2019.145.
王统苗,郭其新,白皓,常国斌,陈国宏. 2022. 基于全基因组重测序对不同鸭遗传资源进行群体结构分析[J]. 中国畜牧杂志,57(11):78-81. [Wang T M,Guo Q X,Bai H,Chang G B,Chen G H. 2022. Analysis of population struc‐ture of different duck genetic resources based on whole-genome resequencing[J]. Chinese Journal of Animal Scien-ce,57(11):78-81.] doi:10.19556/j.0258-7033.20210126-05.
王中铎,何传猛,姚泽彬,李金蓬,陈子阳,邓爱萍,赖卓欣,李银芳,董忠典,郭昱嵩. 2023. 全基因组重测序筛选弓背青鳉三亚群体性别遗传标记[J]. 广东海洋大学学报,43(4):69-75.[ Wang Z D,He C M,Yao Z B,Li J P,Chen Z Y,Deng A P,Lai Z X,Li Y F,Dong Z D,Guo Y S. 2023. Screening of sex genetic markers in the Sanya population of Oryzias curvinotus by whole-genome resequencing[J]. Journal of Guangdong Ocean University,43(4):69-75.] doi:10.3969/j.issn.1673-9159.2023.04.009.
吴昊天,李亚琳,王军,陈晓雯,陆颖,王成辉. 2022. 新疆和西藏棕鳟群体的遗传多样性分析[J]. 南方农业学报,53(5):1425-1433.[ Wu H T,Li Y L,Wang J,Chen X W,Lu Y,Wang C H. 2022. Genetic diversity analysis on brown trout populations in Xinjiang and Tibe[t J]. Journal of Sou-thern Agriculture,53(5):1425-1433.] doi:10.3969/j.issn. 2095-1191.2022.05.025.
徐煜,黄小帅,胡晓娟,徐武杰,苏浩昌,文国樑,曹煜成,杨铿. 2021. 凡纳滨对虾进口亲虾子一代群体的遗传变异分析[J]. 南方农业学报,52(11):3139-3146.[ Xu Y,Huang X S,Hu X J,Xu W J,Su H C,Wen G L,Cao Y C,Yang K. 2021. Genetic variation analysis among first filial genera‐tion of introduced Litopenaeus vannamei stocks[J]. Jour‐nal of Southern Agriculture,52(11):3139-3146.] doi:10. 3969/j.issn.2095-1191.2021.11.026.
叶泰荣,李家乐,李应森. 2007. 鳄龟(Chelydra serpentina)的营养成分分析[J]. 现代渔业信息,22(6):6-9.[ Ye T R,Li J L,Li Y S. 2007. Analysis on nutritional composition of Chelydra serpentina[J]. Modern Fisheries Information,22(6):6-9.] doi:10.3969/j.issn.1004-8340.2007.06.002.
张彦,熊和丽,张斌,相德才,刘韶娜,赵智勇. 2023. 基于全基因组重测序的藏猪脂质沉积相关基因鉴定[J]. 南方农业学报,54(2):325-335. [Zhang Y,Xiong H L,Zhang B,Xiang D C,Liu S N,Zhao Z Y. 2023. Identification of lipid deposition related genes in Tibetan pigs based on whole genome re-sequencing[J]. Journal of Southern Agri‐culture,54(2):325-335.] doi:10.3969/j.issn.2095-1191. 2023.02.001.
朱皓东. 2022. 罗氏沼虾野生和养殖群体形态学及遗传多样性研究[D]. 上海:上海海洋大学.[ Zhu H D. 2022. Mor‐phological and genetic diversity of wild and cultured stocks of Macrobrachium rosenbergi[iD]. Shanghai:Shang‐hai Ocean University.] doi:10.27314/d.cnki.gsscu.2022.00 0228.
Alexander D H,Novembre J,Lange K. 2009. Fast model-based estimation of ancestry in unrelated individuals[J]. Genome Research,19(9):1655-1664. doi:10.1101/gr.094052.109.
Botstein D,White R L,Skolnick M,Davis R W. 1980. Con‐struction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics,32(3):314-331.
Chang C C,Chow C C,Tellier L C,Vattikuti S M,Purcell S M,Lee J J. 2015. Second-generation plink: Rising to the cha-llenge of larger and richer datasets[J]. GigaScience,4(1):s13742-015-0047-8. doi:10.1186/s13742-015-0047-8.
de Solla S R,Martin P A. 2011. Absorption of current use pesti‐cides by snapping turtle (Chelydra serpentina) eggs intreated soil[J]. Chemosphere,85(5):820-825. doi:10. 1016/j.chemosphere.2011.06.080.
He W,Zhao S,Liu X,Dong S,Lv J,Liu D,Wang J,Meng Z. 2013. ReSeqTools:An integrated toolkit for large-scale next-generation sequencing based resequencing analysis[J]. Genetics and Molecular Research,12(4):6275-6283. doi:10.4238/2013.December.4.15.
Holsinger K E,Weir B S. 2009. Genetics in geographically structured populations:Defining,estimating and interpre-ting FS[TJ]. Nature Reviews Genetics,10(9):639-650. doi:10.1038/nrg2611.
Lawniczak C J,Teece M A. 2009. Lipid metabolism during em-bryonic development of the common snapping turtle,Che‐lydra serpentina[J]. Comparative Biochemistry and Phy-siology. Part B:Biochemistry amp; Molecular Biology,153(1):73-80. doi:10.1016/j.cbpb.2009.01.016.
Letunic L,Bork P. 2021. Interactive tree of life( iTOL) v5:An online tool for phylogenetic tree display and annotation[J]. Nucleic Acids Research,49(W1):W293-W296. doi:10.1093/NAR/GKAB301.
Li H,Handsaker B,Wysoker A,Fennel T,Ruan J,Homer N,Marth G,Abecasis G,Durbin R. 2009. The sequence align‐ment/map format and SAMtools[J]. Bioinformatics,25(16):2078-2079. doi:10.1093/bioinformatics/btp352.
Liu F,Qu Y K,Geng C,Wang A M,Zhang J H,Li J F,Chen K J,Liu B,Tian H Y,Yang W P,Yu Y B. 2020. Analysis of the population structure and genetic diversity of the red swamp crayfish (Procambarus clarkii) in China using SSR markers[J]. Electronic Journal of Biotechnology,47:59-71. doi:10.1016/j.ejbt.2020.06.007.
Ma J,Gao X,Li J Y,Gao H J,Wang Z Z,Zhang L P,Xu L Y,Gao H,Li H W,Wang Y H,Zhu B,Cai W T,Wang C Y,Chen Y. 2021. Assessing the genetic background and selec‐tion signatures of Huaxi cattle using high-density SNP array[J]. Animals (Basel),11(12):36-49. doi:10.3390/ANI11123469.
McConnell S,Hamilton L,Morris D,Cook D,Paquet D,Bent‐zen P,Wright J. 1995. Isolation of salmonid microsatellite loci and their application to the population genetics of Canadian east coast stocks of Atlantic salmon[J]. Aquacul‐
ture,137(1-4):19-30. doi:10.1016/0044-8486(95)01111-0.
McKenna A,Hanna M,Banks E,Sivachenko A,Cibulskis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,Daly M,DePristo M A. 2010. The Genome Analysis Toolkit:A MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research,20(9):1297-1303. doi:10.1101/gr.107524.110.
Paterson J E,Steinberg B D,Litzgus J D. 2012. Generally spe‐cialized or especially general? Habitat selection by Snap‐ping turtles (Chelydra serpentina) in central Ontario[J]. Canadian Journal of Zoology,90(2):139-149. doi:10.1139/z11-118.
Patterson N,Price A L,Reich D. 2006. Population structure and eigenanalysis[J]. PLoS Genetics,12(12):e190. doi:10. 1371/journal.pgen.0020190.
Phillips C A,Dimmick W W,Carr J L. 1996. Conservation genetics of the common snapping turtle( Chelydra serpen‐tina)[J]. Conservation Biology,10(2):397-405. doi:10. 1046/j.1523-1739.1996.10020397.x.
Rhen T,Jangula A,Schroeder A,Woodward-Bosh R. 2009. The platelet-derived growth factor signaling system in snap‐ping turtle embryos,Chelydra serpentina:Potential role in temperature-dependent sex determination and testis deve-lopmen[t J]. General and Comparative Endocrinology,161(3):335-343. doi:10.1016/j.ygcen.2009.01.014.
Saha A,Andersson A,Kurland S,Keehnen N L P,Kutschera V E,Hössjer O,Ekman D,Karlsson S,Kardos M,Ståhl G,Allendorf F W,Ryman N,Laikre L. 2022. Whole-genome resequencing confirms reproductive isolation between sym‐patric demesof brown trout (Salmo trutta) detected with allozyme[J]. Molecular Ecology,31(2):498-511. doi:10. 1111/mec.16252.
Schnars J L,Voss M A,Stauffer J R. 2011. An egg injection technique to evaluate the effect of polychlorinated biphe‐nylson the hatching success of the snapping turtle (Chel‐dra serpentina serpentina)[J]. Environmental Toxicology and Chemisiry,30(4):915-919. doi:10.1002/etc.455.
Spinks P Q,Thomson R C,Bradley Shaffer H. 2014. The advantages of going large:Genome-wide SNPs clarify the complex population history and systematics of the threa-tened western pond turtle[J]. Molecular Ecology,23(9):2228-2241. doi:10.1111/mec.12736.
Tamura K,Stecher G,Kumar S. 2021. MEGA11:Molecular evolutionary genetics analysis version 11[J]. Molecular Biology and Evolution,38(7):3022-3027. doi:10.1093/molbev/msab120.
van der Nest M A,Hlongwane N,Hadebe K,Chan W Y,van der Merwe N A,de Vos L,Greyling B,Kooverjee B B,Soma P,Dzomba E F,Bradfield M,Muchadeyi F C. 2020. Breed ancestry divergence admixture,and selection pa-tterns of the Simbra crossbreed[J]. Frontiers in Genetics,11:608-650. doi:10.3389/fgene.2020.608650.
Vervust B,Brecko J,Herrel A. 2011. Temperature effects on snapping performance in the common snapper Chelydra serpentina (Reptilia,Testudines)[J]. Journal of Experi‐mental Zoology,315(1):41-47. doi:10.1002/jez.650.
Wickham H,Navarro D,Pedersen T L. 2017. ggplot2:Elegant graphics for data analysis[M]. New York:Springer.
Zhang S J,Yao Z,Li X M,Zhang Z J,Liu X,Yang P,Chen N B,Xia X T,Lü S J,Shi Q T,Wang E Y,Ru B R,Jiang Y,Lei C Z,Chen H,Huang Y Z. 2022. Assessing genomic diversity and signatures of selection in Pinan cattle using whole-genome sequencing date[J]. BMC Genomics,23(1):460. doi:10.1186/S12864-022-08645-Y.
(责任编辑 兰宗宝)
