普通油茶与小果油茶的叶绿体基因分子鉴定标记
2024-01-30陈璐张剑赵松子陈凯谢昊星张华轩冯立云赵耀戎俊
陈璐 张剑 赵松子 陈凯 谢昊星 张华轩 冯立云 赵耀 戎俊
摘 要:【目的】开发能够准确、便捷、快速鉴定普通油茶与小果油茶的叶绿体基因分子标记,为油茶遗传资源的挖掘与利用提供参考。【方法】基于高通量测序技术组装了普通油茶和小果油茶的叶绿体全基因組,通过比较基因组学技术寻找两者叶绿体全基因组的差异,并根据其差异来开发可用于鉴定普通油茶与小果油茶的分子标记。为验证标记的有效性,分别选取具有代表性的12 个普通油茶居群和6 个小果油茶居群的个体,提取叶绿体全基因组,进行PCR 扩增和凝胶电泳检验。【结果】组装的普通油茶与小果油茶叶绿体基因组序列全长分别为156 977、156 539 bp,两者叶绿体全基因组具有高度的保守性,但小果油茶在atpH 基因与atpI 基因间的非编码区存在长度为452 bp 的大片段缺失。所开发引物的PCR 扩增结果表明,所有普通油茶样本均可扩增出609 bp的条带,而小果油茶扩增出227 bp 的条带,条带差异显著且稳定。【结论】使用所开发的分子标记能够稳定、便捷、精确地鉴定出普通油茶与小果油茶,但是使用时应先确定所鉴定的物种为普通油茶和小果油茶中的一种。
关键词:普通油茶;小果油茶;叶绿体全基因组;遗传分化;分子标记
中图分类号:S601 文献标志码:A 文章编号:1003—8981(2023)03—0206—08
油茶是山茶科Theaceae 山茶属Camellia 中种子油脂含量较高、有栽培经济价值的一类植物的总称[1]。油茶籽油富含以油酸与亚油酸为主的不饱和脂肪酸,其营养价值可以与橄榄油媲美,是一种优质的食用植物油[2-4]。在我国,油茶具有悠久的栽培历史,分布地区广、栽培面积大,其中普通油茶C. oleifera 的栽种面积已经突破446.7 万hm2,为第一大木本油料作物,小果油茶C. meiocarpa 次之[5-6]。
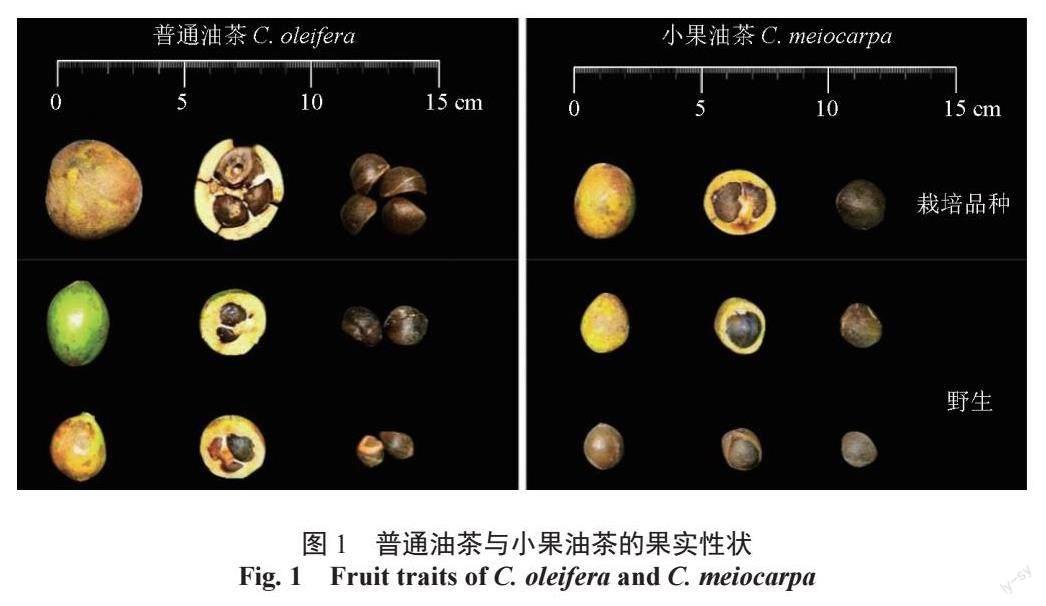
普通油茶与小果油茶的花、叶、果性状及株形等都具有高度的相似性[7-9]。因此,对于两者的亲缘关系存在一些争议。胡先骕[10] 最早命名了小果油茶,而张宏达等[11] 认为小果油茶是普通油茶的1 个变种。经测定,两者存在着明确的染色体倍性差异(普通油茶6 倍体,小果油茶4 倍体)以及较大的遗传分化[12]。形态性状(果实大小、种皮厚度、单果种籽数量等)常被用于辨别两者的种类[13],个体间性状的连续变异,导致两者的中间类型不易区分(图1),且又存在同域分布的现象[14],因此,通过观测常规形态性状难以准确、有效区分两者的种类。叶绿体基因保守且含有较多的遗传信息,拥有较高的分辨率和更好的通用性,因此被广泛应用于系统发育研究等领域[15],有利用叶绿体基因组来分析系统发育关系复杂的山茶属植物的报道[16]。根据叶绿体基因间的差异开发分子标记是一种常用的物种鉴定方法,殷鑫等[17] 以山茶属9 个组9 个物种的叶绿体基因组建立了山茶属cpSSR 分子标记体系,并初步筛选出在种间具有较好多态性的cpSSR 标记。关于油茶叶绿体基因组的研究报道较多,其中‘三华油茶的叶绿体基因组已被发布[18],也有其他一些近缘种的相关报道[19-20]。然而,鲜见关于小果油茶叶绿体全基因组序列的研究报道。
‘赣无1是小果型普通油茶,是较早被选育出来的江西省良种,结实率高,推广面积较大。与‘三华系列相比,‘赣无1果实小,更适宜与小果油茶进行比较。本研究中拟组装出完整的小果油茶和‘赣无1的叶绿体基因组,通过进行比较基因组学分析,挖掘普通油茶与小果油茶在叶绿体基因组层面的差异,并基于此开发能够准确、便捷鉴定两者的叶绿体基因分子标记。
1 材料与方法
1.1 样品采集与叶绿体基因组测序、组装及功能注释
从江西省林业科学院山茶园中采集‘赣无1和小果油茶‘白皮中子的叶片,作为样品。取待测样品干叶片0.02 g,加液氮彻底磨碎,使用新型植物基因组DNA提取试剂盒(TIANGEN,上海)提取样本的全基因组DNA。采用将二代测序平台与三代测序平台结合的方式,获得普通油茶与小果油茶叶绿体全基因组数据。组装之前,对原始数据进行过滤,将带有接头的片段、质量分数小于20 的低测序质量数据去除。之后根据二代加三代混合组装原理,采用以下3 个步骤进行混合组装:使用SPAdes v3.10.1 软件[21] 通过Illumina 和PacBio 数据组装基因组框架;验证组装结果,使叶绿体基因组成环,将gap 补齐;将短读长序列重新比对到组装好的叶绿体基因组骨架上,纠正错误碱基,检查是否存在碱基的插入和删除。
使用在线质体注释软件DOGMA[22] 进行叶绿体基因组的注释,使用软件默认参数预测蛋白质、tRNA 及rRNA,即“Gene Code for Blastx”设为“11Plant plastid”,“Percent identity cutoff for protein coding genes”设为60,“Percent identity cutoff forRNAs”设为80。5 个数据库(KEGG[23]、COG[24]、NR、Swiss-Prot[25] 和GO[26]) 的叶绿体全基因组比对参数为E 值不大于1×10-5、最小对齐长度百分比不小于40%。使用Organellar Genome DRAWv1.2 软件[27] 绘制环状的叶绿体基因组图。
1.2 叶绿体基因组序列比较
分析叶绿体基因组序列的结构,利用在线全局比对软件EMBOSS 中的Stretcher 进行普通油茶和小果油茶的叶绿体全基因组序列比对。利用在线软件IRscope 分析叶绿体基因组的LSC/IRb、IRb/SSC、SSC/IRa、IRa/LSC 4 个边界区域分布情况,以评估普通油茶和小果油茶叶绿体基因组IR区的扩张和收缩情况。
1.3 叶绿体基因分子标记开发及验证
1.3.1 分子标记设计与开发
基于普通油茶、小果油茶及其近缘种(从NCBI数据库中获取)叶绿体基因组比对结果,以普通油茶的叶绿体全基因组序列为参考,针对叶绿体基因组的变异位点,使用在线软件primer3 设计引物[28]。
1.3.2 PCR 扩增、测序
利用设计的引物对普通油茶、小果油茶以及其他近源种的DNA,使用耶拿Biometra Tone96G PCR 扩增仪进行特异性扩增。PCR 反应体系组成为2×FastTaq Premix 10 μL(TOLOBIO, 上海),10 μmol/L 上、下游引物各1 μL,2 μmol/L的DNA 模板1 μL(反应体系模板浓度不宜超过0.2 μmol/L),ddH2O 7 μL。扩增条件:94 ℃ 预变性5 min;94 ℃ 变性30 s,57 ℃ 退火30 s,72 ℃延伸90 s,共40 个循环;最后于72 ℃补平10 min,终止温度为4 ℃。使用1% 琼脂糖凝胶电泳检验扩增产物,确定扩增成功后,进行一代Sanger 测序,验证位点作为分子标记的潜力。
1.3.3 分子标記验证
根据崔相艳等[29] 基于生态位模型预测的油茶潜在分布区,从全国油茶适生区内选取了具有代表性的12 个区域的普通油茶居群以及6 个区域的小果油茶居群,在每个居群中随机挑选3 株个体进行采样,居群信息见表1。所有样本的全基因组DNA 提取、PCR 扩增及凝胶电泳等方法同1.3.2。
2 结果与分析
2.1 普通油茶与小果油茶叶绿体基因组测序质控
使用Illumina Hiseq 2500 平台测序下机得到6.95 G 原始数据,经质控后得到5.48 G 高质量测序数据,读长(read)为150 bp。使用Bowtie2 比对至参考基因组C. sinensis 筛选属于叶绿体基因组的序列,利用Samtools 软件计算得到普通油茶与小果油茶叶绿体基因组测序平均覆盖度为103.025 6,Q20 分别为91.73%、97.15%,Q30 分别为86.39%、93.03%,表明该测序数据达到后续叶绿体基因组研究的要求。三代PacBio 测序平台的质控结果表明,测得的小果油茶与普通油茶样品碱基总量达到预期目标,N50 均超过6 400 bp,获得的读长集中分布在3 000 ~ 10 000 bp,测序质量较高,达到三代组装要求。
2.2 普通油茶与小果油茶叶绿体基因组特征
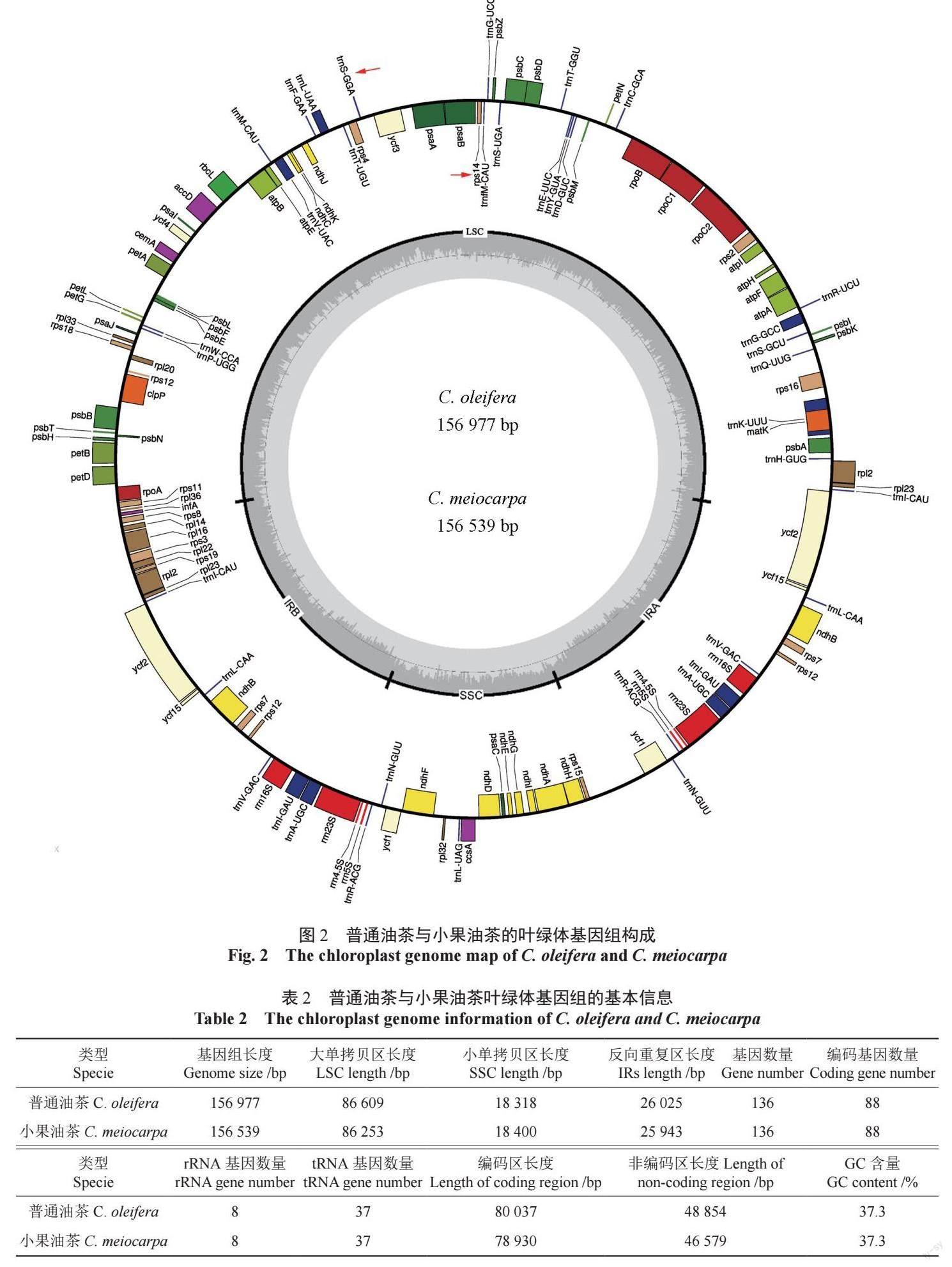
普通油茶、小果油茶叶绿体基因组由1 个环状双链DNA 分子构成(图2)。普通油茶与小果油茶的叶绿体基因组之间有一定的差异,序列全长分别为156 977、156 539 bp。两者叶绿体基因组呈典型的四分体结构,普通油茶的大单拷贝区(LSC)和反向重复区(IRs)均长于小果油茶,而小单拷贝区(SSC)短了82 bp(表2)。两者的叶绿体基因组基因数量、编码基因数量相同,分别为136、88,但是编码区序列长度及非编码区序列长度有较大差异,普通油茶的序列更长。rRNA 与tRNA 数量均保持一致。
利用mVISTA 软件,对普通油茶、小果油茶叶绿体基因组进行比较分析。两者叶绿体基因组间拥有高度保守的结构,不存在叶绿体基因组结构上的变异及重组(图3)。但在部分位点出现了多位点空白现象,其中atpH 基因与atpI 基因间的非编码区存在大片段的缺失,与普通油茶叶绿体全基因组相比,小果油茶存在大片段缺失,缺失片段长度为452 bp。
2.3 普通油茶与小果油茶叶绿体基因分子鉴定标记开发
利用上述缺失的片段和atpH、atpI 基因的保守性,提取普通油茶atpH、atpI 基因及其间隔区序列,设计跨过这段间隔区序列的最优引物,开发成分子标记。设计得到最优引物对,F:CTCCCTCTCCTAACCAACCC,R:CCGCGGCTTATATAGGTGAA,退火温度为57 ℃。经测序,普通油茶‘赣无1与小果油茶‘白皮中子PCR 产物的片段长度分别为609、227 bp。
IR 区对叶绿体基因组整体大小及各部件结构起决定性作用。在叶绿体基因组的IR/SSC 边界部分,假基因ycf1 分别从IRb 延伸到SSC 区,其中小果油茶为29 bp,普通油茶为1 bp。与普通油茶相比,小果油茶的IR 区扩张了72 bp,其IRa 扩张至假基因的5' 端(图4)。
2.4 普通油茶与小果油茶叶绿体基因分子鉴定标记验证
使用上述引物,所有普通油茶与小果油茶样本的叶绿体基因组均能扩增出明亮的条带(图5),表明该分子标记的稳定性较好。所有普通油茶样本PCR 产物的相对分子质量稳定在609 bp 左右。所有小果油茶样本PCR 产物的电泳条带排成1 条直线,相对分子质量在227 bp 左右。小果油茶与普通油茶的PCR 产物长度具有非常显著的区别,分子标记的可靠性、便捷性较高。
3 结论与讨论
本研究中组装的普通油茶与小果油茶叶绿体基因组序列全长分别为156 977、156 539 bp,小果油茶atpH 基因与atpI 基因间的非编码区存在长度为452 bp 的大片段缺失。根据缺失片段开发出分子标记,使用该分子标记能够稳定、便捷、精确地鉴定普通油茶与小果油茶。
3.1 普通油茶与小果油茶叶绿体基因组特征
6 种山茶科植物叶绿体基因组长度为156 607 ~157 166 bp[30],本研究中组装的普通油茶与小果油茶叶绿体基因组序列全长分别为156 977、156 539 bp,且GC 含量与已测定的山茶科植物相近。叶绿体基因组的编码基因数量和rRNA 基因数量与海南油茶叶绿体基因组一致,仅tRNA 基因的数量有细微差异[19]。这表明普通油茶与小果油茶叶绿体基因组的特征具有高度的保守性。
普通油茶与小果油茶的叶绿体基因组在编码区序列长度及非编码区序列长度方面有一定的差异,普通油茶的序列更长。影响叶绿体全基因组大小的主要因素是IR 和SSC 边界区域的扩张和收缩[31]。Yang 等[32] 经研究发现,山茶科植物主要是ycf1 基因发生从IR 区向SSC 区扩张。本研究结果表明,小果油茶ycf1 基因向SSC 区延伸了29 bp,而普通油茶仅为1 bp。陈凯[33] 的研究结果表明,使用根据ycf1 基因差异设计的分子标记不能区分普通油茶与小果油茶,其主要原因可能是两者间扩增的差异较小。对组装的叶绿体基因组进一步比较,结果表明小果油茶与普通油茶叶绿体基因组差异较大的区域主要是在atpH 基因与atpI 基因间的非编码区,相较于普通油茶,小果油茶在此区域存在大片段的缺失,长度为452 bp。本研究中利用该区域的大片段缺失,设计了分子标记,用于鉴定普通油茶与小果油茶。叶绿体基因组的这段缺失在丹寨秃茶C. danzaiensis、西南红山茶C. pitardii、猴子木C. yunnanensis 等物種中也被报道[34-35],油茶组中其他近缘物种的叶绿体基因组也存在缺失情况[34]。因此,在使用本研究中所开发的分子标记时,应先确定所鉴定的物种为普通油茶和小果油茶中的一种。
3.2 普通油茶与小果油茶叶绿体分子标记鉴别的可靠性
本研究中选用了大量不同区域的普通油茶与小果油茶来验证分子标记的可靠性。根据前人研究结果选取了油茶适生区内的典型油茶种群,且着重采集了同域分布区的部分普通油茶与小果油茶种群[36]。普通油茶样点分布于11 个省份,小果油茶取自6 个区域,涵盖了大部分油茶的分布区域。此外,为了进一步保证分子标记鉴别的准确性,在每个样点选用3 株油茶个体作为重复,以排除偶然误差。以所开发的分子标记作为引物进行PCR 扩增,结果表明所有普通油茶叶绿体基因组均可被扩增出609 bp 的条带,小果油茶叶绿体基因组被扩增出227 bp 的条带,两者具有非常明显的差异。以往也有利用分子标记来区分不同油茶品种的报道,例如韩欣等[37] 运用SRAP 标记对赣南油茶良种进行鉴别,但存在条带不清晰且重复性较差的现象,于小玉等[38] 利用ISSR 分子标记技术对66 个油茶品种进行鉴别,也出现某些条带扩增效果不明显的现象,陈永忠等[39] 利用RAPD和李海波等[40] 利用SCAR 分子标记鉴别油茶优良无性系,但存在步骤多、操作繁琐、易受技术设备影响等问题。相较于已报道的其他分子标记,本研究中开发的分子标记能够清晰鉴别普通油茶与小果油茶,且操作简便,鉴定速度快。
参考文献:
[1] 段伟华, 袁德义, 谭晓风, 等. 湖南地区10 个油茶优良无性系花粉特性比较[J]. 湖北农业科学,2012,51(17):3762-3764,3774.
DUAN W H, YUAN D Y, TAN X F, et al. Study on pollencharacteristics of ten excellentCamellia oleifera clones inHunan[J]. Hubei Agricultural Sciences,2012,51(17):3762-3764,3774.
[2] 廖玉芳, 蒋元华, 彭嘉栋, 等. 气候指标对油茶鲜果含油率的影响[J]. 经济林研究,2019,37(4):1-9.
LIAO Y F, JIANG Y H, PENG J D, et al. Effects of climateindexes on oil content inCamellia oleifera fresh fruit[J]. NonwoodForest Research,2019,37(4):1-9.
[3] 吴方圆, 李泽, 刘繁灯, 等. 油茶主栽品种光合特性的比较[J].经济林研究,2018,36(2):7-13.
WU F Y, LI Z, LIU F D, et al. Comparison of photosyntheticcharacteristics ofCamellia oleifera cultivars[J]. Non-wood ForestResearch,2018,36(2):7-13.
[4] 王渊, 谭晓风, 谢鹏, 等. 油茶优良无性系的理化性质及脂肪酸的分析[J]. 中南林业科技大学学报,2011,31(6):70-74.
WANG Y, TAN X F, XIE P, et al. The physical and chemicalproperties of the seed oil and its fatty acid compositions ofCamellia oleifera superior clones species[J]. Journal of CentralSouth University of Forestry & Technology,2011,31(6):70-74.
[5] 陈素传, 季琳琳, 姚小华, 等. 油茶品种果实主要经济性状和营养成分的差异分析[J]. 经济林研究,2022,40(2):1-9.
CHEN S C, JI L L, YAO X H, et al. Variation analysis on themain economic characters and nutrients of fruit fromCamelliaoleifera varieties[J]. Non-wood Forest Research,2022,40(2):1-9.
[6] 谢一青. 小果油茶农家品种花粉形态及亲缘关系分析[J]. 南京林业大学学报( 自然科学版),2016,40(3):26-32.
XIE Y Q. Analysis of pollen morphological difference andrelationship ofCamellia meiocarpa native varieties[J]. Journalof Nanjing Forestry University (Natural Sciences Edition),2016,40(3):26-32.
[7] 黄勇, 谢一青, 李志真, 等. 小果油茶表型多样性分析[J]. 植物遗传资源学报,2014,15(2):270-278.
HUANG Y, XIE Y Q, LI Z Z, et al. Analysis ofCamellia meiocarpaphenotype diversity[J]. Journal of Plant Genetic Resources,2014,15(2):270-278.
[8] 闵天禄, 顾志建, 张文驹, 等. 世界山茶属的研究[M]. 昆明:云南科技出版社,2000.
MIN T L, GU Z J, ZHANG W J, et al. Monograph of the genusCamellia[M]. Kunming: Yunnan Science and TechnologyPress,2000.
[9] 张恒, 申春晖, 陈锐帆, 等. 基于3 种数学方法的粤北油茶果实性状综合评价[J]. 中南林业科技大学学报,2022,42(11):71-79,208.
ZHANG H, SHEN C H, CHEN R F, et al. Comprehensiveevaluation of the fruit traits ofCamellia oleifera in northernGuangdong based on three mathematical methods[J]. Journal ofCentral South University of Forestry & Technology,2022,42(11):71-79,208.
[10] 胡先骕. 中国山茶科小志Ⅰ [J]. 科学通报,1957(6):170.
HU X S. Chinese annals of Theaceae I[J]. Chinese ScienceBulletin,1957(6):170.
[11] 张宏达, 任善湘. 中国植物志: 第49 卷第3 分册[M]. 北京:科学出版社,1990.
ZHANG H D, REN S X. Flora Reipublicae Popularis Sinicae tomus:Vol.49, Pt.3[M]. Beijing: Science Press,1990.
[12] 黄勇. 小果油茶与普通油茶居群遗传结构及种间杂交渐渗[J].应用生态学报,2013,24(8):2345-2352.
HUANG Y. Population genetic structure and interspecificintrogressive hybridization betweenCamellia meiocarpa andC. oleifera[J]. Chinese Journal of Applied Ecology,2013,24(8):2345-2352.
[13] 陈永忠, 许彦明, 张震, 等. 油茶果实主要数量性状分析及育种指标体系筛选[J]. 中南林业科技大学学报,2021,41(3):1-9.
CHEN Y Z, XU Y M, ZHANG Z, et al. Analysis of fruit mainquantitative traits and selection of breeding index inCamelliaoleifera[J]. Journal of Central South University of Forestry &Technology,2021,41(3):1-9.
[14] 秦声远, 戎俊, 张文驹, 等. 油茶栽培历史与长江流域油茶遗传资源[J]. 生物多样性,2018,26(4):384-395.
QIN S Y, RONG J, ZHANG W J, et al. Cultivation history ofCamellia oleifera and genetic resources in the Yangtze RiverBasin[J]. Biodiversity Science,2018,26(4):384-395.
[15] LI X W, YANG Y, HENRY R J, et al. Plant DNA barcoding: fromgene to genome[J]. Biological Reviews,2015,90(1):157-166.
[16] 張立, 程永琴, 姜在民, 等. 中国山茶科植物区系及叶绿体基因组结构进化分析[J]. 西北林学院学报,2020,35(5):47-53.
ZHANG L, CHENG Y Q, JIANG Z M, et al. Structure andphylogeny of chloroplast genomes and spermatophyte flora inChinese Theaceae[J]. Journal of Northwest Forestry University,2020,35(5):47-53.
[17] 殷鑫, 温强, 王建文, 等. 山茶属叶绿体全基因组微卫星特征分析及标记开发[J]. 分子植物育种,2018,16(20):6761-6769.
YIN X, WEN Q, WANG J W, et al. Characterization ofmicrosatellites in complete chloroplast genome of the genusCamellia and marker development[J]. Molecular Plant Breeding,2018,16(20):6761-6769.
[18] WU L L, LI J A, GU Y Y, et al. Complete chloroplast genomesequences and phylogenetic analysis of threeCamellia oleiferacultivars[J]. International Journal of Agriculture and Biology,2020,24(4):743-750.
[19] ZHANG W, ZHAO Y L, YANG G Y, et al. Characterization ofthe complete chloroplast genome sequence ofCamellia oleiferain Hainan, China[J]. Mitochondrial DNA Part B-Resources,2017,2:843-844.
[20] WANG G, LUO Y, HOU N, et al. The complete chloroplastgenomes of three rare and endangered camellias (Camellia huana,C. liberofilamenta andC. luteoflora) endemic to SouthwestChina[J]. Conservation Genetics Resources,2017,9:583-585.
[21] BANKEVICH A, NURK S, ANTIPOV D, et al. SPAdes:a new genome assembly algorithm and its applications tosingle-cell sequencing[J]. Journal of Computational Biology,2012,19(5):455-477.
[22] 李明启. 果实生理[M]. 北京: 科学出版社,1989.
LI M Q. Fruit physiological[M]. Beijing: Science Press,1989.
[23] WIXON J, KELL D. Website review: the kyoto encyclopedia ofgenes and genomes - KEGG[J]. Yeast,2000,17(1):48-55.
[24] KOONIN E V, FEDOROVA N D, JACKSON J D, et al. Acomprehensive evolutionary classification of proteins encoded incomplete eukaryotic genomes[J]. Genome Biology,2004,5(2):R7.
[25] BRIGITTE B, AMOS B, ROLF A, et al. The SWISS-PROTprotein knowledgebase and its supplement TrEMBL in 2003[J].Nucleic Acids Research,2003,31(1):365-370.
[26] CARBON S, DOUGLASS E, DUNN N, et al. The gene ontologyresource: 20 years and still GOing strong[J]. Nucleic AcidsResearch,2019,47:D330-D338.
[27] GREINER S, LEHWARK P, BOCK R. Organellar GenomeDRAW (OGDRAW) version 1.3.1: expanded toolkit for thegraphical visualization of organellar genomes[J]. Nucleic AcidsResearch,2019,47:W59-W64.
[28] UNTERGASSER A, CUTCUTACHE I, KORESSAAR T, et al.Primer3: new capabilities and interfaces[J/OL]. Nucleic AcidsResearch,2012,40(15):e115. Doi:10.1093/nar/gks596.
[29] 崔相艷, 王文娟, 杨小强, 等. 基于生态位模型预测野生油茶的潜在分布[J]. 生物多样性,2016,24(10):1117-1128.
CUI X Y, WANG W J, YANG X Q, et al. Potential distributionof wildCamellia oleifera based on ecological niche modeling[J].Biodiversity Science,2016,24(10):1117-1128.
[30] LI W, ZHANG C P, GUO X, et al. Complete chloroplast genomeofCamellia japonica genome structures, comparative andphylogenetic analysis[J/OL]. PLoS One,2019,14(5):e0216645.Doi:org/10.1371/journal.pone.0216645.
[31] DUGAS D V, HERNANDEZ D, KOENEN E J M, et al.Mimosoid legume plastome evolution: IR expansion, tandemrepeat expansions, and accelerated rate of evolution inclpP[J/OL].Scientific Reports,2015,5:16958. Doi:10.1038/srep16958.
[32] YANG J B, YANG S X, LI H T, et al. Comparative chloroplastgenomes ofCamellia species[J/OL]. PLoS One,2013,8(8):e73053. Doi:10.1371/journal.pone.0073053.
[33] 陳凯. 小果油茶与普通油茶系统发育关系及果实大小分化的转录组分析[D]. 南昌: 南昌大学,2020.
CHEN K.Camellia meiocarpa andCamellia oleiferaphylogenetic relationship and fruit size differentiationtranscriptome analysis[D]. Nanchang: Nanchang University,2020.
[34] ZHANG W, ZHAO Y L, YANG G Y, et al. Determinationof the evolutionary pressure onCamellia oleifera on HainanIsland using the complete chloroplast genome sequence[J/OL].PeerJ,2019,7:e7210. Doi:10.7717/peerj.7210.
[35] HUANG H, SHI C, LIU Y, et al. ThirteenCamellia chloroplastgenome sequences determined by high-throughput sequencing:genome structure and phylogenetic relationships[J]. BmcEvolutionary Biology,2014,14:151.
[36] CUI X Y, LI C H, QIN S Y, et al. High-throughput sequencingbasedmicrosatellite genotyping for polyploids to resolveallele dosage uncertainty and improve analyses of geneticdiversity, structure and differentiation: a case study of thehexaploidCamellia oleifera[J]. Molecular Ecology Resources,2022,22(1):199-211.
[37] 韩欣, 张党权, 王志平, 等. 基于SRAP 的赣南油茶良种分子鉴别研究[J]. 中南林业科技大学学报,2012,32(3):147-151.
HAN X, ZHANG D Q, WANG Z P, et al. SRAP-based molecularidentification of elite cultivars ofCamellia oleifera from southJiangxi province[J]. Journal of Central South University ofForestry & Technology,2012,32(3):147-151.
[38] 于小玉, 喻方圆, 刘建兵, 等. ISSR 在油茶品种鉴别和遗传多样性分析中的应用[J]. 南京林业大学学报( 自然科学版),2013,37(1):61-66.
YU X Y, YU F Y, LIU J B, et al. Identification and genetic diversityanalysis ofCamellia oleifera varieties using ISSR marker[J].Journal of Nanjing Forestry University (Natural SciencesEdition),2013,37(1):61-66.
[39] 陈永忠, 张智俊, 谭晓风. 油茶优良无性系的RAPD 分子鉴别[J]. 中南林学院学报,2005,25(4):40-45.
CHEN Y Z, ZHANG Z J, TAN X F. Identification of oil tea(Camellia oleifera) superior clones by RAPD molecularmarker[J]. Journal of Central South Forestry University,2005,25(4):40-45.
[40] 李海波, 刘勇, 赵佳, 等. 12 个油茶良种的SCAR 分子标记鉴别[J]. 浙江林业科技,2017,37(2):17-23.
LI H B, LIU Y, ZHAO J, et al. Identification of 12 superiorcultivars ofCamellia oleifera by sequence characterized amplifiedregion markers[J]. Journal of Zhejiang Forestry Science andTechnology,2017,37(2):17-23.
[ 本文编校:闻 丽]
