探索结直肠癌血清外泌体有效分子标志物
2024-01-30邹鲁飞
杨 钊,邹鲁飞
结直肠癌(colorectal cancer,CRC)是临床中最为常见的恶性肿瘤之一,其发病率和死亡率目前位居世界第3 位和第4 位[1]。目前针对CRC 主要筛查手段为粪便隐血检查及电子结肠镜,但粪便隐血检查由于受到取样等多种因素影响其准确度受限,而结肠镜检查常因耐受性等因素并未被大部分健康人群所接受。因此, 如何更为精准地筛查CRC 患者仍是临床所面临的难题。近年来飞速发展的液体活检技术为临床更为精准方便筛选肿瘤标志物带来了巨大帮助[2~4]。 因此,基于液体活检技术筛选CRC 有效的生物学标志物将可能极大的帮助CRC 患者的早期诊断。 笔者利用基于液体活检技术的血清外泌体检测寻找CRC 有效生物学标志物。
1 材料与方法
1.1 实验材料
选择外泌体数据库exoRBase 2.0[5](http://www.exorbase.org/) 和基因表达综合集(gene expression omnibus,GEO) 网站(https://www.ncbi.nlm.nih.gov/geo/)、肿瘤免疫评估资源(tumor immune estimation resource,TIMER)网站(https://cistrome.shinyapps.io/timer/)。
1.2 方法
1.2.1 外泌体数据集和验证数据集获取
从外泌体数据库exoRBase 2.0[5](http://www.exorbase.org/)中下载健康人和CRC 患者的血清外泌体测序数据。 从GEO(https://www.ncbi.nlm.nih.gov/geo/)网站中获取GSE192667 和GSE24549 数据集中基因表达谱表达数据及预后信息作为核心基因的外部验证数据集,并据此构建生存风险模型。
1.2.2 基于血清外泌体数据库筛选结直肠癌患者高表达基因
利用外泌体数据库中所包含的测序数据集,筛选共获得19616 个基因, 其中有2520 个基因在CRC组无表达,1534 个基因在健康人群中无表达,1293基因在健康人群和CRC 患者中均无表达。 并利用R软件筛选两组之间的差异基因及其所涉及的基因本体(gene ontology,GO)功能和京都基因与基因组百科全书 (Kyoto Encyclopedia of Genes and Genomes,KEGG)分析。
1.2.3 基于加权基因共表达网络筛选核心分子
加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)是一种近年来被广泛使用的生物信息学筛选方法,其通过无序算法筛选有效的分子标志物, 其可以应用于肿瘤及其他疾病中[6]。首先利用R 软件中的“WGCNA”包构建WGCNA网络模型,基于基因与基因之间关联程度确定软阈值大小[7,8],并将基因表达矩阵转化为拓扑重叠矩阵(topological overlap,TOM)[9,10], 并对基因进行归类分析, 将基于基因关联关系划分基因模块属性(module eigengene,ME),计算模块中每个基因对每个样本贡献度,并累加获得每个模块对样本的贡献度。 基于贡献度绝对值最大模块为核心模块。同时,基于蛋白-蛋白互作用(protein-protein interact,PPI)分析核心模块内所有基因的互作关系,并利用Cytoscape 软件中3 种算法Degree Score、MCODE Score 和Closeness Score 计算各基因的评分,筛选核心模块中核心分子。 并基于核心模块中差异基因确定核心分子。
1.2.4 基于外部网站验证核心分子及其可能涉及机制
为验证所筛选的核心分子是否能较好地区分CRC 患者预后,利用基因表达谱交互分析(Gene ExpressionProfilingInteractive Analysis,GEPIA)网站(http://gepia.cancer-pku.cn/)分析癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中核心基因在各个肿瘤及其正常组织中的表达情况。利用R2 平台(https://hgserver1.amc.nl/cgi-bin/r2/main.cgi/)验证核心基因预后。 利用CancerSEA 网站[11](http://biocc.hrbmu.edu.cn/CancerSEA/)中所包含的CRC 单细胞数据集分析核心基因可能所涉及的功能。利用TIMER网站(https://cistrome.shinyapps.io/timer/)分析核心基因与免疫细胞关联性,进一步分析核心分子可能所涉及通路。
1.2.5 GSE192667 和GSE24549 数据集验证核心分子并构建生存风险模型
利用GEO 数据库中的GSE192667 和GSE24549数据集,验证核心分子在外部数据集中能否有效预测CRC 患者预后。 利用COX 多因素分析计算核心分子在GSE192667 数据集患者中风险评分, 并据此计算每个样本的风险评分;基于风险评分将患者平均分为高风险组和低风险组。 同时利用该评分系统应用于GSE24549 数据集中,再进行后续分析。
1.3 统计学方法
采用GraphPad Prism 7.0、SPSS 24.0 和R(4.0.1)软件对数据进行统计分析和作图。 t 检验用来分析两组间平均数的差异。X-tile 软件基于生存分析结果对核心基因选取最佳截点,Kaplan-Meier(K-M)曲线分析患者预后。 P <0.05 为差异有统计学意义。
2 结果
2.1 健康人群和结直肠癌患者差异基因及GO 功能和KEGG 分析
外泌体数据库中共含有118 例健康人血清外泌体测序数据及35 例CRC 患者。 其中CRC 患者较健康人群上调基因有5078 个, 下调基因有13004 个,以P <0.05 及差异倍数大于1.5 筛选共获得了上调基因有600 个,下调基因有1175 个(图1A、B)。 对所有差异基因进行GO 功能和KEGG 通路分析, 发现CRC 患者其差异基因主要富集于细胞周期、RNA 可变剪接及RNA 修饰过程等(图1 C、D)。
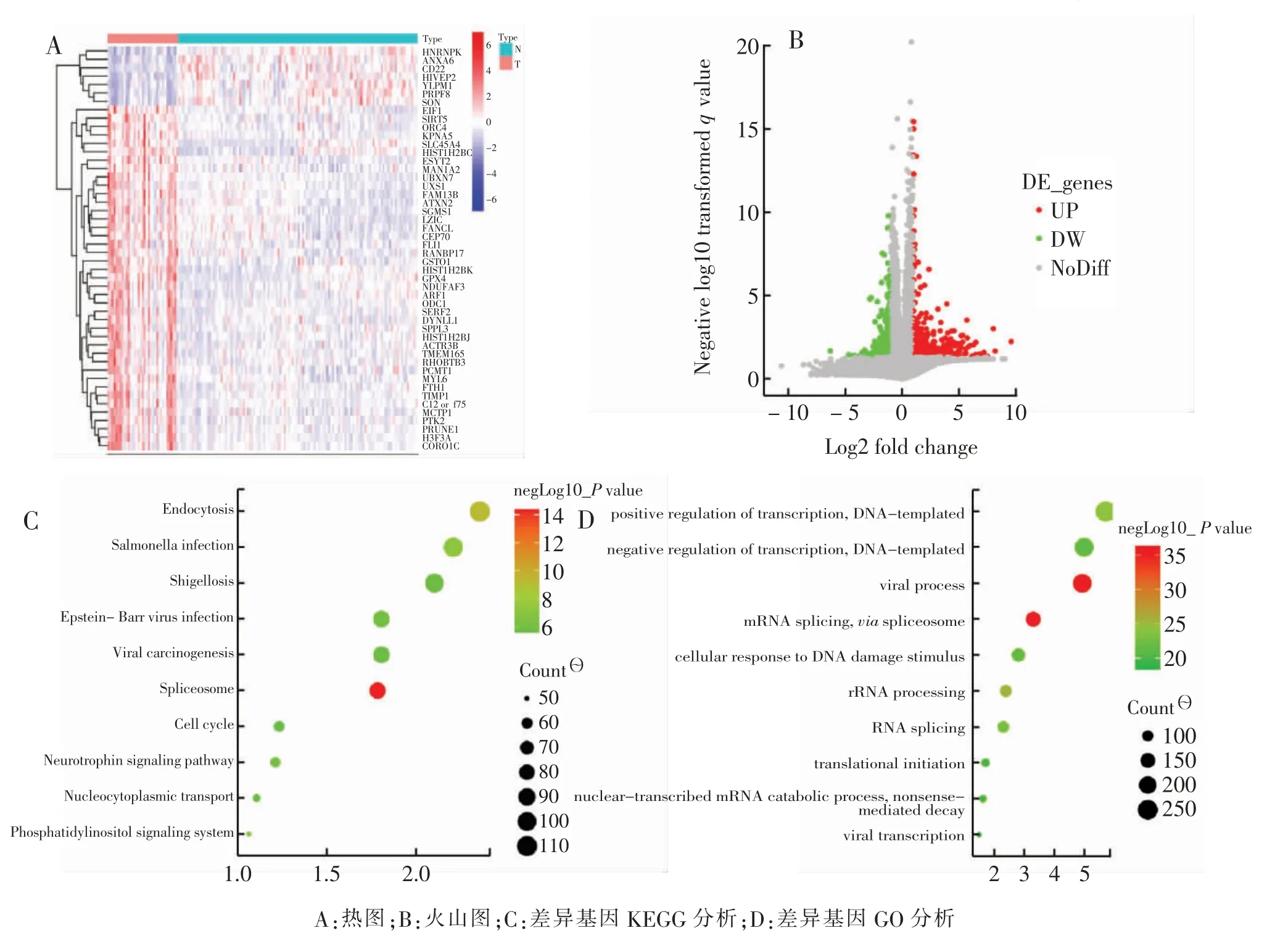
图1 CRC 患者和健康人差异基因分析Fig.1 Differential gene analysis diagrams of CRC patients and healthy
2.2 基于WGCNA 筛选血清外泌体中核心模块及核心基因并初步验证
2.2.1 核心模块与核心基因
基于WGCNA 获得其软阈值为3(图2A、B),共获得了7 个模块及其相应模块贡献度(图3A),并据此认为RED(红色)模块为最相关模块。红色模块中共包含70 个基因,基于GO 和KEGG 分析了其可能涉及的功能和通路(图3B),红色模块基因可能细胞凋亡阶段负调控、受体活性负调控及核转录mRNA 的调控。

图2 软阈值筛选图(A)和WGCNA 模式图(B)Fig.2 Diagrams of soft threshold screening(A)and WGCNA pattern(B)
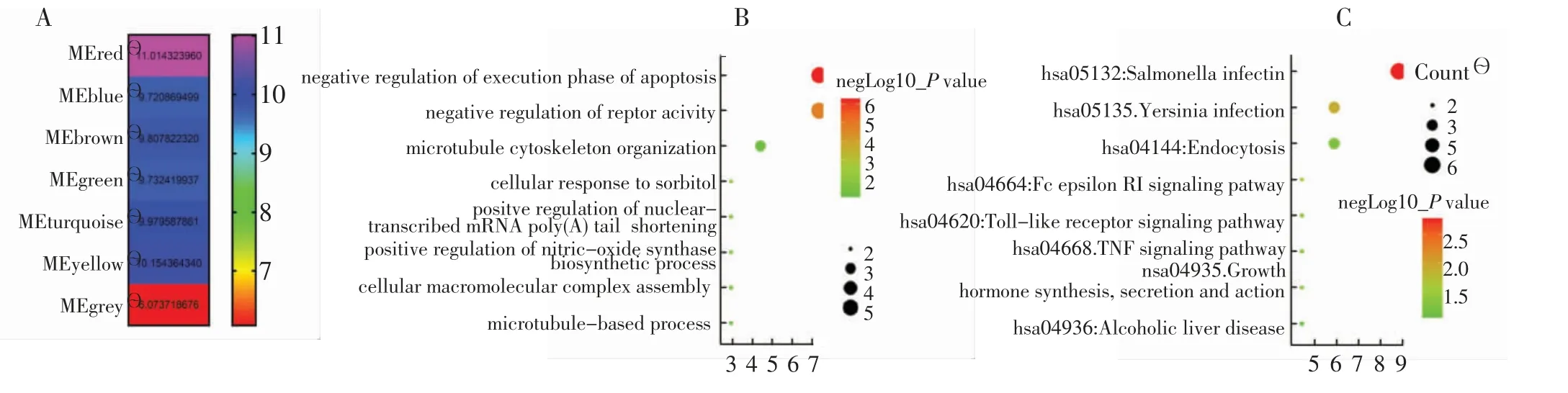
图3 基因模块热图(A)、红色模块GO 分析(B)和红色模块KEGG 分析(C)Fig.3 Diagrams of gene module thermography(A),red module GO analysis(B)and red module KEGG analysis(C)
红色模块中共有70 个基因(图4A、B),从中筛选其差异基因,按照P <0.05,CRC 患者比健康人群上调有6 个、下调有10 个。为寻找红色模块中核心基因,基于PPI 分析及Cytoscape 软件中自带算法(图4C),计算各基因之间DEGREE Score(≥4),同时计算MOCDE Score(≥1)和Closeness Score(≥10),最终共有15个基因位于核心位置(图4D)。 并继续与上调差异基因合并分析,发现甘油醛3-磷酸脱氢酶基因(glyceraldehyde 3-phosphate dehydrogenase gene,GAPDH)和微管蛋白α-1B 基因(tubulin alpha 1b gene,TUBA1B)两个基因可能可以预测CRC 发生。因此,拟进一步探讨并验证上述基因在预测CRC 发生的有效性。
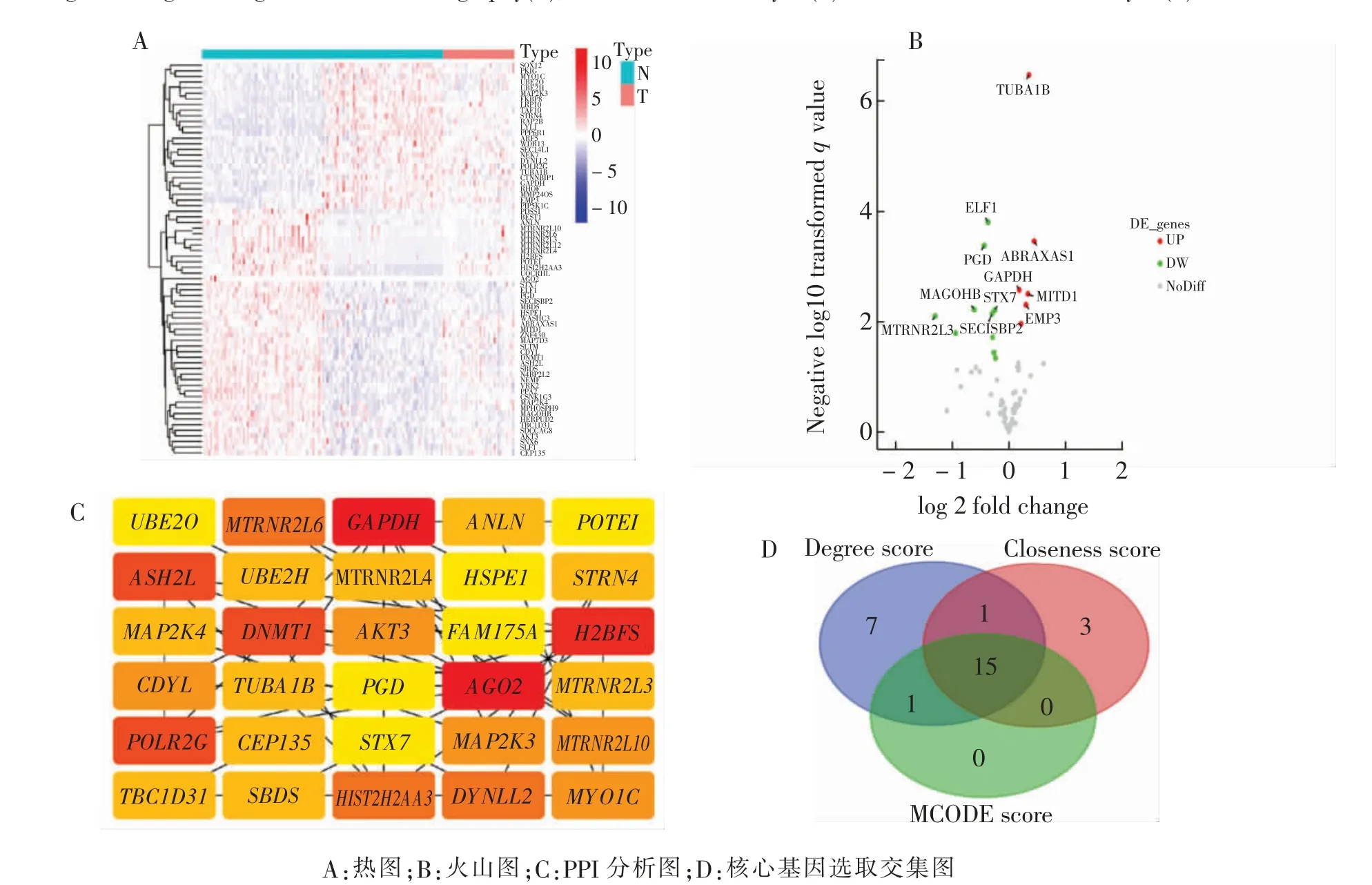
图4 红色模块中健康人群和CRC 患者差异基因分析Fig.4 Diagrams of differential gene analysis in red module between CRC patients and healthy
2.2.2 初步验证
为明确所筛选的基因是否在CRC 患者及健康人群中存在差异, 进一步分析了上述2 个基因在CRC患者与健康人群间差异(图5),其中GAPDH 在健康人群中的表达值为636.60±13.68,在CRC 患者中表达值为815.50±40.88 (P <0.01);TUBA1B 在健康人群中的表达值为1546.00±33.22, 在CRC 患者中表达值为1758.00±60.16(P <0.01)。同时,受试者工作特性(receiver operating characteristic,ROC)曲线被用于分析上述2 个基因能否预测CRC 的发生 (图5),GAPDH 和TUBA1B 能较好地区分CRC 患者和健康人群[GAPDH:曲线下面积(area under curve,AUC) =0.69,P <0.01;TUBA1B:AUC = 0.73,P <0.01]。 上述结果证实,利用WGCNA 结合PPI 所筛选的基因能较好地区分CRC 和健康人群。

图5 核心基因在CRC 和健康人群中验证Fig.5 Diagrams of hub gene verified in CRC patients and healthy
2.3 外部数据库验证核心基因表达及预后
为验证所筛选的核心基因是否在CRC 组织和癌旁组织中存在差异表达,利用TCGA 数据库中所含有的多种肿瘤数据集验证核心基因在不同肿瘤组织及其正常组织中表达差异 (图6),GAPDH 和TUBA1B在CRC 和直肠癌中均较癌旁组织表达高(P <0.05)。同时在其他肿瘤中,也发现了GAPDH 和TUBA1B 在多个肿瘤中呈现高表达,该结果进一步佐证了试验所筛选的基因具有较大的预测意义。为进一步明确核心基因GAPDH 和TUBA1B 是否与CRC 预后相关,对R2 分析平台中CRC 患者数据分析(图7C ~H),高表达GAPDH 和TUBA1B 患者其预后较低表达患者更差。上述结果进一步证实了实验所筛选的基因具有较好的预测意义。

图6 GAPDH(A)和TUBA1B(B)在TCGA 数据库中不同肿瘤组织中癌组织和癌旁组织中表达差异Fig.6 Diagrams of differences expression between GAPDH(A) and TUBA1B(B) for different tumor tissues and adjacent tissues in TCGA database
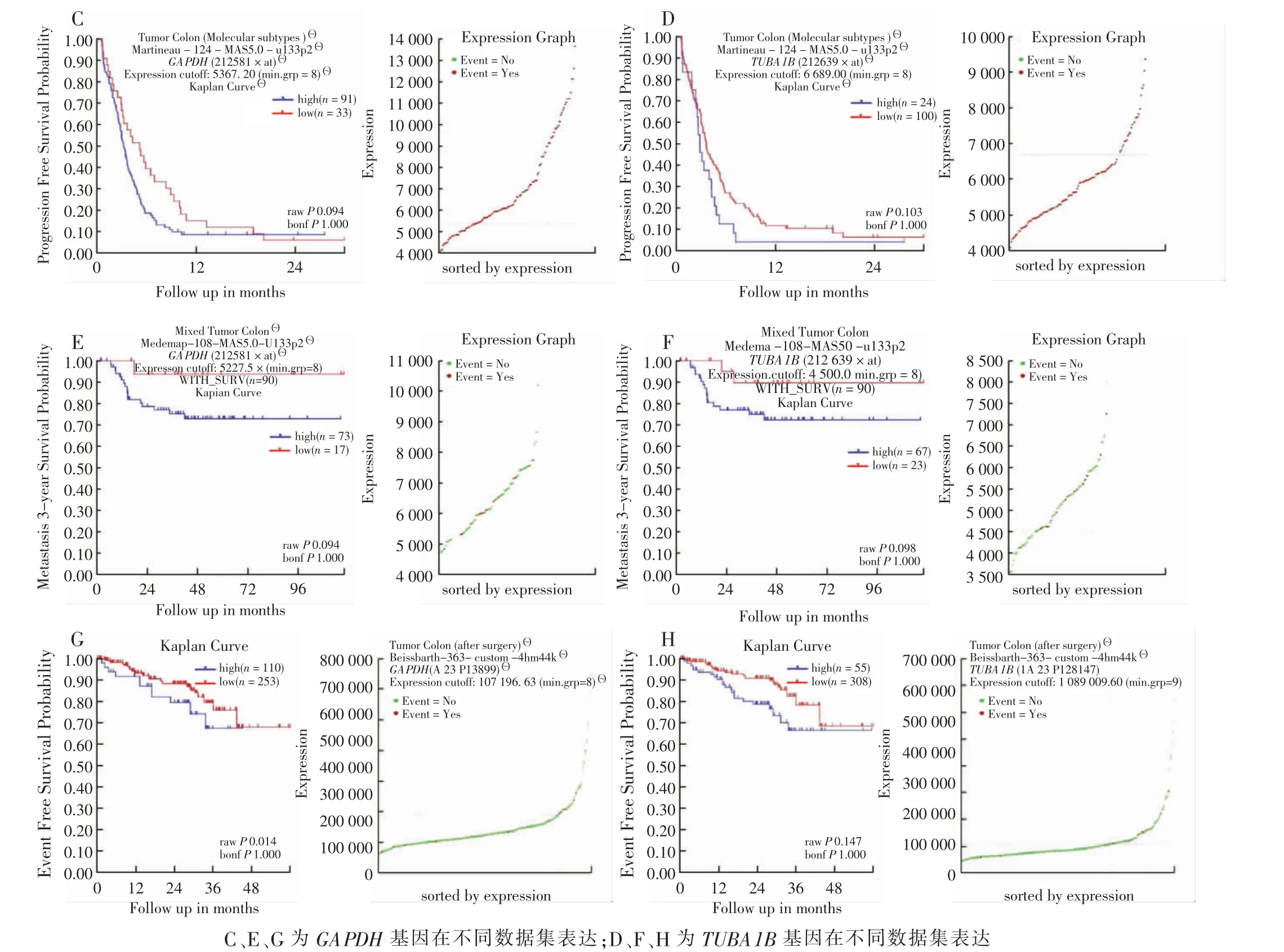
图7 外部数据库验证核心基因GAPDH 和TUBA1B 在R2 平台与CRC 患者预后分析Fig.7 Diagrams of validation hub genes GAPDH and TUBA1B in R2 platform and prognosis analysis of CRC patients in external database
2.4 GSE192667 和GSE24549 数据集验证核心基因并构建生存风险模型
2.4.1 K-M 生存分析验证
利用GEO 数据库中GSE192667 和GSE24549 数据集所含有的CRC 患者基因表达谱数据及预后信息,对所获得的基因GAPDH 和TUBA1B 验证其与预后关联。 其中GSE192667 数据集中含有89 例CRC患者数据,GSE24549 数据集中含有83 例CRC 患者数据。 依据X-tile 软件选取基因最佳截点,并进行K-M生存分析。 结果显示,在GSE24549 中,GAPDH 低表达患者较高表达患者无病生存期更长(P <0.01),TUBA1B 低表达患者较高表达患者无病生存期更长,但差异无统计学意义(P = 0.17);在GSE192667 中,GAPDH 和TUBA1B 低表达患者较高表达患者无病生存期更长(P <0.01)。 见图8。

图8 核心基因在GSE24549 数据集和GSE192667 数据集中患者K-M 生存分析Fig.8 Patient K-M survival analysis curves of hub genes in GSE24549 dataset and GSE192667 dataset
2.4.2 生存风险分析
为更好地对CRC 患者进行分层管理,对GSE192667 患者进行多因素COX 分析并据此结果构建生存风险模型(图9),生存风险模型得分=GAPDH表达量×0.0015+TUBA1B 表达量×(-0.0002)。 据此获得每例患者的评分,并依据上述评分将患者分为高风险组和低风险组。 据此对GSE192667 患者进行预后分析(图10A),高风险组预后更差(P <0.01)。 同时,进一步验证该模型,在GSE24549 数据集中,高风险组预后较低风险组差(图10B。 P <0.01)。
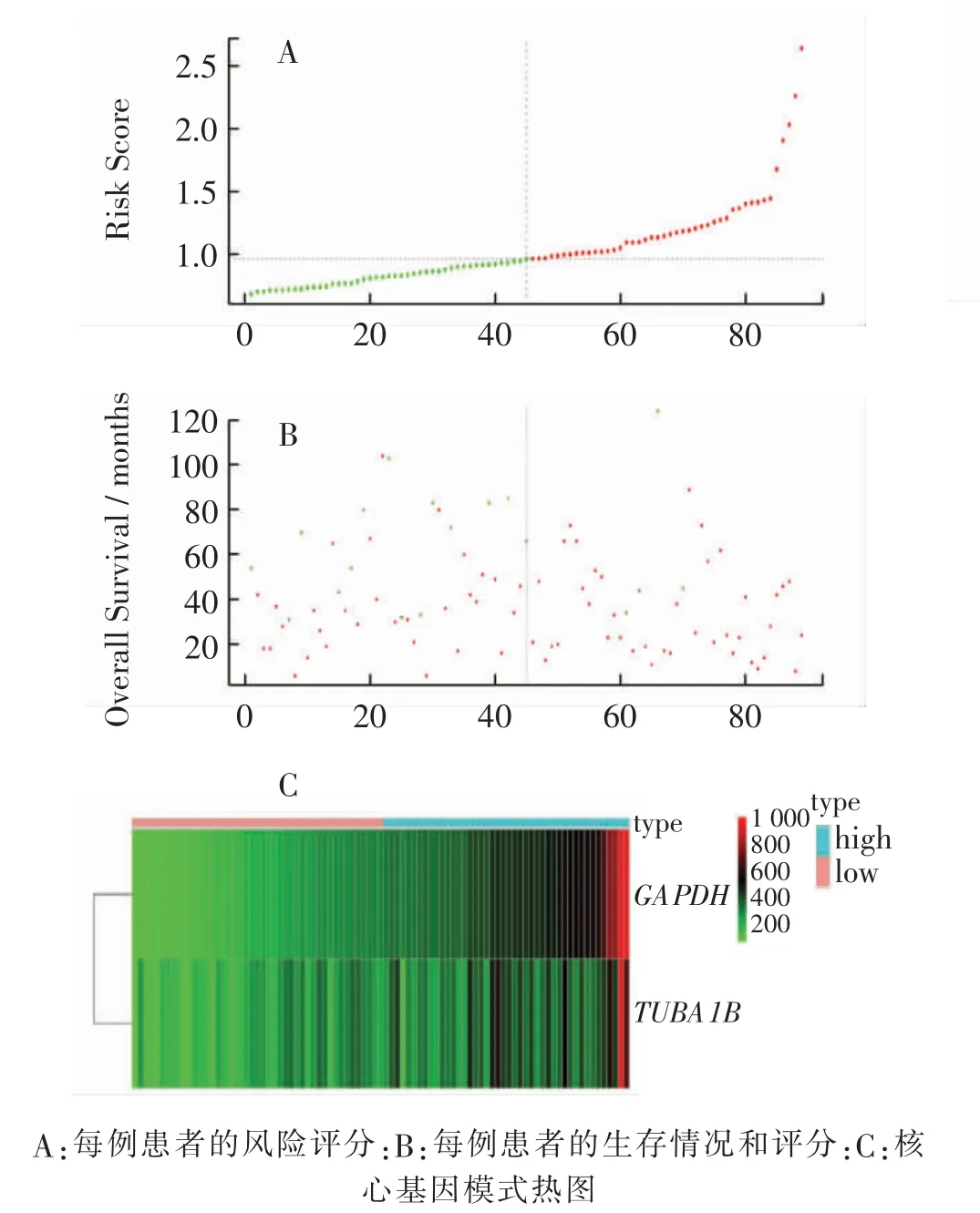
图9 基于核心基因构建生存风险模型Fig. 9 Diagram of construction risk score model based on hub genes

图10 生存风险模型在GSE192667 数据集和GSE24549 验证Fig. 10 Curves of survival risk model verified on GSE192667 dataset and GSE24549
2.5 外部数据集及GSEA 探索核心基因可能涉及通路和功能
为探索核心基因GAPDH 和TUBA1B 可能所涉及的功能和通路, 用CancerSEA 网站中所包含的CRC 单细胞数据集分析核心基因与其相关细胞功能(图11)。 TUBA1B 可能涉及了细胞周期、细胞增殖、DNA 修复及细胞干性;GAPDH 可能涉及了侵袭、DNA 修复及血管生成。 同时,利用基因集富集分析法(gene set enrichment analysis,GSEA)分析外泌体数据集中核心基因可能所涉及的通路。 其中TUBA1B 可能涉及药物代谢酶P450、 核苷酸结合寡聚化结构域(nucleotide binding oligomerization domain,NOD)样受体通路和膀胱癌通路(图12)。GAPDH 可能涉及血管内皮生长因子 (vascular endothelial growth factor,VEGF) 信号通路、CRC 信号通路和丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)信号通路(图13)。 同时利用TIMER 网站分析核心基因与免疫细胞关联性(图14),GAPDH 可能涉及了B 细胞、巨噬细胞(P <0.05)。 而TUBA1B 在结肠癌和直肠癌中并未获得一致结果(P >0.05)。
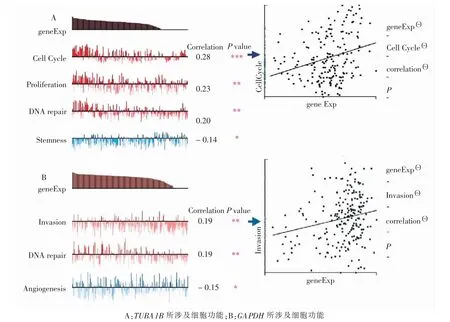
图11 CancerSEA 网站探索核心基因可能相关细胞功能Fig.11 Diagrams of exploring hub genes possibly related cellular function in CancerSEA website
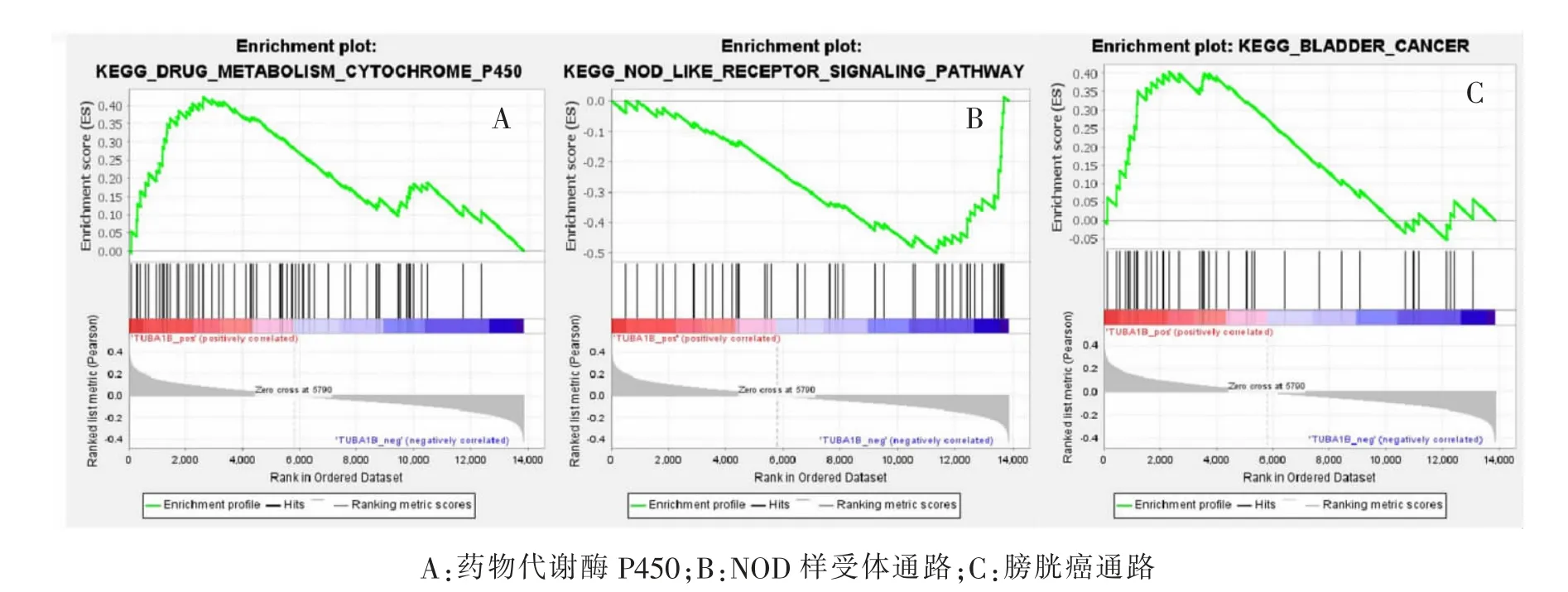
图12 GSEA 分析TUBA1B 所涉及通路Fig.12 Diagrams of TUBA1B involved pathways by GSEA

图13 GSEA 分析GAPDH 所涉及通路Fig.13 Diagrams of GAPDH involved pathways by GSEA
3 讨论
近年来CRC 的发病率和死亡率在中国逐年提升[12],由于CRC 患者前期无明显典型症状,大部分患者发现时即为中晚期,导致了其预后较差[1,13]。目前国内外指南推荐针对CRC 患者的筛查主要为粪便隐血检查及电子结肠镜。 但由于准确度及可接受度导致CRC 筛查难以推广应用。 因此寻找更精准及无创的筛查方法将对CRC 早诊做出巨大的贡献。近年来,飞速发展的液体活检技术为临床对肿瘤患者的筛查提供了巨大的可能。其主要是基于肿瘤形成后分泌一部分特殊物质,包括但不限于外泌体、细胞因子等。基于检测上述特殊物质,将可能使得针对普通人群可以进行快速而有效的筛查。 基于此,笔者利用血清外泌体数据库中健康人群和CRC 患者的血清外泌体测序数据,结合WGCNA 和PPI 筛选基于血清外泌体的核心诊断标志物GAPDH 和TUBA1B。 同时利用了多个外部公共数据库验证了核心基因GAPDH 和TUBA1B在肿瘤组织中较正常组织高表达, 以及高表达GAPDH 和TUBA1B 基因与更差的预后相关。 同时,并分析了核心基因其可能所涉及的细胞功能和通路,为后续进一步研究打下了坚实的基础。 综上所述,所筛选获得的GAPDH 和TUBA1B 基因可以有效地预测CRC 发生及预后,可以作为CRC 有效的血清外泌体分子标志物。
目前针对CRC 患者常用的肿瘤标志物是癌胚抗原(carcinoembryonic antigen,CEA),但不可否认的是,其由于受到多种因素的影响,准确度仍受限[14,15]。因此寻找更为精准的血清学标志物将为CRC 的诊断和随访提供较大的帮助。外泌体是一类直径在30 ~100 nm的小囊泡,其通过胞膜内陷形成囊泡,并将其释放至细胞基质中,达到传递信息的作用[16,17]。外泌体其内包含mRNA、非编码RNA、脂质及蛋白质等多种成分,这些成分在外泌体磷脂双分子层包裹下有更强的稳定性[18,19]。 目前已有研究提示肿瘤细胞其可以通过释放外泌体入血达到调控全身反应作用[20,21],同时,也有研究提示血清外泌体可以作为有效的分子标志物预测多种肿瘤的发生和预后等[18,22,23]。 因此基于液体活检技术检测血清外泌体中某些特殊标志物将对筛查CRC 患者有重要的临床意义。 笔者研究基于该思想,利用血清外泌体数据库分析CRC 患者及健康人群的血清外泌体测序数据,发现GAPDH 和TUBA1B 可以作为有效的分子标志物。 同时,利用外部公共数据库验证了GAPDH 和TUBA1B 基因在TCGA 数据库中不同肿瘤和癌旁组织中以高表达为主, 其中在结肠癌和直肠癌中均获得了一致的结果, 同时在R2 生存平台中也获得了相应的验证, 高表达GAPDH 和TUBA1B 基因患者预后更差。
甘油醛3-磷酸脱氢酶 (glyceraldehyde 3-phosphate dehydrogenase,GAPDH)是一种在糖酵解过程中关键的调控酶,参与了糖酵解过程中多个调控过程[24]。GAPDH 作为应用非常广泛的管家基因, 常被用于基因表达水平分析的内部控制。 现有较多研究提示,GAPDH 参与了体内的一系列生物调控过程, 包括膜融合和运输、细胞凋亡、DNA 修复、转录调控、自噬等[18,25~29]。 现也有研究提示,GAPDH 表达失调可能与肿瘤的发生和发展有关,如肾癌[30]、乳腺癌[31]和鼻咽癌[32]。笔者研究也发现了类似的结果,GAPDH 在CRC患者血清外泌体中较健康人群高表达。同时在多个外部数据库中发现了GAPDH 在CRC 患者中高表达,且高表达患者预后差。并基于单细胞数据库及免疫细胞分析发现GAPDH 基因表达失调在CRC 患者中可能通过影响VEGF 信号通路、CRC 信号通路和MAPK信号通路导致了细胞周期、细胞增殖、DNA 修复等细胞功能发生异常,从而导致了肿瘤的发生和进展。
微管蛋白α-1B(tubulin alpha 1b,TUBA1B)是属于微管蛋白的亚型之一,微管蛋白是细胞骨架的主要成分,有5 种不同形式,其是调节细胞形状、细胞黏附、细胞运动、复制和分裂、驱动细胞内有丝分裂和运输的细胞骨架元件[33,34]。 目前已有部分研究显示,TUBA1B 作为癌基因参与调节了多种肿瘤的发生、发展和预后[35~38]。笔者研究结果也提示TUBA1B 在CRC中作为癌基因, 在CRC 患者血清外泌体及肿瘤组织中高表达, 同时高表达TUBA1B 与更差的CRC 患者预后相关。 基于单细胞数据库及免疫细胞分析发现TUBA1B 基因表达失调在CRC 患者中可能通过影响药物代谢酶P450、NOD 样受体通路和膀胱癌通路导致了侵袭,DNA 修复及血管生成细胞功能发生异常,从而导致了肿瘤的发生和进展。 为更好地对CRC 患者进行分层管理, 笔者基于GSE192667 数据集利用了生存风险模型,并在内部和外部均获得了较好的验证。 综上所述,从内部、外部,利用多种方法验证了笔者所筛选的基因作为有效的分子标志物的可靠性,其可能为CRC 患者的早期筛查及预后随访提供较大的帮助。
据笔者所知, 目前基于血清外泌体筛选CRC 患者有效的分子标志物文献报道较少。笔者利用血清外泌体数据库分析了CRC 患者较健康人群在血清外泌体中主要涉及了细胞周期、RNA 可变剪接及RNA 修饰过程等。 基于WGCNA 和PPI 筛选获得了GAPDH和TUBA1B,并利用内部血清外泌体数据库及外部多个公共数据库从多角度验证了上述基因在CRC 患者中的有效性。但笔者研究作为一项临床前的探索性研究,其应用价值尚需进一步在多中心大样本的临床随机试验中获得验证。
