纳米银胁迫下土壤微生物磷脂脂肪酸的响应特征
2024-01-04宋宇迪樊昊心姚槐应
宋宇迪,樊昊心,姚槐应
武汉工程大学环境生态与生物工程学院,湖北 武汉 430205
纳米银(silver nanoparticles,AgNPs)是指在三维空间中至少有一维的尺寸范围在纳米级别(1~100 nm)的金属银单质[1]。AgNPs 是应用最广泛的工程纳米材料之一。据估计,AgNPs 相关产品的全球产量约为每年450 t[2],其市场规模预计将从2014 年的7.9 亿美元增长到2022 年的25.4 亿美元[3]。AgNPs 与传统银单质相比,具有更为高效的抗菌性能[4]和独特的物理化学性质(催化活性、有效导电性、特定光学性质等),因而广泛应用于医疗[5]、环保[6]、食品[7]、工业生产[8]等领域。此外,AgNPs 在农业生产方面有着广阔的应用前景,可以用于促进植物生长和管理植物病害[9]等。AgNPs 的生产和消费规模日益增加,继而通过各种途径在自然环境中释放和积累[10],因此产生潜在的生态风险。根据建模结果,纳米材料在土壤中的排放量高于在水体和大气中的排放量[11-12]。将含有AgNPs 的污泥等生物固体用作农业肥料、使用污水灌溉农田、不当处理医疗和消费品废弃物[13-14]等,会造成AgNPs 在土壤中,尤其是农田土壤的积累。
微生物是土壤中最为丰富多样的生物种群,在维持生态平衡、驱动生物地球化学循环等方面起着重要作用[15]。微生物群落的变化可以反映土壤污染物的生态胁迫效应[16-17]。环境中的AgNPs会不同程度地改变微生物活性和群落结构。Peixoto 等[18]研究了AgNPs 长期暴露对土壤细菌群落的影响,发现暴露56 d 后细菌群落结构与对照组相比仅有71.9%的相似性。Montes 等[19]发现AgNPs 在预测环境质量分数下使土壤微生物生物量有所下降。Samarajeewa 等[20]通过一系列微生物测试评估了沙壤土中聚乙烯吡咯烷酮包被的AgNPs(3~706 mg·kg-1)对微生物生长、活性和多样性的影响,结果表明AgNPs 对土壤微生物群落活性有显著的抑制作用,而且毒性随着时间的推移变得更加明显。
磷脂是含有磷酸基团的脂质,是生物细胞膜的主要成分。不同微生物具有不同种类的磷脂脂肪酸(phospholipid fatty acid,PLFA),其含量和结构具有种属特征或与其分类密切相关,能够标志某种或某类特定微生物的存在[21]。不同微生物群落具有独特的PLFA 特征谱图,因此PLFA 可以用于对微生物群落进行识别和定量描述,PLFA 的变化反映了环境样品中微生物群落结构的变化[22]。此外,由于磷脂不能作为细胞的贮存物质,微生物细胞死亡后,其中的磷脂化合物会迅速降解,因而PLFA 可以代表微生物群落中的活性微生物[23]。使用PLFA 鉴定微生物种类并定量分析微生物群落生物量和群落结构,克服了传统微生物培养及计数方法的缺点,具有较高的准确性、稳定性和敏感性[24],被广泛应用于土壤微生物群落研究[25]。
目前,关于不同水平AgNPs 胁迫下土壤微生物PLFA 变化的研究较少。因此,本研究以农田土壤为研究体系,对不同水平AgNPs 胁迫下土壤微生物PLFA 含量、丰度及群落结构的变化趋势进行分析,探究土壤微生物PLFA 对AgNPs 胁迫的响应特征。
1 实验部分
1.1 试剂与材料
1.1.1 主要试剂 纳米银粒子(上海麦克林生化科技股份有限公司,纯度为99.5%,粒径为60~120 nm)。使用的化学试剂主要包括:乙酸(分析纯),氢氧化钾(分析纯),一水合柠檬酸(分析纯),二水合柠檬酸三钠(分析纯),氯仿(色谱纯),丙酮(色谱纯),正己烷(色谱纯),甲醇(色谱纯),甲苯(色谱纯);以上试剂均购自国药集团化学试剂有限公司。
1.1.2 供试土壤 土壤采集自甘肃定西的旱地农田。采用对角线布点法采集3 个子样,每个子样点从上至下铲取0~15 cm 表层土壤,充分混匀后采用四分法保留5 kg 土壤。使用孔径为2 mm 的土壤筛清除土壤中的植物残体和砾石,储存在4 ℃下。实验开始前,所有土壤样品在25 ℃下避光预培养7 d,使土壤微生物从干扰中恢复活性。
土壤样品具有以下特性:土壤总有机碳含量为4.70 g·kg-1,总氮含量为0.40 g·kg-1;土壤铵态氮含量为1.19 mg·kg-1,硝态氮含量为65.05 mg·kg-1;土壤pH 值(土水质量比为1.0∶2.5)为8.34;土壤质地为粉壤土,其中砂粒(0.02~2.00 mm)质量分数为28%,粉粒(0.002~0.020 mm)质量分数为68%,粘粒(<0.002 mm)质量分数为4%。
1.2 方 法
1.2.1 实验设计 本研究设计在土壤中添加10、100、1 000 mg·kg-1AgNPs(Ag10 组,Ag100 组,Ag1 000 组),并以未添加AgNPs 的土壤作为对照组(CK 组),共计4 种处理,每种处理设置3 个平行样品。AgNPs 以粉末形式添加到土壤中;首先向空白土壤加入质量分数10 g·kg-1AgNPs,使用搅拌装置持续混合10 min;然后采用10 倍稀释法[26],对含有10 g·kg-1AgNPs 的土壤进行“稀释”,添加空白土壤使其依次达到目标质量分数,每次“稀释”后使用搅拌装置充分混合。建立微宇宙土壤培养体系,将10 g 含有目标质量分数AgNPs 的土壤放入培养瓶,置于生化培养箱(LRH-250,上海一恒科学仪器有限公司)中25 ℃下避光培养56 d。每隔2 d 向土壤中补充无菌去离子水,采用差量法使土壤水分维持在土壤持水量的60%。
1.2.2 微生物PLFA 的提取和检测 将土壤样品在-80 ℃下冷冻12 h,使用冷冻干燥机(FreeZone,美国Labconco 公司)冻干48 h。采用改良的Bligh-Dyer 法提取土壤微生物PLFA[27],包括提取脂类、分离磷脂和甲醇酯化3 个步骤:(1)称取1.00 g 冻干土壤样品,加入7.6 mL 甲醇-氯仿-柠檬酸缓冲液(体积比2.0∶1.0∶0.8)进行萃取,50 r·min-1水平振荡2 h,1 500 r·min-1离心10 min;重复萃取1次。将上清液与6 mL 氯仿和4.8 mL 柠檬酸缓冲液混合,静置4 h,转移下层有机相并使用氮吹仪(DC-24,上海安谱实验科技股份有限公司)吹干。(2)加入5 mL 氯仿溶解氮吹后的样品,并转移到固相萃取柱中,依次加入8 mL 氯仿、10 mL 丙酮和8 mL甲醇洗脱中性脂、糖脂和磷脂,收集磷脂。再加入200 μL 99.36 μmol·L-1十九烷酸甲酯作为内标物质,使用氮吹仪吹干。(3)加入1 mL 甲醇甲苯混合溶液溶解氮吹后的样品,并加入1.0 mL 0.2 mol·L-1氢氧化钾甲醇溶液,37 ℃水浴15 min。加2.0 mL正己烷氯仿混合溶液、0.3 mL 1 mol·L-1乙酸溶液和2.0 mL 无 菌 去 离 子 水,50 r·min-1水 平 振 荡10 min,以1 500 r·min-1速度离心10 min;重复提取1 次。转移上层有机相并使用氮吹仪吹干。
加入正己烷溶解氮吹后的磷脂脂肪酸甲酯,使用MIDI 全自动微生物鉴定系统(Sherlock,美国MIDI 公司)和气相色谱仪(7890B,美国安捷伦科技有限公司)检测微生物PLFA。系统根据各组分的出峰时间和保留时间,与系统谱库中的标准数值相匹配,鉴定PLFA 的种类。PLFA 常见命名格式包括X:YωZ(c/t)、(i/a)X:0、X:0 cy、X:0 10Me等。其中,X表示碳原子数;Y表示双键数;ω 表示甲基末端;Z表示双键到甲基端的距离;c/t分别表示顺式和反式;i/a分别表示异构和反异构支链;cy表示环丙烷脂肪酸;Me 表示甲基[28]。
1.3 数据处理
1.3.1 微生物PLFA 含量和多样性的计算 微生物PLFA 含量采用峰面积归一化法计算:
其中Ai为第i种特征PLFA 的峰面积值,ms为内标物质的物质量,As为内标物质的峰面积值,m为土壤样品干重。
微生物PLFA 的Shannon 多样性指数(H)、Pielou 均匀度指数(J)和Simpson 优势度指数(D)计算公式[29]如下:
其中Pi为第i种特征PLFA 占总PLFA 的比例;S为微生物群落中PLFA 的频次。
1.3.2 统计分析 所有数据使用IBM SPSS Statistics 25 进行统计分析,并使用Origin 2021 进行绘图。采用单因素方差分析,在p<0.050 水平上进行最小显著性差异分析(least significant difference,LSD)。基于Spearman 相关系数分析AgNPs 质量分数与微生物PLFA 之间的相关性。采用主成分分析(principal component analysis,PCA)反映基于PLFA 的微生物群落结构变化。采用Kruskal-Wallis 秩和检验和Wilcoxon 秩和检验检测PLFA 丰度差异,并进行线性判别分析(linear discriminant analysis effect size,LEfSe)。
2 结果与讨论
2.1 微生物PLFA 的种类
通过系统鉴定和计算,识别并筛选出相对含量大于1%的原核生物PLFA 共23 种(见表1)。其中细菌PLFA 共12 种,包括6 种革兰氏阳性菌(gram-positive bacteria,G+)PLFA 和6 种革兰氏阴性菌(gram-negative bacteria,G-)PLFA;真菌PLFA共2 种,包括1 种普通真菌PLFA 和1 种丛枝菌根真菌(arbuscular mycorrhizal fungi,AMF)PLFA;放线菌PLFA 共5 种;通用PLFA 共4 种。
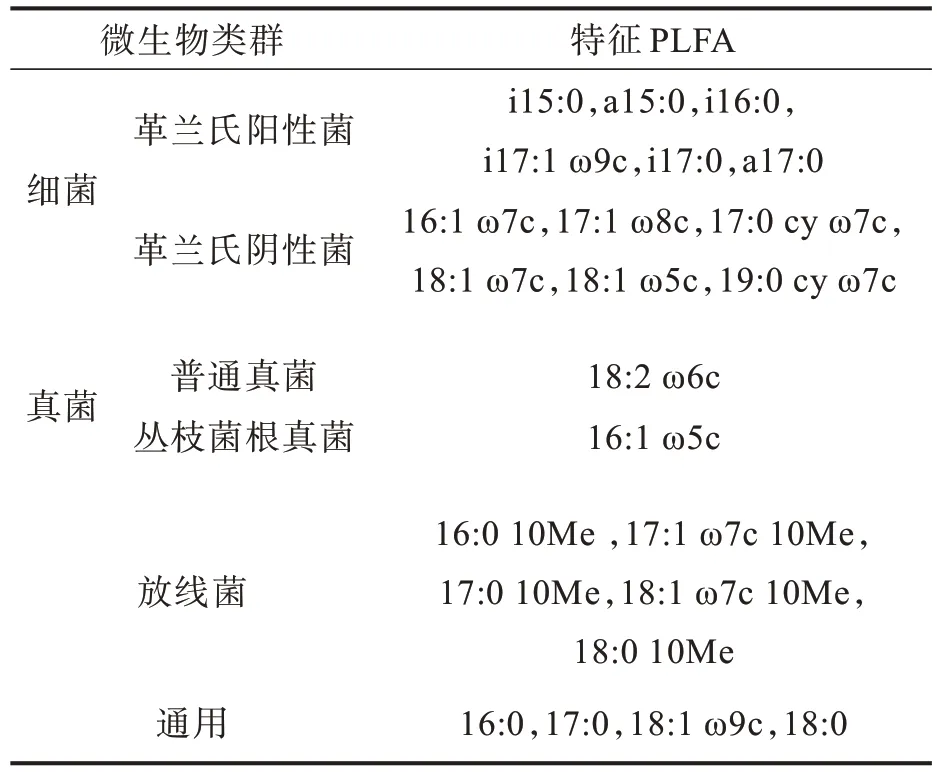
表1 纳米银胁迫下土壤微生物PLFA 种类Tab.1 Species of soil microbial PLFAs under AgNPs stress
2.2 微生物PLFA 的含量
如图1 所示,相关性分析表明,微生物PLFA含量与AgNPs 质量分数之间存在显著负相关(p<0.050)。如图2 所 示,与对 照组相比,Ag10 组、Ag100 组和Ag1 000 组的微生物PLFA 总含量、真菌、放线菌及G-的PLFA 含量分别降低了11.2%~43.0%、14.4%~46.9%、8.6%~52.1%、14.9%~29.4%(p<0.050)。此外,Ag10 组的细菌、G+及AMF 的PLFA 含量与对照组之间没有显著差异(p>0.050),而Ag100 组 和Ag1 000 组 的 相 应 类 群PLFA 含 量 分 别 降 低 了25.9%~39.3%、40.4%~57.3%、33.1%~52.3%(p<0.050)。
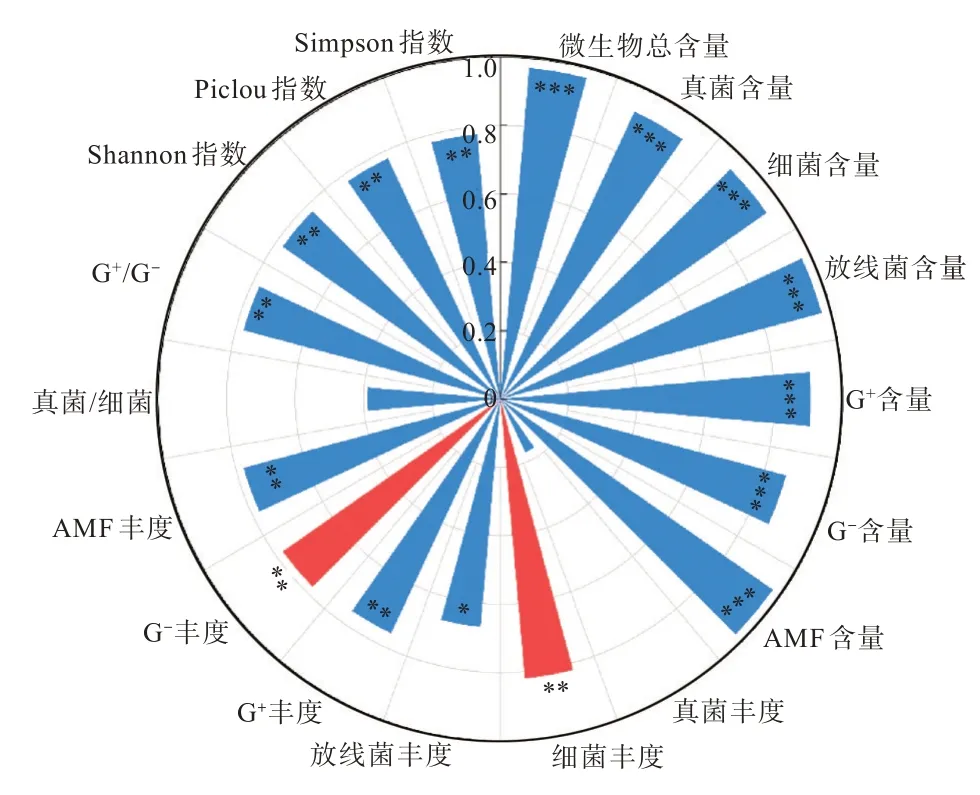
图1 AgNPs 质量分数与土壤微生物PLFA 含量、丰度及比值、多样性指数的Spearman 相关性Fig.1 Spearman correlation between AgNPs mass fraction and content,abundance,ratio,and diversity index of soil microbial PLFA
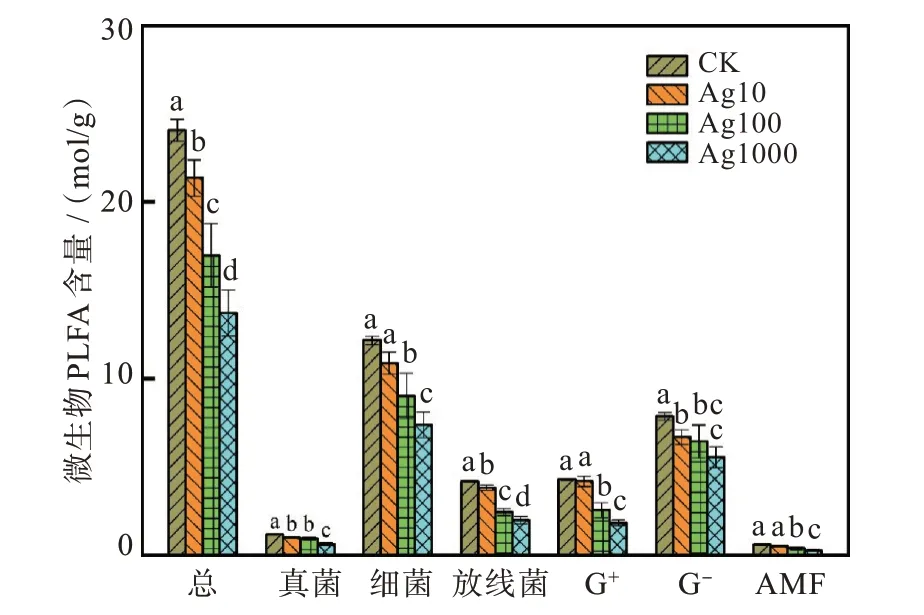
图2 不同水平AgNPs 胁迫下土壤微生物PLFA 含量Fig.2 Content of soil microbial PLFAs under different levels of AgNPs stress
上述结果表明,AgNPs 对微生物整体及各主要类群的生物量存在明显的剂量依赖性抑制作用。Xin 等[30]发现,经过2 个月的培养后,微生物生物量在200 和500 mg·kg-1AgNPs 暴露下均发生显著减 少。Montes 等[19]发现,在暴露于0.015、0.150、1.500 μg·kg-1AgNPs 60 d 后,土壤中G+和放线菌生物量均有所下降。
2.3 微生物PLFA 的丰度及其比值
如图1 所示,相关性分析表明,细菌和G-的PLFA 丰度与AgNPs 质量分数之间存在显著正相关,放线菌、G+和AMF 的PLFA 丰度与AgNPs 质量分数之间存在显著负相关,因此G+/G-丰度比值与AgNPs 质量分数之间存在显著负相关(p<0.050);而真菌PLFA 丰度及真菌/细菌丰度比值与AgNPs 质量分数之间没有显著相关性(p>0.050)。
如图3 所示,与对照组相比,Ag10 组的真菌、细菌、G-和AMF 的PLFA 丰度没有显著差异(p>0.050);Ag100 组的真菌、细菌、G-的PLFA 丰度分别升高了13.4%、4.7%、15.9%(p<0.050),而AMF的PLFA 丰度没有显著差异(p>0.050);Ag1 000组真菌和AMF 的PLFA 丰度则分别降低了7.2%、16.6%,而细菌和G-的PLFA 丰度分别升高了6.4%、23.7%(p<0.050)。此外,Ag10 组放线菌和G+的PLFA 丰度分别升高了3.0%、9.4%,而Ag100组和Ag1 000 组的相应类群PLFA 丰度分别降低了16.2%~16.7%、15.9%~25.1%(p<0.050)。

图3 不同水平AgNPs 胁迫下土壤微生物PLFA 相对丰度Fig.3 Relative abundance of soil microbial PLFAs under different levels of AgNPs stress
如图4 所示,与对照组相比,Ag10 组的G+/G-丰度比值升高了14.2%,而Ag100 组和Ag1 000 组降低了27.3%~39.4%(p<0.050)。同时,Ag10 组和Ag100 组的真菌/细菌的丰度比值与对照组之间没有显著差异(p>0.050),只有Ag1 000 组降低了12.8%(p<0.050)。
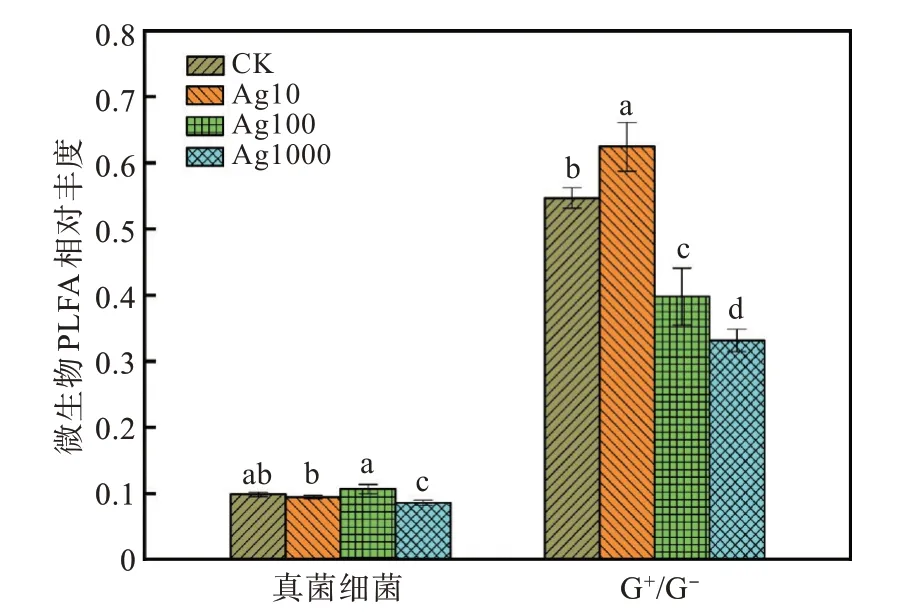
图4 不同水平AgNPs 胁迫下土壤微生物PLFA 相对丰度比值Fig.4 Ratios of soil microbal PLFA values under different levels of AgNPs stress
高质量分数AgNPs(100、1 000 mg·kg-1)暴露下,G-丰度显著升高。Liu 等[31]发现,在1 mg·kg-1AgNPs 胁迫下,G-丰度增加,G+丰度减少。G-的耐受性可能归因于其细胞外膜的脂多糖,在银离子接触细胞时提供了一定的保护作用[32]。此外,Montes 等[33]发现,暴露第60 d 时,与细菌相比,真菌对AgNPs 相对不敏感。这可能是由于真菌作为一种真核生物,对Ag+和重金属更具耐受性。Kumar 等[34]发现在含0.066%AgNPs 的土壤中,细菌PLFA 减少,具有抗氧化特性的Hypocreales 真菌占比从1%增加到70%。
2.4 微生物PLFA 的多样性
如图1 所示,相关性分析表明,微生物PLFA的Shannon 指 数、Pielou 指 数 和Simpson 指 数 与AgNPs 质量分数之间均存在显著负相关(p<0.050)。如图5 所示,与对照组相比,Ag10 组的多样性指数没有显著差异(p>0.050),Ag100 组和Ag1 000 组的Shannon 指数、Pielou 指数和Simpson指数分别降低了1.9%~3.2%、0.9%~1.4%、1.9%~3.2%(p<0.050)。

图5 不同水平AgNPs 胁迫下土壤微生物PLFA 多样性指数Fig.5 Diversity indexes of soil microbal PLFA under different levels of AgNPs stress
如图6 所示,PCA 坐标轴1 和轴2 对微生物群落结构的解释率分别为76.9%和10.8%。对照组与Ag10 组之间的距离较为接近,表明低质量分数AgNPs(10 mg·kg-1)暴露引起的微生物群落组间差异较小。Ag100 组和Ag1 000 组与对照组在坐标轴1 上的距离较远,且2 个处理组之间的距离较为接近,表明高质量分数AgNPs 引起微生物群落结构的显著变化。
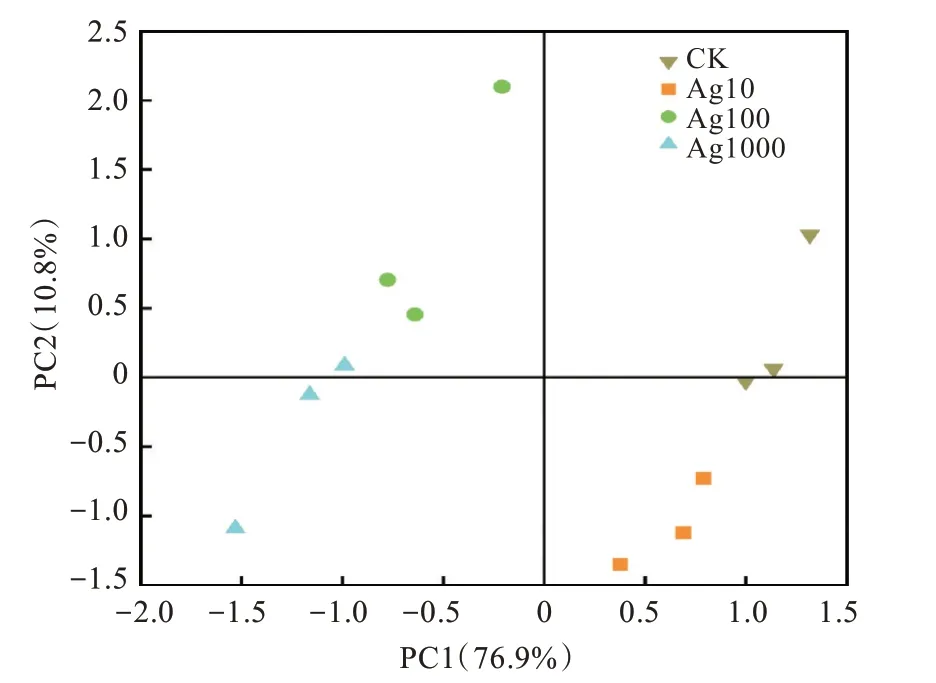
图6 土壤微生物PLFA 的主成分分析Fig.6 Principal component analysis of soil microbial PLFA
随着AgNPs 质量分数增加,微生物多样性逐渐下降,群落组间差异逐渐增大。Zou 等[35]发现,高质量分数AgNPs 处理组的Chao1 指数和Shannon 指数低于对照组;即使在30 d 后,处理组多样性指数仍然未恢复到对照组水平。这种长期影响可能是由于AgNPs 在土壤中的滞留和缓慢释放,AgNPs 的毒性效应随着时间推移而增强[36]。微生物群落结构的变化主要是由于物种丰度发生变化,不同微生物物种对AgNPs 的响应不同,银抗性较强的物种丰度升高,对银敏感的物种丰度下降。此外,群落结构的变化程度与AgNPs 质量分数密切相关。Vasileiadis 等[37]研究发现细菌16S和真菌ITS 基因拷贝数的平均EC50 为89.3 和202.0 mg·kg-1。
2.5 不同处理组间微生物PLFA 的差异
对土壤微生物PLFA 丰度进行LEfSe 分析。如图7 所示,设定线性判别分析值>3.8,p<0.050,共发现8 种具有显著差异的特征PLFA。在对照组中,放线菌标志脂肪酸16:0 10Me 被显著富集;在Ag10 组中,G+标志脂肪酸a15:0 和i16:0及放线菌标志脂肪酸18:0 10Me 被显著富集;在Ag100 组中,真菌标志脂肪酸18:2 ω6c 及放线菌标志脂肪酸18:1 ω7c 10Me 被显著富集;在Ag1 000 组中,G-标志脂肪酸16:1 ω7c 和18:1 ω7c 被显著富集。结果表明,不同质量分数AgNPs 能够显著改变土壤微生物群落结构。
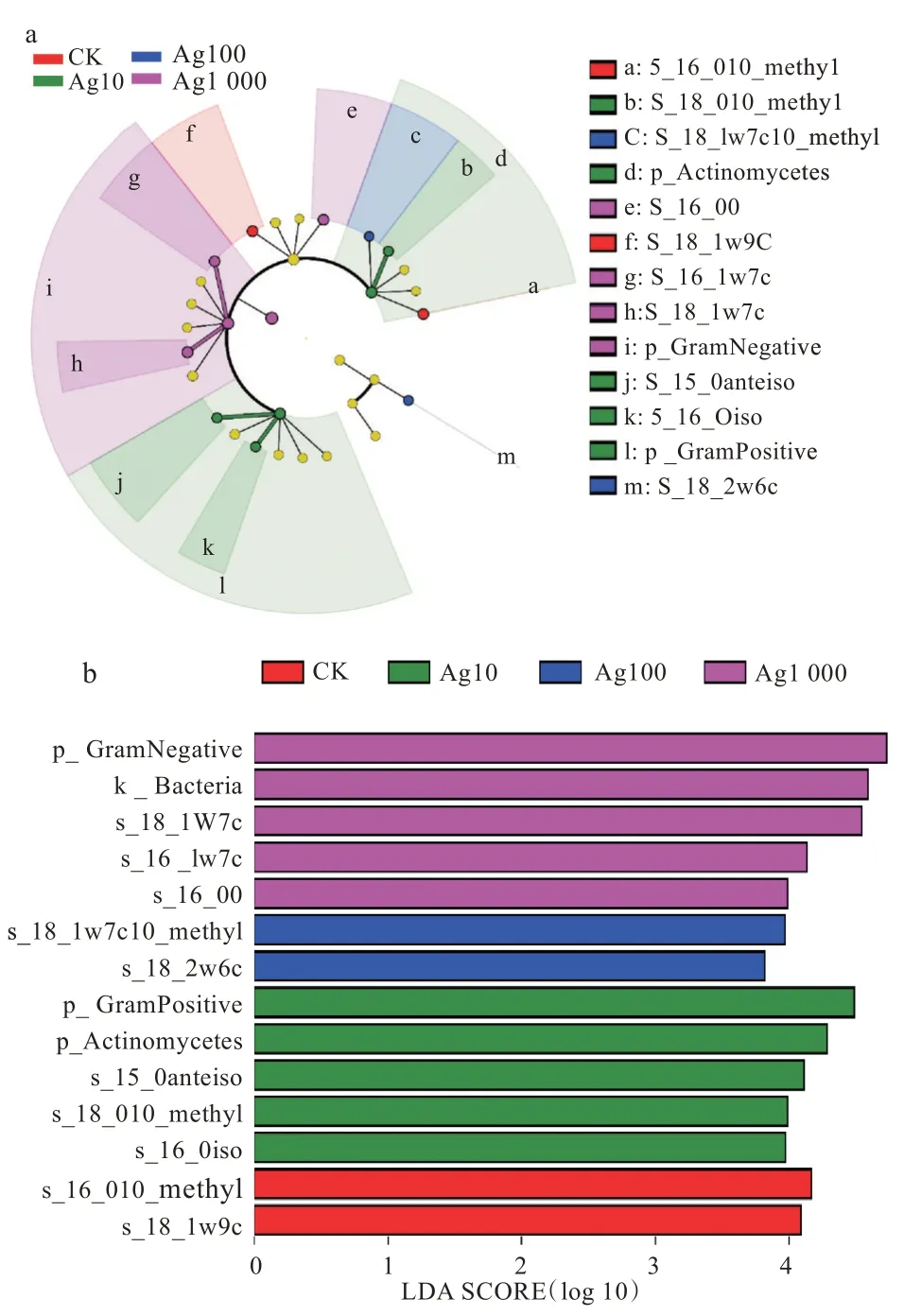
图7 微生物群落组间差异PLFA 的LEfSe 分析:(a)进化分支图,(b)分布柱状图Fig.7 LEfSe analysis of PLFA for differences in microbial communities:(a)cladogram,(b)distribution bar chart
Zhang 等[38]发现,在未种植和种植黄瓜的土壤中,一些脂肪酸及其前体在AgNPs 暴露下显著降低。PLFA 的变化可能是由于AgNPs 诱导生成了大量活性氧。Gunawan 等[39]发现AgNPs 破坏了磷脂酰乙醇胺中的磷酸胺和脂肪酸基团,并认为观察到的细胞膜损伤至少部分是由AgNPs 在其浸出过程中产生的活性氧自由基所造成;细胞内膜磷脂上的包膜靶向可能会引起致死水平的细胞超氧化物和羟基自由基的快速生成。另外,微生物群落中的不同类群可能会对自身细胞膜上的PLFA 作出调整,以应对AgNPs 胁迫。
3 结 论
(1)不同水平AgNPs 胁迫下,土壤微生物PLFA 含量受到剂量依赖性的抑制作用;细菌和G-丰度受到一定的促进作用;G+/G-丰度比受到一定的抑制作用,真菌/细菌丰度比与AgNPs 质量分数没有显著相关性(p>0.050)。
(2)不同水平AgNPs 胁迫下,土壤微生物PLFA 多样性指数降低0.9%~3.2%(p<0.050),微生物群落组间差异随AgNPs 质量分数增加而增大。
(3)G+标志脂肪酸a15:0 和i16:0、真菌标志脂肪酸18:2 ω6c,以及G-标志脂肪酸16:1 ω7c 和18:1 ω7c 的丰度在不同质量分数AgNPs 处理组间具有显著差异(p<0.050)。
[1] REIDY B,HAASE A,LUCH A,et al. Mechanisms of silver nanoparticle release, transformation and toxicity:a critical review of current knowledge and recommendations for future atudies and applications[J].Materials,2013,6(6):2295-2350.
[2] MCGILLICUDDY E,MURRAY I,KAVANAGH S,et al. Silver nanoparticles in the environment:sources,detection and ecotoxicology[J]. Science of the Total Environment,2017,575:231-246.
[3] PACHAPUR V L,LARIOS A D,CLEDON M,et al.Behavior and characterization of titanium dioxide and silver nanoparticles in soils[J]. Science of the Total Environment,2016,563/564:933-943.
[4] LEE S H,SALUNKE B K,KIM B S. Sucrose density gradient centrifugation separation of gold and silver nanoparticles synthesized usingMagnolia kobusplant leaf extracts [J]. Biotechnology and Bioprocess Engineering,2014,19(1):169-174.
[5] VAN HENGEL I A J, RIOOL M, FRATILAAPACHITEI L E,et al. Selective laser melting porous metallic implants with immobilized silver nanoparticles kill and prevent biofilm formation by methicillinresistantStaphylococcus aureus[J]. Biomaterials,2017,140:1-15.
[6] BI Y Q,HAN B,ZIMMERMAN S,et al. Four release tests exhibit variable silver stability from nanoparticlemodified reverse osmosis membranes [J]. Water Research,2018,143:77-86.
[7] KUMAR S, SHUKLA A, BAUL P P, et al.Biodegradable hybrid nanocomposites of chitosan/gelatin and silver nanoparticles for active food packaging applications[J]. Food Packaging and Shelf Life,2018,16:178-184.
[8] MATSUHISA N, INOUE D, ZALAR P, et al.Printable elastic conductors byin situformation of silver nanoparticles from silver flakes[J]. Nature Materials,2017,16(8):834-840.
[9] HANSCH M,EMMERLING C. Effects of silver nanoparticles on the microbiota and enzyme activity in soil[J]. Journal of Plant Nutrition and Soil Science,2010,173(4):554-558.
[10] PROSPOSITO P,BURRATTI L,VENDITTI I. Silver nanoparticles as colorimetric sensors for water pollutants[J].Chemosensors,2020,8(2):26:1-29.
[11] SUN T Y,GOTTSCHALK F,HUNGERBUHLER K,et al. Comprehensive probabilistic modelling of environmental emissions of engineered nanomaterials[J].Environmental Pollution,2014,185:69-76.
[12] ADAM V,CABALLERO-GUZMAN A,NOWACK B. Considering the forms of released engineered nanomaterials in probabilistic material flow analysis[J].Environmental Pollution,2018,243(A):17-27.
[13] WANG P,MENZIES N W,DENNIS P G,et al.Silver nanoparticles entering soils via the wastewatersludge-soil pathway pose low risk to plants but elevated Cl concentrations increase Ag bioavailability[J]. Environmental Science and Technology,2016,50(15):8274-8281.
[14] ANJUM N A,GILL S S,DUARTE A C,et al.Silver nanoparticles in soil-plant systems[J]. Journal of Nanoparticle Research,2013,15(9):1896:1-26.
[15] DELGADO-BAQUERIZO M,REICH P B,TRIVEDI C,et al. Multiple elements of soil biodiversity drive ecosystem functions across biomes [J]. Nature Ecology and Evolution,2020,4(2):210-220.
[16] 管圣迪,苏颖轩,李梦莉,等.老化石油污染土壤微生物群落多样性特征[J]. 化学与生物工程,2023,40(2):37-43.
[17] 徐智,杨玉明,江志强,等.微塑料对生态环境影响的研究进展[J].武汉工程大学学报,2022,44(4):363-370.
[18] PEIXOTO S,OLIVEIRA J M M,HENRIQUES I,et al. Pollution-induced community tolerance framework-disc diffusion method to assess the impact of silver nanoparticles in soils:potential relevance for risk assessment[J]. Applied Soil Ecology,2022,169:104185:1-5.
[19] MONTES de OCA-VASQUEZ G, SOLANOCAMPOS F, VEGA-BAUDRIT J R, et al.Environmentally relevant concentrations of silver nanoparticles diminish soil microbial biomass but do not alter enzyme activities or microbial diversity[J].Journal of Hazardous Materials, 2020, 391:122224:1-10.
[20] SAMARAJEEWA A D, VELICOGNA J R,SCHWERTFEGER D M,et al. Effect of silver nanoparticle contaminated biosolids on the soil microbial community[J]. NanoImpact,2019,14:100157:1-11.
[21] FOSTER A L,MUNK L,KOSKI R A,et al.Relationships between microbial communities and environmental parameters at sites impacted by mining of volcanogenic massive sulfide deposits,Prince William Sound,Alaska[J]. Applied Geochemistry,2008,23(2):279-307.
[22] IBEKWE A M,KENNEDY A C. Phospholipid fatty acid profiles and carbon utilization patterns for analysis of microbial community structure under field and greenhouse conditions[J]. FEMS Microbiology Ecology,1998,26:151-163.
[23] TUNLID A,BAIRD B H,TREXLER M B,et al.Determination of phospholipid ester-linked fatty acids and poly β -hydroxybutyrate for the estimation of bacterial biomass and activity in the rhizosphere of the rape plantBrassica napus(L.)[J]. Canadian Journal of Microbiology,1985,31:1113-1119.
[24] ROSLEV P,IVERSEN N,HENRIKSEN K. Direct fingerprinting of metabolically active bacteria in environmental samples by substrate specific radiolabelling and lipid analysis[J]. Journal of Microbiological Methods,1998,31:99-111.
[25] 齐鸿雁,薛凯,张洪勋. 磷脂脂肪酸谱图分析方法及其在微生物生态学领域的应用[J]. 生态学报,2003,23(8):1576-1582.
[26] PRIESTER J H,GE Y,MIELKE R E,et al.Soybean susceptibility to manufactured nanomaterials with evidence for food quality and soil fertility interruption[J]. Proceedings of the National Academy of Sciences(USA),2012,109(37):E2451-E2456.
[27] BLIGH E G,DYER W J. A rapid method of total lipid extraction and purification[J]. Canadian Journal of Biochemistry and Physiology,1959,37(8):911-917.
[28] 吴建军,蒋艳梅,吴愉萍,等.重金属复合污染对水稻土微生物生物量和群落结构的影响[J]. 土壤学报,2008,45(6):1102-1108.
[29] GARLAND J L,MILLS A L. Classification and characterization of heterotrophic microbial communities on the basis of patterns of communitylevel sole-carbon-source utilization[J]. Applied and Environmental Microbiology,1991,57(8):2351-2359.
[30] XIN X P,ZHAO F L,ZHAO H M,et al. Comparative assessment of polymeric and other nanoparticles impacts on soil microbial and biochemical properties[J].Geoderma,2020,367:114278:1-9.
[31] LIU G F,ZHANG M,JIN Y J,et al. The effects of low concentrations of silver nanoparticles on wheat growth,seed quality,and soil microbial communities[J]. Water Air and Soil Pollution,2017,228(9):348:1-12.
[32] JACOBSON K H,GUNSOLUS I L,KUECH T R,et al. Lipopolysaccharide density and structure govern the extent and distance of nanoparticle interaction with actual and model bacterial outer membranes[J].Environmental Science and Technology,2015,49(17):10642-10650.
[33] MONTES de OCA-VASQUEZ G, SOLANOCAMPOS F,VEGA-BAUDRIT J R,et al. Organic amendments exacerbate the effects of silver nanoparticles on microbial biomass and community composition of a semiarid soil[J]. Science of the Total Environment,2020,744:140919:1-11.
[34] KUMAR N,PALMER G R,SHAH V,et al. The effect of silver nanoparticles on seasonal change in arctic tundra bacterial and fungal assemblages[J].PLoS One,2014,9(6):e99953:1-12.
[35] ZOU X Y,LI P H,WANG X D,et al. Silver nanoparticle and Ag+-induced shifts of microbial communities in natural brackish waters:are they more pronounced under oxic conditions than anoxic conditions?[J]. Environmental Pollution, 2020,258:113686:1-29.
[36] KRAAS M,SCHLICH K,KNOPF B,et al. Longterm effects of sulfidized silver nanoparticles in sewage sludge on soil microflora[J]. Environmental Toxicology and Chemistry,2017,36(12):3305-3313.
[37] VASILEIADIS S,BRUNETTI G,MARZOUK E,et al. Silver toxicity thresholds for multiple soil microbial biomarkers[J]. Environmental Science and Technology,2018,52(15):8745-8755.
[38] ZHANG H L,HUANG M,ZHANG W H,et al.Silver nanoparticles alter soil microbial community compositions and metabolite profiles in unplanted and cucumber-planted soils[J]. Environmental Science and Technology,2020,54(6):3334-3342.
[39] GUNAWAN C,FAIZ M B,MANN R,et al.Nanosilver targets the bacterial cell envelope:the link with generation of reactive oxygen radicals[J]. ACS Applied Materials and Interfaces,2020,12(5):5557-5568.
