基于转录组和WGCNA筛选丝瓜果长和果径发育调控相关基因
2023-12-29陈敏氡王彬刘建汀李永平白昌辉叶新如裘波音温庆放朱海生
陈敏氡,王彬,刘建汀,李永平,白昌辉,叶新如,裘波音,温庆放,朱海生
基于转录组和WGCNA筛选丝瓜果长和果径发育调控相关基因
陈敏氡,王彬,刘建汀,李永平,白昌辉,叶新如,裘波音,温庆放,朱海生
福建省蔬菜遗传育种重点实验室/福建省蔬菜工程技术研究中心/福建省农业科学院作物研究所,福州 350013
【目的】鉴定丝瓜果长和果径发育基因共表达模块并筛选关键调控基因,为后续丝瓜果形控制的分子机制研究提供理论依据。【方法】以丝瓜9个发育阶段(开花前2 d以及花后0、2、4、6、8、10、15和20 d)果实为研究材料,测定各阶段的果长和果径,利用WGCNA方法联合分析转录组与果长和果径数据,鉴定与果长和果径发育相关的共表达基因模块,筛选关键调控基因。【结果】利用WGCNA鉴定出14个共表达模块,与果长和果径显著相关(相关系数的绝对值=0.9)的共表达模块有2个,显著正相关的模块为Turquoise模块,显著负相关的模块为Lightpink4模块。KEGG富集分析发现,Turquoise模块显著富集在内吞作用和苯丙烷生物合成通路,与果实膨大、伸长调控密切相关,可作为研究丝瓜果长和果径的关键基因模块。根据Turquoise模块内基因的连接度以及功能注释,筛选出10个关键调控基因,包括木葡聚糖内转葡糖基酶/水解酶基因、肌动蛋白解聚因子基因、伴侣蛋白基因、扩展蛋白基因(、和)、驱动蛋白基因、生长素反应基因和。RT-qPCR结果显示,10个调控基因的表达量均在果实进入快速生长期(花后8 d)后显著升高,增加倍数约为2—50倍。通过构建基因互作网络,发现部分调控基因与WRKY、bHLH和HSF转录因子家族存在互作关系。【结论】获得了丝瓜果长和果径基因共表达重要模块Turquoise模块,筛选到10个调控基因可作为丝瓜果形控制的潜在候选基因,并发现丝瓜果长和果径的发育调控主要涉及细胞壁的重构、细胞的发育与分化、生长素的调控等过程。
转录组;WGCNA;丝瓜;果长;果径;调控基因
0 引言
【研究意义】丝瓜(L.)为葫芦科丝瓜属作物,是我国夏秋供应市场的主要瓜类蔬菜之一。丝瓜果肉清香软滑、营养丰富且具有较高的药用功能,可用于治疗筋骨酸痛、月经不调或产后乳汁不通等症状,因此深受消费者喜爱[1-2]。不同地区对丝瓜的消费习惯并不一致,南方地区比较倾向于棒形的丝瓜,而北方地区以圆筒形丝瓜和皱皮丝瓜为主。随着现代家庭结构的改变,单瓜重约500 g的短棒、厚肉型丝瓜也逐渐受到消费者的追捧[3]。‘福研1号’[4]、‘双丰1号’[5]、‘早优3号’[6]等短肉型丝瓜新品种相继问世,现已在我国福建、浙江、广东等沿海地区大面积种植,具有广阔的市场前景和巨大的经济潜力。由此可见,果长和果径已成为丝瓜育种的重要考量指标,解析丝瓜果形控制的分子调控机制显得尤为必要。【前人研究进展】果长和果径是丝瓜重要的商品性状,反映丝瓜果实的发育动态,与品质、产量性状紧密相关。前人对于丝瓜果长、果径的遗传研究已有少量文献报道,高军等[7]认为,普通丝瓜的果长遗传由一对加性主基因控制,同时存在多基因的修饰作用。林明宝等[8]研究发现,有棱丝瓜中控制果长性状的最少基因数目为4对。苏小俊等[9]研究显示,普通丝瓜果长、果径的主基因遗传力均较大,宜在分离早代采取表型选择法进行选择。崔竣杰等[10-11]在丝瓜连锁群scaf1上定位到1个贡献率为28.90%的果长QTL位点,表明丝瓜果长受环境影响较大,认为需要在高世代中进行选择。近年来,随着丝瓜全基因组的公布,少数果实发育基因被研究。曾美娟等[12]发现6个生长素酰胺合成酶家族基因(gretchen hagen 3,)在普通丝瓜长、短果品种中差异表达,推测这些基因与丝瓜果长发育相关。随后又发现SUN-domain蛋白家族基因在丝瓜花后12 d短果中的表达量极显著高于长果,可能是育种潜力基因[13]。然而,目前已获得的这些基因依然是对前人研究结果的验证,调控丝瓜果长和果径的主效基因及功能尚未可知。权重基因共表达网络分析(WGCNA)方法能够有效地整合转录组、代谢组和蛋白质组等多组学数据,正在成为发掘新基因功能和探索基因网络与特定性状间关联关系的重要方法[14]。近几年,在脐橙[15]、黄瓜[16]、西瓜[17]等果蔬形态发生研究中均可见利用WGCNA方法的报道。冯贵芝[15]发现介导柑橘果实ABA信号调节过程,在果实膨大期间起主要作用。李情[16]在黄瓜果实形态发生过程共鉴定出235个细胞分裂相关、1 020个细胞伸长相关的差异表达转录本,并发现、在其中起着至关重要的作用。孙蕾[17]发现胞壁酶和激素相关的基因,如聚半乳糖醛酸酶基因、果胶酯酶基因、生长素反应蛋白基因、脱落酸受体基因、乙烯反应转录因子、糖基转移酶基因和扩展蛋白基因等参与调控了西瓜果实的膨大、成熟软化过程。【本研究切入点】WGCNA为果蔬形态调控基因的挖掘提供了新途径,目前尚未见采用WGCNA方法筛选丝瓜果长和果径发育调控基因的报道。【拟解决的关键问题】本研究以普通丝瓜9个发育阶段的果实为研究材料,分析果实在各个发育阶段果长和果径的变化动态,通过WGCNA方法联合分析转录组与果长和果径数据划分基因共表达模块,挖掘与果长和果径相关联的关键模块及调控基因,并构建这些基因的互作调控网络,为后续丝瓜果形控制的遗传转化研究提供潜在候选基因。
1 材料与方法
1.1 材料
试验于2022年在福建省农业科学院作物研究所福清东张基地和福州科研综合研究中心进行,选择丝瓜杂交种‘福研3号’为试验材料。
1.2 方法
1.2.1 果实果长、果径测定 采集开花前2 d(记为花后-2 d)以及花后0、2、4、6、8、10、15和20 d长势均匀的果实各3个,共计27个样品,用游标卡尺测量其果长和果径。
1.2.2 丝瓜果肉RNA的提取、纯化、质检和文库构建及其数据分析 选取花后-2 d至15 d共24个样品进行转录组测序。切下样品中部约5 cm的果肉,液氮中速冻30 min,80℃保存,利用Plant RNA Purification Reagent(Invitrogen)提取总RNA,琼脂糖凝胶电泳分析RNA的完整性及是否存在DNA污染,采用NanoDropOne(Thermo Fisher Scientific,USA)对RNA质量进行初步检测(RIN≥6.5,OD260/280=1.8—2.2,OD260/230≥2.0)。按照Illumina TruSeqTMRNA sample preparation Kit方法构建转录组文库,库检合格后采用Illumina MiSeq PE300(Illumina,USA)进行测序,由北京奥维森基因科技有限公司完成。高通量测序仪得到的图像数据经CASAVA碱基识别转化为序列数据(raw read)。对原始数据进行去除带接头、poly-N及低质量read后获得高质量clean read。并对Q20、Q30、GC含量及重复序列水平进行统计;将clean read用HISAT2软件与丝瓜参考基因组(https://peanutbase. org/data/public/Arachis_hypogaea/Tifrunner.gnm1.KYV3/)进行基因比对。
1.2.3 加权基因共表达网络分析 利用RESM软件对基因表达数据进行背景校正和标准化,过滤表达量过低和变异系数较小的基因[18]。利用R软件WGCNA包构建基因共表达网络分析(https://horvath.genetics. ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/index.html)[19]。为了确保符合无尺度网络分布,利用WGCNA包中的pickSoftThreshold函数计算合适的加权系数,的选取标准即满足相关系数的平方接近0.8,同时还需要保证一定的基因连接度[20]。采用动态切割法识别共表达模块[21]。利用自动网络构建函数blockwiseModules构建网络,其中最小模块大小为50,合并相似性为0.75的模块[20]。
1.2.4 特异性共表达模块的鉴定、KEGG分析和基因表达趋势分析 定义相关系数绝对值在0.5以上且显著性值达到0.05水平的模块为显著模块[22]。利用R语言的clusterProfiler包对显著模块内基因进行功能富集(KEGG)分析,列出显著性前20个代谢通路做气泡图[23]。利用STEM软件对特异性共表达模块基因进行趋势分析,设定模块数量为20,为0.05,以获得显著性和非显著性基因表达模型[24]。
1.2.5 特异性模块核心基因的筛选及互作网络的构建 利用softConnectivity函数计算基因的连接度,筛选出模块内连接度前10的基因,结合已报道的与葫芦科果长和果径相关的基因(基因的连通性在模块排名的前10%),定义这些基因为模块的调控基因[25]。计算调控基因与模块内全部基因的皮尔逊相关性系数,分别挑选与调控基因正负相关性前10的基因绘制网络图,利用Cytoscape v3.6.1进行可视化展示[23]。
1.2.6 RT-qPCR分析 采用通用植物总RNA提取试剂盒(北京百泰克生物技术有限公司)提取丝瓜花后-2 d至15 d共8个发育阶段果实总RNA,利用HiScript III 1st Strand cDNA Synthesis Kit(+gDNA wiper)反转录试剂盒(南京诺唯赞生物科技股份有限公司)合成cDNA。利用Primer Premier 5软件设计定量引物,具体引物序列见表1,以丝瓜为内参基因[26]。按照SYBR Premix Ex TaqTM Ⅱ荧光定量试剂盒(宝生物工程大连有限公司)说明书在ABI7500荧光定量PCR仪(Applied Biosystems,USA)上进行分析。扩增程序为95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火34 s,40个循环。每个处理3个重复,并使用2-ΔΔCT方法计算基因的相对表达量[27],采用Excel软件作图。

表1 本研究所用实时荧光定量PCR引物
2 结果
2.1 丝瓜果实发育过程中果长和果径的生长动态分析
由图1、2可知,丝瓜果实发育过程可以大致分为3个时期。花后-2 d至2 d为果实发育前期,这期间丝瓜果长和果径生长缓慢;花后2—15 d为果实发育中期,这期间果长和果径明显增长、增大,其中花后8—15 d为果实的快速生长期和成熟期,果实膨大和伸长极显著(<0.001);花后15—20 d果实进入发育后期,这期间丝瓜果长和果径基本不发生变化。
2.2 加权基因共表达网络的构建
对丝瓜8个发育阶段(花后-2 d至15 d)的24个丝瓜果实样品进行RNA-seq数据分析,对表达矩阵中变异度较低的基因进行过滤后,选取26 620个基因进行WGCNA分析。当最优软阈值=6时,2>0.8,平均连通性趋于0(附图1),表明该软阈值适合用来构建无尺度网络。根据基因的表达量进行聚类分析,聚类程度高的基因将被分到一个模块(附图2)。本研究共获得14个基因共表达模块,各模块中所包含的基因数量差异明显,其中Turquoise模块中的基因数量最多(6 128个),Honeydew1模块中的基因数量最少(86个)(图3)。
2.3 特异性共表达模块的鉴定
将14个基因共表达模块分别与果长和果径进行相关性分析,结果发现与丝瓜果长和果径发育显著相关的共表达基因模块基本一致。其中,Turquoise模块(=0.9,=3×10-9和=0.9,=1×10-9)和Brown4模块(=0.64,=7×10-4和=0.65,=5×10-4)与果长和果径显著正相关,Lightpink4模块(=-0.9,=2×10-9和=-0.9,=2×10-9)和Ivory模块(=-0.67,=4×10-4和=-0.65,=6×10-4)与果长和果径显著负相关(图4)。
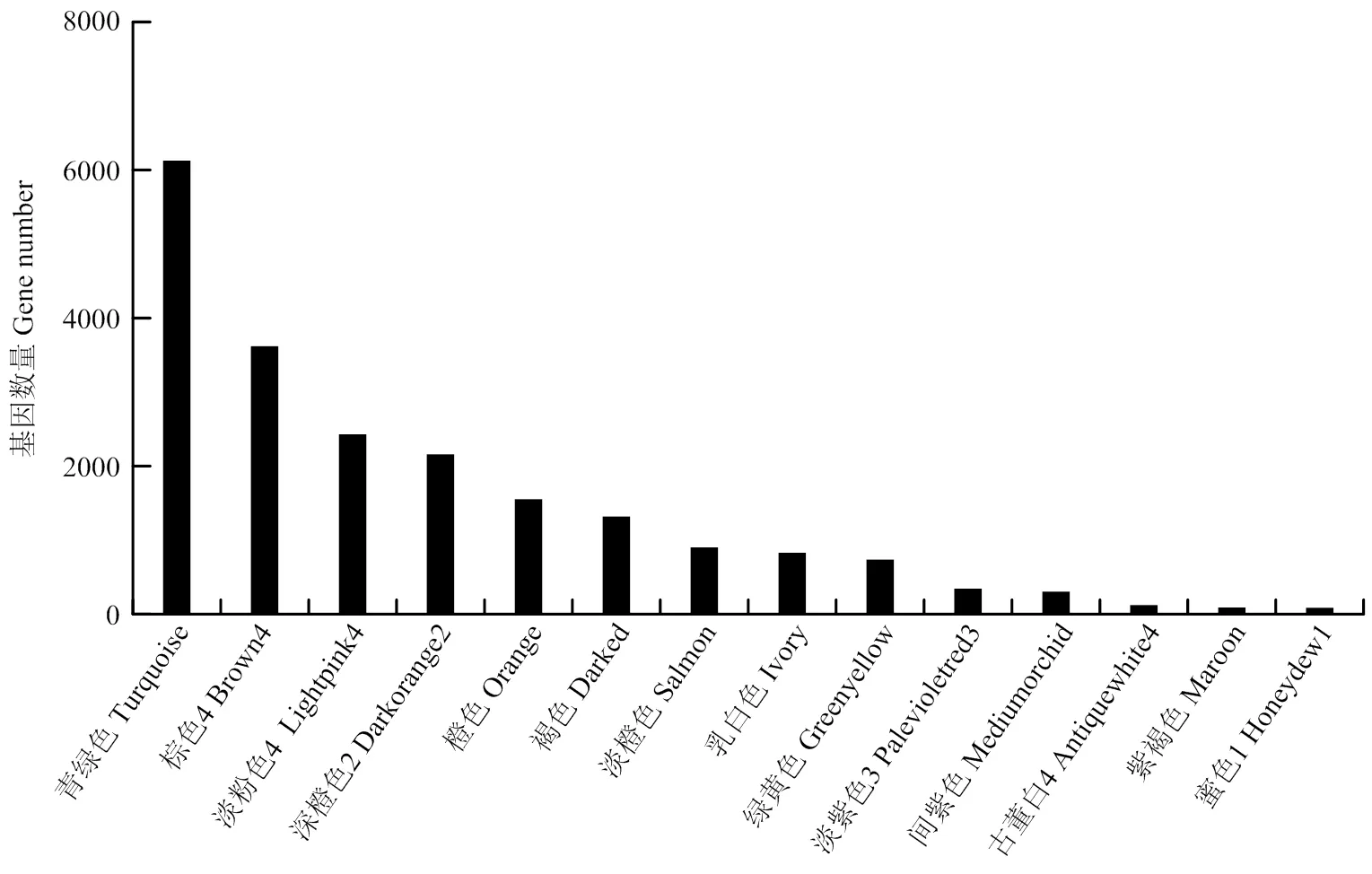
图3 各模块的基因数量
2.4 基因共表达模块的KEGG分析及表达趋势分析
选取相关性最高的Lightpink4和Turquoise基因共表达模块进行KEGG分析。结果显示,Lightpink4模块的基因主要显著富集在氨基酸合成通路、剪接体通路、C5-二元酸代谢通路和丁酸代谢通路(图5-A)。在生物体中,氨基酸合成通路、C5-二元酸代谢通路和丁酸代谢通路是产生能量、脂肪酸、氨基酸的重要途径。剪接体负责pre-mRNA剪接,是基因表达与调控的重要环节之一[28]。2016年,哈佛公卫学院Heintz等[29]研究团队首次将剪接体与生物体的寿命相关联,并提出在未来剪接体可能作为衰老的一种生物标志物或是早期标记。本研究中Lightpink4模块的基因与丝瓜果长和果径显著负相关(=-0.9,=2×10-9和=-0.9,=2×10-9),表明这些基因可能更多地参与到丝瓜果实发育后期至衰老过程。Turquoise模块的基因主要显著富集在内吞作用通路、氨基糖和核苷酸糖代谢通路、植物-病原相互作用通路和苯丙烷生物合成通路(图5-B)。其中内吞作用和苯丙烷生物合成通路内显著富集的基因数量最大(分别为52和41个)。内吞作用是植物细胞吸收水分、矿物质等的常见方式之一。德国弗莱堡大学DÜNSER等[30]课题组发现内吞运输能够同步细胞和液泡表面的增大,推测这可能是植物细胞在细胞扩张速度方面表现出色的原因。苯丙烷生物合成的重要代谢物木质素能够为植物提供机械支撑,同时参与导管的形成以及水分和矿质元素的运输[31]。可见,这两个通路均与果实膨大、伸长过程紧密相关。本研究中Turquoise模块的基因与丝瓜果长和果径显著正相关(=0.9,=3×10-9和=0.9,=1×10-9),表明该模块可作为研究丝瓜果长和果径调控相关基因的重要模块。因此,本研究重点关注Turquoise模块。对Turquoise模块中6 128个基因进行表达趋势分析(图6),Turquoise模块中的基因可以归纳为20种表达趋势模型,其中Profile 0、1、16、19为4个显著基因表达模型(<0.05),Profile 0为显著下调表达模型,包含349个基因;Profile 19为显著上调表达模型,包含2 607个基因。
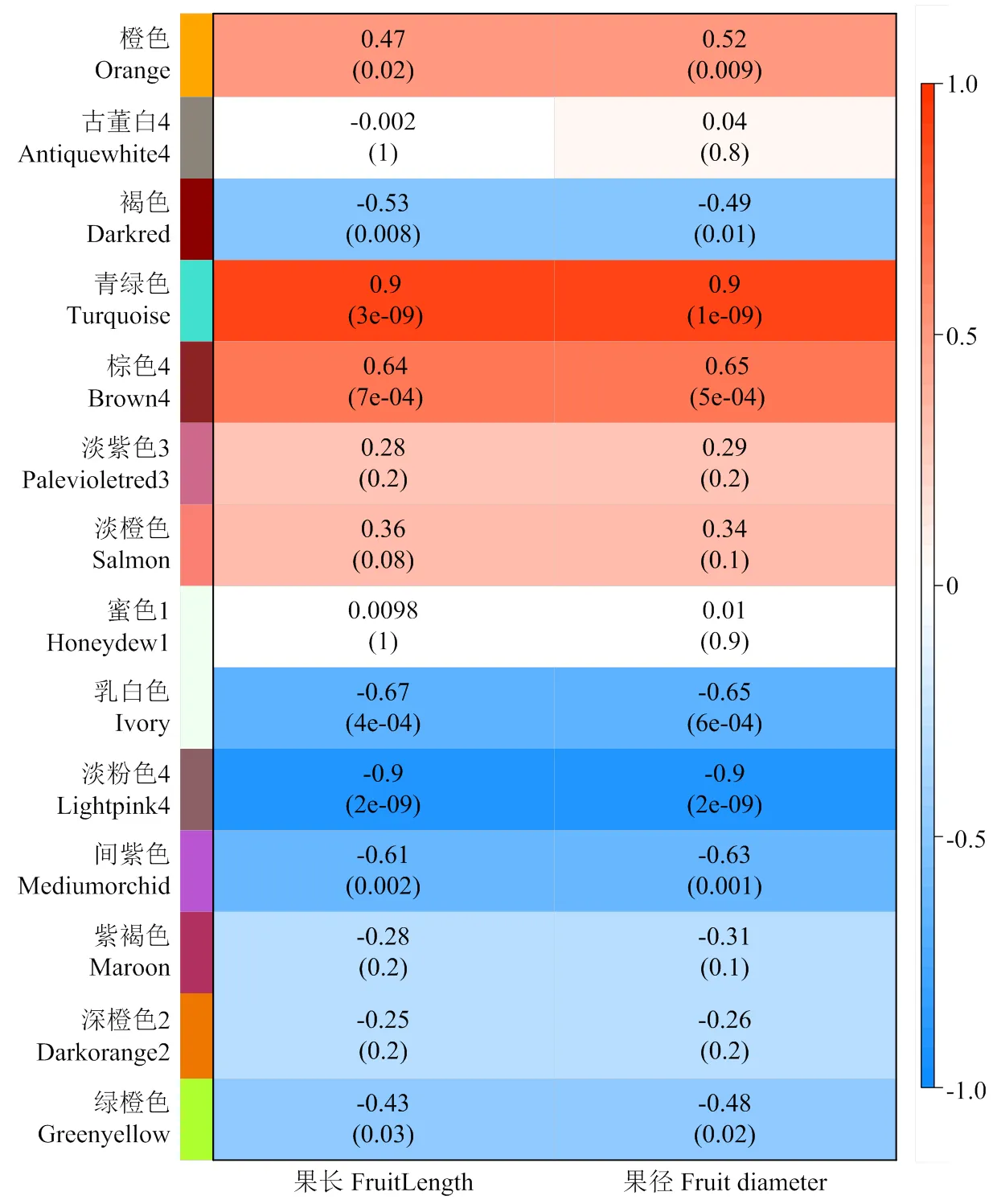
横坐标代表不同表型,纵坐标代表不同模块。图中每组数据表示模块与表型的相关性系数R值及显著性P值(括号内)。红色代表模块与表型具有正相关,蓝色代表模块与表型具有负相关

图5 Lightpink4(A)和Turquoise(B)基因共表达模块的KEGG富集分析
2.5 丝瓜果长和果径发育调控相关基因筛选
选取Turquoise模块内连接度最高的前10个基因作为该模块的核心基因,并通过与葫芦科同源基因的比对和注释来预测核心基因的功能。结果表明,Turquoise模块内连接度最高的前10个基因中有6个基因有注释信息,分别编码木葡聚糖内转葡糖基酶/水解酶蛋白(xyloglucan endotransglucosylase/ hydrolase protein 23,XTH23)(Maker00007620)、肌动蛋白解聚因子(actin-depolymerizing factor 2,ADF2)(Maker00033581)、伴侣蛋白DnaJ10(chaperone protein dnaJ 10,DnaJ10)(Maker00000726)、磷脂酰肌醇(phosphatidylinositol,PI)(Maker00012467)、同型半胱氨酸S-甲基转移酶(homocysteine S- methyltransferase 1,HMT1)(Maker00010036)和钙依赖性蛋白激酶(calcium-dependent protein kinase 1,CDPK1)(Maker00009822)。PI、HMTI和CPK1在植物体中主要参与激活信号传递[32]、甲基传递[33]和钙信号转导[34]等过程。而XTH23、ADF2和DnaJ10在植物细胞壁重组[35]、细胞生长[36]和叶绿体的形成及分化[37]等生命过程起重要作用,与果长和果径发育密切相关,因此,认为这3个基因可作为丝瓜果长和果径发育候选基因。继续在Turquoise模块中筛选葫芦科中已报道与果长和果径发育相关的基因,结果在该模块中共发现7个已报道过的基因,且基因的连通性均在模块排名的前10%,包括4个扩展蛋白基因(Maker00000768、Maker00039481、Maker00026031、Maker00009740)、1个驱动蛋白基因(Maker00024997)以及2个生长素反应蛋白基因(Maker00001372)和(Maker00038241)(表2)。因此,本研究将以上10个基因作为丝瓜果长和果径发育的候选基因。
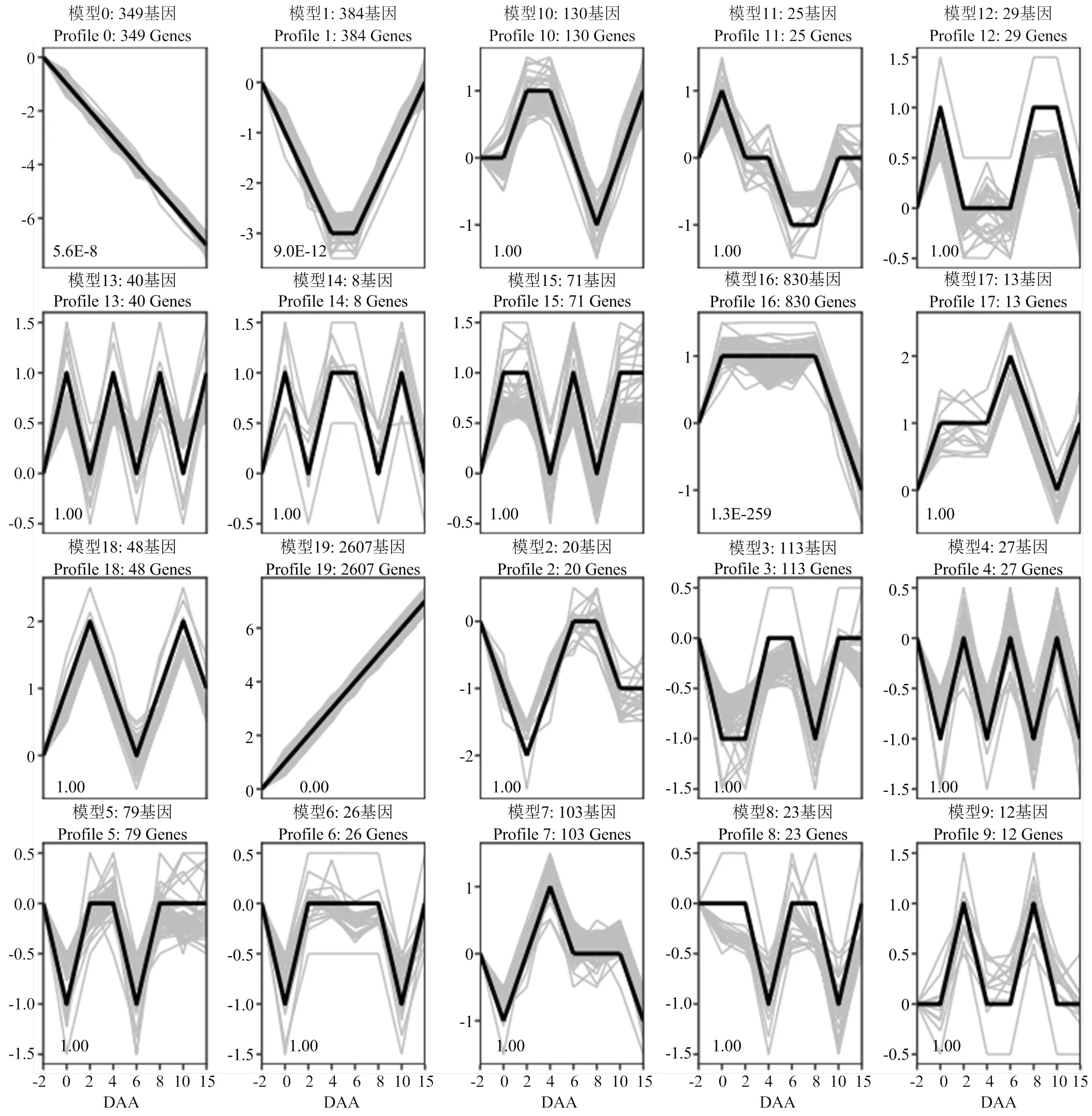
方框内数字为P值 The number in the box denoting the P-value

表2 丝瓜果长和果径发育调控的候选基因
2.6 调控基因的表达模式分析
进一步利用RT-qPCR方法对10个调控基因的表达情况进行验证,其结果与转录组数据基本一致(图7),说明转录组数据结果的可信度较高。RT-qPCR结果显示,10个候选基因大致包括3种表达模式,Maker00000726、Maker00038241、Maker00001372和Maker00024997这4个基因随着果实的生长发育,表达量逐渐升高,其中花后10—15 d后升高明显,表达量增加约2—6倍。Maker00007260和Maker00033581的表达量在发育前期(花后-2 d至2 d)基本不变,进入发育中期后共有2次表达量的显著升高,花后4—8 d表达量增加约2—6倍,花后10—15 d表达量增加显著,约10—50倍。Maker00026031、Maker00000768、Maker00039481和Maker00009740的表达量在花后8 d前基本不变,花后10—15 d表达量显著升高,增加约10—40倍。
2.7 调控基因互作网络的构建
挑选与10个调控基因正负相关性前10的基因绘制网络图(附表1),利用Cytoscape v3.6.1进行可视化展示(图8)。结果显示与(Maker00007620)负相关性最高的基因编码UV-B诱导蛋白(Maker00013740)(相关系数=0.980),正相关性最高的基因编码富亮氨酸重复受体蛋白激酶(Maker00026072)(=0.985);与2(Maker00033581)负相关性最高的基因编码谷氨酰-tRNA还原酶1(Maker00033993)(=0.930),正相关性最高的基因编码DA1相关蛋白2(Maker00009584)(=0.970);与(Maker00000726)负相关性最高的基因编码3-脱氢奎尼酸脱水酶/莽草酸脱氢酶(Maker00027020)(=0.943),正相关性最高的基因编码真核生物翻译起始因子3亚基J(Maker00008454)(=0.986);与(Maker00000768)负相关性最高的基因编码羟酰基谷胱甘肽水解酶(Maker00028749)(= 0.926),正相关性最高的基因编码受体激酶At5g67200(Maker00012477)(=0.965);与(Maker00039481)负相关性最高的基因编码葡萄糖-6-磷酸1-脱氢酶(Maker00001558)(=0.921),正相关性最高的基因编码生长素外排载体(Maker00029480)(=0.939);与(Maker00026031)负相关性最高的基因编码受体蛋白激酶At5g15080(Maker00025844)(=0.934),正相关性最高的基因编码生长素诱导蛋白22d(Maker00039055)(=0.988);与(Maker00024997)负相关性最高的基因编码转运抑制反应蛋白1(Maker00016411)(=0.907),正相关性最高的基因编码TIFY 9蛋白(Maker000)(=0.924);与(Maker00009740)负相关性最高的基因编码E3泛素蛋白连接酶RING1(Maker00022044)(=0.893),正相关性最高的基因编码转录因子bHLH149(Maker00003629)(= 0.912);与(Maker00001372)负相关性最高的基因编码Csa_5G517870蛋白(Maker00029937)(=0.817),正相关性最高的基因编码LOC101210747亚型X1蛋白(Maker00004186)(=0.858);与(Maker00038241)负相关性最高的基因编码非典型聚(A)聚合酶PAPD5(Maker00016990)(=0.968),正相关性最高的基因编码胚芽水解酶7亚型X1(Maker00006161)(=0.977)。此外,在网络图中注释到3类转录因子,分别编码(Maker00039492)、9(Maker00036518)、(Maker0002332)、(Maker00003629)和(Maker00028475)。、、、分别与、、、存在互作关系。前人已在拟南芥[38]、苜蓿[39]、杨树[40]中证实WRKY和bHLH类转录因子参与木质素生物合成的调控。木质素是植物细胞壁的主要成分,、、、均与植物细胞壁合成相关。由此推测这2类转录因子可能也参与到丝瓜果实生长膨大过程中细胞壁的合成、松弛和伸展。调控植物生长发育,在西瓜果实发育[41]和番茄植株矮化[42]过程中均发挥着重要作用。与存在互作关系,推测二者共同参与丝瓜果实细胞的生长、分化。
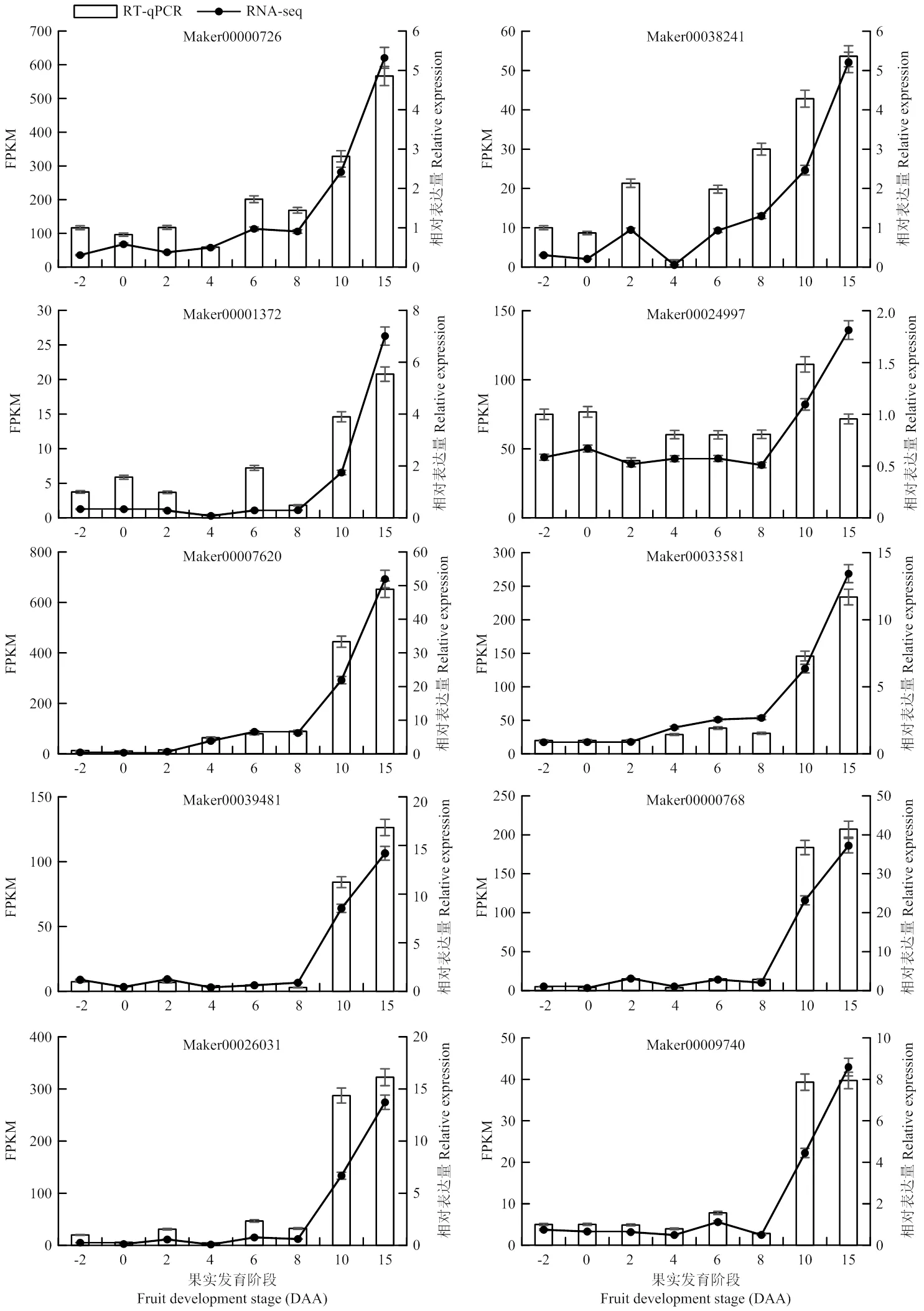
图7 10个调控基因的RT-qPCR和RNA-seq结果
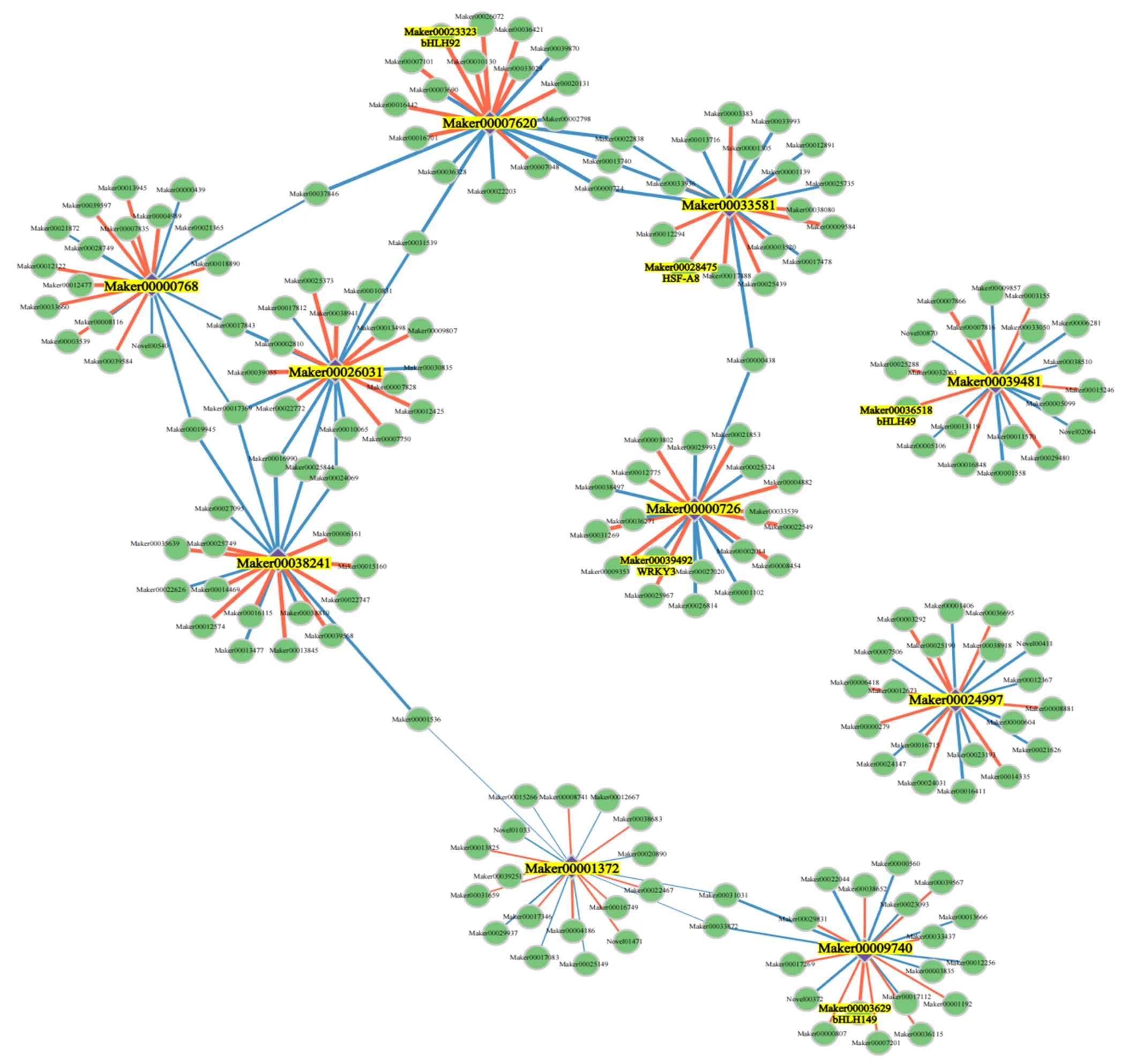
图中连线颜色代表调控基因与关联基因的相关性类型,红色代表正相关,蓝色代表负相关;线条粗细代表相关性数值大小。调控基因及其关联的转录因子用黄色标注
3 讨论
2008年,Langfelder和Horvath[19]提出了加权共表达网络分析(WGCNA)方法[19],国内外学者通过该方法寻找到了许多与植物器官形态建成和发育调控的关键基因[43-45]。因此,本研究将WGCNA方法引入丝瓜果长和果径发育调控基因的筛选。
3.1 丝瓜果长和果径基因共表达重要模块的鉴定
在共表达网络分析中共获得14个基因共表达模块,并发现与丝瓜果长和果径发育显著相关的模块基本一致,推测丝瓜果长和果径发育可能由许多相同的基因调控。通过共表达模块与表型的相关性分析以及模块内基因的KEGG分析,关注到Turquoise模块。该模块中有41个共表达基因显著富集在苯丙烷生物合成途径。苯丙烷类生物合成途径是植物3条主要次生代谢途径之一,该途径中的一个重要产物木质素与植物细胞的分化及器官生长过程密切相关。晏巢等[46]研究显示油茶果皮中木质素积累过程影响了果实发育,木质素积累越早,果实越小,并且种子的形态也与木质素具有直接的关系。朱海英等[47]研究表明,丝瓜果实的发育过程伴随着大量木质素的累积。课题组前期也发现5种木质素合成蛋白(PAL、4-CL、CAD、POD和CCoAOMT)的表达水平在丝瓜果实发育过程显著升高,说明丝瓜果实发育过程中苯丙烷生物合成活跃,木质素大量合成[48]。由此表明,Turquoise模块可作为研究丝瓜果长和果径基因的重要模块。未来可以结合细胞生物学和代谢组学手段研究丝瓜果实内木质素的结构类型、积累规律以及活跃区域等,进一步探究木质素与丝瓜果长和果径发育调控的关系。
3.2 丝瓜果形控制潜在的候选基因及功能预测
共表达模块内具有高连通性的基因被视为核心基因,这些基因通常起重要调控作用[23,49]。本研究对Turquoise模块内连通度前10的基因进行功能注释,筛选出3个与果长和果径发育相关的调控基因,分别编码木葡聚糖内转葡糖基酶/水解酶XTH23、肌动蛋白解聚因子ADF2和伴侣蛋白DnaJ10。XTH是植物细胞壁重构过程中的关键酶,不仅能够松弛、降解细胞壁,而且能参与细胞壁的强化和合成[50]。有研究表明,XTH在调控黄瓜[51]、番茄[52]、柿子[53]、甜樱桃[54]等果实伸长膨大、成熟及软化过程中发挥着重要作用。本研究中,(Maker00007620)是Turquoise模块内连通性最高的基因。RT-qPCR显示丝瓜果实进入快速生长期(花后8 d)后,的表达量显著升高,花后10—15 d基因表达量较发育前期(花后-2 d至2 d)增加约33—48倍。可见,在调控丝瓜果长和果径发育过程中起着重要调控作用。ADF是植物细胞的结构、生长、发育和分化中必不可少的因素。水稻的和拟南芥的与花粉的形成和花粉管的伸长有关[55-56]。杨梅的ADF蛋白对果实的质地变化起重要作用[57]。Qiao等[58]发现玉米的异源过表达提高了转基因拟南芥种子的大小,推测该基因可能在玉米籽粒发育中发挥重要作用。本研究中,(Maker00033581)同样在丝瓜果实的快速生长期大量表达,花后10—15 d基因表达量较发育前期增加约7—11倍,提示该基因可能也与丝瓜果实的伸长及膨大相关。DnaJ是多种作物叶绿体的形成和分化过程中必不可少的重要因子[36]。果实叶绿体的丰度和功能直接影响着果实发育和品质。本研究中,(Maker00000726)的表达量在丝瓜果实的发育中期显著升高,推测该基因可能在该过程中调控果实叶绿素形成,而叶绿素作为同化器官合成大量同化物用于丝瓜果实发育。
此外,本研究还在Turquoise模块中筛选到7个已在葫芦科中报道过与果长和果径发育相关的基因,它们分别编码4个扩展蛋白Expansin、1个驱动蛋白Kinesin以及2个生长素反应蛋白SAUR和Aux/IAA。Expansin与XTH的功能相似,也在植物细胞壁的松弛与重构中发挥重要作用[35]。郝西等[59]鉴定了黄瓜扩展蛋白基因家族,发现该家族基因在黄瓜果实发育过程中表达丰度较高。刘昌[60]在甜瓜中克隆到,发现敲除该基因的T1代果实与野生型相比出现果实变小的表型,表明能促进甜瓜果实成长期的膨大生长。本研究筛选到4个(Maker00026031、Maker00000768、Maker00039481和Maker00009740),发现这4个基因的表达量在果实进入快速生长期(花后8 d)前基本不变,随后表达量均显著升高,表明它们在果实果长、果径的发育过程发挥重要作用。其中Maker00039481和Maker00009740编码的和在果实成熟期(花后10—15 d)出现第二次的高表达,推测它们在果实成熟过程也起着重要作用。研究发现植物具有明显的时空效应,有些的表达与果实发育、膨大相关,而有些是果实成熟软化的特异基因[61]。Kinesin参与细胞分裂、生长和形态发生等植物生长发育过程。有研究报道,在黄瓜[62]、西瓜[63]等果实发育早期的细胞分裂和细胞膨大过程中起重要作用。本研究中,(Maker00024997)的表达量随着丝瓜果实发育逐渐升高,并且在果实快速生长期显著升高,可见很可能参与丝瓜果实整个生长发育期的细胞快速分裂过程。生长素是植物最重要的激素之一。/和作为两类主要的生长素早期诱导基因在葫芦科果实发育调控中发挥关键作用[64]。袁静贤[65]从西瓜中鉴定到4个/与果实发育早期细胞分裂呈正相关,7个/与果实膨大呈正相关。于越等[66]前期通过EMS诱变方法获得黄瓜短果突变体,对其进行转录组分析发现一个关键的上调表达差异基因,该基因属于Aux/IAA家族。LUO等[67]在冬瓜果实中发现一个特异性高表达的,发现在拟南芥中过表达该基因会导致角果变长,由此推测该基因在冬瓜果长调控中起重要作用。本研究中,(Maker00038241)和(Maker00001372)的表达量均随着果实发育逐渐升高,并在果实快速生长期显著高表达,可见生长素及其响应基因对丝瓜果实发育过程具有十分重要的调节作用。通过对以上10个调控基因的功能分析,发现丝瓜果长和果径的发育调控主要涉及细胞壁的重构、细胞的发育与分化、生长素的调控等过程,同时可能也与叶绿素的形成和分化相关。与此同时,研究还发现绝大多数的调控基因与多种作物果实的成熟软化存在着密不可分的关系,因此,推测这些基因可能也参与丝瓜果实的成熟软化过程,但其具体调控方式还需要进一步深入研究。
4 结论
本研究基于转录组和WGCNA构建了丝瓜果长和果径发育相关基因共表达网络,共鉴定出14个共表达模块,获得了关键基因调控模块Turquoise模块,筛选到、、、、、、、和等10个调控基因可作为丝瓜果形控制潜在的候选基因。
[1] 李佳欣, 冯玉. 丝瓜不同部位药理作用研究进展. 食品工业科技, 2021, 42(10): 355-361.
LI J X, FENG Y. Research progress on pharmacological action of different parts of(L.) Roem. Science and Technology of Food Industry, 2021, 42(10): 355-361. (in Chinese)
[2] OBOH I O, ALUYOR E O.-an emerging cash crop. African Journal of Agricultural Research, 2009, 4(8): 684-688.
[3] 王青青, 王天文, 高安辉. 丝瓜种质资源与育种研究进展. 现代园艺, 2019(21): 33-35.
WANG Q Q, WANG T W, GAO A H. Research progress on germplasm resources and breeding of luffa. Xiandai Horticulture, 2019(21): 33-35. (in Chinese)
[4] 李大忠, 朱海生, 康玉妹, 温庆放, 李永平, 康建坂. 普通丝瓜新品种‘福研1号’选育. 亚热带植物科学, 2016, 45(1): 87-89.
LI D Z, ZHU H S, KANG Y M, WEN Q F, LI Y P, KANG J B. Breeding of a new sponge gourd cultivar ‘Fuyan 1’. Subtropical Plant Science, 2016, 45(1): 87-89. (in Chinese)
[5] 林奕韩, 陈捷凯, 林爱惜. 丝瓜新品种双丰1号肉丝瓜的选育. 广东农业科学, 2010, 37(7): 46-47.
LIN Y H, CHEN J K, LIN A X. Breeding of a new luffa variety Shuangfeng No.1 meat luffa. Guangdong Agricultural Sciences, 2010, 37(7): 46-47. (in Chinese)
[6] 王安乐, 朱海泉, 邓稳桥, 张望隆. 丝瓜新品种早优3号的选育. 中国蔬菜, 2007(11): 27-28.
WANG A L, ZHU H Q, DENG W Q, ZHANG W L. A new sponge gourd F1hybrid - ‘Zaoyou No. 3’. China Vegetables, 2007(11): 27-28. (in Chinese)
[7] 高军, 徐海, 苏小俊, 袁希汉. 普通丝瓜果长遗传规律分析. 江苏农业科学, 2007, 35(5): 123-125.
GAO J, XU H, SU X J, YUAN X H. Analysis on genetic rule of fruit length in luffa. Jiangsu Agricultural Sciences, 2007, 35(5): 123-125. (in Chinese)
[8] 林明宝, 林师森. 有棱丝瓜果长遗传效应的初步研究. 华南农业大学学报, 2000, 21(2): 8-9, 24.
LIN M B, LIN S S. Prelimary study on the heredity of fruit length of angular sponge gourd. Journal of South China Agricultural University, 2000, 21(2): 8-9, 24. (in Chinese)
[9] 苏小俊, 徐海, 高军, 郑子松, 宋波, 陈龙正, 袁希汉, 陈劲枫. 普通丝瓜果实性状的遗传分析. 江苏农业学报, 2009, 25(5)1112-1118.
SU X J, XU H, GAO J, ZHENG Z S, SONG B, CHEN L Z, YUAN X H, CHEN J F. Genetic analysis on fruit characters of luffa. Jiangsu Journal of Agricultural Sciences, 2009, 25(5)1112-1118. (in Chinese)
[10] 崔竣杰, 程蛟文, 谭澍, 李卫鹏, 汪国平, 胡开林. 丝瓜果长及果柄长的遗传规律分析. 广东农业科学, 2014, 41(8): 52-56.
CUI J J, CHENG J W, TAN S, LI W P, WANG G P, HU K L. Genetic analysis of fruit length and fruit stalk length in luffa. Guangdong Agricultural Sciences, 2014, 41(8): 52-56. (in Chinese)
[11] 崔竣杰, 吕振, 杨天文, 王静, 洪宇, 曹毅. 丝瓜高密度bin标记遗传图谱构建与果长QTL定位. 浙江农业学报, 2023, 35(3): 590-597.
CUI J J, LÜ Z, YANG T W, WANG J, HONG Y, CAO Y. Construction of high-density bin marker genetic map and QTL mapping for fruit length inspp. Acta Agriculturae Zhejiangensis, 2023, 35(3): 590-597. (in Chinese)
[12] 曾美娟, 刘建汀, 李祖亮, 陈敏氡, 叶新如, 王彬, 朱海生, 温庆放. 普通丝瓜基因家族全基因组鉴定及表达分析. 江苏农业科学, 2022, 50(18): 82-87.
ZENG M J, LIU J T, LI Z L, CHEN M D, YE X R, WANG B, ZHU H S, WEN Q F. Genome-wide identification and expression analysis ofgene family in. Jiangsu Agricultural Sciences, 2022, 50(18): 82-87. (in Chinese)
[13] 曾美娟, 陈敏氡, 刘建汀, 叶新如, 王彬, 林锦辉, 朱海生, 温庆放. 普通丝瓜基因的克隆及表达分析. 中国细胞生物学学报, 2022, 44(6): 1031-1038.
ZENG M J, CHEN M D, LIU J T, YE X R, WANG B, LIN J H, ZHU H S, WEN Q F. Cloning and expression analysis ofgene from. Chinese Journal of Cell Biology, 2022, 44(6): 1031-1038. (in Chinese)
[14] ZHANG B, HORVATH S. A general framework for weighted gene co-expression network analysis. Statistical Applications in Genetics and Molecular Biology, 2005, 4: 17.
[15] 冯贵芝. 基于时空转录组的脐橙果实发育和成熟调控机理的研究[D]. 武汉: 华中农业大学, 2021.
FENG G Z. Study on the regulation mechanism of navel orange fruit development and ripening based on spatio-temporal transcriptome [D]. Wuhan: Huazhong Agricultural University, 2021. (in Chinese)
[16] 李情. 黄瓜果实早期发育中基因的时空表达与调控[D]. 北京: 中国农业科学院, 2019.
LI Q. Temporal and spatial expression and regulation of genes in early development of cucumber fruit [D]. Beijing: Chinese Academy of Agricultural Sciences, 2019. (in Chinese)
[17] 孙蕾. 西瓜中心果肉硬度主效QTL及候选基因分析[D]. 哈尔滨: 东北农业大学, 2021.
SUN L. Analysis of major QTL and candidate genes for firmness of watermelon central pulp [D]. Harbin: Northeast Agricultural University, 2021. (in Chinese)
[18] TRAPNELL C, ROBERTS A, GOFF L, PERTEA G, KIM D, KELLEY D R, PIMENTEL H, SALZBERG S L, RINN J L, PACHTER L. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature Protocols, 2012, 7(3): 562-578.
[19] LANGFELDER P, HORVATH S. WGCNA: An R package for weighted correlation network analysis. BMC Bioinformatics, 2008, 9: 559.
[20] 马娟, 曹言勇, 王利锋, 李晶晶, 王浩, 范艳萍, 李会勇. 利用WGCNA鉴定玉米株高和穗位高基因共表达模块. 作物学报, 2020, 46(3): 385-394.
MA J, CAO Y Y, WANG L F, LI J J, WANG H, FAN Y P, LI H Y. Identification of gene co-expression modules of maize plant height and ear height by WGCNA. Acta Agronomica Sinica, 2020, 46(3): 385-394. (in Chinese)
[21] 周雨青, 杨永飞, 葛常伟, 沈倩, 张思平, 刘绍东, 马慧娟, 陈静, 刘瑞华, 李士丛, 赵新华, 李存东, 庞朝友. 基于WGCNA的棉花子叶抗冷相关共表达模块鉴定. 中国农业科技导报, 2022, 24(4): 52-62.
ZHOU Y Q, YANG Y F, GE C W, SHEN Q, ZHANG S P, LIU S D, MA H J, CHEN J, LIU R H, LI S C, ZHAO X H, LI C D, PANG C Y. Identification of cold-related co-expression modules in cotton cotyledon by WGCNA. Journal of Agricultural Science and Technology, 2022, 24(4): 52-62. (in Chinese)
[22] 王泽涵, 于文涛, 王鹏杰, 刘财国, 樊晓静, 谷梦雅, 蔡春平, 王攀, 叶乃兴. 茶树秃房与茸房种质花器官差异表达基因的WGCNA分析. 园艺学报, 2023, 50(3): 620-634.
WANG Z H, YU W T, WANG P J, LIU C G, FAN X J, GU M Y, CAI C P, WANG P, YE N X. WGCNA analysis of differentially expressed genes in floral organs of tea germplasms with ovary-glabrous and ovary-trichome. Acta Horticulturae Sinica, 2023, 50(3): 620-634. (in Chinese)
[23] 郭永春, 王鹏杰, 金珊, 侯炳豪, 王淑燕, 赵峰, 叶乃兴. 基于WGCNA鉴定茶树响应草甘膦相关的基因共表达模块. 中国农业科学, 2022, 55(1): 152-166. doi: 10.3864/j.issn.0578-1752.2022. 01.013.
GUO Y C, WANG P J, JIN S, HOU B H, WANG S Y, ZHAO F, YE N X. Identification of co-expression gene related to tea plant response to glyphosate based on WGCNA. Scientia Agricultura Sinica, 2022, 55(1): 152-166. doi: 10.3864/j.issn.0578-1752.2022.01.013. (in Chinese)
[24] ERNST J, BAR-JOSEPH Z. STEM: A tool for the analysis of short time series gene expression data. BMC Bioinformatics, 2006, 7(1): 191.
[25] 荐红举, 张梅花, 尚丽娜, 王季春, 胡柏耿, Vadim Khassanov, 吕典秋. 利用WGCNA筛选马铃薯块茎发育候选基因. 作物学报, 2022, 48(7): 1658-1668.
JIAN H J, ZHANG M H, SHANG L N, WANG J C, HU B G, KHASSANOV V, LÜ D Q. Screening candidate genes involved in potato tuber development using WGCNA. Acta Agronomica Sinica, 2022, 48(7): 1658-1668. (in Chinese)
[26] 朱海生, 陈敏氡, 温庆放, 蓝新隆, 李永平, 王彬, 张前荣, 吴卫东. 丝瓜基因克隆及其作为内参基因的应用. 核农学报, 2016, 30(1): 35-41.
ZHU H S, CHEN M D, WEN Q F, LAN X L, LI Y P, WANG B, ZHANG Q R, WU W D. Cloning ofgene fromand its application as an internal standard. Journal of Nuclear Agricultural Sciences, 2016, 30(1): 35-41. (in Chinese)
[27] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCTmethod. Methods, 2001, 25(4): 402-408.
[28] 施一公. 剪接体的结构与分子机理研究. 科学通报, 2020, 65(27): 2952-2953.
SHI Y G. Structural and mechanistic investigations of the spliceosome. Chinese Science Bulletin, 2020, 65(27): 2952-2953. (in Chinese)
[29] HEINTZ C, DOKTOR T K, LANJUIN A, ESCOUBAS C, ZHANG Y, WEIR H J, DUTTA S, SILVA-GARCÍA C G, BRUUN G H, MORANTTE I, HOXHAJ G, MANNING B D, ANDRESEN B S, MAIR W B. Splicing factor 1 modulates dietary restriction and TORC1 pathway longevity in. Nature, 2017, 541(7635): 102-106.
[30] DÜNSER K, SCHÖLLER M, RÖßLING A K, LÖFKE C, XIAO N N, PAŘÍZKOVÁ B, MELNIK S, RODRIGUEZ-FRANCO M, STÖGER E, NOVÁK O, KLEINE-VEHN J. Endocytic trafficking promotes vacuolar enlargements for fast cell expansion rates in plants. eLife, 2022, 11: e75945.
[31] 于明革, 杨洪强, 翟衡. 植物木质素及其生理学功能. 山东农业大学学报(自然科学版), 2003, 34(1): 124-128.
YU M G, YANG H Q, ZHAI H. Lignin and physiological function in plant. Journal of Shandong Agricultural University (Natural Science Edition), 2003, 34(1): 124-128. (in Chinese)
[32] 肖玫, 赵旭琴, 许迎科, 李汉兵. 磷脂酰肌醇信号在GLUT4囊泡转运中的调控作用. 中国细胞生物学学报, 2018, 40(1): 139-145.
XIAO M, ZHAO X Q, XU Y K, LI H B. The regulatory role of phosphatidylinositol signaling in GLUT4 vesicle trafficking. Chinese Journal of Cell Biology, 2018, 40(1): 139-145. (in Chinese)
[33] 张载励, 丁晨, 王爽, 梁明朗, 李涛, 苏来曼·买买提, 胡康棣. 番茄、拟南芥和葡萄中同型半胱氨酸S-甲基转移酶基因家族功能研究. 现代农业科技, 2023(3): 66-71.
ZHANG Z L, DING C, WANG S, LIANG M L, LI T, SULAIMAN MAIAITI, HU K D. Study on function of homocysteine S-methyltransferase gene family in tomato,and grape. Modern Agricultural Science and Technology, 2023(3): 66-71. (in Chinese)
[34] 何乐平, 张蕾. 植物钙依赖性蛋白激酶及其相关蛋白激酶(CDPKs/CRKs)的研究进展. 安徽农业科学, 2020, 48(18): 26-30, 51.
HE L P, ZHANG L. Research progress of plant calcium-dependent protein kinases(CDPKs) and CDPK-related kinases(CRKs). Journal of Anhui Agricultural Sciences, 2020, 48(18): 26-30, 51. (in Chinese)
[35] 贾鑫磊, 何贝轩, 郭丹丹, 郭美丽. 膨胀素和木葡聚糖内转葡糖基酶/水解酶基因的功能研究进展. 植物生理学报, 2018, 54(11): 1659-1668.
JIA X L, HE B X, GUO D D, GUO M L. Research progress in the function of expansins and xyloglucan endotransglucosylase/hydrolase. China Industrial Economics, 2018, 54(11): 1659-1668. (in Chinese)
[36] 王楠, 吕香玲, 李亮, 吴金凤, 尚佳微, 郝转芳, 李凤海. 植物肌动蛋白解聚因子ADF的研究进展. 西北植物学报, 2015, 35(11): 2349-2354.
WANG N, LÜ X L, LI L, WU J F, SHANG J W, HAO Z F, LI F H. Research progress of ADF/cofilin in plant. Acta Botanica Boreali-Occidentalia Sinica, 2015, 35(11): 2349-2354. (in Chinese)
[37] 樊芳菲, 杨暹, 康云艳, 柴喜荣, 蒙林平. 植物DnaJ蛋白的研究进展. 分子植物育种, 2018, 16(6): 2028-2034.
FAN F F, YANG X, KANG Y Y, CHAI X R, MENG L P. Research progress on DnaJ proteins in plants. Molecular Plant Breeding, 2018, 16(6): 2028-2034. (in Chinese)
[38] YAN L, XU C H, KANG Y L, GU T W, WANG D X, ZHAO S Y, XIA G M. The heterologous expression inof sorghum transcription factor SbbHLH1 downregulates lignin synthesis. Journal of Experimental Botany, 2013, 64(10): 3021-3032.
[39] WANG H Z, AVCI U, NAKASHIMA J, HAHN M G, CHEN F, DIXON R A. Mutation of WRKY transcription factors initiates pith secondary wall formation and increases stem biomass in dicotyledonous plants. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(51): 22338-22343.
[40] 王依依. 杨树、葡萄、拟南芥和水稻WRKY Ⅲ基因家族比较及表达分析[D]. 合肥: 安徽农业大学, 2016.
WANG Y Y. Comparison and expression analysis of WRKY Ⅲ gene family in poplar, grape,and rice [D]. Hefei: Anhui Agricultural University, 2016. (in Chinese)
[41] HAMEED SABA. 西瓜热激转录因子家族基因的鉴定及其在果实发育过程中的表达分析[D]. 武汉: 华中农业大学, 2019.
HAMEED S. Identification of heat shock transcription factor family genes in watermelon and their expression analysis during fruit development [D]. Wuhan: Huazhong Agricultural University, 2019. (in Chinese)
[42] 王华容. 番茄热激转录因子SlHSFC1过表达导致转基因植株矮化的机理研究[D]. 重庆: 西南大学, 2023.
WANG H R. Study on the mechanism of dwarfing transgenic plants caused by overexpression of tomato heat shock transcription factor SlHSFC1 [D]. Chongqing: Southwest University, 2023. (in Chinese)
[43] WANG R C, SHU P, ZHANG C, ZHANG J L, CHEN Y, ZHANG Y X, DU K, XIE Y, LI M Z, MA T, ZHANG Y, LI Z G, GRIERSON D, PIRRELLO J, CHEN K S, BOUZAYEN M, ZHANG B, LIU M C. Integrative analyses of metabolome and genome-wide transcriptome reveal the regulatory network governing flavor formation in kiwifruit (). The New Phytologist, 2022, 233(1): 373-389.
[44] CHEN L, TIAN N, HU M Q, SANDHU D, JIN Q F, GU M Y, ZHANG X Q, PENG Y, ZHANG J L, CHEN Z Y, LIU G Z, HUANG M D, HUANG J N, LIU Z H, LIU S Q. Comparative transcriptome analysis reveals key pathways and genes involved in trichome development in tea plant (). Frontiers in Plant Science, 2022, 13: 997778.
[45] CHEN Z Y, LIAO M L, YANG Z J, CHEN W Y, WEI S H, ZOU J, PENG Z S. Co-expression network analysis of genes and networks associated with wheat pistillody. PeerJ, 2022, 10: e13902.
[46] 晏巢, 姚小华, 殷恒福, 王开良, 尹润富, 滕建华. 油茶果实生长发育动态和木质素积累规律. 江西农业大学学报, 2020, 42(4): 788-801.
YAN C, YAO X H, YIN H F, WANG K L, YIN R F, TENG J H. Fruit development dynamics and lignin accumulation law of oil tea. Acta Agriculturae Universitatis Jiangxiensis (Natural Sciences Edition), 2020, 42(4): 788-801. (in Chinese)
[47] 朱海英, 李人圭, 王隆华, 黄祥辉, 颜季琼. 丝瓜果实发育中木质素代谢及有关导管分化的生理生化研究. 华东师范大学学报(自然科学版), 1997(1): 87-94.
ZHU H Y, LI R G, WANG L H, HUANG X H, YAN J Q. Physiological and biochcmical studies on lignin mctabolism and its treachary element differentiation of fruit of. Journal of East China Normal University (Natural Science), 1997(1): 87-94. (in Chinese)
[48] 陈敏氡, 王彬, 朱海生, 李永平, 刘建汀, 温庆放. 丝瓜果实发育过程的蛋白质差异表达分析. 中国细胞生物学学报, 2018, 40(11): 1806-1818.
CHEN M D, WANG B, ZHU H S, LI Y P, LIU J T, WEN Q F. Proteomic analysis of differentially expressed proteins induring fruit development. China Industrial Economics, 2018, 40(11): 1806-1818. (in Chinese)
[49] 彭佳伟, 张叶, 寇单单, 杨丽, 刘晓飞, 张学英, 陈海江, 田义. ‘仓方早生’桃及其早熟芽变不同发育时期果实的转录组分析. 中国农业科学, 2023, 56(5): 964-980. doi:10.3864/j.issn.0578-1752.2023.05. 012.
PENG J W, ZHANG Y, KOU D D, YANG L, LIU X F, ZHANG X Y, CHEN H J, TIAN Y. Transcriptome analysis of peach fruits at different developmental stages in peach kurakato wase and early-ripening mutant. Scientia Agricultura Sinica, 2023, 56(5): 964-980. doi:10.3864/j.issn.0578-1752.2023.05.012. (in Chinese)
[50] 杜丽萍, 沈昕, 陈少良, 胡赞民. 细胞壁重构关键酶木葡聚糖内转糖苷酶/水解酶(XTH)的研究进展. 农业生物技术学报, 2010, 18(3): 604-609.
DU L P, SHEN X, CHEN S L, HU Z M. Research advances on a key cell wall remodeling enzyme xyloglucan endotransglucosylase/ hydrolase(XTH). Journal of Agricultural Biotechnology, 2010, 18(3): 604-609. (in Chinese)
[51] 傅丰庆. 黄瓜果实发育调控的分子机理研究[D]. 杭州: 浙江大学, 2008.
FU F Q. Study on molecular mechanism of cucumber fruit development regulation [D]. Hangzhou: Zhejiang University, 2008. (in Chinese)
[52] TSUCHIYA M, SATOH S, IWAI H. Distribution of XTH, expansin, and secondary-wall-related CesA in floral and fruit abscission zones during fruit development in tomato (). Frontiers in Plant Science, 2015, 6: 323.
[53] HAN Y, ZHU Q G, ZHANG Z K, MENG K, HOU Y L, BAN Q Y, SUO J T, RAO J P. Analysis of xyloglucan endotransglycosylase/ hydrolase () genes and diverse roles of isoenzymes during persimmon fruit development and postharvest softening. PLoS One, 2015, 10(4): e0123668.
[54] ZHAI Z F, FENG C, WANG Y Y, SUN Y T, PENG X, XIAO Y Q, ZHANG X, ZHOU X, JIAO J L, WANG W L, DU B Y, WANG C, LIU Y, LI T H. Genome-wide identification of the xyloglucan endotransglucosylase/hydrolase (XTH) and polygalacturonase (PG) genes and characterization of their role in fruit softening of sweet cherry. International Journal of Molecular Sciences, 2021, 22(22): 12331.
[55] LI L, LI Y X, SONG S F, DENG H F, LI N, FU X Q, CHEN G H, YUAN L P. An anther development F-box (ADF) protein regulated by tapetum degeneration retardation (TDR) controls rice anther development.Planta, 2015, 241(1): 157-166.
[56] 彭世清, 黄冬芬. 拟南芥肌动蛋白解聚因子4基因()在烟草中表达引起植株形态变化. 植物生理与分子生物学学报, 2006, 32(1): 52-56.
PENG S Q, HUANG D F. Expression of anactin- depolymerizing factor 4 gene () in tobacco causes morphological change of plants. Journal of Plant Physiology and Molecular Biology, 2006, 32(1): 52-56. (in Chinese)
[57] 陈义勇. 不同发育时期杨梅果肉差异表达蛋白及其功能研究[D]. 福州: 福建农林大学, 2014.
CHEN Y Y. Study on differentially expressed proteins inpulp at different developmental stages and their functions[D]. Fuzhou: Fujian Agriculture and Forestry University, 2014. (in Chinese)
[58] QIAO D H, DONG Y B, ZHANG L, ZHOU Q, HU C H, REN Y L, LI Y L. Ectopic expression of the maizeZmADFgene inrevealing its functions in kernel development. Plant Cell, Tissue and Organ Culture, 2016, 126(2): 239-253.
[59] 郝西, 理向阳, 腊贵晓, 代丹丹, 杨铁钢. 黄瓜扩展蛋白基因家族的鉴定与生物信息学分析. 分子植物育种, 2015, 13(10): 2280-2289.
HAO X, LI X Y, LA G X, DAI D D, YANG T G. Identification and bioinformatic analysis of the expansin gene family in cucumber. Molecular Plant Breeding, 2015, 13(10): 2280-2289. (in Chinese)
[60] 刘昌. 甜瓜扩展蛋白基因家族成员的鉴定及CmEXPA8和CmEXPA12基因在果实发育中功能的初步分析[D]. 呼和浩特: 内蒙古大学, 2018.
LIU C. Identification of members of melon expansin gene family and preliminary analysis of the functions of CmEXPA8 and CmEXPA12 genes in fruit development [D]. Hohhot: Inner Mongolia University, 2018. (in Chinese)
[61] 张安, 曹清河, 周志林, 赵冬兰, 李昂, 李元元, 唐君. 植物细胞壁松弛因子: Expansin研究进展. 江苏农业科学, 2013, 41(6): 11-13.
ZHANG A, CAO Q H, ZHOU Z L, ZHAO D L, LI A, LI Y Y, TANG J. Research progress of plant cell wall relaxation factor-Expansin. Jiangsu Agricultural Sciences, 2013, 41(6): 11-13. (in Chinese)
[62] YANG X Y, WANG Y, JIANG W J, LIU X L, ZHANG X M, YU H J, HUANG S W, LIU G Q. Characterization and expression profiling of cucumber kinesin genes during early fruit development: Revealing the roles of kinesins in exponential cell production and enlargement in cucumber fruit. Journal of Experimental Botany, 2013, 64(14): 4541-4557.
[63] TIAN S J, JIANG J, XU G Q, WANG T, LIU Q Y, CHEN X E, LIU M, YUAN L. Genome wide analysis of kinesin gene family inreveals an essential role in early fruit development. BMC Plant Biology, 2021, 21(1): 210.
[64] 李艳艳, 齐艳华. 植物Aux/IAA基因家族生物学功能研究进展. 植物学报, 2022, 57(1): 30-41.
LI Y Y, QI Y H. Advances in biological functions of Aux/IAA gene family in plants. Bulletin of Botany, 2022, 57(1): 30-41. (in Chinese)
[65] 袁静贤. 生长素相关转录因子家族基因在西瓜果实膨大过程中的调控作用分析[D]. 武汉: 华中农业大学, 2015.
YUAN J X. Regulation of auxin-related transcription factor family genes in watermelon fruit expansion [D]. Wuhan: Huazhong Agricultural University, 2015. (in Chinese)
[66] 于越, 陈海燕, 周龙, 郭冬雪, 宫思宇, 郝宁, 杜亚琳, 武涛. 黄瓜果实发育相关基因CsIAA29的克隆与表达分析. 华北农学报, 2020, 35(S1): 11-17.
YU Y, CHEN H Y, ZHOU L, GUO D X, GONG S Y, HAO N, DU Y L, WU T. Cloning and expression analysis of CsIAA29 gene related to fruit development in cucumber. Acta Agriculturae Boreali-Sinica, 2020, 35(S1): 11-17. (in Chinese)
[67] LUO C, YAN J Q, HE C X, LIU W R, XIE D S, JIANG B. Genome-wide identification of the SAUR gene family in wax gourd () and functional characterization of BhSAUR60 during fruit development. International Journal of Molecular Sciences, 2022, 23(22): 14021.
Screening Regulatory Genes Related to Luffa Fruit Length and Diameter Development Based on Transcriptome and WGCNA
CHEN MinDong, WANG Bin, LIU JianTing, LI YongPing, BAI ChangHui, YE XinRu, QIU BoYin, WEN QingFang, ZHU HaiSheng
Fujian Key Laboratory of Vegetable Genetics and Breeding/Fujian Engineering Research Center for Vegetables/Crops Research Institute, Fujian Academy of Agricultural Sciences, Fuzhou 350013
【Objective】 The aim of this study was to identify the co expression modules of luffa fruit length and diameter development and to screen key regulatory genes, so as to provide the theoretical basis for subsequent research on the molecular mechanism of fruit shape control in luffa. 【Method】 The luffa fruits in 9 fruit development stages (2 days before anthesis, and 0, 2, 4, 6, 8, 10, 15, and 20 days after anthesis) were applied as research materials. The fruit length and diameter of each stage were measured. The WGCNA method was used to jointly analyze transcriptome and fruit length and diameter data, to identify co-expressed gene modules of fruit length and diameter development, and to screen out key regulatory genes.【Result】A total of 14 co expression modules were identified by WGCNA, among which two modules (Turquoise and Lightpink4) were significantly correlated with fruit length and diameter (absolute value of correlation coefficient=0.9); Turquoise module was significantly positively correlated, while Lightpink4 module was significantly negatively correlated. KEGG enrichment analysis found that the Turquoise module was significantly enriched in endocytosis and phenylpropanoid biosynthesis pathways, which were closely related to fruit enlargement and growth regulation, and could be used as a key gene module for studying fruit length and diameter in luffa. According to the connectivity and functional annotation of genes in Turquoise module, ten key regulatory genes were screened, including xyloglucan endotransglucosylase/hydrolase gene, actin-depolymerizing factor gene, chaperone protein gene, expansin gene (,and), kinesin gene, auxin response genes, and. The RT-qPCR results showed that the expression levels of ten regulatory genes significantly increased after the fruit entered the rapid growth period (8 day after anthesis), with an increase of 2-50 times approximately. Through constructing a gene interaction network, it was found that some candidate genes interacted with the,, andtranscription factor families.【Conclusion】The Turquoise module, an important co expression module of luffa fruit length and diameter was obtained, and ten potential candidate genes for luffa fruit shape control were screened. It was found that luffa fruit length and diameter development regulation mainly involved the processes of cell wall reconstruction, cell development and differentiation, and auxin regulation.
transcriptome; WGCNA;L.; fruit length; fruit diameter; regulatory genes

10.3864/j.issn.0578-1752.2023.22.012
2023-04-10;
2023-09-05
福建省自然科学基金(2021J01494)、福建省农业科学院英才项目(YC2021004)、福建省农业科学院科技创新团队项目(CXTD2021003-1)、国家大宗蔬菜产业技术体系福州综合试验站项目(CARS-23-G51)、福建省种业创新与产业化工程项目(zycxny2021009)、福建省农业高质量发展超越“5511”协同创新工程项目(XTCXGC2021003)
陈敏氡,E-mail:1053870789@qq.com。通信作者朱海生,E-mail:zhs0246@163.com。通信作者温庆放,E-mail:fjvrc@163.com
(责任编辑 赵伶俐)
