甘蓝花叶病病毒种类鉴定及BrYV遗传变异分析
2023-12-20仲健新李建国武崇高焦泰至何志兰陈雅寒杨成德
仲健新 李建国 武崇高 焦泰至 何志兰 陈雅寒 杨成德


摘 要 为了明确引起甘蓝花叶病的病毒种类,利用小干扰RNA(small interfering RNA,siRNA)高通量测序技术,并结合分子生物学方法查找甘蓝样品中的病毒序列,发现存在芜菁花叶病毒(turnip mosaic virus,TuMV)、黄瓜花叶病毒(cucumber mosaic virus,CMV)、蚕豆萎蔫病毒2(broad bean wilt virus2,BBWV2)、烟草花叶病毒(tobacco mosaic virus,TMV)和芸薹黄化病毒(brassica yellows virus,BrYV)等5种病毒。采用RT-PCR技术进行验证,结果显示样品检测到BrYV、TuMV和CMV共3种病毒。对获得的BrYV外壳蛋白基因(coat protein,cp)进行测序和系统进化分析,获得1条576 bp的BrYV cp基因全长序列,与GenBank中BrYV分离物cp基因核苷酸序列同源率为88.0%~96.4%,氨基酸序列同源率为90.1%~95.8%。基于cp基因序列構建系统发育树,BrYV-GL为BrYV-C基因型,与中国内蒙古油菜BrYV(GenBank登录号EF079907)分离物cp基因序列聚集在一起,亲缘关系较近。BrYV cp基因共检测到4个潜在重组事件,可为后续甘蓝花叶病的研究和防治提供参考。
关键词 甘蓝;芸薹黄化病毒(BrYV);外壳蛋白(CP);RT-PCR
甘蓝(Brassica oleracea L.var. capitata L.)是十字花科芸薹属植物,叶球中含有丰富的营养物质,具有产量高、对生长环境适应性和抗逆性强等特点,在中国各蔬菜种植区广泛分布[1-2]。2018年全国种植面积约98.3万 hm 占世界的41%,产量3 384万 t,占世界的49%(FAO)。近年来,由于种植规模的不断扩大,甘蓝病毒病发生严重,成为制约产业发展的主要因素之一[3]。据报道,侵染甘蓝的病毒主要有:芜菁花叶病毒(turnip mosaic virus,TuMV)、芜菁黄化病毒(turnip yellows virus,TuYV)、黄瓜花叶病毒(cucumber mosaic virus,CMV)、甘蓝曲叶病毒(cabbage leaf curl virus,CaLCuV)、芸薹黄化病毒(brassica yellows virus,BrYV)、花椰菜花叶病毒(cauliflower mosaic virus,CaMV)、烟草花叶病毒(tobacco mosaic virus,TMV)、芜菁黄花叶病毒(turnip yellow mosaic virus,TYMV)、蚕豆萎蔫病毒2(broad bean wilt virus2,BBWV2)和长叶车前花叶病毒(ribgrass mosaic virus,RMV)等,萝卜隐状病毒2(raphanus sativus cryptic virus2,RsCV2)和莴苣花叶病毒(lettuce mosaic virus,LMV)只在甘蓝种子中被检测到[4-14]。其中芸薹黄化病毒(brassica yellows virus,BrYV)属于马铃薯卷叶病毒属(Polerovirus),黄症病毒科(Luteoviridae),是一种新发现的多角体病毒,2015年首次在中国烟草中被发现和鉴定,可自然侵染豇豆、草莓和烟草等多种植物,引起黄化和卷叶症状[15-17]。BrYV基因组由一个正义单链RNA组成,包含7个开放阅读框(ORF0,ORF1,ORF2,ORF3a,ORF3,ORF4和ORF5),其中P3是其外壳蛋白基因(coat protein,cp)[15,18-20]。根据序列比较和系统发育分析,已经鉴定到3种BrYV基因型(BrYV-A、BrYV-B和BrYV-C)[21]。
小干扰RNA(small interfering RNA,siRNA)深度测序技术于2009年被Jan和Ana等首次提出并在被病毒感染的甘薯中进行应用,该方法可以作为一种常规手段来检测各类DNA或RNA病毒[22]。目前,siRNA已成为新病毒检测的强有力工具,在未知病毒种类、序列以及特征信息等情况下,通过高通量测序结合生物信息学分析,获得病毒的基因组序列[23]。可以通过组装来快速检测和发现病毒,同时该方法具有适应性强、灵敏度高和适应范围广等特点,对病毒信息的了解更加全面,从而有助于未知病毒种类的发现[24]。目前siRNA测序技术在植物病毒鉴定中应用广泛,如陈雅寒等[25]在表现萎黄症状的杏树样品中发现存在亚洲李属病毒1(asian prunus virus1,APV1)和亚洲李属病毒3(asian prunus virus3,APV3);龚明霞等[26]在广西辣椒病毒病样中发现10种病毒,其中小米椒内源RNA病毒1(capsicum frutescens endornavirus1,CFEV1)、辣椒潜隐病毒2(pepper cryptic virus2,PCV2)、烟草轻型绿花叶病毒(tobacco mild green mosaic virus,TMGMV)和辣椒黄脉病毒(pepper vein yellows virus,PeVYV)在广西辣椒上为首次发现,CFEV1在中国为首次发现。
本研究中利用siRNA技术对甘肃省兰州市临洮县采集到的2株甘蓝样品叶片进行深度测序,通过生物信息学方法进行病毒序列分析,然后采用RT-PCR方法进一步对检测结果进行验证,并对甘蓝BrYV分离物的cp全长序列进行扩增和遗传进化分析,旨在进一步研究其发生及危害情况,为抗BrYV甘蓝蔬菜品种选育和制定BrYV的防控措施提供依据。
1 材料与方法
1.1 试验材料
1.1.1 样品采集
2020年7月在甘肃省临洮县采集到2株表现为花叶和皱缩等疑似病毒病的甘蓝样品,放入密封袋中,液氮速冻后于-80 ℃冰箱保存,备用。
1.1.2 主要试剂及设备 主要试剂:Omega Plant RNA Kit(R6827)、TaKaRa Prime ScriptTMRT reagent Kit(Perfect Real Time)、TaKaRa Ex Taq○RDNA Polymerase和TaKaRa Mini BEST Agarose GEL DNA Extraction Kit试剂盒等。
主要设备:Centrisart○RD-16C通用台式离心机(sartorius,德国)、iBrightTM 1000系列成像系统(Invitrogen,美国)、2010 Geno/Grinder高通量动植物组研磨机(SPEX,美国)、Thermal Cycler PCR仪(MiniAmp,美国)和Unano-2000微量核酸分析仪(UMI,中国)等。
1.2 试验方法
1.2.1 样品总RNA提取及siRNA测序 按照Plant RNA Kit(R6827)說明书进行病叶总RNA提取,提取的RNA用35 μL RNase Free ddH2O溶解,利用Unano-2000微量核酸分析仪测定RNA浓度,RNA样品于-80 ℃保存,备用。
RNA送至北京百迈客生物科技有限公司完成siRNA的分离、高通量测序以及cDNA文库的构建,测序平台为Illumina Hiseq 2500。
1.2.2 样品病毒序列分析 对测序后获得的原始reads进行过滤,去除测序获得片段的接头和低质量的序列,选取长度18~26 bp的sRNA,利用Bowtie 软件与 GenBank Virus RefSeq核酸数据库比对,初步鉴定甘蓝样品感染病毒的情况。使用Vevet 软件对筛选后的有效sRNA进行序列组装拼接获得contigs,利用NCBI Nr(non-redundant protein sequences)、NCBI Nt(non-redundant nucleotide sequences)、GenBank Virus RefSeq Nucleotide和GenBank Virus RefSeq Protein等数据库对获得的contigs进行比对和分类注释,筛选和鉴定引起甘蓝花叶病的病毒种类。采用Blast算法比对,设定阈值e-value=1×10-5[27]。
1.2.3 样品病毒RT-PCR检测 cDNA合成:取1 μg RNA作为模板,加入1 μL Random 6 mars,1 μL dNTP Mixture,用RNase Free ddH2O补足至10 μL,65 ℃ 5 min;冰上迅速冷却,后依次加入5×Prime ScriptⅡ Buffer 4 μL、RNase Inhibitor 0.5 μL、Prime ScriptⅡ RNase 1 μL、RNase Free ddH2O 4.5 μL,总体积20 μL;30 ℃ 10 min,95 ℃ 5 min,冰上冷却。将所得cDNA于-20 ℃保存,备用。
用于以上病毒检测的5对特异性引物见表1。所用引物均由西安擎科生物科技有限责任公司合成。RT-PCR反应体系为:cDNA产物 2 μL、10×Ex Taq Buffer 2.5 μL、dNTP Mix 2.0 μL、10 μmol/L上下游引物各1 μL、Ex Taq酶 0.2 μL、用ddH2O补足至25 μL。反应条件为: 94 ℃变性3 min,然后是94 ℃ 30 s,X ℃退火 30 s(X值取自表1“退火温度”),72 ℃延伸 1 min,共35个循环,最后 72 ℃ 10 min。所得PCR产物中加入4.2 μL 6×Loading Buffer,经2%琼脂糖凝胶电泳,于紫外灯下观察电泳结果并照相记录。
1.2.4 BrYV cp全长序列克隆测序 将上述步骤扩增的BrYV cp基因序列经琼脂糖凝胶电泳检测合格后,使用DNA回收试剂盒(TaKaRa)对产物进行切胶回收。纯化产物与载体pMD19-T Vector(TaKaRa)连接,并转化至大肠杆菌DH5α感受态细胞,均匀涂布于含有50 μg/mL Amp抗性的LB固体培养基,置于37 ℃培养箱中过夜培养。挑取单克隆菌落过夜摇菌,菌落PCR筛选阳性克隆,鉴定为阳性的菌液提取质粒。质粒测序由西安擎科生物科技有限责任公司完成。
1.2.5 BrYV cp序列系统发育和同源性分析 测序后获得的原始序列采用DNAMAN 6.0软件进行校正和拼接,将得到的BrYV cp全长基因序列提交NCBI核酸数据库进行BLASTn(http:∥www.ncbi.nlm.nih.gov/BLAST/)序列比对分析。采用Vector NTI Advance11(Thermo Fisher Scientific,USA)和SDT软件分析序列间核苷酸和氨基酸同源性,利用CLUSTAL W算法比对序列[30];采用MEGA 7.0以邻接法构建系统发育进化树,自展校正值设置为1 000,显示系统进化树的阈值为50%。
1.2.6 BrYV cp 序列重组分析 采用RDP4软件进行重组分析,利用RDP、BOOTSCAN、MACCHI、GENECONV、CHIMAERA、3SEQ和SISCAN等7种方法对获得的BrYV分离物cp基因全长序列与从NCBI数据库中获得的14条BrYV cp基因核苷酸序列进行重组检测分析,确定分离物的重组来源,分子类型选择线性。参考Dhir等[31]的方法,以P≤0.05为标准,若序列被3种以上方法同时检测到为有意义的重组事件;若序列被3种以下方法或者P>0.05检测到可看作为假定的潜在重组事件。
2 结果与分析
2.1 样品sRNAs测序结果
将甘蓝叶片混合样品提取总RNA,sRNAs进行测序,获得12 568 877条原始数据,将原始数据中低质量的reads、N含量超过10%的reads、去掉接头后过长或过短的reads过滤去除掉,将sRNAs片段在18~26 bp的提取出来,共获得 4 879 886条sRNA。将所得sRNAs与GenBank Virus RefSeq核酸数据库进行比对。其中 1 566 293条sRNAs与BrYV相匹配,占匹配病毒总 sRNAs数的32.09%;1 461 095条sRNAs与TuMV相匹配,占匹配病毒总sRNAs数的 29.94%;798 143条sRNAs与CMV相匹配,占匹配病毒总sRNAs数的15.36%;557 109条 sRNAs与BBWV 2相匹配,占匹配病毒总sRNAs数的11.42%;349 867条sRNAs与TMV相匹配,占匹配病毒总sRNA数的7.17%(表2)。筛选结果表明,甘蓝样品中病毒种类主要是BrYV、CMV、TMV、TuMV和BBWV2共5种病毒。
用velvet软件对筛选后的sRNA进行序列拼接获得1 295条contigs,利用数据库进行比对和分类注释(表2)。结果表明,注释到NCBI Nr、NCBI Nt、GenBank Virus RefSeq Nucleotide 和Protein等4个病毒数据库的contigs分别为114、58、41和50条。对这些比对结果进行分析,其中注释为TuMV的contigs为14条,注释为BrYV的contigs为10条,注释为CMV的contigs为6条,注释为BBWV 2和TMV的contigs各为2条。这与sRNA病毒种类初步筛选一致。
2.2 样品病毒检测结果
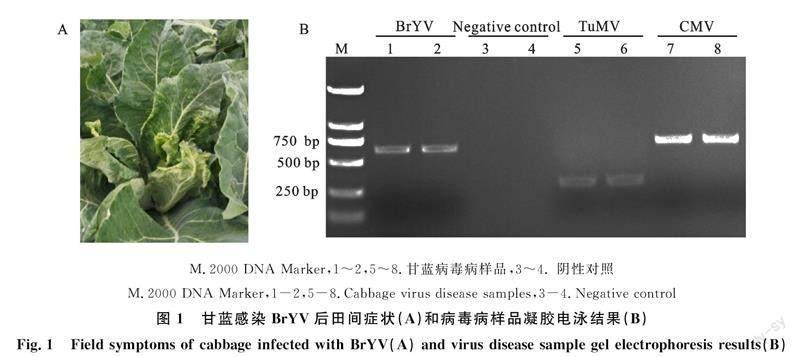
甘蓝感染BrYV后,叶片表现出花叶和皱缩症状(图1-A)。利用特异性引物以合成的cDNA作为模板进行RT-PCR检测,结果表明2份疑似病毒病的甘蓝样品中,扩增得到3条特异性条带(图1-B),测序结果显示BrYV cp序列片段大小为576 bp;TuMV特异性片段大小为377 bp;CMV cp序列片段大小为760 bp,BBWV2和TMV均未检测到。
2.3 BrYVcp序列分析
对测序后所获得的BrYV cp原始序列采用DNAMAN 6.0软件进行校正与拼接,获得1条576 bp的甘蓝病毒样品分离物cp基因全长序列,命名为GL。采用BLASTn工具比对发现GL分离物与14个序列同源,核苷酸序列同源率为88.0%~96.4%;氨基酸序列同源率为90.1%~95.8%。说明测序所获得的BrYV分离物和GenBank数据库中获得的14个BrYV株系具有很高的核苷酸和氨基酸同源性。
2.4 BrYV cp系统发育树分析
将BrYV-GL的全长cp基因核苷酸序列与从美国NCBI数据库中所随机检索到的14个cp基因核苷酸序列,使用MEGA 7.0软件以邻接法构建系统发育进化树(图2),自展校正值设定为 1 000,显示系统发育树时阈值为50%。结果表明, BrYV-GL cp基因与已报道的中国内蒙古油菜BrYV(GenBank登录号EF079907)分离物cp基因序列聚集在一起,亲缘关系较近。系统发育结果表明临洮BrYV-GL cp基因属于BrYV-C基因型。
2.5 BrYV cp重组分析
采用RDP4软件中RDP、GENECONV、BOOTSCAN、MAXCHI、CHIMAERA、SISCAN和3SEQ等7种方法,对BrYV-GL序列与NCBI获得的14条BrYV cp基因序列进行基因重组分析,结果显示,BrYV cp基因中检测到4个潜在的重组事件(表3)。重组事件1重组位点在 150~537 nt,获得4种算法支持,被认为是有意义的重组事件。最低P值为4.883×10-8(SiScan),分别以BrYV-GL和以中国油菜分离物BrYV-BJS(GenBank登录号HQ388351)为亲本。剩余3个潜在重组事件重组位点分别为55~523 nt、41~113 nt和40~149 nt。
3 讨 论
本研究采用sRNA深度测序技术从甘肃临洮采集的2份病样中共发现5种病毒,虽然测序结果提供了BBWV2和TMV的contigs,但是RT-PCR没有检测到这些病毒,这与siRNA测序和比对的过程中存在假阳性有关[25]。在测序过程中可能会有多个核苷酸依次加入。当遇到同聚物时,很难测量其实际长度,导致插入和删除等测序错误。在比对过程中,同一DNA片段可能被map到多个位置,导致数据错误[32];所参考的文献中的引物特异性不强,与目的片段有多处结合位点导致了套峰的情况。因此,本研究在根据sRNA测序结果进行病毒的鉴定和分类中存在一定误差。
BrYV为近年来发现的一种杆状新病毒,通过蚜虫以持久循回非增殖的方式进行传播,现已在中国普遍流行[33]。Zhang等[34]对中國22个省十字花科作物中的BrYV、CMV和TuMV病毒做了详细调查,结果发现BrYV-C在所调查地区发病率最高,为中国最主要的基因型,并且BrYV 3种基因型可单一侵染,也可2种以上混合侵染,还可与其他2种病毒混合侵染。本研究通过对BrYV的外壳蛋白基因序列进行一致性分析、系统发育树分析以及重组分析,明确其与BrYV(GenBank登录号EF079907)之间的进化关系,并且还显示其与韩国分离物(GenBank登录号KF923236)有很近的亲缘关系。这说明来自不同寄主,不同地区的分离物在分子生物学和生物学特性上存在较大相关性。并基于系统进化分析结果对临洮甘蓝BrYV分离物进行基因型鉴定,但由于受样品数量的影响,临洮地区是否存在BrYV-A和BrYV-B基因型,以及其是否存在混合侵染与其他寄主,有待进一步研究。本研究结果对BrYV的田间识别,选育抗病品种,开展综合防治具有指导意义。同时今后还需要通过采集不同地区、不同时间和不同种类蔬菜作物以及扩大采集样品数量,进行更多病毒检测,为更好地了解临洮当地BrYV的3种基因型的流行学信息以及其他病毒分布,为该地区蔬菜作物病毒病害的防控奠定基础。
参考文献 Reference:
[1]中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京:科学出版社,1993:16.
Editorial Committee of Flora of China,Chinese Academy of Sciences. Flora Reipublicae Popularis Sinicae[M].Beijing:Science Press,1993:16.
[2]杨丽梅,方智远,庄 木,等. “十二五”我国甘蓝遗传育种研究进展[J]. 中国蔬菜,2016(11):1-6.
YANG L M,FANG ZH Y,ZHUANG M,et al. Advances of research on cabbage genetics and breeding during “The Twelfth Five-year Plan” in China[J]. China Vegetables,2016(11):1-6.
[3]ZHAO L,FENG CH H,HAO X G,et al. Detection and molecular variability of turnip mosaic virus(TuMV) in Shaanxi,China[J]. Journal of Phytopathology,2014,162(7-8):519-522.
[4]MILOSEVIC D,IGNJATOV M,VLAJI S,et al. First report of turnip yellows virus on cabbage in Serbia[J]. Plant Disease,2019,104(3):997-998.
[5]MANDAL B,LANGSTON D B,PAPPU H R,et al. First report of cabbage leaf curl virus (Family Geminiviridae) in Georgia[J]. Plant Disease,2001,85(5):561-561.
[6]张艳超,侯明生,蔡 丽. 湖北省蔬菜病毒病主要毒原种类检测[J]. 华中农业大学学报,2017,36(6):31-38.
ZHANG Y CH,HOU M SH,CAI L. Detection of main viral species of vegetable virus diseases in Hubei Province[J]. Journal of Huazhong Agricultural University,2017,36(6):31-38.
[7]SEVIK,ALI M. Viruses infecting Brassica crops in the Black Sea Region of Turkey[J]. Acta Agriculturae Scandinavica. Section B-Soil and Plant Science,2016,66(7):553-557.
[8]ZHANG X Y,XIANG H Y,ZHOU C J, et al. Complete genome sequence analysis identifies a new genotype of Brassica yellows virus that infects cabbage and radish in China[J]. Archives of Virology,2014,159(8):2177-80.
[9]劉 欢. 常见蔬菜病毒病病原鉴定及多重RT-PCR的建立与应用[D]. 陕西杨凌:西北农林科技大学,2016.
LIU H. The identification of pathogens causing common vegetable virus diseases and establishment of multiplex RT-PCR detection system[D]. Yangling Shaanxi:Northwest A&F University,2016.
[10]LEE S,RHO J Y. Development of a specific diagnostic system for detecting turnip yellow mosaic virus from Chinese cabbage in Korea[J]. Indian Journal of Microbiology,2016,56(1):103-107.
[11]LI C,ZHANG Y,CHEN Y,et al. First report of broad bean wilt virus 2 infecting non-heading Chinese cabbage in China[J]. Plant Disease,2017,101(8):1559-1560.
[12]RYU S Y,HONG J S,RHEE S J, et al. Brief report:genome sequence and construction of an infectious cDNA clone of ribgrass mosaic virus from Chinese cabbage in Korea[J]. Virus Genes,2012,44(2):345-348.
[13]JEONG K S,SOH E H. One-step multiplex RT-PCR method for simultaneous detection of seed transmissible bacterium and virus occurring on Brassicaceae crop seeds[J]. Research in Plant Disease,2011,17(1):52-58.
[14]JUN M,SHIN S,LEE S J,et al. First report of raphanus sativus cryptic virus 2 on cabbage seeds (Brassica rapa subsp. pekinensis) imported from Italy[J]. Virusdisease,2021,32(4):834-836.
[15]XIANG H Y,DONG S W,Shang Q X,et al. Molecular characterization of two genotypes of a new polerovirus infecting brassicas in China[J]. Archives of Virology,2011,156(12):2251-2255.
[16]WANG Q,MAO J J,XIANG H Y,et al. First report of Brassica yellows virus on tobacco in China[J]. Plant Disease,2015,99(8):1192-1192.
[17]YOSHIDA N,TAMADA T. Host range and molecular analyses of beet leaf yellowing virus,beet western yellows virus-JP and brassica yellows virus in Japan[J]. Plant Pathology, 2019,68(6):1045-1058.
[18]ZHOU C J,ZHANG X Y,LIU S Y,et al. Synergistic infection of BrYV and PEMV2 increases the accumulations of both BrYV and BrYV-derived siRNAs in Nicotiana benthamiana[J]. Scientific Reports,2017,7(1):1-13.
[19]ZHANG X Y,ZHAO T Y,LI Y Y,et al. The conserved proline18 in the Polerovirus P3a is important for Brassica yellows virus systemic infection[J]. Frontiers in Microbiology,2018,9:613.
[20]PETER K A,GILDOW F,PALUKAITIS P,et al. The C terminus of the Polerovirus P5 readthrough domain limits virus infection to the Phloem[J]. Journal of Virology,2009,83(11):5419-5429.
[21]ZHANG X Y,XIANG H Y,ZHOU C J,et al. Complete genome sequence analysis identifies a new genotype of Brassica yellows virus that infects cabbage and radish in China[J]. Archives of Virology,2014,159(8):2177-2180.
[22]KREUZE J F,PEREZ A,UNTIVEROS M,et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs:A generic method for diagnosis,discovery and sequencing of viruses[J]. Virology,2009,388(1):1-7.
[23]蘇 秀. 小RNA深度测序在几种木本植物病毒鉴定中的应用[D]. 杭州:浙江大学,2015.
SU X. Application of small RNAs deep sequencing technology for identifying viruses from woody plants[D].Zhejiang:Zhejiang University,2015.
[24]李 洋,王 昊,张 晨,等. 基于小RNA(sRNA)深度测序技术进行病毒鉴定和发现的研究进展[J]. 病毒学报,2015,31(4):457-462.
LI Y,WANG H,ZHANG CH,et al. Advance in deep sequencing of Small RNAs for virus identification and discovery[J]. Chinese Journal of Virology,2015,31(4):457-462.
[25]陈雅寒,马 强,孙平平,等. 杏衰退萎黄病病毒的 siRNA高通量测序和RT-PCR鉴定[J]. 园艺学报,2020,47(4):725-733.
CHEN Y H,MA Q,SUN P P,et al. Identification of viruses causing apricot decline and leaf chlorosis disease by siRNA high-throughput sequencing and RT-PCR detection[J]. Acta Horticulturae Sinica,2020,47(4):725-733.
[26]龔明霞,赵 虎,王 萌,等. 广西辣椒病毒的sRNA深度测序和RT-PCR鉴定[J]. 园艺学报,2022,49(5):1060-1072.
GONG M X,ZHAO H,WANG M,et al. Identification of viruses infecting peppers in Guangxi by small RNA deep sequencing and RT-PCR[J]. Acta Horticulturae Sinica,2022,49(5):1060-1072.
[27]柴阿丽,陈利达,曹金强,等. 利用siRNA高通量测序和RT-PCR技术鉴定引起茄子斑驳紫花病的病毒种类[J]. 园艺学报,2019,46(3):508-518.
CHAI A L,CHEN L D,CAO J Q,et al. Identification of viruses causing eggplant purple mottle flower disease by siRNA high-throughput sequencing and RT-PCR detection[J]. Acta Horticulturae Sinica,2019,46(3):508-518.
[28]刘 勇,李 凡,李月月,等. 侵染我国主要蔬菜作物的病毒种类、分布与发生趋势[J]. 中国农业科学,2019, 52(2):239-261.
LIU Y,LI F,LI Y Y,et al. Identification,distribution and occurrence of viruses in the main vegetables of China[J]. Scientia Agricultura Sinica,2019,52(2):239-261.
[29]王莉爽,李 淳,陈小均,等. 贵州芸薹黄化病毒的分子检测及基因型鉴定[J]. 南方农业学报,2021,52(5):1149-1157.
WANG L SH,LI CH,CHEN X J,et al. Molecular detection and genotype identification of Brassica yellows virus in Guizhou[J]. Journal of Southern Agriculture,2021,52(5):1149-1157.
[30]MUHIZI M B,ARVIND V,MD PATRICK,et al. SDT:A virus classification tool based on pairwise sequence alignment and identity calculation[J]. PLOS ONE,2014,9(9):108277.
[31]DHIR S,ZAIDI A A,HALLAN V. Molecular Characterization and recombination analysis of the complete genome of apple chlorotic leaf spot virus[J]. Journal of Phytopathology,2013,161(10):704-712.
[32]MIERLO J T V,CLEEF K,RIJ R. Small silencing RNAs:Piecing together a viral genome[J].Cell Host & Microbe,2010,7(2):87-89.
[33]彭艳梅,张晓艳,王 颖,等. 芸薹黄化病毒3种基因型在我国的发生分布检测[C]∥中国植物病理学会2015年学术年会论文集. 北京:中国农业出版社,2015.
PENG Y M,ZHAO X Y,WANG Y,et al. Detection of occurrence and distribution of three genotypes of Brassica yellow virus in my country [C]∥Proceedings of the 2015 Annual Conference of the Chinese Society of Phytopathology.Beijing:China Agriculture Press,2015.
[34]ZHANG X Y,PENG Y M,XIANG H Y,et al. Incidence and prevalence levels of three aphid-transmitted viruses in crucifer crops in China[J]. Journal of Integrative Agriculture,2022,21(3):7.
Identification of Viruses Causing Cabbage Mosaic Disease and Genetic Variation in BrYV
ZHONG Jianxin,LI Jianguo,WU Chonggao,JIAO Taizhi,HE Zhilan,CHEN Yahan and YANG Chengde
Abstract To identify viral pathogens associated with cabbage mosaic viruses,we conducted a high-throughput sequencing of the small interfering RNA (siRNA) of the leaf samples. Based on the siRNA sequences,potential viral pathogens were detected,including the turnip mosaic virus (TuMV),cucumber mosaic virus (CMV),broad bean wilt virus2(BBWV2),tobacco mosaic virus (TMV) and brassica yellows virus (BrYV). To confirm the existence of the viruses by RT-PCR,BrYV,TuMV and CMV were detected. Moreover,cloning and phylogenetic analysis were carried out on the coat protein (cp) of the BrYV. The cp fragments of the BrYV was 576 bp. Pairwise comparison analysis indicated that the nucleotide and amino acid sequence identities of the BrYV cp genes with other published BrYV BrYV isolates in NCBI were from 88.0%-96.4% and 90.1%-95.8%,respectively. A phylogenetic tree was reconstructed for BrYV using the cp gene sequence,indicating a close relationship between BrYV-GL (classified as BrYV-C genotype) and an isolate from Inner Mongolia (GenBank accession no. EF079907).Four potential evidence of recombination was found among the 15 complete cp sequences. The results of this study may provide a theoretical reference for future research and control of the disease.
Key words Cabbage; Brassica yellow virus;Coat protein; RT-PCR
Received2022-07-27Returned 2022-08-18
Foundation item National Natural Science Foundation of China (No. 32160628); Natural Science Foundation of Gansu Province (No.21JR7RA820); Universities Innovation Foundation of Gansu Province (No.2021B-117); the Start-up Funding of High-level Talent Researchers in Gansu Agricultural University (No. GAU-KYQD-2019-22).
First author ZHONG Jianxin,male,master student. Research area:resource utilization and plant protection,functional components.E-mail:2294088150@qq.com
Corresponding author CHEN Yahan,female,Ph.D,associate professor. Research area:plant virology,functional components.E-mail:yhchen1018@nwafu.edu.cn
YANG Chengde,male,Ph.D,professor. Research area:plant pathology,functional components.E-mail:yangcd@gsau.edn.cn
(責任编辑:郭柏寿 Responsible editor:GUO Baishou)
收稿日期:2022-07-27修回日期:2022-08-18
基金项目:国家自然科学基金(32160628);甘肃省自然科学基金(21JR7RA820);甘肃省高等学校创新基金(2021B-117);甘肃农业大学人才启动金(GAU-KYQD-2019-22)。
第一作者:仲健新,男,硕士研究生,研究方向为植物病理学。E-mail:2294088150@qq.com
通信作者:陈雅寒,女,博士,副教授,研究方向为植物病毒学。E-mail:yhchen1018@nwafu.edu.cn
杨成德,男,博士,教授,研究方向为植物病理学。E-mail:yangcd@gsau.edn.cn
