基于F1群体的玉米株高与穗位高的全基因组关联分析
2023-12-20梁航宇叶凡董远李婷段迎新刘斌斌李周帅徐淑兔张兴华薛吉全
梁航宇 叶凡 董远 李婷 段迎新 刘斌斌 李周帅 徐淑兔 张兴华 薛吉全
摘 要 株高、穗位高是影响玉米耐密性的重要株型性状,明确玉米株高、穗位高的遗传基础有利于株型改良。基于NCⅡ遗传交配设计,以陕A群和陕B群选育的85个自交系组配的246份F1群体为材料,进行株型表型评价,同时结合55 951个高质量SNPs标记,对株高、穗位高以及穗位系数进行全基因组关联分析。结果表明:株高、穗位高、穗位系数的遗传力分别为77.68%、77.04%、73.78%,且三者之间存在显著正相关。同时,通过全基因组关联分析检测到7、5、4个SNP与株高、穗位高和穗位系数显著相关,解释1.47%~ 25.27%表型变异。通过候选基因功能注释,共筛选到10个候选基因,涉及植物的生长发育、生物合成、响应非生物胁迫等过程,针对3个共定位区间和热点区间锚定plt1、atp2、ZC3H46、emp21等为候选基因。可见,通过株型表型鉴定及相关基因的挖掘,可为陕A群和陕B群两个玉米群体的株型改良提供理论基础。
关键词 玉米;杂交种;株高;穗位高;全基因组关联分析
作为中国第一大粮食作物,玉米生产对保障国家粮食安全具有重要战略意义[1]。当前,提高种植密度和生产机械化程度已成为实现玉米高产、稳产最重要的技术措施[2]。合理密植可充分利用光能,合成更多物质,实现玉米生育期内源、流和库的协调发展,光合产物的合理分配,可使群体内植株间的竞争达到最小状态,从而有效地提高产量。而实现密植的关键因素之一是株型相关性状,包括株高、穗位高和穗位系数等,植株过高会降低种植密度、抗倒伏能力和收获指数,过低则影响群体通透性、易患病虫害、降低生物产量;穗位过高易造成植株倒伏,过低则不利于光合产物向穗部的有效运转[3]。通过改良株型可以调节个体间的构型和空间排列方式,使群体和个体之间协调发展,从而使玉米获得高产[4]。因此,探究玉米株型性状的遗传结构,培育具有合适株型的玉米品种显得尤为重要。
全基因组关联分析(Genome-Wide Association Study,GWAS)是作物复杂数量性状遗传解析的有效可行策略[5]。近年来,国内外学者选用不同的群体定位了许多与株高、穗位高等株型性状显著关联的SNP。Weng等[6]利用MLM模型对284份玉米自交系对株高进行全基因组关联分析,鉴定了204个显著关联的 SNP位点和105个携带编码区的基因组区域;Vanous等[7]在252个双单倍体(Double Haplotype,DH)中鉴定了18个与株高相关的候选基因,其中一些与株高相关的基因已经被克隆并经过了验证。Zhang等[8]使用由300个玉米F1杂交种组成的群体进行了全基因组关联分析,确定了9个重要的SNPs标记和两个株高相关候选基因Zm00001d018617和Zm00001d023659,为杂交种中玉米株高的遗传基础提供了新的见解。目前已克隆的调控玉米株高和穗位高性状的基因多数均与植物激素有关,如通过赤霉素合成、生长素运输途径参与玉米株高和穗位高调控的基因Anther ear1(AN1)、ZmPIN1a等[9-10]。
玉米生产上使用的是F1单交种,有必要开展以F1为材料的株型性状遗传解析工作,当前相关研究进展较少。玉米生物学与遗传育种课题组前期基于陕A群和陕B群选育的玉米自交系开展了株高、穗位高和茎秆强度的全基因组关联分析,挖掘了44个与抗倒伏性状显著关联位点,同时结合KA105和KB020构建的重组自交系群体(RIL)QTL定位结果,筛选了6个候选基因[11]。为丰富研究内容,本研究以基于NCⅡ设计获得的246个F1杂交组合为材料,通过调查多环境下的表型数据进行F1群体的株型性状及其配合力评估,并结合高通量分子标记进行全基因组关联分析(GWAS),以期筛选到适宜株型优良的杂交种、初步解析株型及其配合力的遗传基础,为进一步的株型改良奠定理论基础。
1 材料与方法
1.1 试验材料
本研究材料源于西北农林科技大学玉米生物学与遗传育种课题组在高密、干旱、低氮条件下构建的陕A群、陕B群2个杂种优势群选育的85个自交系,采用 NC-Ⅱ 遗传交配设计,按3×61+3×21共组配246份杂交组合,即选取陕A群选育的3份骨干自交系为测验种,与陕B群选育的61份自交系组配获得183个F1杂交种;和选取陕B群选育的3份骨干自交系为测验种,与陕A群选育的21份自交系进行组配获得63个F1杂交种,具体种植方案为:2018年在陕西杨凌和陕西榆林种植2个重复,采用α-格子试验设计,密度90 000株·hm-2,4行区,行长 5 m,行距0.6 m。其他管理同當地大田生产管理一致。该群体已开展了产量和籽粒大小的配合力评估及遗传解析[12-13]。
1.2 表型测定与分析
玉米生理成熟至收获前调查试验群体中每个材料的5个单株的株高(plant height,PH)和穗位高(ear height,EH),并计算穗位系数(穗位高/株高,EH/PH)。5个单株的表型平均值代表这个重复的平均值,2个重复的平均值代表该环境下的表型值,利用IBM SPSS Statistics 26.0软件和R语言(https://cran.r-project.org/)对原始数据进行描述性统计、方差分析和相关性分析。
广义遗传力(broad-sense heritability,H2)按照Knapp等[14]提出的公式H2=σg2/(σg2+σge2/n+σe2/nr)计算,其中σg2为遗传方差,σge2为基因型与环境互作方差,σe2为误差方差,n为环境数,r为重复。利用R包的lme4中的混合线性模型来计算最佳线性无偏预测值(Best Linear Unbiased Prediction,BLUP),用于后续的关联分析。
1.3 基因型检测与全基因组关联分析
本试验使用基因型检测和质控与李周帅等[12]研究一致,即基因型使用Affymetrix maize 6H90K芯片(北京康普森生物技术有限公司)进行基因分型,质控后获得55 951个高质量SNP标记进行全基因组关联分析。
全基因组关联分析采用TASSEL[15]软件的混合线性模型(Mixed Linear Model,MLM),以群体结构-主成分(Principle Component Analysis,PCA)为固定效应,亲缘关系Kinship作为随机效应。李周帅等[12]利用SimpleM算法(http://simplem.sourceforge.net/)获得720个有效标记,本研究将阈值设为P≤0.1/N,即 -lgP=3.86,用于筛选显著的SNP位点。同时对显著SNP位点按照r2>0.6的标准筛选连锁位点,保留其中P值较小的SNP位点。并结合公共数据库maizeGDB (https://www.maizegdb.org/),以距显著SNP物理位置最近的基因为候选基因,并进行功能注释。
2 结果与分析
2.1 表型数据分析
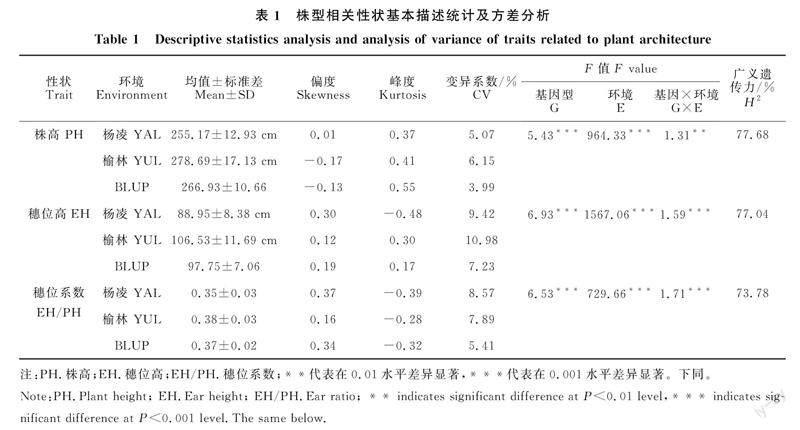
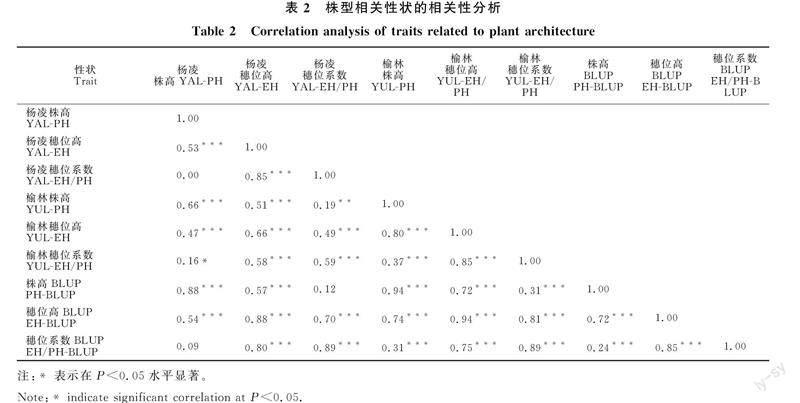
经统计分析,榆林点株高、穗位高和穗位系数3个性状的均值均高于杨凌,且3个性状在榆林点的变异系数均较杨凌高,表明榆林点与杨凌点相比具有更丰富的表型变异。方差分析表明,株高、穗位高、穗位系数在基因型、环境、基因型环境互作均存在极显著差异。该群体中株高、穗位高以及穗位系数的广义遗传力分别为77.68%、 77.04%和73.78%(表1),说明这3个性状主要受遗传因素影响。相关性分析表明,同一性状不同环境间显著相关,不同性状之间也存在显著相关:各性状在榆林和杨凌 2个环境中均显著正相关,其中穗位系数的相关系数最低,为 0.59;各性状 BLUP 值与各环境下表型均值的相关系数超过了 0.88。穗位高与穗位系数之间的相关系数为0.85,而株高与穗位系数之间相关性较弱,仅为0.24(表2)。为了后续的全基因组关联分析,使用BLUP值作为表型值,株高、穗位高以及穗位系数的变异系数都有不同程度的下降,降低了环境效应的影响。
2.2 全基因组关联分析
2.2.1 株型相关性状的关联分析
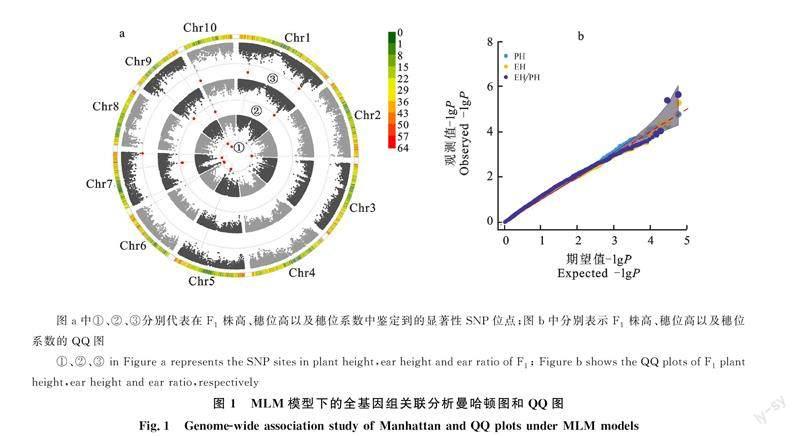
利用TASSEL中MLM模型(PCA+K)对株高、穗位高以及穗位系数的BLUP值进行全基因组关联分析,以-lgP≥3.86作为阈值进行筛选,共检测到16个显著关联的SNP标记,其中在株高、穗位高以及穗位系数中检测到的SNP个数分别为7、5和4个(图1),解释1.47%~25.27%表型变异(表3)。发现各性状均能检测到表型解释率大于10%的主效位点:与株高显著关联的 SNP 解释的表型变异介于1.60%~13.64%,其中位于9号染色体的Affx-291376942能够解释大于10%的表型变异,为主效SNP。与穗位高显著关联的 SNP 解释的表型变异介于2.99%~15.89%,位于7号染色体的Affx-291446022及位于10号染色体的Affx-291379977均能解释大于10%的表型变异,为主效SNP。与穗位系数显著关联的 SNP 解释的表型变异介于1.47%~25.27%,除了位于7号染色体的Affx-291386395之外,均为主效SNP,且位于10号染色体的Affx-291379977能解释25.27%的表型变异。
比较不同性状检测到的SNP位点,获得3个共定位SNP位点。其中,位于7号染色体上的SNP标记Affx-291432370在株高与穗位高中共定位到;位于1号染色体上的SNP标记Affx-291415291以及位于10号染色体上的SNP位点Affx-291379977在穗位高以及穗位系数中共定位到。结果说明株高、穗位高、穗位系数之间可能存在共同的遗传基础。
2.2.2 共定位位点的遗传效应分析 为了更好地比较位点的遗传效应,比较分析株高、穗位高、穗位系数中检测到的3个共定位位点,发现株高与穗位高共定位的SNP位点Affx-291432370在株高与穗位高中的优异等位基因均为CC或CT;穗位高与穗位系数共定位到的2个显著SNPs,其中SNP标记Affx-291379977的优异等位基因为CT,Affx-291415291的优异等位基因为GG。即这3个共定位位点纯合为有利等位基因,可适当降低株高、穗位高和穗位系数,说明可利用分子标记辅助选择育种进行株型改良。
2.2.3 候选基因预测 结合MaizeGDB数据库 (http://www.maizegdb.org/),比对参考基因组Maize B73 RefGen_v3,以距显著SNP物理位置最近的基因为候选基因,经过筛选,共获得了10个有功能注释的候选基因(表4),它们大多与植物的生长发育、生物合成以及响应非生物胁迫等有关,包括涉及调控生长素合成与运输的GRMZM2G458082(hagtf31)和GRMZM2 G137869(ofp37),涉及调控细胞伸长生长过程的GRMZM2G324471(grp),参与木质素生物合成过程的GRMZM2G305526(lac11),参与能量代谢的GRMZM2G113408(atp2)等。
3 讨 论
3.1 F1杂交种群体在复杂性状遗传解析中的作用
复杂数量性状遗传解析的方法主要有连锁分析和关联分析,其中全基因组关联分析的应用实现了快速的复杂数量性状机理解析,如干旱、株高、产量和品质等。Guo等[16]基于209份不同种质在2种水分条件下对玉米种子根长进行全基因组关联分析,鉴定响应干旱胁迫相关的基因,最终筛选出了7個与抗旱性相关的候选基因;Yang等[17]利用513个具有热带、亚热带和温带背景的玉米品系对17个玉米农艺性状进行了关联分析,定位到一个位于第3染色体的SNP (Chr.3:[CM(21]162709488) ,该SNP可解释9.61%的表型变异;Zhang等[18]结合QTL定位与GWAS对玉米产量相关性状进行剖析,并结合共表达网络,发现编码丝氨酸/苏氨酸蛋白激酶的基因Zm00001d016656与多种环境中的5个性状有关;Ma等[19]使用FarmCPU和CMLM 2种关联分析模型对8个产量相关性状的BLUE值进行GWAS,共鉴定出22个显著SNP,其中FarmCPU模型检测到17个关联位点,高于CMLM。当前,大多数的GWAS研究都以纯合自交系或者重组自交系群体等作为关联群体,群体中缺乏杂合位点的效应评估,即无法评估显性效应;同时生产上应用的材料主要为杂交种,基于自交系的GWAS分析结果并不能直接用于剖析F1群体的遗传基础,因此利用F1群体开展遗传研究十分必要。郑灵[20]提出利用品种间成对杂交产生的F1群体作为关联群体的思路,经过模拟研究对其有效性进行了验证,结果表明F1群体作为关联群体是可行的。基于F1群体的全基因组关联分析方法,已经在多个物种中得到了广泛应用[21-23]。本研究利用85个自交系材料组配了246个F1杂交种,通过亲本的基因型进而推测出杂交组合F1的基因型[24],基于此,开展了株高、穗位高和穗位系数的全基因组关联分析,检测到16个关联位点。课题组前期于芮苏等[11]开展的全基因组关联分析利用的是陕A群、陕B群的自交系材料,检测到的关联位点与本研究利用F1群体检测到的位点共定位区间少,说明在遗传研究中,不能单独的使用纯系群体开展研究,特别是对显性效应明显的表型。本研究结果进一步丰富了陕A群和陕B群选育玉米自交系的株型相关性状的遗传解析。
3.2 株型相关性状的调控机制解析
前人已报道的关于调控玉米株高和穗位高相关的基因主要包括激素类相关基因、营养成分运输调控、光敏色素类基因等。如VT2基因可编码色氨酸氨基转移酶,该酶在生长素生物合成途径中将色氨酸转化为吲哚-3-丙酮,从而参与玉米株高调控[25];SXD1基因编码生育酚环化酶,其突变体叶中花青素和淀粉积累异常,不能正常输出蔗糖,最终表现为植株表现矮小[26];ELM1基因通过光敏色素合成途径参与玉米株高调控,其编码一个 phytochromobilin 合成酶,突变会使植株节间伸长,株高变高[27]。
本研究结合MaizeGDB数据库 (http://www.maizegdb.org)一共鉴定到了10个具有功能注释的基因,如位于7号染色体上的GRMZM2G458082(hagtf31),其编码类N-乙酰基转移酶HLS1,HLS1是顶端弯钩发育过程中必不可少的调控因子,参与拟南芥叶中的糖和生长素信号传导,并可能通过控制类GH3基因参与IAA稳态的负反馈调控[28];位于1号染色体上的GRMZM2G305526(lac11)在穗位系数中被检测到,漆酶(LAC)是植物催化木质素单体聚合合成所必需的,木质素的积累对维持细胞壁结构和完整性至关重要[29-30],推测其为玉米植株抗倒特性密切相关的候选基因。
3.3 与前人研究结果共定位分析
本研究筛选到的与株高显著相关的SNP(Affx-291431456、Affx-291380774)分别位于SNP热点区间0.003~9.158 Mb(Chr:10)、 160.902~166.929 Mb(Chr:6)[31],在此区间确定了2个候选基因:GRMZM2G101958(plt1)为AP2/ERF型转录因子,目前普遍认为生长素浓度信号是通过对 PLTs 基因的表达调控转变为 PLTs 蛋白浓度梯度调控根尖的生长发育。AP2/ERF与赤霉素(GA),细胞分裂素(CTK)和油菜素类固醇介导的生长和发育过程有关,并调节植物激素的生物合成和信号传导[32-33]。GRMZM2G113408(atp2)是一种ATP合酶,在质膜能量中起着重要作用,作为细胞中的能量通货,在调节植物细胞功能和营养物质运输等各种生命活动[34]。本研究筛选到的与穗位高显著相关的SNP(Affx-291419522)位于bin9.03區间,与张体付等[35]、李凯等[36]、于芮苏等[11]定位到的QTL同属一个bin区间,且该区间属于株高与穗位高的QTL热点区间[31]。
另外,在本研究中共筛选到了3个共定位位点,并对其进行候选基因功能注释,发现GRMZM5G822449(ZC3H46)编码CCCH型锌指蛋白,在拟南芥中过表达AtC3H49和AtC3H20基因,转基因植株前期成长缓慢,后期生长迅速,成熟植株较野生型植株大[37];GRMZM5G849971(emp21)编码一个典型的PPR-DYW蛋白,亚细胞定位分析显示其仅定位于线粒体,emp21的功能缺失严重抑制了线粒体复合物Ⅰ和Ⅴ的组装和活性,与能量代谢紧密相关,且是玉米种子发育的关键基因[38]。
4 结 论
本研究选用来源于陕A群、陕B群的85份玉米自交系基于NCⅡ遗传交配设计组成了246份F1群体,结合株型相关性状,通过全基因关联分析挖掘到16个关联位点,其中有3个共定位位点。在热点区间锚定了plt1、 atp2为候选基因,在共定位区间锚定了ZC3H46、emp21为候选基因用于进一步的机理解析研究。本研究的结果说明利用杂交群体开展遗传研究的方法是可行的,并初步解析了株型相关性状的遗传基础,为陕A群和陕B群的株型改良提供了理论基础。
参考文献 Reference:
[1]TANG J H,TENG W T,YAN J B,et al.Genetic dissection of plant height by molecular markers using a population of recombinant inbred lines in maize [J].Euphytica,2007,155(1/2):117-124.
[2]别 海,郑 斐,王保林,等.利用玉米RIL群体分析株型性状对两种密度的响应 [J].玉米科学,2019,27(6):13-18.
[DW]BIE H,ZHENG F,WANG B L,et al.Analysis of plant architecture underlying the plant density response by RIL population in maize[J].Journal of Maize Sciences,2019,27(6):13-18.
[3]胡德升,谢旭东,张向歌,等.利用单片段代换系测交群体定位玉米株高和穗位高的杂种优势位点 [J].河南农业大学学报,2016,50(1):8-13.
[DW]HU D SH,XIE X D,ZHANG X G,et al.Heterotic loci detection for plant height and ear height in maize using CSSLs test population
[J].Journal of Henan Agricultural University,2016,50(1):8-13.
[4]吉春容,李世清,冯宏昭,等.不同株型夏玉米冠层叶片气孔特性的差异 [J].西北农林科技大学学报(自然科学版),2008,36(5):57-63.
[DW]JI CH R,LI SH Q,FENG H ZH,et al.Study on change rule of stomata characteristics of different maize types [J].Journal of Northwest A& University(Natural Science Edition),2008,36(5):57-63.
[5]HUANG X,HAN B.Natural variations and genome-wide association studies in crop plants[J].Annual Review of Plant Biology,2014,65(1):531-551.
[6]WENG J,XIE C,HAO Z,et al.Genome-wide association study identifies candidate genes that affect plant height in Chinese elite maize(Zea mays L.) inbred lines [J].Plos One,2011,6(12):e29229.
[7]VANOUS A,GARDNER C,BLANCO M,et al.Association mapping of flowering and height traits in germplasm enhancement of maize doubled haploid (GEM-DH) lines [J].Plant Genome,2018,11(2):1-14.
[8]ZHANG Y,WAN J,HE L,et al.Genome-wide association analysis of plant height using the maize F1 population [J].Plants-Basel,2019,8(10):432.
[9]BENSEN R J,JOHAL G S,CRANE V C,et al.Cloning and characterization of the maize An1 gene [J].The Plant Cell,1995,7(1):75-84.[ZK)]
[10][ZK(#]LI Z X,ZHANG X R,ZHAO Y J,et al.Enhancing auxin accumulation in maize root tips improves root growth and dwarfs plant height [J].Plant Biotechnology Journal,2018,16(1):86-99.
[11]于芮苏,田小康,刘斌斌,等.玉米抗倒伏相关性状QTL的关联和连锁分析 [J].作物学报,2022,48(1):138-150.
[DW]YU R S,TIAN X K,LIU B B,et al.Dissecting the genetic architecture of lodging related traits by genome-wide association study and linkage analysis in maize[J].Acta Agronomica Sinica,2022,48(1):138-150.
[12]李周帅,董 远,李 婷,等.基于杂交种群体的玉米产量及其配合力的全基因组关联分析 [J].中国农业科学,2022,55(9):1695-1710.
[DW]LI ZH SH,DONG Y,LI T,et al.Genome wide association analysis of maize yield and combining ability based on hybrid population [J].Scientia Agricultura Sinica,2022, 55(9):1695-1710.
[13]李 婷,王亞鹏,董 远,等.基于杂交群体解析玉米籽粒大小相关性状及其配合力的分子遗传机制 [J].作物学报,2022,48(10):2451-2462.
[DW]LI T,WANG Y P,DONG Y,et al.Molecular genetic mechanism of grain size related traits and their combining ability in maize based on hybrid population analysis [J].Acta Agronomica Sinica,2022,48(10):2451-2462.
[14]KNAPP S J,STROUP W W,ROSS W M.Exact confidence intervals for heritability on a progeny mean basis [J].Crop Science,1985,25(1):192-194.
[15]BRADBURY P J,ZHANG Z,KROON D E,et al.TASSEL:software for association mapping of complex traits in diverse samples [J].Bioinformatics,2007,23(19):2633-2655.
[16]GUO J,LI C,ZHANG X,et al.Transcriptome and GWAS analyses reveal candidate gene for seminal root length of maize seedlings under drought stress [J].Plant Science,2020,292:110380.
[17]YANG N,LU Y,YANG X,et al.Genome wide association studies using a new nonparametric model reveal the genetic architecture of 17 agronomic traits in an enlarged maize association panel [J].Plos Genetics,2014,10(9):e1004573.
[18]ZHANG X,REN Z,LUO B,et al.Genetic architecture of maize yield traits dissected by QTL mapping and GWAS in maize [J].Crop Journal,2022,10(2):436-446.
[19]MA J,CAO Y.Genetic dissection of grain yield of maize and yield-related traits through association mapping and genomic prediction [J].Frontiers in Plant Science,2021,12:690059.
[20]鄭 灵.白玉米地方种质遗传多样性及基于F1家系关联分析初探 [D].成都:四川农业大学,2011.
[DW]ZHENG L.Genetic diversity of white jade rice landrace and its application based on F1 preliminary study on family relationship analysis [D].Chengdu:Sichuan Agricultural University,2011.
[21]LI P,ZHOU H,YANG H,et al.Genome-wide association studies reveal the genetic basis of fertility restoration of CMS-WA and CMS-HL in xian/indica and aus accessions of rice (Oryza sativa L.) [J].Rice,2020,13(1):11.
[22]MEI Y,YU J,XUE A,et al.Dissecting genetic network of fruit branch traits in upland cotton by association mapping using SSR markers [J].Plos One,2017,12(1):e0162815.
[23]BINEAU E,RAMBLA J L,PRIEGO-CUBERO S,et al.Breeding tomato hybrids for flavour:comparison of gwas results obtained on lines and F1 hybrids [J].Genes,2021,12(9):1443.
[24]王恒生.基于玉米F1群体的株高遗传结构解析及调控网络构建[D].合肥:安徽农业大学,2018.
[DW]WANG H SH.Genetic structure analysis and regulation network construction of plant height of population based on maize F1 [D].Hefei:Anhui Agricultural University,2018.
[25]PHILLIPS K A,SKIRPAN A L,LIU X,et al.vanishing tassel2 encodes a grass-specific tryptophan aminotransferase required for vegetative and reproductive development in maize [J].Plant Cell,2011,23(2):550-566.
[26]RUSSIN W A,EVERT R F,VANDERVEER P J,et al.Modification of a specific class of plasmodesmata and loss of sucrose export ability in the sucrose export defective1 maize mutant [J].The Plant Cell,1996,8(4):645-658.
[27]SAWERS R J H,LINLEY P J,FARMER P R,et al.Elongated mesocotyl1,a phytochrome-deficient mutant of maize [J].Plant Physiology,2002,130(1):155-163.
[28]OHTO M A,HAYASHI S,SAWA S,et al.Involvement of HLS1 in sugar and auxin signaling in Arabidopsis leaves [J].Plant & cell Physiology,2006,47(12):1603-1611.
[29]ZHAO Q,NAKASHIMA J,CHEN F,et al.Laccase is necessary and nonredundant with peroxidase for lignin polymerization during vascular development in Arabidopsis [J].Plant Cell,2013,25(10):3976-3987.
[30]RANOCHA P,CHABANNES M,CHAMAYOU S,et al.Laccase down-regulation causes alterations in phenolic metabolism and cell wall structure in poplar [J].Plant Physiology,2002,129(1):145-155.
[31]马雅杰,高悦欣,李依萍,等.玉米株高和穗位高的遗传基础与分子机制 [J].中国生物工程杂志,2021,41(12):61-73.
[DW]MA Y J,GAO Y X,LI Y P,et al.Progress on genetic analysis and molecular dissection on maize plant height and ear height [J].China Biotechnology,2021,41 (12):61-73.
[32]DING Z,FRIML J.Auxin regulates distal stem cell differentiation in Arabidopsis roots [J].Proceedings of the National Academy of Sciences of the United States of America,2010,107(26):12046-12051.
[33]XIE Z,NOLAN T M,JIANG H,et al.AP2/ERF transcription factor regulatory networks in hormone and abiotic stress responses in Arabidopsis [J].Frontiers in Plant Science,2019,10:228.
[34]ELMORE J M,COAKER G.The role of the plasma membrane H+-atpase in plant-microbe interactions [J].Molecular Plant,2011,4(3):416-427.
[35]張体付,梁帅强,吕远大,等.基于三重测交群体解析玉米株高与穗位高杂种优势QTL [J].核农学报,2017,31(5):837-843.
[DW]ZHANG T F,LIANG SH Q,L Y D,et al.QTL analysis of heterosis for plant height and ear position using triple testcross population in maize[J].Journal of Nuclear Agricultural Sciences,2017,31(5):837-843.
[36]李 凯,张晓祥,管中荣,等.玉米株高和穗位高的全基因组关联分析 [J].玉米科学,2017,25(6):1-7.
[DW]LI K,ZHANG X X,GUAN ZH R,et al.Genome-wide association analysis of plant height and ear height in maize [J].Journal of Maize Sciences,2017,25(6):1-7.
[37]LEE S J,JUNG H J,KANG H,et al.Arabidopsis zinc finger proteins AtC3H49/AtTZF3and AtC3H20/AtTZF2are involved in ABA and JA responses [J].Plant and Cell Physiology,2012,53(4):673-686.
[38]WANG Y,LIU X Y,YANG Y Z,et al.Empty pericarp21 encodes a novel PPR-DYW protein that is required for mitochondrial RNA editing at multiple sites,complexes I and V biogenesis,and seed development in maize [J].Plos Genetics,2019,15(8):e1008305.
Genome-wide Association Study of Maize Plant Height and Ear Height Based on F1 Population
LIANG Hangyu YE Fan DONG Yuan LI Ting DUAN Yingxin LIU Binbin LI Zhoushuai XU Shutu ZHANG Xinghua1,2 and XUE Jiquan1,2
Abstract Plant and ear height are key plant architecture traits that affect density tolerance of maize.Understanding the genetic basis of plant and ear height can aid in enhancing the plant architecture,particularly in maize hybrids.This study evaluated plant and ear height in 246 F1 hybrids constructed from 85 inbred lines selected from Shaan A and Shaan B group,using the NCII genetic mating design.This study also conducted a genome-wide association study using 55 951 high-quality SNPs markers to identify the genetic basis of plant height (PH),ear height (EH),and EH/PH.The heritability of plant height,ear height and ear ratio was high,ranging from 77.68%,77.04% and 73.78%,respectively,and there was a high significant positive correlation among them.The genome-wide association study identified 7,5 and 4 SNPs associated with plant height,ear height and EH/PH ,respectively.These SNPs explained 1.47%-25.27% of phenotypic variation.Functional annotation of candidate genes revealed 10 potential candidate genes involved in plant growth and development,biosynthesis process and response to abiotic stress process.This study also identified three co-localization intervals and hot spot intervals where genes such asplt1,atp2,ZC3H46and emp21 were anchored.These results provide a theoretical basis for enhancing plant architecture in the Shaan A and Shaan B maize populations.
Key words Maize; Hybrids; Plant height; Ear height; Genome-wide association study
Received2022-05-23Returned 2022-09-28
Foundation item China Agricultural Research System(Maize) (No.CARS-02).
First author LIANG Hangyu,male,master student.Research area:maize genetics and breeding. E-mail:liang198124@163.com
Corresponding author XUE Jiquan,male,professor.Research area:maize genetics and breeding. E-mail:xjq2934@163.com
(责任编辑:成 敏 Responsible editor:CHENG Min)
收稿日期:2022-05-23修回日期:2022-09-28
基金项目:现代农业产业技术体系专项(玉米) (CARS-02)。
第一作者:梁航宇,男,硕士研究生,研究方向为玉米遗传育种。E-mail:liang198124@163.com
通信作者:薛吉全,男,教授,研究方向为玉米遗传育种。E-mail:xjq2934@163.com
