基于小RNA 深度测序技术的苜蓿病毒病鉴定与分析
2023-12-08杜江马振男王晨燕张丽王德富牛颜冰
杜江,马振男,王晨燕,张丽,王德富,牛颜冰*
(1. 山西农业大学生命科学学院,山西 太谷 030801;2. 山西农业大学草业学院,山西 太谷 030801)
紫花苜蓿(Medicago sativa)为豆科草本植物,是一种高营养成分的牧草,在农牧业生产中发挥着巨大的作用[1-2]。我国是畜牧大国,因此对于紫花苜蓿的需求量极为庞大,据统计紫花苜蓿在我国的种植面积呈现出逐年增长的趋势[3]。伴随着紫花苜蓿种植面积的不断扩大,越来越多的紫花苜蓿病毒病也随之被检出。紫花苜蓿的病毒病害不仅严重影响了紫花苜蓿的产量和品质,还对紫花苜蓿产业的良性发展产生了巨大危害。
对危害紫花苜蓿的病毒病原进行调查,发现主要有苜蓿花叶病毒(alfalfa mosaic virus, AMV)、豇豆花叶病毒(cowpea mosaic virus, CPMV)、白三叶草花叶病毒(white clover mosaic virus, WCMV)、番茄花叶病毒(tomato mosaic virus, ToMV)、菜豆黄花叶病毒(bean yellow mosaic virus, BYMV)、黄瓜花叶病毒(cucumber mosaic virus, CMV)、豌豆线条病毒(pea streak virus, PeSV)、苜蓿卷叶病毒(alfalfa leaf curl virus, ALCV)、菜豆卷叶病毒(bean leafroll virus, BLRV)和烟草花叶病毒(tobacco mosaic virus, TMV)等多达47 种植物病毒[4-7]。研究发现,有些感病紫花苜蓿往往是被病毒复合侵染,如在甘肃、内蒙古、河南、陕西的紫花苜蓿上同时检测到了AMV、苜蓿矮化病毒(alfalfa dwarf virus, ADV)和PeSV 的侵染;在甘肃不同品种的紫花苜蓿上同时检测到了AMV 和WCMV 的侵染,并且有研究者发现病毒的侵染会显著影响紫花苜蓿的过氧化物酶(peroxidase, POD)、多酚氧化酶(polyphenol oxidase, PPO)、苯丙氨酸解氨酶(phenylalanine ammonialyase, PAL)的活性[6,8]。目前对于紫花苜蓿病毒病害的研究发展迅猛,但在部分重要紫花苜蓿种植区,紫花苜蓿病毒病的病原仍不清楚,限制了对该病害的研究和防治。
本研究于2021 年5 月在山西农业大学植物园(37°25′N,112°34′E)进行病害调查时,发现种植面积约为0.5 hm2的多年生紫花苜蓿有大量植株叶片有卷曲、皱缩、花叶的症状出现。为明确引起山西农业大学植物园内紫花苜蓿卷曲、皱缩和花叶症状的病毒病原,本研究采用小RNA(small RNA,sRNA)深度测序技术并结合分子生物学的方法,分离鉴定侵染山西农业大学植物园内紫花苜蓿的病毒病原,研究结果将为山西省紫花苜蓿病毒病的防治工作提供一定的参考依据。
1 材料与方法
1.1 供试材料
在山西农业大学植物园内,采集有代表性的4 株具有典型病毒病症状的紫花苜蓿样品,及1 株无症状样品分别置于-80 ℃超低温冰箱中保存。
1.2 小RNA 深度测序
取等量4 株样品混合后于2021 年5 月送至南京集思慧远生物科技有限公司进行小RNA 的建库及测序。利用Bwa(0.7.17-r1188)软件和Velvet(1.1.07)软件对小RNA 测序数据进行组装和序列比对分析。
1.3 RNA、DNA 提取及PCR 验证
RNA、DNA 提取及PCR 检测于2021 年8-11 月进行。采用TRIzol(TaKaRa BIO INC)提取供试样品总RNA,利用PrimeScriptTMRT reagent Kit with gDNA Eraser(TaKaRa BIO INC)反转录合成cDNA。利用PlantZol 试剂盒(TransGen Biotech)提取样品总DNA,提取好的RNA 和DNA 保存于-80 ℃超低温冰箱。按照试剂盒说明书,配制含有5×gDNA Eraser Buffer 2 μL,gDNA Eraser 1 μL,总RNA 1 μg,加RNase Free H2O 至10 μL,42 ℃保温5 min 后,置于冰上迅速冷却;然后向混合液中加入5×Prime Script Buffer 4 μL 的混合液,RT Primer Mix 1 μL,PrimeScript RT Enzyme Mix 1 μL,加入RNase Free H2O 至20 μL,混匀后在37 ℃保温15 min,最后85 ℃反应5 s 后得到第1 链cDNA。
以合成的第1 链cDNA 或DNA 作为模板,用表1 所示的病毒特异性引物进行PCR 扩增。PCR 反应体系如下:第1 链cDNA 或DNA 模板1 μL,PCR 引物(10 μmol·L-1)各2.0 μL,PrimeSTAR HS(Premix)(TaKaRa BIO INC)25 μL,最后加入RNase Free H2O 至50 μL。PCR 扩增程序:98 ℃ 10 s,55 ℃ 5 s,72 ℃ 0.5~3.0 min,共30 个循环。PCR 扩增产物用1 %的琼脂糖凝胶电泳分析。将目标条带回收后进行连接、转化试验,通过菌落PCR 鉴定后随机挑选3 个阳性克隆送至生工生物工程(上海)股份有限公司测序。
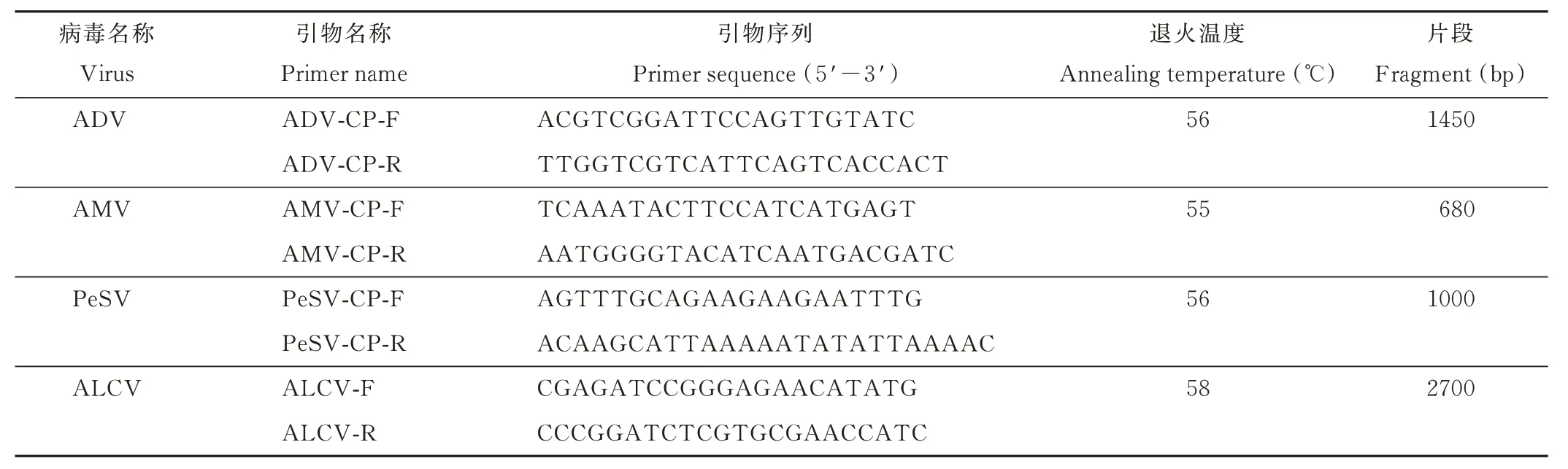
表1 本研究中使用的引物Table 1 Primers used in this study

表2 AMV-SX 与其他AMV 分离物的CP 基因核苷酸序列及氨基酸序列一致性Table 2 Nucleotide and amino acid sequence identities of CP gene between SX and other AMV isolates
1.4 数据分析
将上述阳性克隆送至生工生物工程(上海)股份有限公司进行测序,利用NCBI 中的BLAST 软件(http://blast.ncbi.nlm.nih.gov/Blast.)进行同源性比对分析,利用DNAMAN 9.0 软件和SDT version 1.2 软件对序列进行拼接和比对分析,使用MEGA 7.0 软件以邻接法(Neighbor-joining)构建系统进化树,可信度设置为1000 次自导复制验证。采用Excel 2016 绘制柱状图。
2 结果与分析
2.1 紫花苜蓿样品采集及小RNA 深度测序鉴定
采集的紫花苜蓿病害样品叶片呈典型的花叶、皱缩、卷叶等症状(图1)。紫花苜蓿样品经小RNA 深度测序后,共获得11037662 条原始序列(raw reads),去除raw reads 中低质量序列共获得长度在18~28 nt 的clean reads数为7882612 条,占clean reads 的百分比为71.42%(图2)。将获得的contigs 过滤掉宿主基因组序列后与NCBI数据库进行比对,共匹配到3 种植物病毒:苜蓿花叶病毒、苜蓿矮化病毒和豌豆线条病毒,共计117 条contigs 序列,其中AMV 相关序列49 条contigs,ADV 相关序列11 条contigs,PeSV 相关序列57 条contigs。所有组装序列分别在AMV、ADV 和PeSV 的病毒基因组上标示(图3),其中PeSV 相关序列比例最高,占49%,其他依次为AMV(42%)和ADV(9%)。在对高通量测序结果中一些较短的contigs 进行序列比对时,发现有5 条contigs 能够比对到苜蓿卷叶病毒。

图1 田间紫花苜蓿感病症状Fig.1 Symptoms of M. sativa plant in fields

图2 sRNA 长度分布Fig.2 Small RNA length distribution
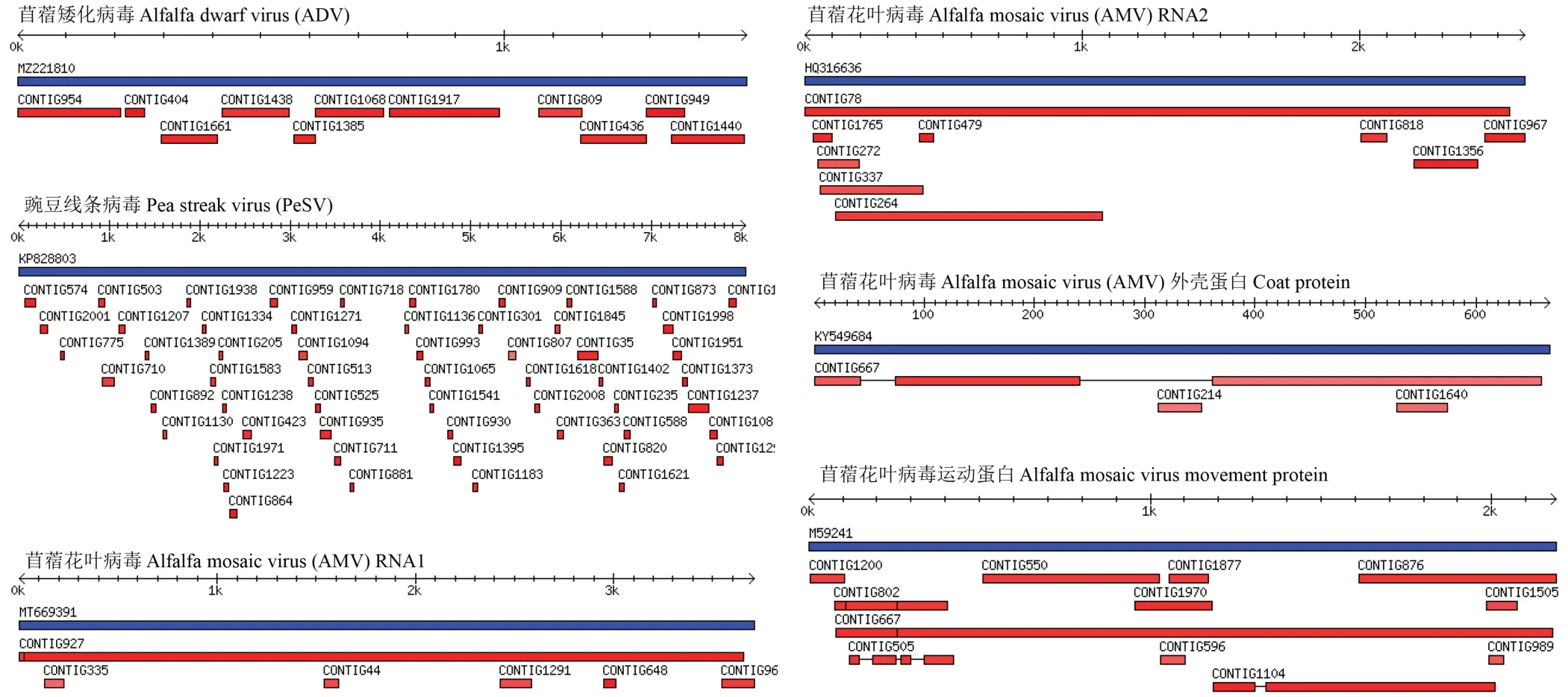
图3 紫花苜蓿3 种病毒组装序列在基因组中的位置和分布Fig.3 Position and distribution of 3 M. sativa viruses assembly contigs in the genome
2.2 PCR 法检测紫花苜蓿病样病毒
由于ADV、AMV 和PeSV 是RNA 病毒,遂以紫花苜蓿样品RNA 反转录的cDNA 为模板,分别使用特异性引物 ADV-CP-F/ADV-CP-R、AMV-CP-F/AMV-CP-R 和PeSV-CP-F/PeSV-CP-R 进行PCR 扩增;而ALCV 为DNA 病毒,以紫花苜蓿样品DNA 为模板,使用特异引物ALCV-F/ALCV-R 进行PCR 扩增。结果得到与4 种苜蓿病毒病AMV、PeSV、ALCV和ADV 大小相近的目的条带(图4),将回收的目的条带进行测序。测序显示与小RNA 结果一致,说明小RNA 测序结果具有可靠性。
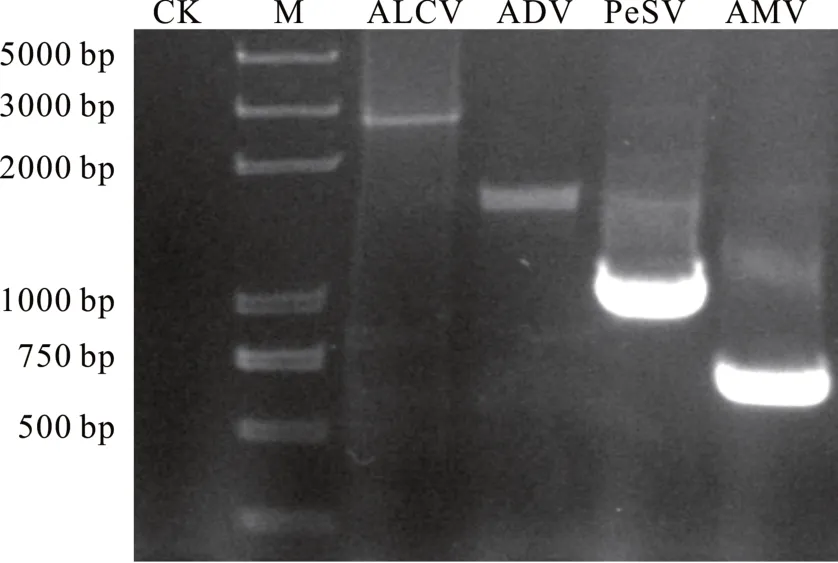
图4 感病样品的PCR 扩增Fig.4 PCR amplification from the sample
2.3 AMV 分离物(SX)外壳蛋白系统发育进化分析
将扩增获得的AMVCP基因序列上传至GenBank 获得登录号OP748369,命名为苜蓿花叶病毒山西紫花苜蓿分离物(AMV-SX),并且将其CP基因与来自中国、阿根廷、伊朗、意大利、塞尔维亚和美国等18 个国家的AMV 外壳蛋白分离物进行氨基酸和核苷酸的相似性分析(表 2)。结果表明,AMV-SXCP基因编码的氨基酸与其他AMVCP基因编码的氨基酸同源性为97.25%~99.54%,氨基酸同源性最高的分离物为AMV-ACat(MW835977);AMV-SXCP基因与其他AMVCP基因核苷酸同源性为96.85%~99.24%,核苷酸同源性最高的分离物为AMV-ACat(MW835977)。
为了进一步明确AMV-SX 外壳蛋白序列的分子进化关系,利用MEGA 7.0 软件对AMV-SXCP基因编码的氨基酸序列与已报道的其他AMV 分离物进行系统发育分析。本研究获得的AMV 山西紫花苜蓿分离物 SX 与 AMV 阿根廷苜蓿分离物 ACat(MW835977)亲缘关系最近,聚为一簇(图5);但是与我国其他地区分离物如Yuanyang-1/H2(OL706257)、See-1(MT093211)距离较远,亲缘关系较远。
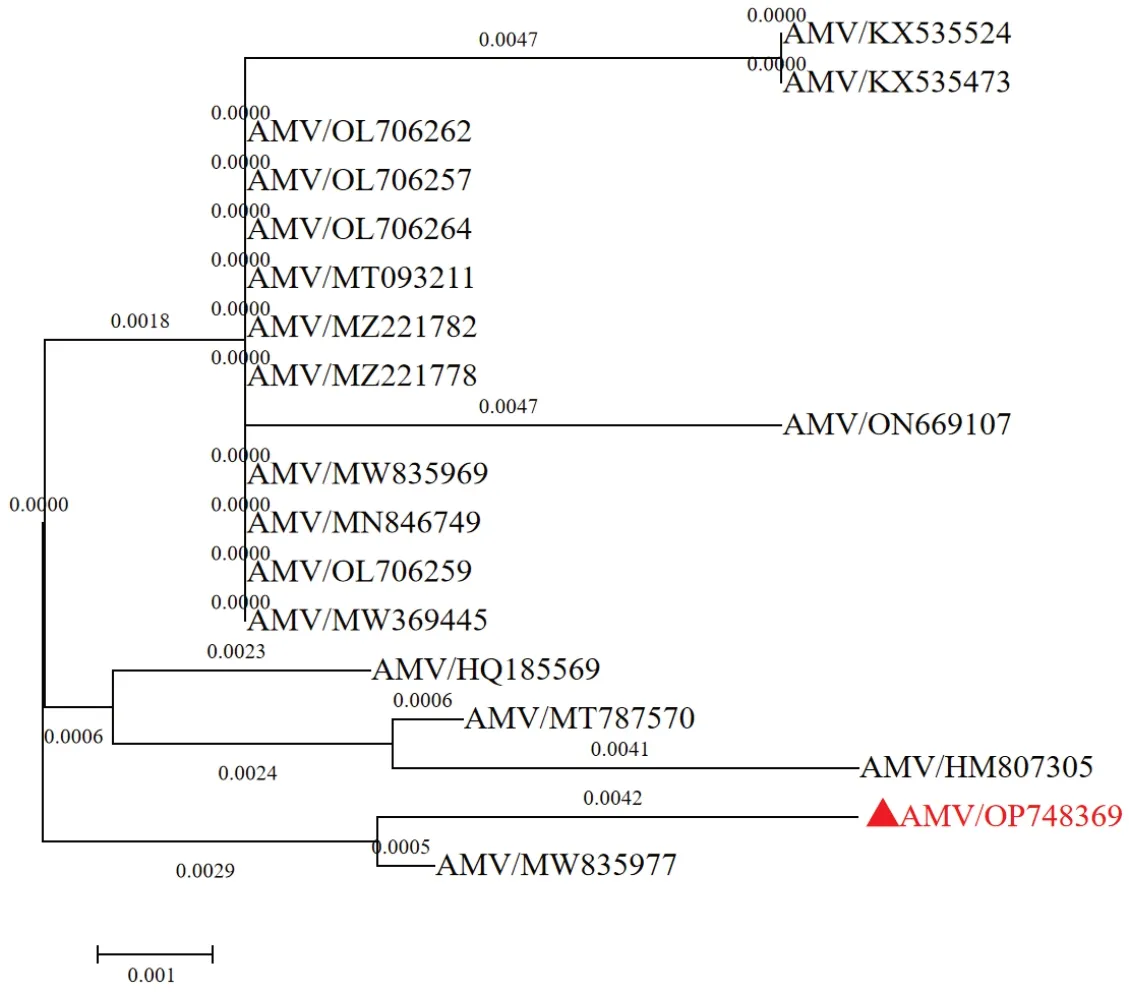
图5 AMV-SX 外壳蛋白氨基酸系统发育分析Fig.5 Phylogenetic analysis of AMV-SX CP amino acid
2.4 ADV 分离物(SXJZ)外壳蛋白系统发育进化分析
同样将扩增获得的ADV 外壳蛋白序列上传至GenBank 获得登录号OP957285,命名为苜蓿矮化病毒山西紫花苜蓿分离物(ADV-SXJZ),并且将CP基因与来自中国、阿根廷的27 个ADV 外壳蛋白分离物进行氨基酸相似性分析。结果表明,ADV-SXCP基因编码的氨基酸与其他ADVCP基因编码的氨基酸同源性为91.34%~99.43%,氨基酸同源性最高的ADV 分离物为N1(MZ221810)(图6)。
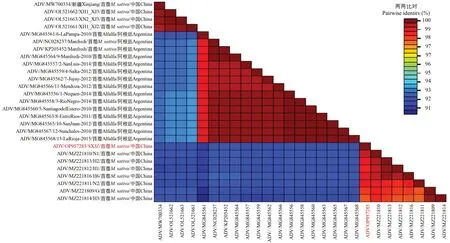
图6 ADV-SXJZ 外壳蛋白氨基酸相似性比对Fig.6 Pairwise amino acid sequence identity of ADV-SXJZ CP to other ADV
为了分析ADV 山西紫花苜蓿分离物(SXJZ)CP基因编码的氨基酸与其他ADV 分离物的分子进化关系,采用MEGA 7.0 软件进行系统发育分析(图7)。ADV 山西紫花苜蓿分离物(SXJZ)CP基因编码的氨基酸与中国ADV 分离物聚集在一个大的分支,并且与分离物ADV-N1(MZ221810)亲缘关系最近,聚为一簇。

图7 ADV-SXJZ 外壳蛋白氨基酸系统发育分析Fig.7 Phylogenetic analysis of ADV-SXJZ CP amino acid
2.5 ALCV 分离物(SXTG)全基因组序列一致性和系统发育分析
利用设计的背靠背引物(ALCV-F/ALCV-R)扩增获得ALCV-SXTG 基因组全长序列,测序拼接后长度为2751 nt,这是首次在山西的紫花苜蓿样品上扩增获得ALCV 全基因序列。ALCV 序列上传GenBank 获得登录号为OP748371。分析发现该基因组共编码7 个开放阅读框,分别为V4(113~403 nt),V2(118~264 nt),V3(225~677 nt),CP(523~1257 nt)、复制相关蛋白(replication-associated protein,1350~1733 nt,1870~2493 nt)、RepA(1697~2493 nt)和C3(1866~2363 nt)。对ALCVSXTG 全长基因与来自阿根廷、西班牙、伊朗、法国、意大利和中国的29 个ALCV 分离物进行核苷酸相似性比较,结果表明ALCV-SXTG 全长基因与ALCV 其他株系核苷酸同源性为75.88%~97.34%(图8),其中与ALCV 阿根廷苜蓿分离物Colonia Dora(MG792026)的序列一致性最高,为97.34%,与ALCV 西班牙苜蓿分离物ES36(MH603849)的序列一致性最低,为75.88%。

图8 ALCV-SXTG 核苷酸相似性比对Fig.8 Pairwise amino acid sequence identity of ALCV-SXTG to other ALCV
为了进一步明确ALCV-SXTG 的分子进化关系,利用MEGA 7.0 软件对ALCV-SXTG 与已报道的其他ALCV 分离物进行系统发育分析(图9)。 ALCV-SXTG 与来自中国和阿根廷的ALCV 分离物Colonia Dora (MG792026)亲缘关系较近,聚为一大簇;而与法国、西班牙、伊朗的苜蓿ALCV 分离物距离较远,亲缘关系较远。

图9 ALCV-SXTG 基因全长系统发育分析Fig. 9 Phylogenetic analysis on genome sequence of ALCVSXTG and other reported isolates
2.6 PeSV 分离物(SXJZ)外壳蛋白系统发育进化分析
将PCR 扩增获得的PeSV-SX 外壳蛋白序列上传至GenBank 获得登录号OQ108501,命名为豌豆线条病毒山西紫花苜蓿分离物(PeSVSXJZ),并且CP基因编码的氨基酸与其他PeSV分离物外壳蛋白编码的氨基酸相似性达到100%。而与其他香石竹潜隐病毒属(Carlavirus)成员相似性较低(图10)。
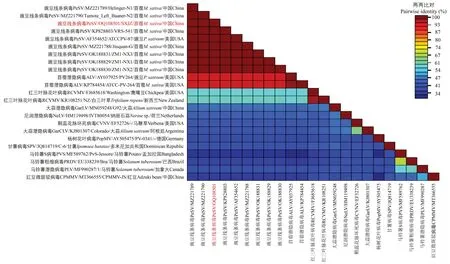
图10 PeSV-SXJZ 外壳蛋白核苷酸相似性比对Fig.10 Pairwise amino acid sequence identity of PeSV-SXJZ CP gene to other PeSV
进一步的分子进化关系显示,山西紫花苜蓿PeSV分离物SXJZ 与已报道的其他PeSV 分离物聚集于一簇,而与其他Carlavirus成员距离较远(图11)。
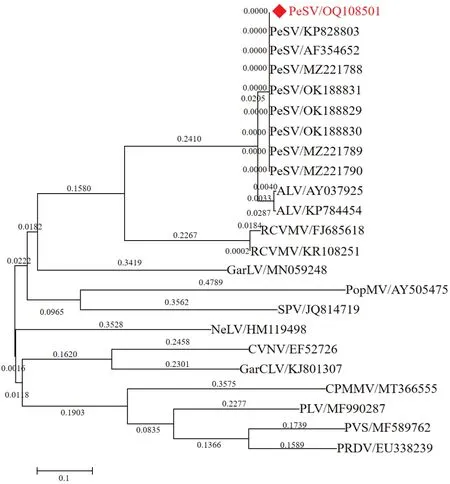
图11 PeSV-SXJZ 外壳蛋白氨基酸系统发育分析Fig. 11 Phylogenetic analysis of genome sequence of PeSV-SXJZ CP amino acid
3 讨论
紫花苜蓿是营养价值极高的一类牧草,在畜牧业生产中有着极为重要的作用,因此了解紫花苜蓿病毒病的发生状况及防治对策迫在眉睫。本研究通过sRNA 深度测序技术,发现山西农业大学植物园内种植的紫花苜蓿被苜蓿卷叶病毒、豌豆线条病毒、苜蓿花叶病毒和苜蓿矮化病毒4 种病毒复合侵染。这是首次在山西地区种植的紫花苜蓿样品中检测到上述4 种病毒的复合侵染。
AMV 是雀麦花叶病毒科(Bromoviridae)苜蓿花叶病毒属(Alfamovirus)成员[9]。AMV 寄主广泛,可以侵染上百种双子叶植物,除了通过蚜虫传播外,还能够以种子、花粉及汁液摩擦等方式传播[10]。紫花苜蓿是AMV 最为常见的寄主,紫花苜蓿被其侵染后会产生叶片的皱缩、花叶甚至是严重矮化等症状[11]。Trucco 等[4]从采自阿根廷的紫花苜蓿病样中扩增到了AMV 的全基因序列。我国也有研究人员发现甘肃张掖地区表现出黄斑花叶、叶柄扭曲及整株矮化症状的紫花苜蓿病原是AMV 和番茄花叶病毒[11]。本研究从采自山西农业大学植物园内感病的紫花苜蓿中同样检测到AMV的侵染,对外壳蛋白序列进行比对和进化分析发现,AMV 山西紫花苜蓿分离物与阿根廷紫花苜蓿分离物ACat(MW835977)亲缘关系最近,但是与我国其他地区AMV 分离物亲缘关系较远,说明本研究AMV 分离物与其他地区AMV分离物来源不同,分析可能为带病种子传播所致。
ADV 是负义链RNA 病毒,为弹状病毒科(Rhabdoviridae) 细 胞 质 弹 状 病 毒 属(Cytorhabdovirus)成员[12]。Bejerman 等[13]发现引起阿根廷紫花苜蓿节间缩短、叶片耳突等症状的病毒病原为ADV。随后Samarfard 等[14]也从阿根廷的病样中检测到ADV 的侵染。热甫卡提等[15]利用小RNA 测序技术从36 份采自新疆表现矮化、丛枝的紫花苜蓿样品中检测到ADV 的侵染。构建的系统进化树显示ADV 山西紫花苜蓿分离物SXJZ(OP957285)与同样来自中国的ADV 紫花苜蓿分离物N1(MZ221810)、H6(MZ221816)、H2(MZ221813)、H1(MZ221812)、N2(MZ221811)、H3(MZ221814)和G(MZ221809)聚为一支,亲缘关系较近。本研究是首次从山西紫花苜蓿样品中检测到ADV 的侵染。
苜蓿卷叶病毒是双生病毒科(Geminiviridae)大戟潜隐病毒属(Capulavirus)成员[16]。Guo 等[17]2019 年在河南种植的紫花苜蓿上检测到ALCV 的侵染。Davoodi 等[16]通过系统发育分析发现,ALCV 最有可能起源于伊朗,并在中东多样化,然后传播到地中海盆地,之后再传播到阿根廷。将ALCV 山西紫花苜蓿分离物SXTG(OP748371)全基因组序列进行相似性和进化分析,结果显示SXTG(OP748371)与ALCV 阿根廷苜蓿分离物Colonia Dora(MG792026)亲缘关系最近,达到97.34%,与ALCV 中国分离物和ALCV 阿根廷分离物聚集为一个大支,而与ALCV 法国、西班牙和伊朗分离物亲缘关系较远。进一步的研究表明,ALCV 侵染往往伴随有AMV的复合侵染现象[8]。本研究利用小RNA 测序技术和RT-PCR 的方法同时在山西紫花苜蓿上检测到AMV 和ALCV 的侵染,这与Guo 等[17]的研究结果是一致的。本研究是首次在山西紫花苜蓿上检测到ALCV 的侵染。
PeSV 是乙型线形病毒科(Betaflexiviridae)香石竹潜隐病毒属(Carlavirus)成员[18]。有研究发现在美国豌豆(Pisum sativum)广泛种植区检测到PeSV 的侵染,不仅会导致豌豆的茎和叶柄产生紫褐色条纹,还造成静脉坏死和畸形[19]。目前已在多种豆科植物上,如紫花苜蓿、红三叶草和蚕豆上检测到了PeSV 的侵染[19]。PeSV 山西紫花苜蓿分离物(SXJZ)进化分析表明,本研究中扩增到的PeSV-SXJZ 与其他的PeSV 分离物聚集为一个分支,并且PeSV-SXJZ 外壳蛋白氨基酸序列与其他PeSV 分离物的外壳蛋白氨基酸序列相似性达到了100%。本研究中PeSV 是首次从山西采集的紫花苜蓿上检测到该病毒的侵染。
紫花苜蓿病毒的复合侵染现象非常普遍,Alshahwan 等[20]在沙特阿拉伯紫花苜蓿样品中检测到了AMV、CMV、WCMV 和BLRV 的复合侵染。Massumi 等[21]对伊朗田间紫花苜蓿病毒病害检测发现有AMV、BYMV、花生矮化病毒(peanut stunt virus, PSV)和BLRV 4 种不同的病毒。本研究中,利用小RNA 测序技术并结合RTPCR 的方法对采自山西农业大学植物园内的感病紫花苜蓿样品进行病毒病原鉴定,共检测出AMV、ADV、ALCV 和PeSV 四种病毒。这是首次在山西省内的紫花苜蓿上检测到AMV、ALCV、ADV 和PeSV 四种病毒的复合侵染。本研究结果不仅丰富了侵染紫花苜蓿的病毒病原信息,还将会为紫花苜蓿病毒病的有效防控及抗病毒育种提供一定的理论依据。
4 结论
本研究利用小RNA 深度测序技术结合RT-PCR/PCR 的方法对采自山西农业大学植物园中表现花叶、皱缩、卷曲的紫花苜蓿样品进行病毒病原的鉴定,发现导致样品症状产生的病毒病原为AMV、PeSV、ADV 和ALCV四种病毒。本研究是首次从种植于山西的紫花苜蓿上检测到AMV、ADV、ALCV 和PeSV 的复合侵染,该研究结果有助于深入了解侵染紫花苜蓿的病毒病分子进化,为紫花苜蓿抗病毒育种提供了一定的理论基础。
