基于液相色谱-串联质谱分子网络的牡蛎共生真菌Myceliophthora lutea ML-1发酵液的化学成分分析
2023-12-02陈春颖丁木子王凤舞朱小丽
陈春颖, 陶 超, 丁木子, 王凤舞, 朱小丽, 申 丽
(1. 扬州大学医学院, 江苏 扬州, 225009; 2. 江苏省中西医结合老年病防治重点实验室, 江苏 扬州, 225009;3. 青岛农业大学 食品科学与工程学院, 山东 青岛, 266109)
微生物来源的天然药物是新药及其先导化合物的重要来源,特殊生态环境下的微生物与特殊的次生代谢产物密切相关,因此微生物源新药或先导化合物的研究更多地集中于特殊生境微生物[1-2]。生活在海洋环境中的真菌和细菌,在寡营养、低温、高压、高盐、无光照等恶劣条件下,常形成特殊的代谢途径,易产生结构新颖、活性显著的次生代谢产物[3]。研究[4]发现,海洋真菌和细菌与海洋动物(节肢动物、刺胞动物、软体动物等)存在密切的共生关系,海洋共生微生物很可能是多种海洋生物活性成分的真正产生者。海洋动物共生菌作为海洋微生物药物的重要研究方向之一,为新药研发提供了丰富的物质基础。毁丝霉(Myceliophthora)为半知菌类真菌,在自然界中广泛分布。Myceliophthora属真菌根据分子鉴定和表型可分为4个分支,即Myceliophthora、Corynascus、Crassicarpon和Thermothelomyce[5], 其中Crassicarponthermophilum和Thermothelomycespp. 为嗜热真菌,其能产生非常丰富的酶系。目前,关于Myceliophthora属真菌的研究大多集中在其所产生的耐热木聚糖酶[6]、淀粉酶[7]、纤维素酶[8]等热稳定性酶方面,次生代谢产物相关研究则极少。前期实验[9]从长岛海域牡蛎中分离得到共生真菌MyceliophthoraluteaML-1(M.luteaML-1), 本研究基于液相色谱-串联质谱(LC-MS/MS)分子网络技术研究其发酵液的化学成分,以期明确M.luteaML-1的次生代谢产物谱,为挖掘新颖结构药物先导化合物提供依据。
1 材料与方法
1.1 仪器与材料
Atlantis T3色谱柱(2.1 mm×150 mm, 3 μm), 美国安捷伦科技有限公司; 色谱甲醇,美国TEDIA化学试剂有限公司; 色谱甲酸,美国ACS恩科化学公司; D-山梨醇,国药集团化学试剂有限公司; D-(+)-麦芽糖一水合物,国药集团化学试剂有限公司; 酵母膏,国药集团化学试剂有限公司; L-色氨酸,阿拉丁生化科技有限公司; 海盐,中国盐业集团有限公司; 其他试剂为分析纯。
TripleTOF 4600液-质联用仪(美国AB SCIEX有限公司); SHP-250恒温培养箱(上海精宏实验设备有限公司); SKY-200B恒温振荡摇床(上海苏坤实业有限公司); DW-HL388超低温冰箱(中科美菱低温科技有限责任公司)等。
1.2 菌株发酵和发酵产物提取方法[10]
M.luteaML-1是从长岛海域牡蛎中分离得到的共生真菌[9](Genbank号: ON005439), 菌株保存于扬州大学医学院天然药物化学研究室。使用真菌1号培养基(山梨醇20 g、麦芽糖20 g、味精10 g、KH2PO40.5 g、MgSO4·7H2O 0.3 g、色氨酸0.5 g、酵母膏3 g、海盐30 g、水1 000 mL)进行菌株小量发酵。从M.luteaML-1冻存管中挑取少量菌体接入PDA平板, 28 ℃培养2~3 d; 挑取菌块接入500 mL三角瓶中(200 mL PD培养基), 28 ℃、120转/min培养3 d; 再将种子液接入1 000 mL三角瓶中(400 mL真菌1号培养基), 28 ℃、120转/min培养15 d。发酵液经乙酸乙酯萃取3次,合并萃取液,减压蒸馏去除溶剂,得到粗浸膏。发酵液粗浸膏经色谱甲醇溶解、0.22 μm微孔滤膜过滤后,进行LC-MS/MS分析。
1.3 发酵液粗浸膏的LC-MS/MS分析[10]
高效液相色谱(HPLC)条件: Atlantis T3柱(2.1 mm× 150 mm, 3 μm), 流动相A(0.1%甲酸溶液),流动相B(甲醇),梯度洗脱(0~10 min, 80%→20% A; 10~20 min, 20%→1% A; 20~32 min, 1%→1% A), 流速0.3 mL/min, 柱温40 ℃, 进样量5 μL。
四极杆飞行时间串联质谱(Q-TOF-MS/MS)条件: 电喷雾离子源; 正离子模式; 气帘气压力35 kV; 雾化温度550 ℃; 雾化气55 Psi, 辅助气55 Psi; 喷雾电压5 500 V; 去簇电压80 eV; 碰撞电压35 eV; 全扫描模式,一级母离子扫描范围100~2 000, 二级子离子扫描范围50~2 000。
1.4 经典分子网络(CMN)构建和化学成分识别
原始质谱数据经MSconvert软件转换为.mzXML格式文件,再上传至全球天然产物社会分子网络(GNPS)平台构建CMN。参数设置: 碎片离子质量误差值为0.02 Da, 最小余弦值为0.6, 最小碎片匹配峰为6。将生成的CMN导入Cytoscape软件进行可视化,同时可利用GNPS平台“MS/MS Library”检索功能快速识别潜在化学成分,设置最小匹配峰为6和最小匹配分数为0.7。
1.5 特征峰分子网络(FBMN)构建和相对定量
原始质谱数据经MSconvert软件转换为.mzXML格式文件后,导入MZmine 2软件进行原始谱图的识别、提取、对齐、去噪、分组等处理,再以.mgf和.csv格式文件导出,并上传至GNPS平台的“FBMN”模块中构建FBMN。参数设置: 前体离子质量误差值为0.02 Da, 碎片离子质量误差值为0.02 Da。将生成的FBMN导入Cytoscape软件进行可视化。FBMN可通过LC-MS特征丰度(峰面积或峰高度)更准确地估计相对离子强度,因此能进行化学成分的相对定量[11]。FBMN中,节点大小可直观反映化合物的相对含量。
2 结 果
2.1 基于CMN的发酵液化学成分识别和结构预测
应用CMN进行大规模数据集的元分析,可以方便快捷地分析LC-MS2数据。对M.luteaML-1发酵液粗浸膏的LC-MS/MS谱进行处理后,上传到GNPS平台构建CMN, 见图1、图2。该分子网络共有1 282个节点,其中节点数≥3的分子簇有39个,见图2。利用GNPS平台的"MS/MS Library"功能搜库,将初步匹配的化合物与平台二级质谱库数据进行比对,结果识别出潜在的化学成分39个; 在此基础上,根据分子簇中各节点间的质荷比和MS2碎片离子关系又进一步预测出10个化学成分的结构,见图2、表1。已识别和预测出的化合物包括18个脂肪酸类衍生物(分子簇A, 化合物1~18)、8个蒽醌类化合物(分子簇B, 化合物19~20,23~26; 2个单节点簇,化合物21和22)、12个环二肽类化合物(分子簇C, 化合物27~38)及11个其他类型化合物(化合物39~49), 见图2。研究[11]发现,在非靶向LC-MS2数据采集过程中,同一前体离子在色谱洗脱过程中经常被多次碎片化,虽然MS-Cluster通常能够将这些光谱聚类到CMN中的1个节点中,但在某些情况下,会产生代表同一化合物的多个节点。此外,由于算法的原因,分析结果会出现结构类似物并未聚集在同一个分子簇的现象[12]。

图1 牡蛎共生真菌M.lutea ML-1发酵液粗浸膏的总离子流图
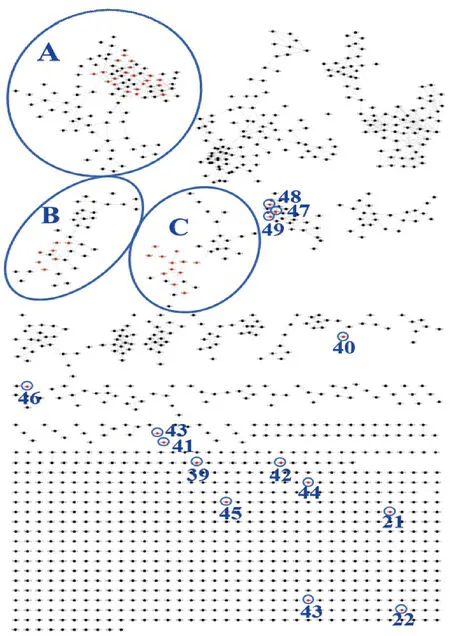
A: 脂肪酸类化合物; B: 蒽醌类化合物; C: 环二肽类化合物。红色节点为已识别或预测出的化合物。
直接利用GNPS平台的"MS/MS Library"功能, 快速发现菌株ML-1发酵液中存在15个脂肪酸类化合物(化合物1~15), 其中化合物1~10分别为Linolenic acid(1)、13-hydroxyoctadeca-6, 9, 11-trienoic acid(2)、9, 12-Octadecadienoicacid(3)、1-stearoyl-sn-glycerol(4)、Monopalmitin(5)、2, 3-Dihydroxypropyl 9, 12, 15-octadecatrienoate(6)、1-Monolinolein(7)、elaidic acid(8)、13-Docosenamide(9)、Oleamide(10)。分析分子网络图发现, 节点m/z293.248 0([M+H-H2O]+,tR=14.51 min)、m/z339.289 0([M+H-H2O]+,tR=16.98 min)、m/z295.264 9([M+H-H2O]+,tR=17.58 min)、m/z381.301 2([M+H-H2O]+,tR=17.76 min)和m/z263.236 7([M+H-H2O]+,tR=16.4 min)分别与化合物methyl 13-hydroxyoctadeca-9, 11-dienoate、Glyceryl monooleate、methyl 12-hydroxyoctadec-9-enoate、1-oleoyl-2-acetoyl-sn-glycerol和10, 12-Octadecadienoic acid具有相同的二级质谱, 故确定其分别为methyl 13-hydroxyoctadeca-9, 11-dienoate(11)、Glyceryl monooleate(12)、methyl 12-hydroxyoctadec-9-enoate(13)、1-oleoyl-2-acetoyl-sn-glycerol(14)和10, 12-Octadecadienoic acid(15); 比对二级质谱发现,节点m/z357.300 6([M+H]+,tR=16.95 min)与m/z339.289 0([M+H-H2O]+,tR=16.98 min)为同一化合物Glyceryl monooleate(12)。在此基础上,依据分子网络提示的相邻节点之间的结构关系进行其他节点化合物的结构预测,结果又识别出3个化合物。节点m/z309.241 3([M+H]+,tR=14.35 min)比节点m/z295.225 7([M+H]+,tR=12.22 min, 化合物2)多1个CH2, 同时比节点m/z293.248 0([M+H-H2O]+,tR=14.51 min, 化合物11)少2个H, 结合二级质谱可确定其为化合物Methyl 13-oxo-9, 11-octadecadienoate(16); 节点m/z341.306 1([M+H-H2O]+,tR=18.26 min)比节点m/z339.289 0([M+H-H2O]+,tR=16.98 min,12)多2个H原子, 结合二级质谱推测其为化合物1-Monostearin(17); 节点m/z280.264 5([M+H]+,tR=15.27 min)与节点m/z281.248 1([M+H]+,tR=16.4 min, 化合物3)质荷比相差1, 同时比m/z282.279 9([M+H]+,tR=16.43 min, 化合物10)少2个H原子,可能是化合物3结构中的羧基变为酰胺基,结合二级质谱确定其为化合物9, 11-Octadecadienamide(18)。分子簇中某些节点分子的提示信息较少,仅靠二级碎片无法确定结构,有待进一步鉴定。见表1-1、图3。
表1-1 牡蛎共生真菌M.lutea ML-1发酵液的脂肪酸类化学成分

图3 分子网络中的脂肪酸类化合物分子簇
运用"MS/MS Library"检索功能,快速发现菌株ML-1发酵液分子网络中的蒽醌类化合物分子簇。节点m/z543.127 7([M+H]+,tR=10.77 min)、m/z节点575.119 4([M+H]+,tR=7.95 min)、m/z节点285.075 9([M+H]+,tR=13.73 min)和节点m/z315.050 4([M+H]+,tR=9.11 min)分别与GNPS平台谱库收录的蒽醌类化合物Rugulosin A、(-)-Luteoskyrin、1, 8-dihydroxy-3-methoxy-6-methylanthracene-9, 10-dione和Endocrocin的二级质谱基本一致,故确定上述节点化合物为Rugulosin A(19)、(-)-Luteoskyrin(20)、1, 8-dihydroxy-3-methoxy-6-methylanthracene-9, 10-dione(21)和Endocrocin(22)。依据相邻节点之间的结构关系,对节点化合物进行结构预测,又识别出4个蒽醌类化合物: ① 节点m/z559.124 1([M+H]+,tR=9.21 min)比节点m/z543.127 7(Rugulosin A, 化合物19)质荷比大16(多1个O原子),其结构有3种可能,分别为Rugulosin A(19)的C-6、C-8、C-11羟基化衍生物; 其比节点m/z575.119 4[(-)-Luteoskyrin, 化合物20]质荷比小16(少1个O原子),也存在3种结构可能,即 (-)-Luteoskyrin(20)的C-2、C-5、C-8脱羟基化衍生物; 综合上述结构信息,并结合二级质谱裂解规律,推测其为化合物19的C-8位羟基化衍生物(+)-Deoxyluteoskyrin(23)。② 同理推测节点m/z591.113 7([M+H]+,tR=5.55 min, 化合物24)是节点m/z575.119 4(-)-Luteoskyrin, 化合物20)的一氧化物,其有4种可能结构,即(-)-Luteoskyrin(20)的C-2、C-4a、C-6、C-11羟基化衍生物; 以分子式C30H22O13检索SciFinder数据库发现, (-)-Luteoskyrin(20)的C-4a羟基化衍生物(-)-4α-Oxyluteoskyrin已有报道,但化合物24是否为该化合物有待进一步确定。③ 节点m/z[M+H]+573.101 9(C31H24O11,tR=10.07 min, 化合物25)和节点m/z[M+H]+589.099 5(C31H24O12,tR=8.63 min, 化合物26)分别为化合物(+)-Deoxyluteoskyrin(化合物23, C30H22O11)和化合物(-)-Luteoskyrin(化合物20, C30H22O12)的甲基化衍生物。用化合物25、26的分子式检索SciFinder数据库,未发现符合二蒽醌结构特征的化合物,提示这2个化合物可能为新二蒽醌类化合物。因二蒽醌化合物的质谱裂解规律比较复杂,仅靠二级质谱信息暂时无法确定化合物25、26的具体结构。见图4、表1-2。

表1-2 牡蛎共生真菌M.lutea ML-1发酵液的蒽醌类化学成分

图4 分子网络图中的蒽醌类化合物所在分子簇
运用"MS/MS Library"功能进行化合物匹配,快速发现菌株ML-1发酵液分子网络中的环二肽类化合物分子簇,经与GNPS平台谱库收录的二级质谱进行比对,快速识别出9个节点化合物,分别为cyclo(Leu-Pro)(27)、cyclo(Val-Pro)(28)、cyclo(Phe-Pro)(29)、cyclo(Leu-Phe)(30)、cyclo(Phe-4-hydroxy-Pro)(31)、cyclo(L-Leu-trans-4-hydroxy-L-Pro)(32)、cyclo(Val-Phe)(33)、cyclo(Leu-Val)(34)和cyclo(Tyr-Pro) (35)。根据分子网络提示的节点化合物的分子式以及相邻节点的结构关系,结合环二肽类化合物的裂解规律[13-15], 进一步将节点m/z263.139 2([M+H]+,tR=3.92 min)、节点m/z185.128 5([M+H]+,tR=3.77 min)和节点m/z227.176 9([M+H]+,tR=7.46 min)分别鉴定为化合物cyclo(Val-Tyr)(36)、cyclo(Ala-Leu)(37)和cyclo(Leu-Ile)(38), 见表1-3、图5。

表1-3 牡蛎共生真菌M.lutea ML-1发酵液的环二肽类化学成分

图5 分子网络图中环二肽类化合物所在分子簇
除脂肪酸类、蒽醌类、环二肽类化合物,本研究还识别出11个其他结构类型化合物,具体包括: 邻苯二甲酸酯类, dibutyl phthalate(39)和dioctyl phthalate(40); 麦角甾醇类, ergosterol-5, 8-peroxide(41); 香豆素类, 1H-2-Benzopyran-1-one(42); 喹啉类, 4-Hydroxy quinolie(43)和4-Quinolinecarboxylic acid(44); 吡啶类, 3-Pyridinecarboxylic acid(45); 仲胺, N-benzyl-1-phenylmethanamine(46); 聚醚类, Nonaethylene glycol(47)、EG-10(48)和undecaethylene glycol(49)。见表1-4。

表1-4 牡蛎共生真菌M.lutea ML-1发酵液的其他类型化学成分
2.2 基于FBMN的发酵液化学成分分析
利用GNPS平台的“Feature Networking”模块构建菌株发酵液的FBMN, 见图6。FBMN共有649个节点,比CMN少633个,结合分子网络快速识别出41个化合物; 节点数≥3的分子簇有14个,比CMN少25个。FBMN构建前的原始质谱数据处理,在剔除仪器、杂质离子和系统误差等因素影响的同时,还会过滤掉一些质谱信息,因此FBMN的节点和分子簇比CMN少。构建FBMN需使用适当的处理步骤和参数,否则会影响分子网络的分析结果; 此外, CMN使用光谱计数或前体离子计数,而FBMN可通过LC-MS特征丰度(峰面积或峰高度)更准确地估计相对离子强度,从而实现分子网络化学成分的相对定量[11]。本研究利用FBMN进行发酵液化学成分的相对定量,结果发现,节点m/z317.245 5(tR=18.63 min)、m/z319.225 0(tR=12.96 min)、m/z763.179 0(tR=24.75 min)、m/z837.198 8(tR=26.91 min)、m/z279.232 5(tR=12.96 min)、m/z321.240 5(tR=13.81 min)、m/z393.298 1(tR=18.01 min)、m/z303.229 7(tR=16.91 min)、m/z235.097 1(tR=6.56 min)和m/z211.144 5(tR=4.69 min)为相对含量排名前10位的化学成分。其中,节点m/z279.232 5(tR=12.96 min)、m/z235.097 1(tR=6.56 min)和m/z211.144 5(tR=4.69 min)分别被鉴定为脂肪酸类化合物Linolenic acid(1)的顺反异构体(两者的二级质谱相同,但保留时间有明显差异)、香豆素类化合物1H-2-Benzopyran-1-one(42)和环二肽类化合物cyclo(Leu-Pro)(27), 其余7个节点分子的结构有待鉴定,见图6、图7。该分析结果明确了菌株ML-1采用真菌1号培养基发酵时的主要次生代谢产物。
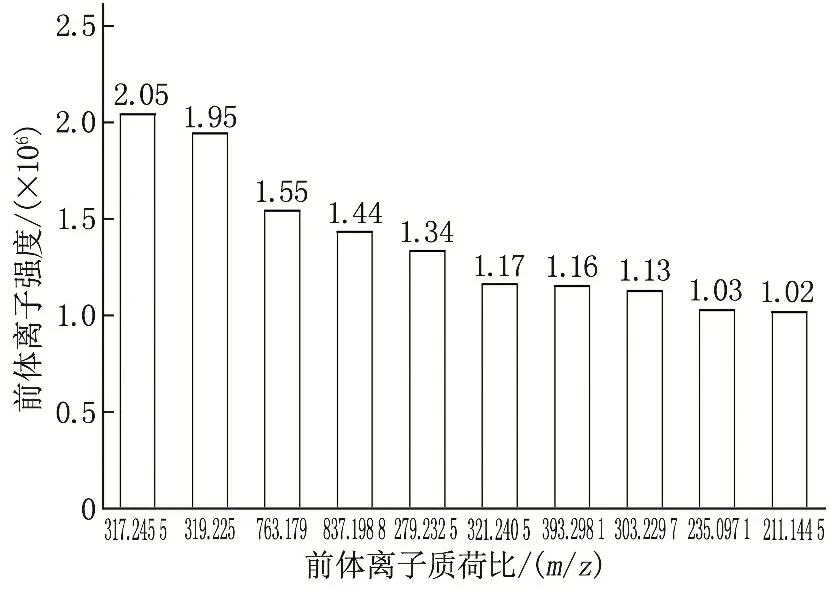
图7 M.lutea ML-1发酵液中的主要次生代谢产物(相对含量排名前10位)
3 讨 论
Index fungorum网站(https://www.indexfungorum.org/Names.asp)统计显示,Myceliophthora属已被报道的种共有19个,去掉同物异名和无效种,被承认的种仅14个,即M.fergusii、M.guttulate、M.heterothallica、M.hinnulea、M.indica、M.lutea、M.novoguineensis、M.officinarum、M.sepedonium、M.setosus、M.sexualis、M.similis、M.thermophila和M.verrucosa[16]。但目前为止,仅M.thermophila、M.lutea和M.heterothallica有次生代谢产物的相关研究报道。GAO Y L等[17]从M.thermophilaATCC 42464中分离得到4个具有反式稠合十氢萘骨架的聚酮-氨基酸杂合化合物myceliothermophin A、B、E、F, 其中新化合物myceliothermophin F对人结肠癌细胞株DLD-1、人肝癌细胞株Hep3B、人肝癌细胞株HepG2、人胃癌细胞株HGC-27有显著抑制作用,半数抑制浓度(IC50)分别为0.48、0.89、0.80、0.33 μg/mL。NOZAWA O等[18-19]从M.luteaTF-0409中分离得到2个新的δ-内酯化合物Waols A、Waols B, 其具有广谱体外细胞毒活性,对阿霉素耐药人白血病细胞株HL-60/ADR、人白血病细胞株HL-60、小鼠白血病细胞株P388、人膀胱癌细胞株T-24、人宫颈癌细胞株Hela、人肺癌细胞株A549具有显著抑制活性,IC50分别为0.1、0.2、4.0、0.5、1.0、1.0 μg/mL和0.2、0.2、4.0、0.5、1.0、1.0 μg/mL。此外, Waols A和Waols B仅对金黄色葡萄球菌有较弱的抑制活性。SMETANINA O F等[20]从库页湾(鄂霍次克海)海洋沉积物来源的真菌M.lutea中分离得到2个新化合物isoacremine D和acremine A, 其中acremine A在光作用下会转化为spiroacremines A和spiroacremines B。这4个化合物对海胆精细胞具有体外细胞毒活性,浓度分别为40、50、15、30 μg/mL时,能使海胆精子的受精能力下降50%; isoacremine D在200 μg/mL时对金黄色葡萄球菌有抑菌活性,而acremine A、spiroacremines A和spiroacremines B对测试的革兰氏阳性菌和革兰氏阴性菌没有抗菌活性。深入开展Myceliophthora属真菌的次生代谢产物研究,能够为挖掘该属真菌的化学多样性和生物活性多样性提供科学依据,并为真菌资源的开发利用奠定基础。
2014年,基于LC-MS/MS的GNPS平台正式开放使用,这是一个基于分子网络和数据共享的信息平台,能帮助研究者快速识别天然化合物的分子特征,已成为研究微生物次级代谢组学的重要技术手段。本研究利用CMN对牡蛎共生真菌M.luteaML-1发酵液粗浸膏的化学成分进行分析,共识别和预测出49个化合物,包括18个脂肪酸类化合物、8个蒽醌类化合物、12个环二肽类化合物和11个其他类化合物,其中可能有2个新二蒽醌类化合物,初步明确了该菌株的次生代谢产物谱。本研究进一步利用FBMN进行相对定量分析发现了真菌1号培养基的主要次生代谢产物,目前已识别出其中的3个化合物,该结果为后续化学成分的导向分离提供了思路,也为通过优化发酵条件提高微量化合物的产量提供了依据。但由于GNPS平台收录的二级质谱数据有限,且可能存在新颖结构成分,目前仅从M.luteaML-1发酵液中识别及预测出不足5%的节点分子,后续考虑将“种子化合物”与菌株发酵产物共同构建分子网络,利用“种子化合物”的结构线索帮助识别出更多化学成分,从而阐明菌株的次级代谢产物谱,为挖掘发现结构新颖、活性显著的药物先导化合物奠定基础。
