甘蔗ShPR10基因的克隆及其编码蛋白与甘蔗线条花叶病毒P1蛋白的互作研究
2023-11-23黄佳艳冯小艳沈林波王文治胡海燕张树珍
黄佳艳 冯小艳 沈林波 王文治 胡海燕 张树珍
(1. 海南大学热带作物学院,海口 570228;2. 中国热带农业科学院热带生物技术研究所,海口 571101)
植物病程相关蛋白(pathogenesis related pro‑teins, PRs)是一类在植物受到逆境胁迫后诱导产生并积累的蛋白质的总称[1]。根据蛋白结构、理化性质和生物活性等特征,将PR蛋白分成17个家族,即PR1-PR17[2]。其中PR10是一类分子量小、等电点呈酸性、无信号肽的胞内蛋白[3]。PR10蛋白不仅参与植物对炎热、寒冷、干旱、盐碱、细菌、真菌等逆境胁迫的响应,还在植物抵抗病毒侵染过程中发挥重要作用[1-3]。辣椒(Capsicum annuum)CaPR10蛋白受烟草花叶病毒(Tobacco mosaic virus,TMV)诱导表达水平显著升高并发生磷酸化,增强了CaPR10的核酸酶活性,促使CaPR10对入侵的病毒RNA进行切割,从而达到抗病毒的作用[4]。烟草(Nicotiana tabacum)NtPR10基因应答TMV侵染,感、抗烟草品种在感染TMV后,其NtPR10表达水平整体呈现相反的变化趋势,表明NtPR10可能在TMV侵染过程中发挥作用[5]。岷江百合(Lilium regale)编码的LrPR10基因受黄瓜花叶病毒(Cucu‑mber mosaic virus, CMV)诱导上调表达,接种CMV 4 d后,LrPR10的表达水平达到最大值,为处理前的58倍,表明LrPR10可能在岷江百合抗CMV防御反应中发挥作用[6]。烟草NbPR10a基因受烟草曲茎病毒(Tobacco curly shoot virus, TbCSV)诱导上调表达,在烟草植株中沉默NbPR10a基因导致TbCSV积累量显著增加,表明NbPR10a具有抵抗TbCSV侵染的作用[7]。
甘蔗(Saccharum spp.)是重要的糖料作物和能源作物,甘蔗糖约占我国食糖总产量的90%[8-9]。甘蔗线条花叶病毒(Sugarcane streak mosaic virus,SCSMV)是引起甘蔗花叶病的主要病原之一,其侵染甘蔗导致叶片褪绿斑驳、植株矮化、分蘖变少,继而影响甘蔗的品质和产量[10-11]。SCSMV在我国各大蔗区广泛分布,对我国甘蔗产业的发展造成严重威胁[11]。其隶属马铃薯Y 病毒科(Potyviridae)禾本科病毒属(Poacevirus),基因组大小约10 kb,编码1个多聚蛋白,该蛋白进一步被蛋白酶水解成11个成熟的功能蛋白,从N端到C端依次是P1、HC‑Pro、P3、P3N‑PIPO、6K1、CI、6K2、VPg、NIa‑Pro、NIb和CP[12-14]。其中,P1蛋白是RNA沉默抑制子,能够抑制寄主的RNA沉默防御机制,从而帮助SCSMV成功侵染寄主[14-15]。
本课题组在前期研究中,以SCSMV编码的P1蛋白为诱饵,采用酵母双杂交(yeast two hybrid,Y2H)技术筛选获得一个与SCSMV P1蛋白互作的甘蔗PR10蛋白,命名为ShPR10[15]。在本研究中,首先利用同源克隆技术克隆ShPR10基因的完整开放阅读框(open reading frame, ORF)序列,并对其编码蛋白进行生物信息学和亚细胞定位分析;然后利用Y2H和双分子荧光互补(bimolecular fluorescence complementation, BiFC)技术验证ShPR10与SCSMV P1的互作关系;最后采用农杆菌共浸润瞬时表达系统和Western blot技术分析ShPR10对SCSMV P1沉默抑制子活性的影响。研究结果揭示ShPR10在甘蔗应答SCSMV侵染过程中的作用,为甘蔗抗SCSMV育种奠定基础。
1 材料与方法
1.1 材料
1.1.1 植物材料 甘蔗品种为ROC22,种植于中国热带农业科学院热带生物技术研究所海南文昌试验基地。野生型本氏烟草(Nicotiana benthamiana)种植于相对湿度60%、光照16 h/d、温度25℃的培养箱中。
1.1.2 主要试剂 EZNA Plant RNA Kit购自Omega BIO‑TEK公司;RevertAid First‑Strand cDNA Synthesis Kit购自Thermo Scientific公司;Nimble Cloning试剂盒购自海南壹田生物科技有限公司;HiPure Gel Pure DNA Mini Kit购自Magen公司;2× Magic Green Taq Mix购自TOLOBIO公司;大肠杆菌(Escherichia coli)DH5α感受态细胞购自北京全式金生物技术有限公司;Y2H Gold酵母(Saccharomyces cerevisiae)感受态细胞和农杆菌(Agrobacterium tumefaciens)GV3101感受态细胞购自上海唯地生物技术有限公司;Bsa I内切酶、2× EasyClone Mix和pBWA(V)HS‑ccdB载体购自武汉伯远生物科技有限公司;pNC‑GADT7、pNC‑BiFC‑Ecn、pNC‑BiFC‑Enn、pNC‑Cam33FN和pNC‑Cam33HN载体由中国热带农业科学院热带生物技术研究所言普研究员馈赠;P1‑18T、ShPR10‑18T、GFP‑18T、pGBKT7‑P1和pCam3304‑35S‑GFP质粒为课题组构建并保存。
1.1.3 主要仪器 Biometra TAdvanced PCR仪购自德国Analytikjena公司;琼脂糖凝胶电泳仪购自美国Bio‑Rad公司;凝胶成像分析仪器购自上海天能科技有限公司;LSM800激光共聚焦显微镜购自德国卡尔蔡司光学有限公司;长波紫外灯购自上海路阳仪器有限公司。
1.2 方法
1.2.1 RNA提取和cDNA第一链合成 称取 0.1 g ROC22叶片,用液氮研磨成粉末,按照EZNA Plant RNA Kit说明书的步骤提取甘蔗总RNA。cDNA第一链合成用RevertAid First‑Strand cDNA Synthesis Kit进行,产物置于‑20℃保存。
1.2.2 ShPR10基因的克隆及生物信息学分析 以酵母双杂交文库筛选获得的ShPR10基因序列和NCBI数据库中玉米(Zea mays, EU976710.1)、野生二粒小麦(Triticum dicoccoides, XM_037558368)及一粒小麦(Triticum monococcum, JX424309.1)的PR10序列为参考,设计ShPR10基因ORF序列的特异扩增引物ShPR10‑F和ShPR10‑R(表1)。以ROC22叶片cDNA为模板,参照冯小艳等[16]的方法克隆ShPR10基因,并送生工生物工程(上海)股份有限公司测序。

表1 本研究使用的引物Table 1 Primers used in this study
利用ProtParam(http://expasy.org/tools/protparam.html)预测ShPR10编码蛋白的理化性质;利用NP‑SA‑PRABI(https://www.so.com/s?ie=utf‑8&src=360se7_addr&q=NPSA‑PRABI)、TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)和 SignalP 5.0(http://www.cbs.dtu.dk /services /Signal P/)分别预测ShPR10的二级结构、跨膜特性和信号肽;利用Blastp在线工具查找ShPR10的同源氨基酸序列,使用DNAMAN软件进行同源氨基酸序列比对分析,并使用MEGA 11.0软件构建进化树。
1.2.3 ShPR10的亚细胞定位 以GFP‑18T质粒为模板,GFP‑p‑F和GFP‑p‑R(表1)为引物扩增绿色荧光蛋白GFP基因。以ShPR10‑18T质粒为模板,ShPR10‑p‑F和ShPR10‑p‑R(表1)为引物扩增ShPR10基因。两个基因扩增产物分别通过琼脂糖凝胶电泳后,切下目的条带置于同一EP管中进行回收。回收产物与经Bsa I酶切线性化的pBWA(V)HS‑ccdB载体进行重组,重组体系:线性化的pBWA(V)HS‑ccdB载体5 μL,回收产物5 μL,2× EasyClone Mix 10 μL,37℃反应30 h获得重组质粒pBWA(V)HS‑GFP‑ShPR10。将该重组质粒转入GV3101农杆菌感受态细胞中,参考尹梦莹等[17]的方法进行农杆菌侵染实验,将带有重组质粒的农杆菌注入本氏烟草叶片,48 h后在激光共聚焦显微镜下观察叶片中荧光蛋白的位置。
1.2.4 Y2H验证ShPR10与SCSMV P1的互作 以ShPR10‑18T质粒为模板,ShPR10‑JT‑F和ShPR10‑JT‑R(表1)为引物扩增ShPR10基因。按照Nimble Cloning试剂盒说明将ShPR10基因连接到pNC‑GADT7载体上,获得猎物质粒pNC‑GADT7‑ShPR10。将猎物质粒pNC‑GADT7‑ShPR10与课题组保存的诱饵质粒pGBKT7‑P1共同转化Y2H Gold酵母菌株,转化产物涂布于SD/‑Trp/‑Leu平板上,28℃培养至长出单菌落。挑取单菌落于SD/‑Trp/‑Leu液体培养基中培养至OD600值为1.0,用ddH2O将菌液进行10倍系列稀释,取4 μL分别点到SD/‑Trp/‑Leu和SD/‑Trp/‑Leu/‑His/‑Ade/X-α-Gal平板上,3-5 d后观察菌落生长情况。以pGBKT7‑53 和 pGADT7‑T 组合为阳性对照,pGBKT7‑Lam 和pGADT7‑T 组合为阴性对照,pGBKT7 和pNC‑GADT7‑ShPR10组合、pGBKT7‑P1和pNC‑GADT7 组合为自激活验证。
1.2.5 BiFC验证ShPR10与SCSMV P1的互作 以P1‑18T质粒为模板,P1‑JT‑F和P1‑JT‑R(表1)为引物扩增P1基因。以ShPR10‑18T质粒为模板,ShPR10‑JT‑F和ShPR10‑JT‑R1(表1)为引物扩增ShPR10基因。参照Nimble Cloning试剂盒说明将ShPR10和P1基因分别连接到pNC‑BiFC‑Ecn和pNC‑BiFC‑Enn载体上,获得重组质粒pNC‑BiFC‑Ecn‑ShPR10和pNC‑BiFC‑Enn‑P1。将重组质粒转入GV3101农杆菌,参照朱海龙等[18]的方法将含有pNC‑BiFC‑Ecn‑ShPR10和pNC‑BiFC‑Enn‑P1的农杆菌等比例混合后注射本氏烟草叶片,48 h后在激光共聚焦显微镜下观察拍照。以pNC‑BiFC‑Ecn和pNC‑BiFC‑Enn‑P1组合、pNC‑BiFC‑Ecn‑ShPR10和pNC‑BiFC‑Enn组合为对照。
1.2.6 ShPR10对SCSMV P1沉默抑制子活性的影响 以P1‑18T质粒为模板,P1‑JT‑F和P1‑JT‑R1(表1)为引物扩增P1基因。以ShPR10‑18T质粒为模板,ShPR10‑JT‑F和ShPR10‑JT‑R(表1)为引物扩增ShPR10基因。按照Nimble Cloning试剂盒说明将P1和ShPR10基因分别连接到植物表达载体pNC‑Cam33FN(带Flag标签)和pNC‑Cam33HN(带HA标签),获得重组质粒pNC‑Cam33FN‑P1和pNC‑Cam33HN‑ShPR10。将pNC‑Cam33FN‑P1、pNC‑Cam33HN‑ShPR10和课题组保存的pCam3304‑35S‑GFP质粒分别转化GV3101农杆菌。参考Chen等[19]方法将携带pCam3304‑35S‑GFP、pNC‑Cam33FN‑P1和pNC‑Cam33HN‑ShPR10质粒的3种农杆菌等比例混合后注射本氏烟草叶片,以携带pCam3304‑35S‑GFP、pNC‑Cam33FN‑P1和pNC‑Cam33HN空质粒的3种农杆菌混合注射为对照。此外,为了说明ShPR10本身不具有沉默抑制子的活性,将携带pCam3304‑35S‑GFP、pNC‑Cam33HN‑ShPR10和pNC‑Cam33FN空质粒的3种农杆菌混合注射烟草叶片。注射4 d后,在长波紫外灯下观察注射区域GFP的荧光强度并采用数码相机拍照记录。在紫外灯下剪下注射区域的烟草叶片,参考Chen等[19]方法进行总蛋白提取和Western blot检测GFP、P1和ShPR10蛋白的表达水平,使用Image J软件对蛋白条带进行定量分析。
2 结果
2.1 ShPR10基因的克隆
以甘蔗ROC22叶片cDNA为模板,ShPR10‑F和ShPR10‑R为引物进行扩增,获得一条600 bp左右的条带(图1‑A),与预期大小相符。回收该条带将其连接至T载体并送测序。结果(图1‑B)显示,克隆获得甘蔗ShPR10基因的完整ORF,其长度为570 bp,编码189个氨基酸,其氨基酸序列中含有以“GxGGxG”为特征的P‑loop基序。
2.2 ShPR10序列分析
ProtParam分析表明,ShPR10的分子量为21.17 kD,等电点为4.77,为酸性蛋白;不稳定系数为70.86,为不稳定蛋白;脂溶指数为48.47,总平均亲水性是‑0.93,可能是亲水性蛋白。NPSA‑PRABI预测表明(图2),ShPR10中无规则卷曲占比为51.85%,α-螺旋占比为35.98%,延伸链占比为7.41%,β-转角占比为4.76%。TMHMM 2.0分析表明(图3),ShPR10蛋白无跨膜结构域。SignalP 5.0分析表明(图4),ShPR10蛋白不含信号肽,是非分泌蛋白。
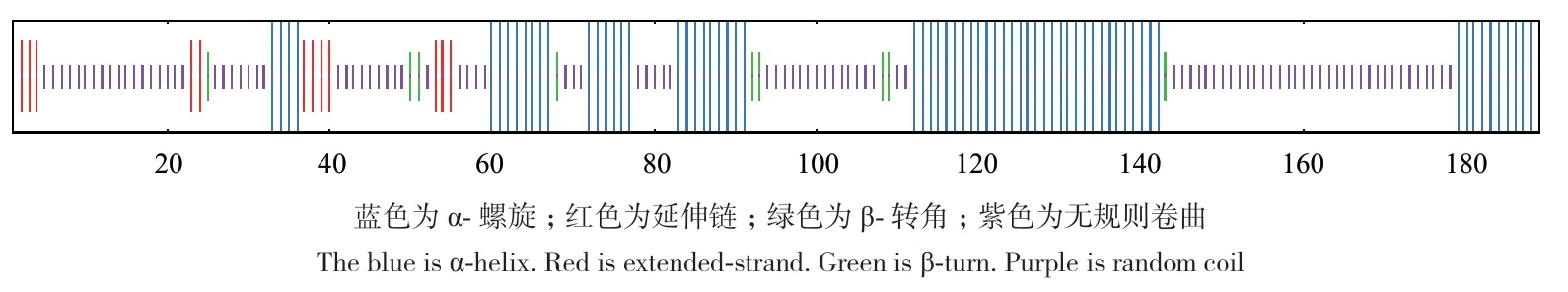
图2 ShPR10蛋白二级结构预测Fig. 2 Secondary structure prediction of ShPR10 protein
图3 ShPR10蛋白跨膜结构预测Fig. 3 Transmembrane structure prediction of ShPR10 protein
图4 ShPR10蛋白信号肽预测Fig. 4 Signal peptide prediction of ShPR10 protein
氨基酸序列同源比对分析结果(图5)显示,甘蔗ShPR10与玉米ZmPR10、柳枝稷(Panicum virgatum)PvPR10、普通小麦(Triticum aestivum)TaPR10、光稃稻(Oryza glaberrima)OgPR10、短花稻(Oryza brachyantha)ObPR10、多年生黑麦草(Lolium perenne)LpPR10、乌拉尔图小麦(Triti‑cum urartu)TuPR10、硬直黑麦草(Lolium rigidum)LrPR10和野生二粒小麦TdPR10的氨基酸序列具有较高相似度,分别为91.53%、82.01%、76.56%、76.50%、76.17%、75.92%、75.52%、75.13%和72.40%。为进一步分析甘蔗ShPR10蛋白与其他物种PR10蛋白的进化关系,使用MEGA 11.0软件构建系统进化树,结果(图6)表明,甘蔗ShPR10蛋白与玉米ZmPR10的亲缘关系最近。

图5 甘蔗ShPR10与其他物种PR10的氨基酸序列比对Fig. 5 Amino acid sequence alignment between ShPR10 protein from sugarcane and PR10 from other species
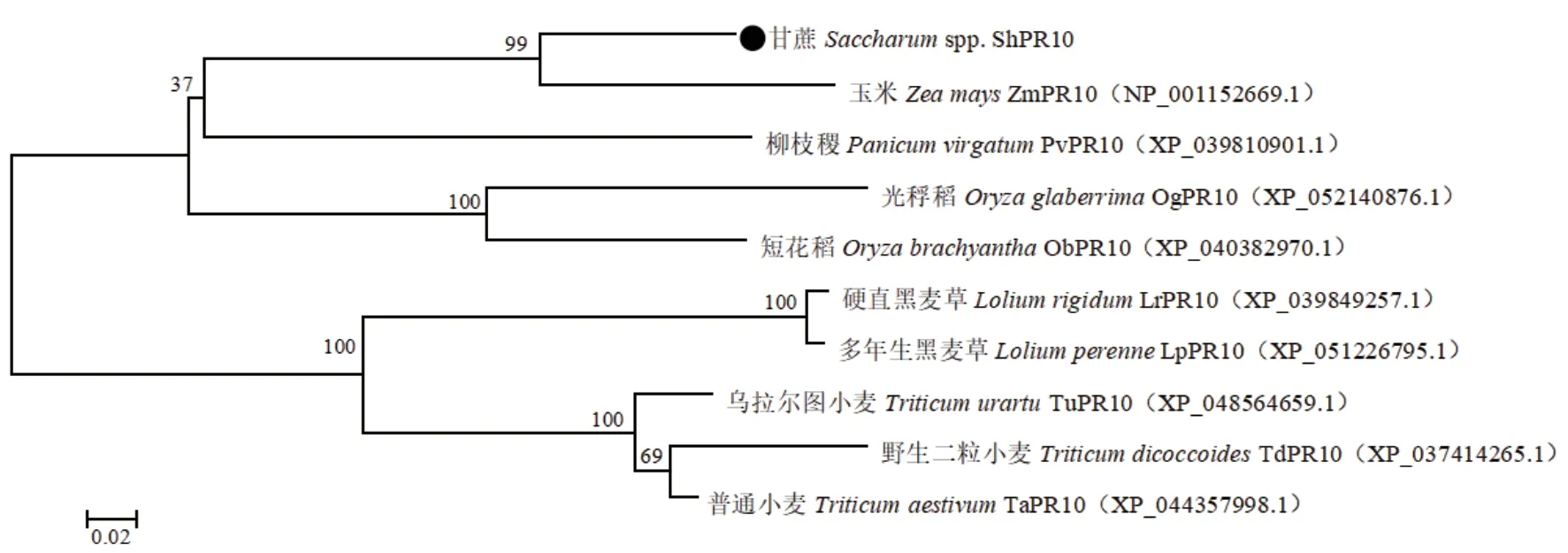
图6 甘蔗ShPR10与其他物种PR10蛋白的系统进化树分析Fig. 6 Phylogenetic tree analysis of ShPR10 protein from sugarcane and PR10 proteins from other species
2.3 ShPR10的亚细胞定位
将GFP基因融合在ShPR10基因的5′端,成功构建亚细胞定位载体pBWA(V)HS‑GFP‑ShPR10。将该载体通过农杆菌介导侵染烟草表皮细胞,在激光共聚焦显微镜下观察GFP‑ShPR10融合蛋白的绿色荧光在烟草细胞内的分布情况。结果(图7)显示,绿色荧光主要分布在细胞质和细胞核,表明ShPR10主要定位于细胞质和细胞核。
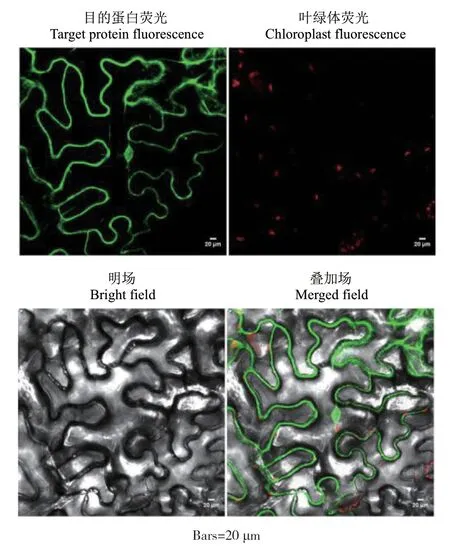
图7 ShPR10在本氏烟草表皮细胞中的定位Fig. 7 Localization of ShPR10 in N. benthamiana epidermal cells
2.4 ShPR10与SCSMV P1的互作验证
Y2H试验结果显示(图8),所有组合在SD/‑Trp/‑Leu培养基上正常生长,表明所有组合均成功转化酵母菌。转入pGBKT7和pNC‑GADT7‑ShPR10组合、pGBKT7‑P1 和pNC‑GADT7组合的酵母菌与阴性对照组(pGBKT7‑Lam和 pGADT7‑T组合)一样,在SD/‑Trp/‑Leu/‑His/‑Ade/X-α-Gal培养基上不生长,表明pNC‑GADT7‑ShPR10和pGBKT7‑P1质粒无自激活活性。转入pGBKT7‑P1和pNC‑GADT7‑ShPR10组合的酵母菌和阳性对照(pGBKT7‑53和 pGADT7‑T组合)一样在SD/‑Trp/‑Leu/‑His/‑Ade/X-α-Gal培养基上长出蓝色菌落,表明ShPR10在酵母细胞中与P1互作。

图8 Y2H验证ShPR10与SCSMV P1的互作Fig. 8 Verification of the interaction between ShPR10 and SCSMV P1 by Y2H assay
BiFC试验结果显示(图9),含pNC‑BiFC‑Ecn‑ShPR10和pNC‑BiFC‑Enn‑P1质粒的农杆菌共注射烟草,在烟草细胞中产生绿色荧光信号,而阴性对照组(pNC‑BiFC‑Ecn和pNC‑BiFC‑Enn‑P1组合、pNC‑BiFC‑Ecn‑ShPR10和pNC‑BiFC‑Enn组合)无荧光信号。以上结果表明ShPR10和P1在烟草细胞中互作。
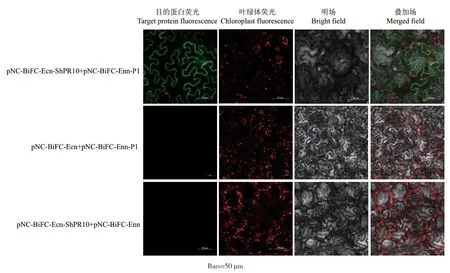
图9 BiFC验证ShPR10与SCSMV P1的互作Fig. 9 Verification of the interaction between ShPR10 and SCSMV P1 by BiFC assay
2.5 ShPR10对SCSMV P1沉默抑制子活性的影响
为了分析ShPR10对P1沉默抑制子活性的影响,将携带pCam3304‑35S‑GFP、pNC‑Cam33FN‑P1和pNC‑Cam33HN‑ShPR10质粒的3种农杆菌混合后共注射本氏烟草叶片,以携带pCam3304‑35S‑GFP、pNC‑Cam33FN‑P1和pNC‑Cam33HN空质粒的3种农杆菌共注射为对照,注射4 d后在紫外灯下观察GFP荧光强度。结果显示,对照组注射区域表现出很强的GFP荧光(图10‑A,右下),证明P1具有沉默抑制子活性,而pCam3304‑35S‑GFP、pNC‑Cam33FN‑P1和pNC‑Cam33HN‑ShPR10共注射区域的GFP荧光强度明显减弱(图10‑A,左下),表明ShPR10削弱了P1的沉默抑制子活性。此外,为了说明ShPR10本身不具有沉默抑制子活性,将携带pCam3304‑35S‑GFP、pNC‑Cam33HN‑ShPR10和pNC‑Cam33FN空质粒的3种农杆菌共注射烟草叶片,结果观察到非常微弱的GFP荧光(图10‑A,上中),说明ShPR10不具有沉默抑制子活性。
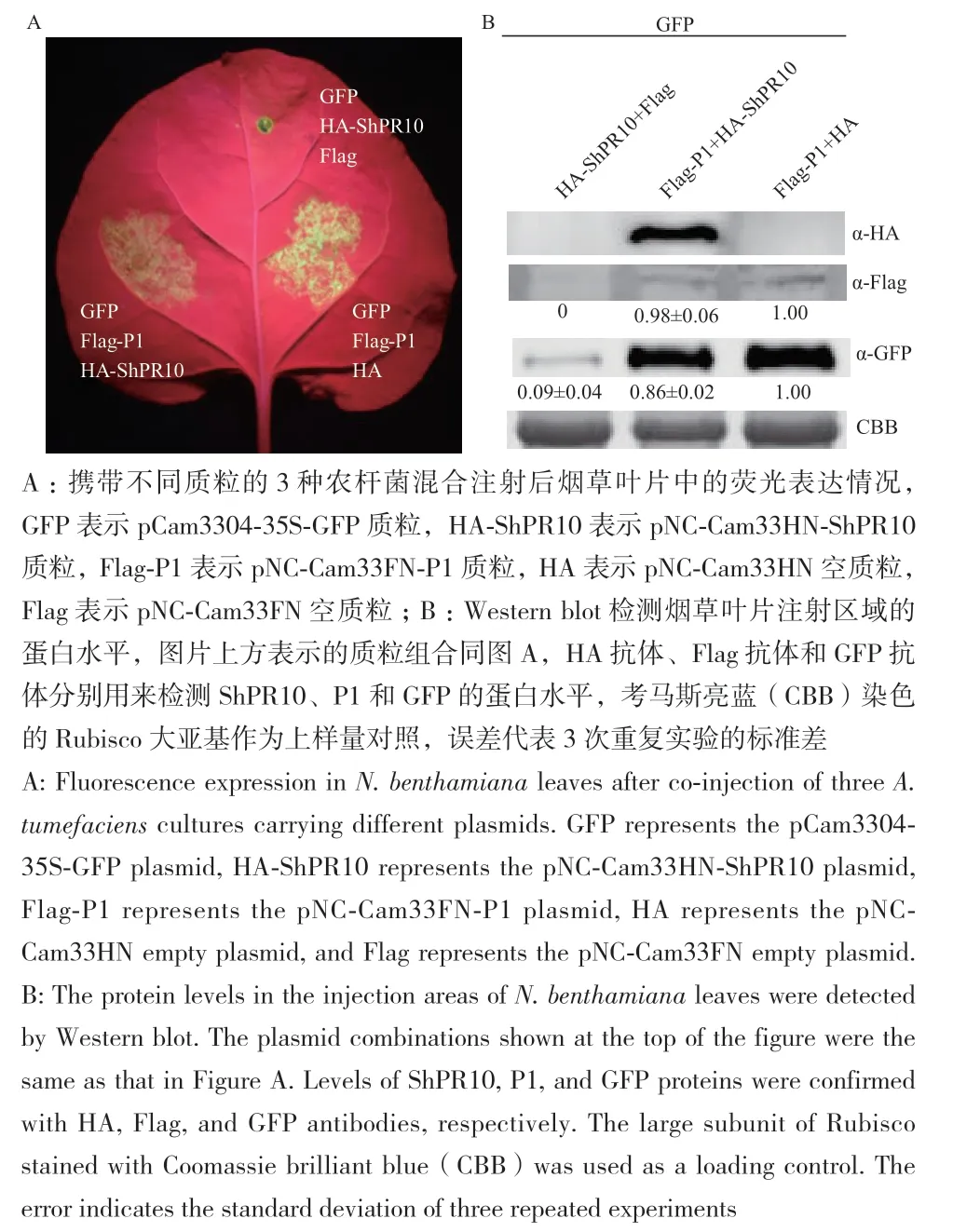
图10 ShPR10对SCSMV P1沉默抑制子活性的影响Fig. 10 Effect of ShPR10 on the activity of SCSMV P1 silencing suppressor
Western blot检测注射区域的蛋白水平(图10‑B)显示,pCam3304‑35S‑GFP、pNC‑Cam33FN‑P1和pNC‑Cam33HN‑ShPR10共注射区域的GFP蛋白水平低于对照组,这与图10‑A中该区域的GFP荧光强度弱于对照组的结果相一致。Flag抗体检测P1蛋白水平,结果显示该区域中P1的蛋白水平与对照组相比无明显差异。HA抗体检测ShPR10蛋白水平,结果显示该区域ShPR10正常表达。以上结果说明,ShPR10的表达削弱了P1的沉默抑制子活性,但对P1蛋白的含量无明显影响。
3 讨论
3.1 ShPR10具有PR10的基本特征和P‑loop基序
PR10蛋白具有分子量在17-22 kD、等电点呈酸性、不含信号肽、定位于胞内等特征[3,20]。本研究克隆获得的甘蔗ShPR10基因,其编码蛋白分子量为21.17 kD,等电点为4.77,无信号肽,定位于细胞质和细胞核,符合PR10的基本特征。大部分PR10蛋白含有一个以“GxGGxG”为序列特征的P‑loop基序,该基序与核酸酶活性密切相关[1]。从草莓(Fragaria ananassa)[21]、辣椒[4]、葡萄(Vitis vini‑fera L.)[22]、玉米[23]、花生(Arachis hypogaea L.)[24]、牛茄子(Solanum surattense)[25]、麻风树(Jatropha curcas)[26]和可可树(Theobroma cacao)[27]中分离到的PR10蛋白都具有一个P‑loop基序,体外实验也都证实这些PR10蛋白具有核酸酶活性。PR10的核酸酶活性对于植物抵御病原微生物入侵至关重要。辣椒CaPR10具有核酸酶活性,能对入侵的病毒RNA进行切割,从而起到抗病毒的作用[4]。可可树利用其编码蛋白TcPR10的核酸酶活性降解可可丛枝病菌(Moniliophthora perniciosa)的RNA,从而发挥抗菌作用[27]。花生AhPR10具有核酸酶活性,能抑制尖孢镰刀菌(Fusarium oxysporum)和立枯丝核菌(Rhizoctonia solani)的生长,将AhPR10的第54位氨基酸由赖氨酸突变成天冬酰胺后,AhPR10的核酸酶活性和抗菌作用均消失,表明AhPR10的核酸酶活性对其抗菌作用至关重要[24]。本研究从甘蔗中克隆获得的ShPR10基因,其编码蛋白含有一个P‑loop基序,推测该蛋白具有核酸酶活性,可能在甘蔗抵抗病原微生物入侵过程中发挥作用。
3.2 ShPR10定位在细胞质和细胞核
PR10蛋白家族成员众多,在亚细胞水平的分布也多种多样[3]。例如,辣椒[28]、玉米[23]和大豆(Glycine max)[29]编码的PR10蛋白定位在细胞质中;杨树(Populus simonii×P. nigra)[30]和刚毛柽柳(Tamarix hispida)[31]编码的PR10蛋白定位在细胞核上;人参(Panax ginseng C. A. Meyer)编码的PgPR10‑1蛋白在细胞质和细胞核中均有分布[32];葡萄编码的VpPR10.2蛋白主要分布在细胞壁、细胞质和叶绿体,在细胞核中也有少量分布[22];花旗松(Pseudotsuga menziesii)编码的DF‑PR10蛋白定位在细胞壁和细胞质中[33];罂粟(Papaver somniferum)编码的PR10蛋白Ps‑NCS2定位在内质网上[34]。本研究对甘蔗ShPR10蛋白进行亚细胞定位分析,结果显示ShPR10定位在细胞质和细胞核中,与人参PgPR10‑1蛋白的亚细胞定位相同。蛋白的功能与其所在的亚细胞位置密切相关,相同亚细胞定位的PR10蛋白可能行使相似的生物学功能。人参PgPR10‑1蛋白能增强植株对假单胞菌(Pseudomonas syringae)、尖孢镰刀菌和贵腐霉菌(Botrytis cinerea)的抗性[32],由此推测甘蔗ShPR10蛋白可能也具有抗菌活性。
3.3 ShPR10与SCSMV P1互作并削弱P1的沉默抑制活性
RNA沉默是植物抵抗病毒侵染的重要机制[35]。为了对抗这一机制,病毒进化出RNA沉默抑制子来干扰植物的RNA沉默[36]。而植物又可通过其编码蛋白与病毒RNA沉默抑制子相互作用,削弱抑制子的沉默抑制活性,进而增强植株对病毒的抗性[19,37-38]。例如,玉米编码的紫黄质脱环氧化酶ZmVDE与甘蔗花叶病毒(Sugarcane mosaic virus,SCMV)编码的HC‑Pro抑制子互作并削弱HC‑Pro的抑制子活性,从而减少SCMV在植物体内的积累[19];烟草编码的钙调素样蛋白rgs‑CaM与多种病毒编码的RNA沉默抑制子的dsRNA结合区域结合,并通过自噬样蛋白降解途径降解RNA沉默抑制子,从而削弱RNA沉默抑制子的活性,增强寄主对病毒的抗性[37];甜橙(Citrus sinensis)编码的脱水素蛋白CtCOR15与柑橘衰退病毒(Citrus tristeza virus,CTV)编码的p20抑制子互作,CtCOR15蛋白的表达削弱了p20的沉默抑制活性[38]。本课题组前期以SCSMV编码的RNA沉默抑制子P1为诱饵,从甘蔗cDNA文库中筛选获得一个与P1互作的甘蔗ShPR10蛋白[15]。本研究利用Y2H和BiFC技术进一步证实了P1和ShPR10的互作关系。此外,本研究还分析了ShPR10对P1沉默抑制活性的影响,结果显示ShPR10的表达削弱了P1的沉默抑制活性,但对P1蛋白的含量无明显影响,表明ShPR10不是通过改变P1蛋白的含量来影响P1的沉默抑制活性。RNA沉默抑制子可以通过结合siRNA、dsRNA和RNA沉默通路上的关键蛋白来发挥其抑制作用,ShPR10与P1的互作可能阻碍了P1与siRNA、dsRNA和RNA沉默通路蛋白的结合,从而削弱P1的沉默抑制活性[39]。
4 结论
从甘蔗叶片中克隆获得ShPR10基因的完整ORF,其编码蛋白含有一个P‑loop基序,分子量为21.17 kD,等电点为4.77,无信号肽和跨膜结构域,定位在细胞质和细胞核。ShPR10与SCSMV编码的P1蛋白互作,ShPR10的表达削弱了P1的沉默抑制子活性,但对P1蛋白的含量无明显影响。
