基于线粒体Ctyb基因的滆湖3种黄颡鱼遗传多样性分析
2023-11-17李大命刘燕山唐晟凯谷先坤殷稼雯
李大命,刘 洋,2,刘燕山,唐晟凯,谷先坤,殷稼雯
(1.江苏省淡水水产研究所/江苏省内陆水域渔业资源重点实验室,江苏南京 210017;2.南京师范大学海洋科学与工程学院,江苏南京 210046)
黄颡鱼属(Pelteobagrus)为鲇形目(Siluriformes)鲿科(Bagridae)鱼类,在江苏省该属鱼类有黄颡鱼(Pelteobagrusfulvidraco)、光泽黄颡鱼(Pelteobagrusnitidus)、长须黄颡鱼(Pelteobagruseupogon)和瓦氏黄颡鱼(Pelteobagrusvachelli)4种,一般统称黄颡鱼[1]。黄颡鱼属鱼类具有肉质鲜美、营养丰富、无肌间刺等特点,因此广受消费者欢迎,市场价格较高[2]。多年来,由于水体环境污染不断加剧,滥捕滥捞现象日益严重,导致黄颡鱼的栖息环境受到严重破坏,黄颡鱼野生资源数量锐减,开展黄颡鱼野生资源保护及相关研究显得十分必要[3]。
遗传多样性是生物多样性的重要组成部分,是物种多样性、生态系统多样性和景观多样性的基础,也是生命进化和适应的基础。开展鱼类遗传多样性的研究可以反映鱼种的进化历史,为分析其进化潜力提供参考;同时,还有利于分析物种稀有或濒危的原因,为鱼种保护提供参考[4-5]。鱼类线粒体具有DNA分子小、结构简单、进化速度快、母系遗传、重组率低等特点,使其成为分子标记在鱼类进化遗传学、群体遗传结构、分子生态学和保护生物学等研究领域的理想分子标记[6-7]。其中,细胞色素b(cytochrome b,Cytb)基因进化速度适中,有通用引物可用于扩增和测序,是检测鱼类遗传多样性和系统发育的常用分子手段[8-10]。
滆湖位于江苏省常州市西南,湖面面积146 km2,平均水深1.17 m,具有饮用、灌溉、航运和渔业等多种功能。近几十年,由于捕捞强度过大、开捕年龄偏低、湖泊富营养化和环境污染加剧,导致滆湖渔业资源明显衰退,鱼类小型化现象严重[11-13]。黄颡鱼是滆湖的常见鱼类,经济价值较高,是渔民重要的捕捞对象。已有研究者利用Cytb基因和D-loop区序列研究了滆湖黄颡鱼的遗传多样性[14-15],但有关滆湖光泽黄颡鱼和长须黄颡鱼的遗传多样性尚未见报道。笔者用线粒体Cytb基因作为分子标记,探索滆湖黄颡鱼、光泽黄颡鱼和长须黄颡鱼的遗传多样性及进化历史,以期为滆湖3种黄颡鱼的野生种质资源保护及合理利用提供科学依据。
1 材料与方法
1.1 样品采集和处理2019—2020年在滆湖设置3个渔获物采样点,开展渔业资源调查。共采集黄颡鱼49尾、光泽黄颡鱼40尾、长须黄颡鱼32尾,现场测量体长和体重。剪取样本背部肌肉组织,保存于无水乙醇中固定,置于4 ℃冰箱中保存备用。
1.2 PCR扩增和测序利用TaKaRa公司的DNA试剂盒提取肌肉组织的基因组DNA,最后用TE溶液溶解DNA。取少量DNA采用1.0%琼脂糖凝胶电泳检测DNA的完整性,使用微量紫外分光光度计检测其吸光值。
Cytb基因扩增和测序的引物为通用引物L14724和H15915[16],引物L14724为5′-GACTTGAAAAACCACCGTTG-3′,H15915为5′-CTCCGATCTCCGGATTACAAGAC-3′,引物由生工生物工程(上海)股份有限公司合成。PCR反应体系(50 μL)如下:模板DNA 2 μL(100 ng),2×PCR Mix 25 μL(Taq酶2.5 U,dNTPs 10 μmol,MgCl20.1 mmol),上、下游引物(10 μmol/L)各2 μL,用水补足50 μL。PCR反应程序如下:94 ℃预变性4 min;94 ℃变性45 s,58 ℃退火50 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min。PCR产物经琼脂糖凝胶电泳检测后送至生工生物工程(上海)股份有限公司进行双向测序。
1.3 数据处理和分析利用DNAStar软件包中SeqMan、EditSeq程序对测序结果进行拼接,获得Cytb基因序列。利用ClustalX 1.83软件[17]对Cytb同源序列进行多重比对分析。采用MEGA 7.0软件[18]统计碱基含量,基于Kimura双参数模型计算种内及种间遗传距离,采用邻接法(neighbor-joining,NJ)构建3种黄颡鱼单倍型的分子系统发育树。利用DnaSP 6.0软件[19]统计多态位点(variable site,V)、单倍型数量(haplotype number,N),计算单倍型多样性(haplotype diversity,h)、核苷酸多样性(nucleotide diversity,π)和平均核苷酸差异数(K)。
2 结果与分析
2.1Ctyb基因序列组成通过PCR扩增、测序和分析,获得了3种黄颡鱼的Cytb基因全序列。结果显示,3种黄颡鱼的Cytb基因长度均为1 138 bp,编码379个氨基酸,其中黄颡鱼Cytb基因的平均碱基含量为A 27.6%、T 26.6%、C 32.1%和 G 13.7%;光泽黄颡鱼的平均碱基含量为A 29.3%、T 28.4%、C 29.1%和 G 13.2%;长须黄颡鱼的碱基平均含量分别为A 27.4%、T 27.4%、C 31.5%和 G 13.7%。由此可见,3种黄颡鱼均表现出碱基组成偏倚性,碱基A+T的含量大于碱基G+C的含量。所有序列均无碱基插入或缺失。
2.2Ctyb基因多态性49尾黄颡鱼Cytb基因序列共检测到23个多态位点,其中单一信息位点10个、简约信息位点13个;49尾黄颡鱼定义17种单倍型(HapPf1~HapPf17),单倍型多样性为0.903,核苷酸多样性为0.002 40。40尾光泽黄颡鱼Cytb基因序列共检测到25个多态位点,其中单一信息位点9个、简约信息位点16个;40尾光泽黄颡鱼定义17种单倍型(HapPn1~HapPn17),单倍型多样性为0.936,核苷酸多样性为0.004 59。32尾长须黄颡鱼Cytb基因序列共检测到20个多态位点,其中单一信息位点12个、简约信息位点8个;32尾黄颡鱼定义11种单倍型(HapPe1~HapPe11),单倍型多样性为0.829,核苷酸多样性为0.002 25(表1)。整体来看,3种黄颡鱼的遗传多样性较为丰富,均呈现出高单倍型多样性和低核苷酸多样性模式,遗传多样性从高到低依次为光泽黄颡鱼、黄颡鱼和长须黄颡鱼。
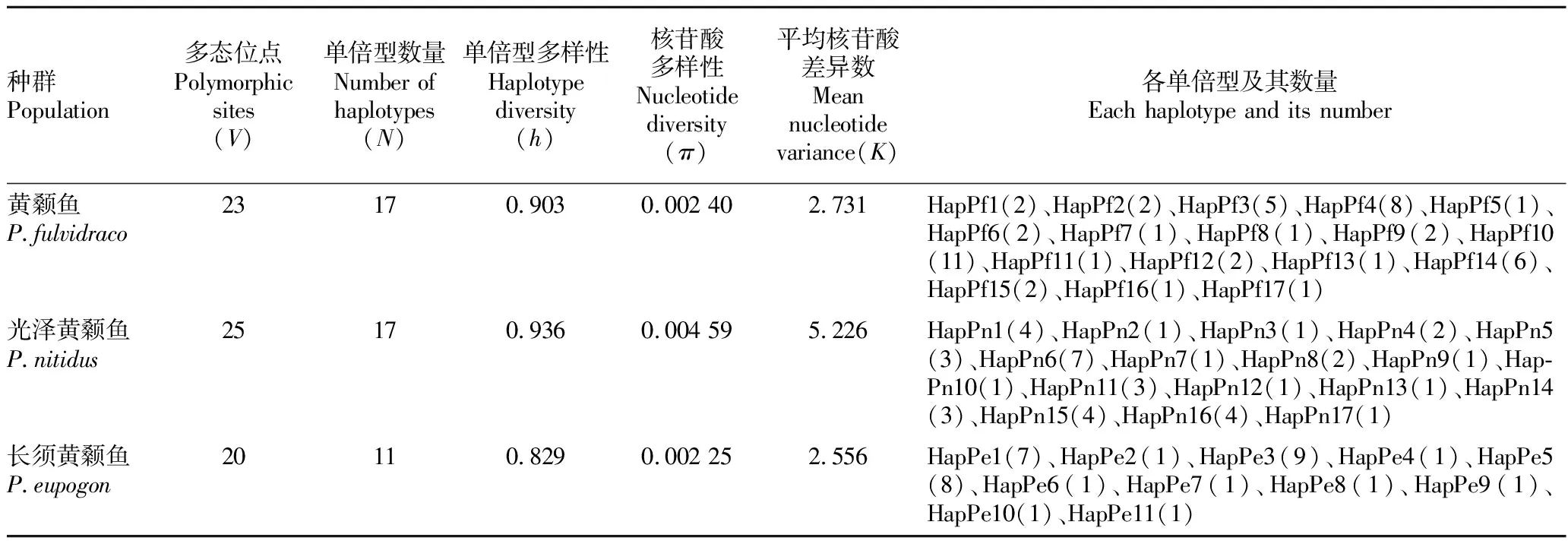
表1 3种黄颡鱼的遗传多样性参数 Table 1 Genetic diversity parameters of three species of Pelteobagrus
2.3 遗传结构和遗传关系分析采用MEGA 7.0软件计算3种黄颡鱼的种内和种间遗传距离,结果表明黄颡鱼种内遗传距离为0~0.006,平均值为0.002;光泽黄颡鱼种内遗传距离为0~0.012,平均值为0.005;长须黄颡鱼种内遗传距离为0~0.008,平均值为0.002。3种黄颡鱼种间遗传距离为0.078~0.138,其中长须黄颡鱼与黄颡鱼的遗传距离最小,长须黄颡鱼与光泽黄颡鱼的遗传距离最大(表2)。
以长吻鮠(Leiocassislongirostris)为外类群,采用邻接法构建3种黄颡鱼的分子系统发育树(图1),结果表明3种黄颡鱼均聚为独立分支,且节点的置信度水平较高,分支的置信限水平均为100%。黄颡鱼和长须黄颡鱼最先聚集在一起,然后与光泽黄颡鱼聚集在一起,表明黄颡鱼和长须黄颡鱼的亲缘关系较近,与光泽黄颡鱼的亲缘关系较远。
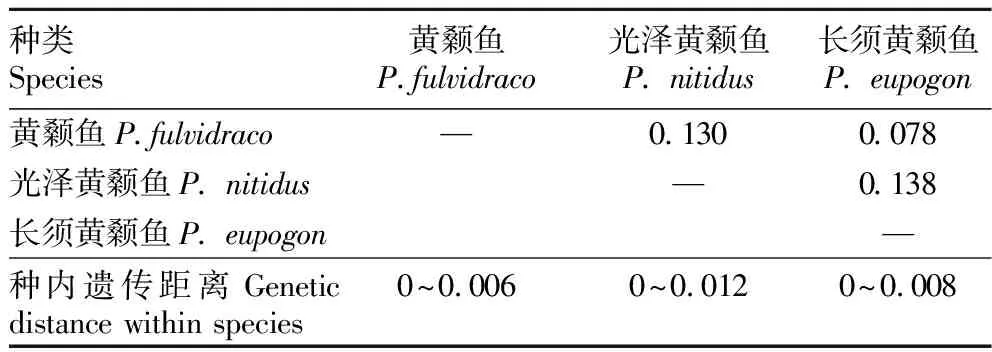
表2 3种黄颡鱼的种内及种间遗传距离Table 2 Intraspecific and interspecific genetic distance of three species of Pelteobagrus
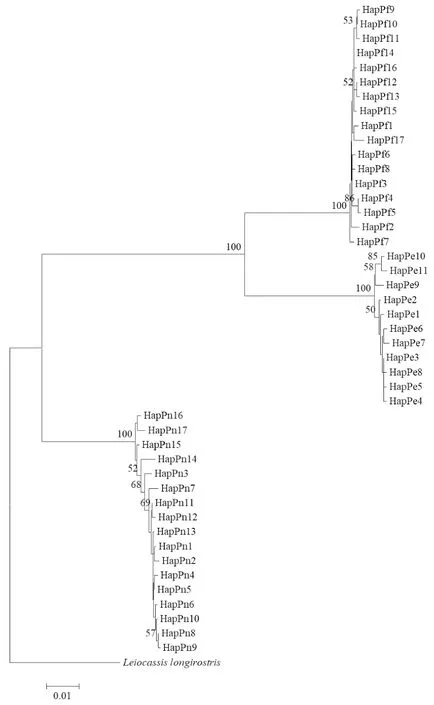
图1 基于Cytb基因的黄颡鱼单倍型分子系统发育树Fig.1 The haplotype molecular phylogenetic tree of Pelteobagrus based on Cytb gene
2.4 种群历史动态采用Arlequin 3.5软件对3种黄颡鱼进行Tajiam’sD和Fu’sFs中性检验,结果显示3种黄颡鱼的中性检验值均为负值,且大部分统计结果具有显著差异(P<0.05)(表3)。另外,3种黄颡鱼的核苷酸不配对分布图总体上呈明显的单峰型(图2),均说明3种黄颡鱼经历过明显的种群扩张。
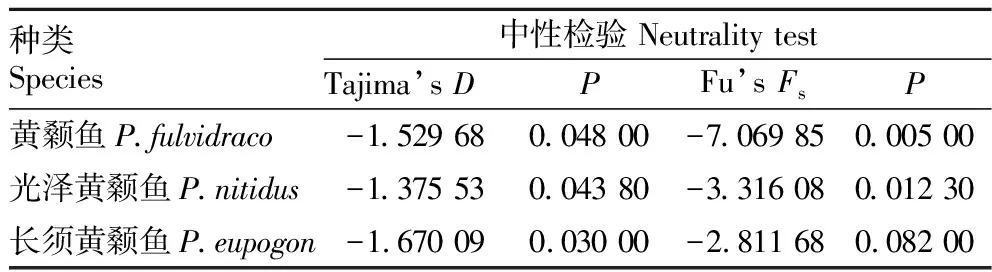
表3 3种黄颡鱼的中性检验结果Table 3 The neutrality test results of three species of Pelteobagrus
3 讨论
3.1 3种黄颡鱼遗传多样性物种的遗传多样性反映一个物种对环境的适应能力、生存能力和进化潜力,其遗传多样性越丰富,对环境的适应能力就越大,生存和进化的能力也越强。一般来说,单倍型多样性和核苷酸多样性是衡量某一物种遗传多样性的2个常用指标[20]。该研究结果表明,黄颡鱼、光泽黄颡鱼和长须黄颡鱼的单倍型多样性分别为0.903、0.936和0.829,核苷酸多样性分别为0.002 40、0.004 59和0.002 25。3种黄颡鱼的遗传多样性差异可能与有效种群数量、生态习性、环境压力等因素有关。根据Grant等[21]提出的鱼类遗传多样性标准,3种黄颡鱼的遗传多样性均具有高单倍型多样性和低核苷酸多样性的特点,其遗传多样性处于中等水平,这与我国黄颡鱼其他水系地理群体的遗传多样性相一致。比如,长江、淮河水系安徽区段黄颡鱼的单倍型多样性为0.930、核苷酸多样性为0.002 36[22],长江中下游5个湖泊黄颡鱼的单倍型多样性为0.945、核苷酸多样性为0.004 19[14],珠江流域黄颡鱼的单倍型多样性为0.849、核苷酸多样性为0.048 17[23],我国6个水系黄颡鱼的单倍型多样性为0.857、核苷酸多样性为0.002 3[24]。从整体来看,我国黄颡鱼的遗传多样性较为丰富,具有较强的环境适应能力、生存能力和进化潜力,但种群数量已大幅度减少,因此需要采取增殖放流、修复栖息环境等措施来恢复黄颡鱼有效种群大小,进一步提高遗传多样性。
3.2 3种黄颡鱼种群进化动态通常采用以下2种方法检验一个群体在历史上是否发生过扩张:一种方法是中性检验,采用Tajima’sD和Fu’sFs检验推测种群历史时如果Tajima’sD和Fu’sFs值呈负值,且在统计学上差异达到显著水平,则说明序列中含有比中性进化模型更多的核苷酸位点变化,可能预示着该群体在历史上有过扩张[25];另一种方法是核苷酸不配对分布,若其核苷酸不配对分布图呈单峰曲线则指示近期群体发生过扩张[26]。
通过Arlequin 3.5软件对3种黄颡鱼Cytb基因序列进行中性检测,获得Tajima’sD和Fu’sFs值,结果表明3种黄颡鱼的Tajima’sD和Fu’sFs值均小于0,且大部分统计检验结果具有显著差异(P<0.05)。另外,3种黄颡鱼群体的核苷酸不配对分布图均呈明显的单峰型,表明这3种黄颡鱼可能经历了近期的群体扩张。库喜英等[24]研究表明我国黄颡鱼群体在10.1万~14.1万年前发生过扩张。另外,3种黄颡鱼的遗传多样性模式也暗示着3种黄颡鱼群体可能是由小的有效种群经过一段时间的稳定后发生了扩张,形成了高单倍型多样性和低核苷酸多样性的特点[21]。
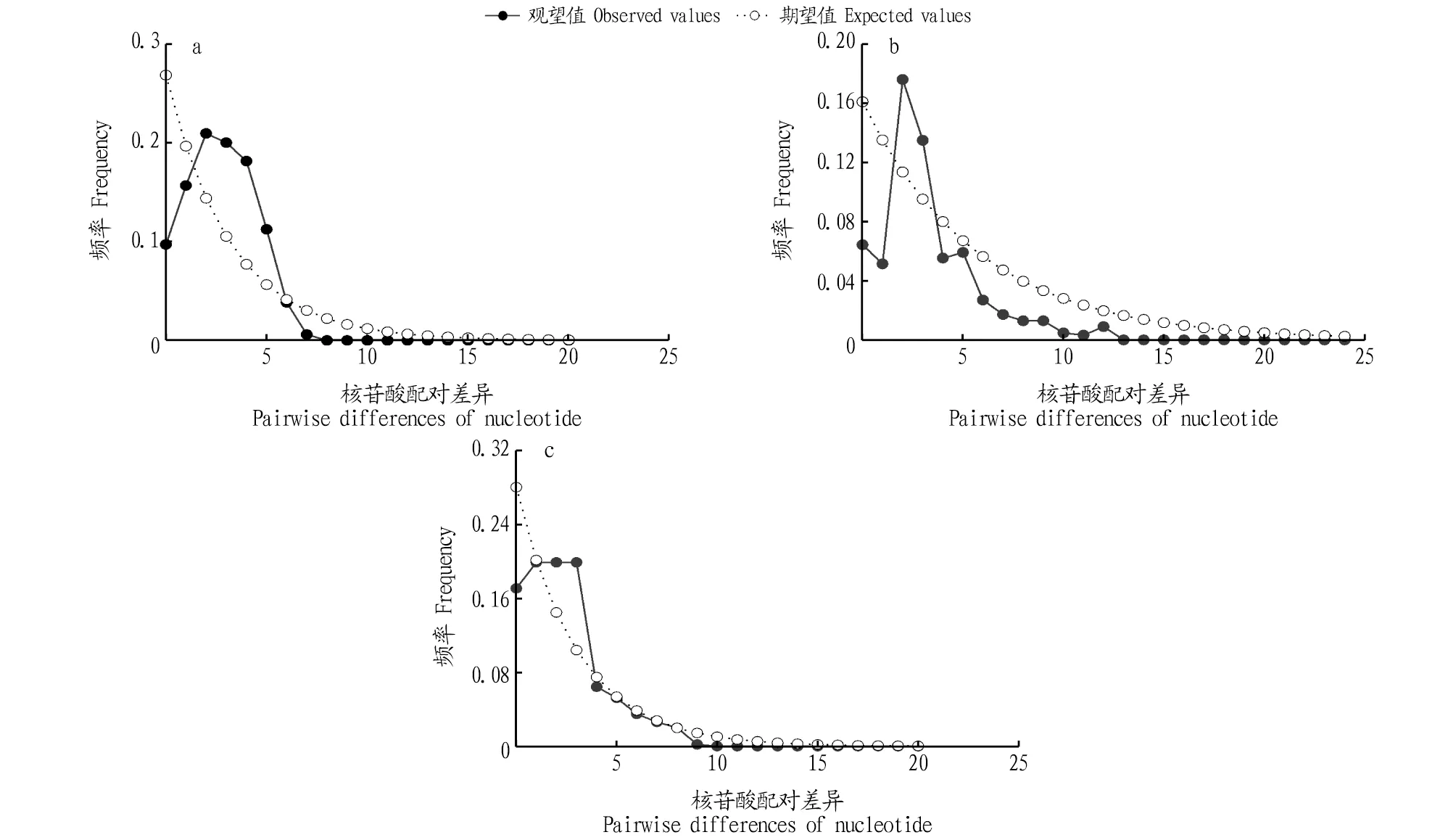
注:a.黄颡鱼;b.光泽黄颡鱼;c.长须黄颡鱼。Note:a.P.fulvidraco;b.P.nitidus;c.P.eupogon.图2 3种黄颡鱼的核苷酸不配对分布图Fig.2 The nucleotide pairwise difference distribution of three species of Pelteobagrus
3.3 3种黄颡鱼遗传结构及遗传差异该研究结果显示3种黄颡鱼的种内遗传距离均较小,黄颡鱼、长须黄颡鱼种内遗传距离分别为0~0.006和0~0.008,平均值均为0.002;光泽黄颡鱼种内遗传距离为0~0.012,平均值为0.005,表明种群内个体间不存在遗传分化。这是因为滆湖水面开阔,湖区间不存在地理隔离,3种黄颡鱼在滆湖内均是基因自由交流的群体。从单倍型组成来看,3种黄颡鱼的单个体单倍型占比较大,比如黄颡鱼种群的单个体单倍型占比为41.2%,2个体单倍型占比为35.3%;光泽黄颡鱼种群的单个体单倍型占比为47.1%,2个体单倍型占比为11.8%;长须黄颡鱼种群的单个体单倍型占比为72.7%。这说明3种黄颡鱼的有效种群数量小,遗传结构不稳定,易于波动[27]。
该研究中3种黄颡鱼的种间遗传距离为0.078~0.138,其中长须黄颡鱼与黄颡鱼的遗传距离最小、与光泽黄颡鱼的遗传距离最大,说明长须黄颡鱼与黄颡鱼的亲缘关系较近、与光泽黄颡鱼的亲缘关系较远,这与丁言伟[28]采用线粒体ND4基因序列对黄颡鱼属的分子系统发育研究结果相一致,而毛慧玲等[29]基于形态学参数的4种黄颡鱼聚类结果与该研究结果不一致,说明黄颡鱼属的物种分类及亲缘关系较为复杂,还有待进一步研究。梁宏伟等[30]探究了线粒体COI基因条形码在鲿科鱼类物种鉴定中的应用,其中黄颡鱼、长须黄颡鱼和光泽黄颡鱼的种间遗传距离为0.043 6~0.111 6,且光泽黄颡鱼和长须黄颡鱼的种间遗传距离不到这2种黄颡鱼种内遗传距离平均值的10倍。该研究结果显示,基于Cytb基因的3种黄颡鱼种间遗传距离为这3种黄颡鱼种内遗传距离平均值的10倍以上,因此线粒体Cytb基因比COI基因更适合作为鉴别黄颡鱼的分子标记。
