2019—2020年桂林市食源性沙门菌耐药分析及PFGE分型研究
2023-11-08王春旭王彦文黄辉唐勇邓洁妤贺漓漓
王春旭,王彦文,黄辉,唐勇,邓洁妤,贺漓漓
(广西桂林市疾病预防控制中心检验科,广西桂林 541001)
沙门菌作为导致食源性疾病的重要致病菌之一,广泛分布于自然界,因易污染食品、水等外环境以及存在于一些带菌的禽畜类动物中,通过食物链传递给人类,危害公共卫生健康[1-2]。我国每年约有上亿人因感染沙门菌而患病,占食源性疾病致病菌首位[3-5]。桂林市历年食源性风险监测中也以沙门菌检出为最多[6-7]。为了进一步揭示桂林市沙门氏菌的血清型分布及其耐药性和基因型,桂林市疾病预防控制中心实验室对2019年检出的32株沙门氏菌和2020年检出的37株沙门氏菌进行了血清型的鉴定和菌株的耐药性分析,同时采用脉冲场凝胶电泳法(PFGE)对其进行分子分型研究。
1 材料与方法
1.1 材料所分析的沙门菌菌株来自2019年收集的32株(其中21株为桂林市食源性致病菌监测的分离株,11株为同年腹泻患者检出菌株)和2020年收集的37株(其中21株为桂林市食源性致病菌监测的分离株,16株为同年腹泻患者检出菌株);所有菌株均经由广西壮族自治区疾病预防控制中心微生物实验室进行复核和血清学分型鉴定。血清型的质控菌株为鼠伤寒沙门菌ATCC14028(中国普通微生物菌种保藏管理中心提供),药敏试验质控菌株为大肠埃希菌ATCC25922(中国普通微生物菌种保藏管理中心提供),脉冲场凝胶电泳使用的分子量标准菌株为沙门菌H9812(广西壮族自治区疾病预防控制中心提供)。
1.2 仪器及试剂全自动细菌鉴定仪(法国生物梅里埃公司);全自动药敏试验菌液接种判读仪(Thermo赛默飞世尔);CHEF Mapper XA脉冲场电泳仪(美国Bio-Rad公司);Gel Doc XR+全自动凝胶成像系统(美国Bio-Rad公司)。沙门菌显色培养基(法国科玛嘉CHROMagar,生产批号P002060);诱导培养基(法国科玛嘉CHROMagar,生产批号20201201);沙门菌诊断血清(丹麦SSI公司,生产批号20200701);革兰阴性需氧菌药敏检测板(中国上海星佰生物技术有限公司,生产批号102012);蛋白酶K(德国Merck公司,生产批号VL558866);GelRed染液(美国Biotium公司,生产批号19G0326);Seakem Gold琼脂糖(美国Cambrex公司,生产批号0000990000);Xba I限制性内切酶(NEW England公司,生产批号AL12042A)。
1.3 方法
1.3.1 食源性疾病病例信息与相关样本采集对于食源性疾病病例个案信息按照《2019年国家食品污染物和有害因素风险监测工作手册》[8]收集,同时采集患者的新鲜粪便或肛拭子标本进行沙门菌检验,对阳性的沙门菌分离株再进行PFGE分子分型分析和药敏试验。
1.3.2 食品样品的采集与检验食品样品参照 《国家食品安全风险监测工作手册》进行采集,并参照GB 4789.4—2016《食品安全国家标准 食品微生物学检验 沙门菌检验》[9]鉴定沙门菌,同时依据White-Kauffmann-LeMinor抗原表[10]判定其血清型。
1.3.3 药物敏感试验药敏试验参照标准为2020年的CLSI(Clinical and Laboratory Standards Institute)标准[11],折点判断(S:敏感;I:中介;R:耐药)。多重耐药(multi drug resistance,MDR)定义:至少同时耐3种和(或)以上的不同类型抗生素菌株。
1.3.4 沙门菌分子分型根据国家食源性疾病溯源网络(TraNet China)沙门菌PFGE标准化分型方法进行沙门菌分子分型,具体方法为:待测沙门菌先经琼脂糖包埋,然后进行蛋白酶K裂解,最后用限制性内切酶Xba I(50U)进行酶切,从而得到限制性DNA片段,进行CHEF-mapper脉冲场凝胶电泳(以沙门菌标准株H9812为分子量标记)。最后的结果标示为经GelRed染色后成像的图谱导入Bionumerics 6.6软件进行聚类分析后的结果。
2 结 果
2.1 桂林市2019—2020年沙门菌血清型分布2019年的32株沙门菌和2020年的37株沙门菌,分别为B、C、D、E 4个群,共20个血清型,主要为鼠伤寒沙门菌,占菌株总数的27.54%(19/69),其次是肠炎沙门菌占14.49%(10/69),科瓦利斯沙门菌占13.04%(9/69),肯塔基沙门菌占5.80%(4/69),其余各型菌株数量较少,分型结果及比例见表1。
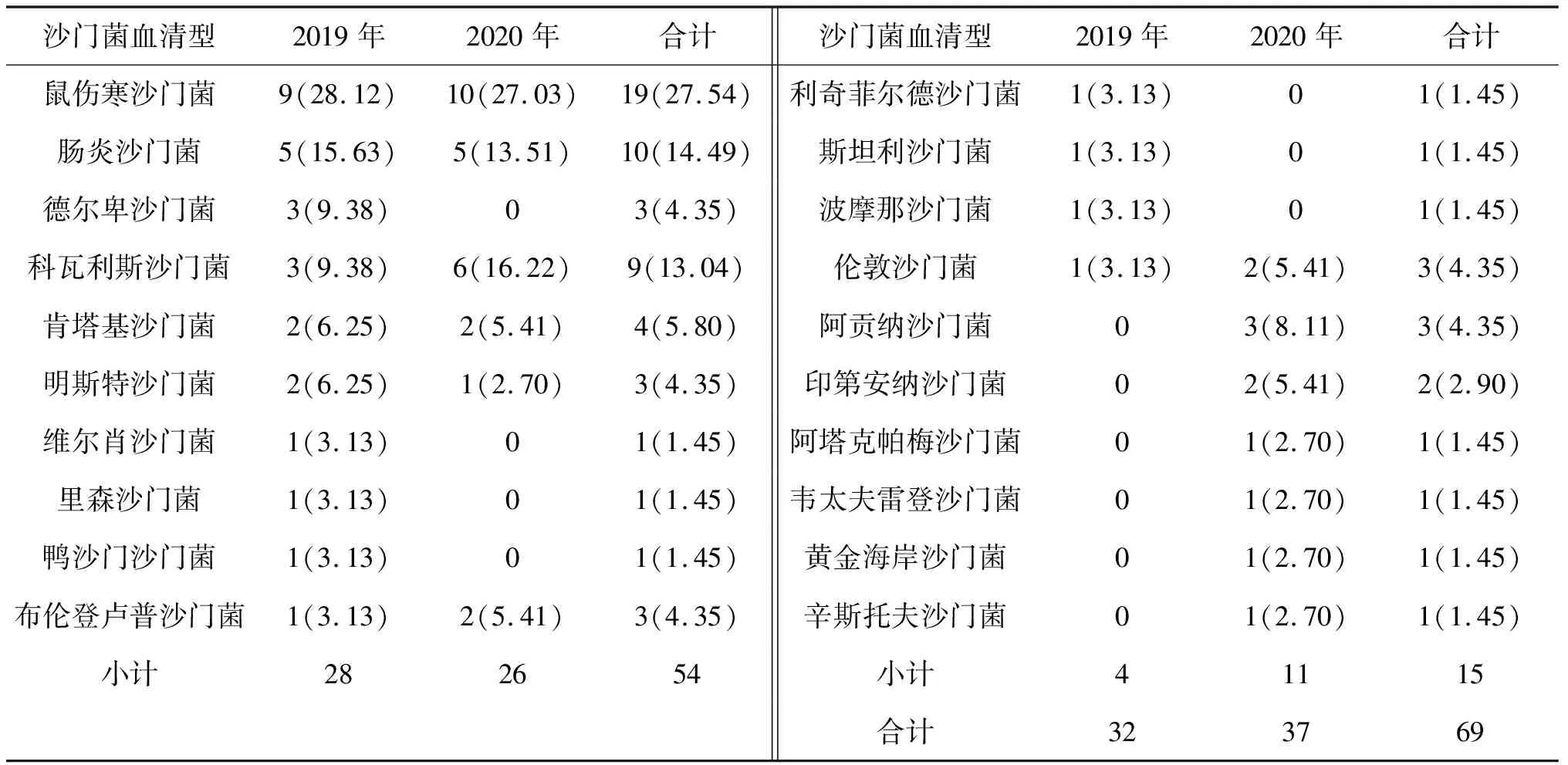
表1 2019—2020年桂林市食源性沙门菌血清型分布[n(%)]
2.2 沙门菌耐药情况
2.2.1 2019—2020年沙门菌耐药情况如表2所示,受试的12种抗生素中,69株菌株对磺胺异噁唑、四环素、氨苄西林、萘啶酸、氯霉素、复方新诺明、头孢曲松、环丙沙星、阿奇霉素、头孢西丁、氨苄西林/舒巴坦的耐药率分别为82.61%、75.36%、60.87%、52.17%、42.03%、28.99%、23.19%、14.49%、13.04%、5.80%、4.35%,对美罗培南100.00%敏感。

表2 2019—2020年沙门菌12种抗生素药敏结果[株(%)]
2.2.2 2019—2020年沙门菌耐药谱分布如表3所示,2019年的32株菌共产生17个耐药谱。其中9株鼠伤寒沙门菌对TET 100%耐药、对AMP 88.89%(8/9)耐药、对Sul 66.67%(6/9)耐药,AMP-TET-NAL和AMP-Sul-TET-AXO为鼠伤寒沙门菌的优势耐药谱,分别有3株菌存在同型耐药情况;肠炎沙门菌优势耐药谱为AMP-Sul-NAL,5株菌株100.00%对此耐药。3株德尔卑沙门菌优势耐药谱为TET-NAL;3株科瓦利斯沙门菌对Sul-TET 100.00%耐药。

表3 2019年32株不同血清型的沙门菌耐药谱分布
如表4所示,2020年的37株菌共产生24个耐药谱。其中10株鼠伤寒沙门菌对Sul 100.00%耐药、对TET 90.00%(9/10)耐药、对AMP 80.00%(8/10)耐药,AMP-Sul-TET为鼠伤寒沙门菌的优势耐药谱,有3株菌存在同型耐药情况;科瓦利斯沙门菌优势耐药谱为Sul-TET,有5株菌株对此耐药;5株肠炎沙门菌100%对AMP和NAL耐药;3株阿贡纳沙门菌全部对Sul耐药。

表4 2020年37株不同血清型的沙门菌耐药谱分布
2.3 PFGE分析结果
2.3.1 2019年32株沙门菌PFGE聚类分析对32株沙门菌酶切并进行脉冲场凝胶电泳后,DNA片段得到较好地分离,可见大小不一的电泳条带,然后进行聚类分析,根据电泳产生条带的位置和数量的不同,依据国家食源性疾病溯源网络手册,获得电泳图谱,通过Bionumerics 6.6软件采用不加权算数平均组对方法(UPG-MA),自动将这些PFGE带型分为多个大小不一的聚类群,获得不同菌株间的遗传克隆关系。菌株间相似度在49.40%~100.00%,呈现多样性(图1)。

图1 2019年32株沙门菌PFGE聚类分析图
2.3.2 分别对2019年4种菌型进行PFGE聚类分析聚类分析结果所示,9株鼠伤寒沙门菌共有6个不同的PFGE带型(图2),按照90%的相似度,这些PFGE带型又可分为4个大小不一的聚类群;5株肠炎沙门菌共有4个不同的 PFGE带型(图3),按照90%的相似度,这些PFGE带型又可分为2个大小不一的聚类群;3株德尔卑沙门菌共有3个不同的PFGE带型(图4),按照90%的相似度,这些PFGE带型又可分为2个大小不一的聚类群;3株科瓦利斯沙门菌共有3个不同的PFGE带型(图5),按照85%的相似度,这些PFGE带型可以分为2个大小不一的聚类群。
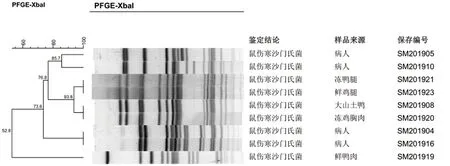
图2 2019年9株鼠伤寒沙门菌PFGE聚类分析图
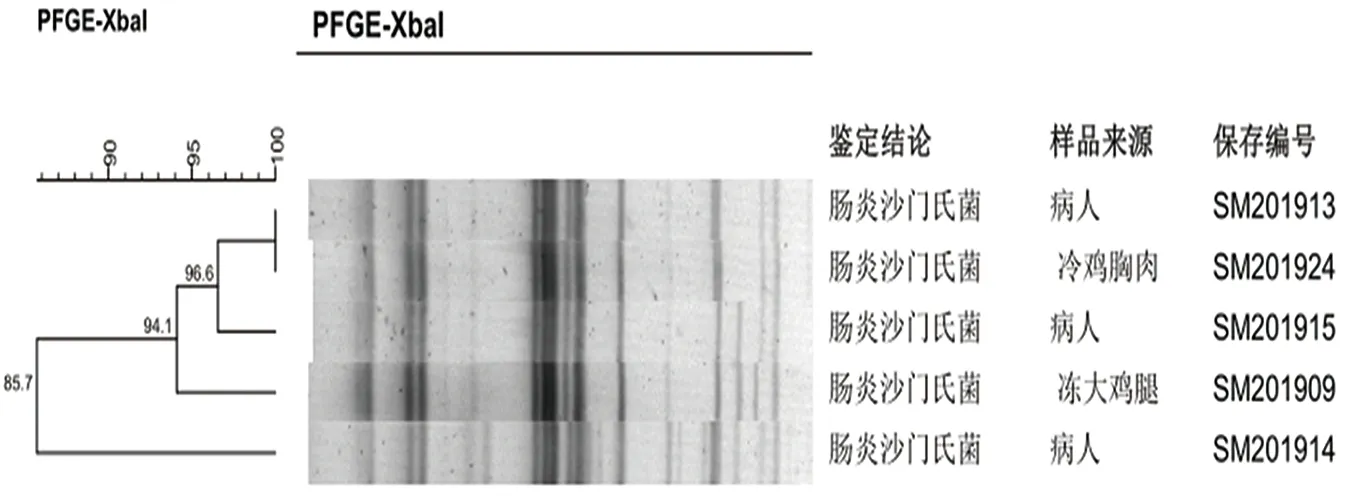
图3 2019年5株肠炎沙门菌PFGE聚类分析图
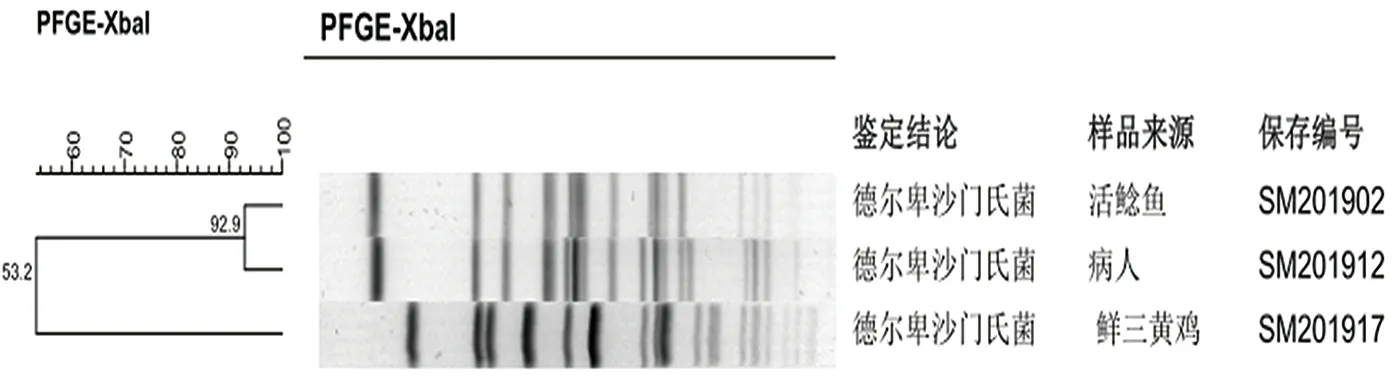
图4 2019年3株德尔卑沙门菌PFGE聚类分析图

图5 2019年3株科瓦利斯沙门菌PFGE聚类分析图
2.3.3 2020年34株沙门菌PFGE聚类分析对2020年的37株沙门菌酶切并进行脉冲场凝胶电泳后,有3株菌发生降解,其余34株沙门菌的DNA片段得到较好地分离,可见大小不一的电泳条带,然后对34株沙门菌进行聚类分析,根据电泳产生条带的位置和数量的不同,这些PFGE带型可以分为多个大小不一的聚类群,菌株间相似度在49.60%~100.00%,呈现多样性(图6)。
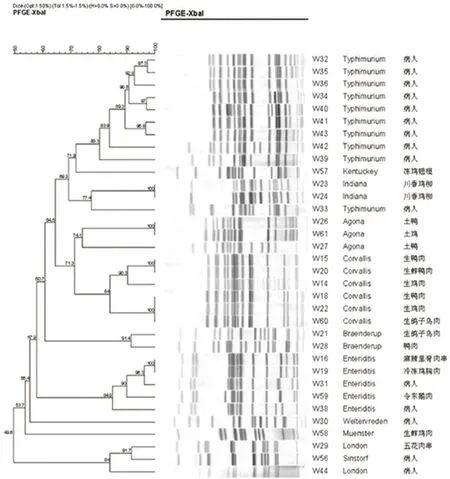
图6 2020年34株沙门菌PFGE聚类分析图
2.3.4 分别对2020年3种菌型进行PFGE聚类分析对10株鼠伤寒沙门菌进行聚类分析,共分为10个不同的PFGE带型(图7),按照90%的相似度,这些PFGE带型又可以分为5个大小不一的聚类群;对6株科瓦利斯沙门菌进行聚类分析,共分为3个不同的PFGE带型(图8),按照90%的相似度,这些PFGE带型又可以分为2个大小不一的聚类群;对5株肠炎沙门菌进行聚类分析,共分为4个不同PFGE带型(图9),按照90%的相似度,这些PFGE带型又可以分为2个大小不一的聚类群。

图7 2020年10株鼠伤寒沙门菌PFGE聚类分析图

图8 2020年6株科瓦利斯沙门菌PFGE聚类分析图
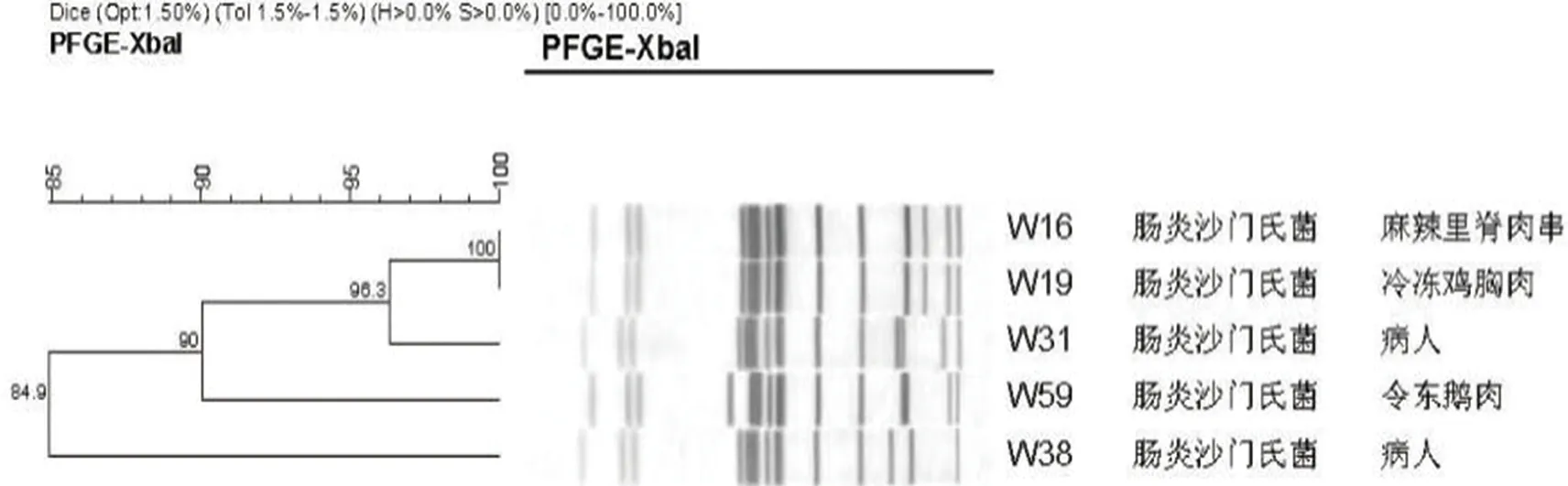
图9 2020年5株肠炎沙门菌PFGE聚类分析图
3 讨 论
沙门菌是导致公共卫生安全事件频发,威胁人畜健康的一种主要的食源性致病菌,不同血清型沙门菌的致病性存在差异。本研究显示,桂林市2019年、2020年食源性风险监测和食物中毒突发公共卫生事件中检出的沙门菌优势血清型主要是鼠伤寒沙门菌,占27.54%,其次为肠炎沙门菌,占14.49%。据文献报道[2,10,12,13],在我国多地引起食源性食物中毒的最常见沙门菌血清型是鼠伤寒沙门菌、肠炎沙门菌等,与本研究结论一致。同时,桂林市分离检出的沙门菌分属于B、C、D、E 4个群20个血清型,说明桂林市食品中及腹泻样本沙门菌呈现多态性,但存在着优势菌。
研究显示,沙门菌的耐药性日益严重且与临床疾病治疗关系密切,同时涉及外环境中如养殖业禽畜动物抗生素耐药,引起食源污染影响到人源的问题,因此受到医疗卫生机构和农业部门等的重视[14]。本研究发现桂林市沙门菌同一血清型的菌种其耐药谱有很大相似性,如本研究发现2019年桂林市9株鼠伤寒沙门菌对四环素100.00%耐药,对氨苄西林88.89%耐药,对磺胺异噁唑66.67%耐药;2020年桂林市10株鼠伤寒沙门菌对磺胺异噁唑100.00%耐药、对四环素90.00%耐药、对氨苄西林80.00%耐药。这一耐药情况与上海市鼠伤寒沙门菌82.30%对四环素耐药率[15]、安徽鼠伤寒沙门菌60.50%对四环素耐药率[16]情况类似,但桂林市鼠伤寒沙门菌四环素耐药率已达90.00%~100.00%,值得相关医疗机构警惕。国内各地报道鼠伤寒沙门菌耐药最高的抗生素种类不同,原因有待进一步探讨。同时实验研究发现肠炎沙门菌和德尔卑沙门菌、科瓦利斯沙门菌存在同型优势耐药谱。
PFGE通过对细菌分子分型开展同源溯源分析,是目前国内外鉴别菌株以及溯源的金标准[17-18],也是实践中应用较为广泛的溯源分析技术。经过对2019—2020年桂林市69株沙门菌进行PFGE分子分型分析,69株沙门菌有多种带型,基因型呈多样性,存在优势的流行基因型。此外,带型无明显的地域、时间、聚集特征,提示沙门菌在桂林市食源性风险监测项目以及突发公共卫生事件呈散发存在。但由于分离菌株数量较少,且缺乏年份时序性研究,尚需不断建立并完善菌株及分子分型数据库,为预警以及干预食源性疾病暴发做好技术和数据储备。
本研究建立了2019—2020年桂林市食源性风险监测项目以及突发公共卫生事件中分离检出的沙门菌的分子分型数据库,本数据库为今后及时发现和食源性疾病暴发的处置奠定了基础。研究发现2019—2020年桂林市沙门菌存在鼠伤寒和肠炎优势菌,菌株存在耐药现象。鼠伤寒沙门菌耐药较为严重,结果提示在本地区食源性致病菌均可能存在耐药基因或耐药菌株扩散现象。收集的沙门菌数据资源充实了本市一级区域性食源性致病菌数据资源库,为今后开展疾病和食源性疾病的监测、暴发溯源、控制预警以及建立和应用大数据提供科学的依据。
