基于转录组测序的辅助鉴定南方大豆皱叶症分子标记开发
2023-11-02陈文杰梁江韦清源汤复跃郭小红陈渊
陈文杰 梁江 韦清源 汤复跃 郭小红 陈渊




摘要:【目的】基于轉录组测序(RNA-Seq)开发辅助鉴定南方大豆皱叶症(SSCLD)的分子标记,为快速鉴定生产上遇到的大豆皱叶是否为SSCLD提供技术支撑。【方法】利用转录组测序技术分析遗传背景相近的皱叶大豆和正常叶大豆材料在皱叶症诱导环境下的差异表达基因(DEGs),从中去除核苷酸序列高度相似的同源序列,筛选出表达差异较大的DEGs进行分子标记开发,利用皱叶大豆材料和逆境处理试验评价分子标记的表达专一性,并利用不同类型的皱叶表型样本对分子标记进行表达专一性及可靠性验证。【结果】7株皱叶植株样本和5株正常叶植株样本的测序碱基错误率为0.0226~0.0238,Q20和Q30分别在99.00%和99.90%以上的碱基数量占总碱基数量均大于95.00%,GC含量为45.60%~46.24%,表明转录组测序数据质量较好。皱叶大豆材料较正常叶大豆材料有1063个DEGs上调表达,85个DEGs下调表达。根据基因的表达量和同源性,共筛选8个DEGs用作开发分子标记的候选基因,均表现为明显上调表达。8个候选基因的实时荧光定量PCR检测结果与转录组数据基本相符。此外,所筛选基因中有6个基因表达在不同程度上受干旱、水渍、冷害、荫蔽和盐胁迫等逆境胁迫诱导,其中,种植于皱叶症土壤中的皱叶型株系叶片GLYMA_18G033400基因的相对表达量均高于其在正常土壤中受干旱、水渍、冷害、荫蔽和盐等逆境胁迫后的相对表达量。根据GLYMA_18G033400基因序列设计分子标记qCL-18G033400,并用该分子标记对19份不同皱叶类型的叶片样品进行实时荧光定量PCR检测,结果发现,以ΔCt值小于10.00作为SSCLD的判定标准,分子标记鉴定结果同田间鉴定结果一致。【结论】利用转录组测序技术开发的分子标记qCL-18G033400可辅助鉴定SSCLD。
关键词:大豆;皱叶症;分子标记;开发;转录组;辅助鉴定
中图分类号:S565.1 文献标志码:A 文章编号:2095-1191(2023)06-1587-11
Development of molecular markers based on transcriptome
sequencing for assisting the identification of southern soybean crinkle leaf disease
CHEN Wen-jie1, LIANG Jiang1, WEI Qing-yuan1, TANG Fu-yue1,
GUO Xiao-hong1, CHEN Yuan2*
(1Cash Crops Research Institute,Guangxi Academy of Agricultural Sciences,Nanning, Guangxi 530007,China;
2Key Laboratory of Guangxi Academy of Agricultural Sciences, Nanning, Guangxi 530007,China)
Abstract:【Objective】Molecular markers based on transcriptome sequencing(RNA-Seq) for assisting the identification of southern soybean crinkle leaf disease(SSCLD) was developed to provide technical support for quickly identifying whether the soybean leaves encountered in production were SSCLD. 【Method】Transcriptome sequencing technology was used to analyze differentially expressed genes between crinkle leaf and normal leaf soybean materials with similar genetic background in the crinkle leaf-induced soil environment. Homologous sequences with highly similar nucleotide sequences were removed, DEGs with large expression differences were selected for molecular marker development, the expression specificity of molecular markers was evaluated by using crinkle leaf soybean material and stress treatment test, and the expression specificity and reliability of molecular markers were verified by different types of crinkle leaf phenotype samples. 【Result】The sequencing base error rate of seven crinkle leaf plants and five normal leaf plants were 0.0226-0.0238, the numbers of bases in Q20 and Q30 were 99.00% and 99.90%, and the GC contents were 45.60% to 46.24%, indicating good quality of transcriptome sequencing data. Transcriptome data showed that 1063 genes were up-regulated and 85 genes were down-regulated in leaves from the crinkle leaf type material,compared to that from normal leaf type material. Based on the expression level and homology of the genes, eight genes were screened according to the expression level and the identification of the homology in soybean genome,and both of them were up-regulated genes. Real-time fluorescence quantitative PCR analysis of the eight candidate genes generally agreed with the transcriptome data.The expression levels of six of these genes showed varying degrees of interference by diverse abiotic stress,including dry stress,water logging stress,cold stress,shade and salt stress,and the expression level of GLYMA_18G033400 in crinkle leaves from the crinkle leaf-induced environment was higher than that from normal soil under dry stress,waterlogging stress,cold stress,shade and salt stress. Molecular marker qCL-18G033400 was designed according to the GLYMA _ 18 G033400 gene sequence, and used to perform real-time fluorescence quantitative PCR on 19 leaf samples of different crinkle leaf types. The results showed that ΔCt value less than 10.00 was used as the determination criterion of SSCLD, and the identification results were consistent with the field identification results. 【Conclusion】Molecular marker qCL-18G033400 deve-loped using transcriptome sequencing can assist in the identification of SSCLD.
Key words: soybean; crinkle leaf disease; molecular marker; development; transcriptome; auxiliary identification
Foundation items:National Natural Science Foundation of China(32060490, 32260451); Special Construction Project of China Agriculture Research System (CARS-04-CES30); Guangxi National Science Foundation(2019GXNSFAA185009)
0 引言
【研究意义】大豆[Glycine max (L.) Merr.]是豆科大豆属的一年生草本植物,是我国重要的粮、油、饲兼用作物,在我国国民生产中具有重要的作用。南方大豆产区是我国重要的高蛋白大豆产区之一,为大豆蛋白加工提供优质的原料。近年来广西、广东、福建和贵州等地大豆生产中出现类似但非大豆病毒病引起的叶片皱缩病症,称为南方大豆皱叶症(Southern soybean crinkle leaf disease,SSCLD),发生严重时大豆可减产40%左右(陈文杰等,2022a),严重影响该产区大豆的生产。引发大豆皱叶的因素有病毒、除草剂、金属离子、基因突变等,其中病毒、除草剂等引起的大豆皱叶与 SSCLD的叶片形态上存在相似性,受大豆品种等差异因素的影响,很难准确辨别生产上遇到的皱叶类型是否为SSCLD(陈文杰等,2022b)。然而,SSCLD叶片表型具有表达专一性,推测SSCLD叶片中存在特异表达基因,该基因可用于开发辅助鉴定SSCLD的分子标记。因此,开发辅助鉴定南方大豆皱叶症分子标记,以期快速判定皱叶类型是否为SSCLD,对大豆皱叶症检测和防治及安全生产具有重要的研究意义。【前人研究进展】鉴定由病毒引起的皱叶症状时,可根据病毒保守序列设计特异分子标记,再对其进行PCR扩增进行判断。目前此类鉴定方法已很成熟,广泛用于豆类(涂丽琴等,2019;吉颖等,2022)、马铃薯(魏瑶等,2020)、辣椒(龚明霞等,2022)和黄瓜(车海彦等,2020)等作物上。随着酶联免疫吸附法(Enzyme-linked immunosorbent assay,ELISA)和胶体金免疫层析法(Gold immunochromatography assy,GICA)等免疫学检测技术的快速发展,使得病毒病的检测快速便捷(王佳等,2021;董旭旭等,2022)。因此,病毒感染所導致的皱叶症(Samertwanich et al.,2001;Ramon et al.,2014;王大刚等,2018;Yang et al.,2019)可通过免疫学检测法和分子标记法(赵小慧等,2021)检测,但SSCLD并非由病毒感染所致,无法利用这2种方法检测。本研究团队前期根据田间皱叶形态建立SSCLD鉴定方法(陈文杰等,2020)。该方法需将被鉴定大豆种质的种子种植于稳定诱发SSCLD的土壤中,根据大豆不同时期叶片形态表现进行鉴定。对于其他地区生产上出现的皱叶症状,单从形态上有时较难判定是否为SSCLD类型。【本研究切入点】目前针对大豆皱叶的研究还比较少,具体何种因素导致SSCLD尚不清楚。SSCLD表型上具有表达专一性,推测SSCLD发生时会有特异表达基因,这些基因可用于开发分子标记进行辅助鉴定SSCLD,但目前鲜见开发辅助鉴定SSCLD分子标记的研究报道。【拟解决的关键问题】利用转录组测序(RNA-Seq)技术分析遗传背景相近的皱叶大豆和正常叶大豆材料在皱叶症诱导环境下的差异表达基因(Differentially expressed genes,DEGs),从中去除核苷酸序列高度相似的同源序列,筛选出表达差异较大的DEGs进行分子标记开发,利用皱叶大豆材料和逆境处理试验评价分子标记的表达专一性,并利用不同类型的皱叶表型样本对分子标记进行表达专一性及可靠性验证,以期开发分子标记用于辅助鉴定SSCLD。
1 材料与方法
1. 1 试验材料
供试的大豆品种为桂春8号(正常叶,皱叶症级为0)和粤春2017-1(皱叶,皱叶症级为7)有性杂交衍生的杂合异质系材料GY_C(皱叶症诱导环境下表现皱叶)和GY_N(皱叶症诱导环境表现正常叶)(陈文杰等,2022a)以及近杂合异质系材料F2:7,即从F2代取杂合单株进行株行种植,收获皱叶单株,再进行株行种植,然后选择分离的株行收获皱叶单株继续进行株行种植,按照此方法一直种至F2:6代的植株,单株收获F2:6代中皱叶植株,得到F2:6代皱叶单株的种子F2:7,以上材料均由广西农业科学院经济作物研究所提供。主要试剂:总RNA提取试剂盒(RNAsimple Total RNA Kit)和反转录试剂盒[FastKing RT Kit (With gDNase)]均购自天根生化科技(北京)有限公司;2×Es Taq MasterMix购自于康为世纪生物科技有限公司;2×ChamQTM Univarsal SYBR qPCR Master Mix购自于南京诺唯赞生物科技股份有限公司。主要设备仪器:LightCycler 480II实时荧光定量PCR仪(Roche,美国)
1. 2 试验方法
1. 2. 1 F2:7代皱叶症田间鉴定试验 F2:6代皱叶单株的种子F2:7于2020年3月8日播种于广西农业科学院明阳基地(东经108°14′11″,北纬22°36′30″,海拔105.64 m,红壤土),多年试验证明该地块为大豆皱叶症表现稳定的地块。选择皱叶和正常叶分离的株行(图1),于始花期分别取7株皱叶植株(C_3、C_6、C_10、C_11、C_18、C_23和C_24)和5株正常叶植株(N1_1、N1_3、N1_5、N1_6和N1_7)的顶部嫩叶2 g左右,分别用锡箔纸包好后置于液氮保存,随后送至上海美吉生物医药科技有限公司进行转录组测序。
采用Illumina TruseqTM RNA sample prep Kit进行文库构建,即提取总RNA后Oligo dT富集mRNA,然后对富集后的mRNA进行片段化处理,随后利用反转录酶反转录合成cDNA,片段两端连接adaptor后利用Illumina NovaSeq 6000平台进行测序。测序完成后,图像信号经Base Calling得到Raw bases,数据质控得到Clean data,使用TopHat2 (http://tophat.cbcb.umd.edu/)与大豆参考基因组Wlliams82.a4.v1(https://phytozome-next.jgi.doe.gov/info/Gmax_Wm82a4_v1)进行对比得到mapped data(Kim et al.,2013),使用Cufflinks(http://cole-trapnelllab.github.io/cufflinks/)将mapped reads进行组装拼接(Trapnell et al.,2010),使用RSEM获得每个样本基因/转录本的Read Counts(Li and Deweg,2011),然后对其进行TPM(Transcripts per million reads)转换,进而得到标准化的基因/转录本表达水平。TPM转换方式:先将读数计数除以每个基因的长度(以千碱基为单位),得到每千碱基Reads(Reads per kilobase,RPK)。计算各样本中所有的RPK值,然后将其除以1000000,得到每百万缩放因子(Per million scaling factor),最后将RPK值除以每百万缩放因子即可得到TPM。
1. 2. 2 逆境处理试验 为验证在皱叶和正常叶中表达差异的基因是否具专一性,2021年7—12月利用皱叶材料GY_C进行干旱(Drought stress treatment,DST)、水渍(Water logging treatment,WLT)、冷害(Cold stress treatment,CST)、荫蔽(Shading treatment,ST)和盐胁迫(Salt stress treatment,SST)等逆境处理试验。试验采用盆栽方法,逆境处理使用土为营养土(皱叶材料表现正常),设置1个营养土对照(CKN)和1个皱叶土壤对照(CKC),重复3次。皱叶种子播种于花盆(口径×底径×高为21.5 cm×17.3 cm×14.8 cm),每盆播种4~5粒,待真叶期每盆留2株,随后分别进行干旱、水渍、冷害、荫蔽和盐胁迫处理。干旱胁迫处理参考王兴荣等(2021)的方法,即第6片复叶期开始处理,停止正常供水3 d后取顶部嫩叶。水渍处理参考孙慧敏等(2010)的方法,即第3片复叶期开始水渍处理15 d后取顶部嫩叶。冷害处理参考盖志佳等(2019)的方法,即第6片复叶期开始处理,3 ℃培养箱中处理12 h后取顶部嫩叶。荫蔽处理参考刘婷等(2016)的方法,即第6片复叶期在透光率为50%的遮阳网下处理5 d后取顶部嫩叶。盐胁迫参考严勇亮等(2021)的方法,即第6片复叶期开始处理,置于1.5%的NaCl溶液中处理3 d后取顶部嫩叶。CKN和CKC均于第6片复叶期取顶部嫩叶。嫩叶用锡箔纸包好置于液氮中保存备用。用RNAsimple Total RNA Kit提取嫩叶样品总RNA,然后用FastKing RT Kit (With gDNase)进行反转录合成cDNA。
1. 2. 3 皱叶样品采集及田间鉴定试验 2021年7—9月分别在南宁、贵港和贺州等地取不同大豆皱叶类型的大豆植株,取嫩叶后提取其总RNA,然后反转录合成cDNA备用。同时用标牌标记好植株成熟后收获该单株,2022年春季在广西农业科学院明阳基地田间鉴定各材料的皱叶症级,鉴定方法参考陈文杰等(2020),即始花期连续调查10株的皱叶症级,根据以下公式计算皱叶病症指数(DI):
DI(%)=[∑(N×R)/(M×T)]×100
式中,N表示病害某一级别的植株数;R表示病害的相对病级数值;M表示病害的最高病级数值;T表示调查的总株数。然后根据皱叶症指数划定皱叶症分级:0级为高抗(HR),DI=0;3級为抗(R),0<DI≤30%;5级为中抗(MR),30%<DI≤60%;7级为感(S),60%<DI≤90%;9级为高感(HS),DI>90%。
1. 3 DEGs筛选
为了筛选DEGs,对各基因的TPM值进行t检验,筛选皱叶和正常叶的DEGs。筛选条件:TPM-Cmin>TPM-Nmax或TPM-Cmax<TPM-Nmin、TPM-Cmin/TPM-Nmax≥5或TPM-Nmin/TPM-Cmax≥5。其中,TPM-Cmin表示某基因中皱叶个体TPM最小值;TPM-Nmax表示某基因中正常叶个体TPM最大值;TPM-Cmax表示某基因中皱叶个体TPM最大值;TPM-Nmin表示某基因中正常叶个体TPM最小值。
1. 4 DEGs同源性比对分析
因大豆属于古四倍体,大多数基因属于同源序列,导致扩增该基因难以保证其表达专一性。因此,设计分子标记时需删掉候选基因中核苷酸序列高度相似的基因。将筛选得到的基因cDNA预测序列提交到NCBI数据库上进行BLAST比对,以获得大豆基因组内的核苷酸序列高度相似的序列,从候选基因中删除这些序列。核苷酸序列高度相似的基因判定规则:Query cover≥80.00%且Percent identity≥80.00%;不存在高度相似基因满足条件:NQP<1。NQP代表Query cover≥80.00%且Percent identity≥80.00%基因的个数。
1. 5 分子标记开发及表达验证
利用Primer 5.0设计筛选得到候选基因的定量引物,引物扩增产物长度控制在100~250 bp。通过温度梯度PCR检测扩增引物的扩增效率和最适退火温度(表1)。挑选扩增条带清晰且不存在非表达专一性扩增的引物进行实时荧光定量PCR检测。以皱叶症诱导环境中的GY_C和GY_N株系叶片cDNA为模板,以GmActin为内参基因,引物见表1,用上述筛选出的引物验证分子标记表达的差异性。利用2×Es Taq MasterMix试剂盒进行引物筛选。温度梯度PCR反应体系25.0 μL: 2×Es Taq MasterMix 12.5 μL,10 nmol/L上、下游引物各1.0 μL,4 ng/μL cDNA模板1.0 μL,ddH2O补足至25.0 μL;扩增程序:96 ℃预变性2 min;96 ℃ 30 s,退火温度(见表1)30 s,72 ℃ 10 s,进行36个循环,72 ℃延伸5 min。利用2×ChamQTM Universal SYBR qPCR MasterMix试剂盒对候选基因进行实时荧光定量PCR检测,操作流程参考其说明书。
1. 6 分子标记表达专一性评价
以干旱、水渍、冷害、荫蔽和盐胁迫处理及相应对照CKN和CKC的GY_C株系叶片cDNA为模板,Actin11为内参基因,对筛选出的优质分子标记进行实时荧光定量PCR检测,进一步筛选出受逆境影响小的基因中的分子标记。
1. 7 分子标记可靠性验证
以Actin11为内参基因,用筛选出的分子标记对南宁、贵港和贺州等地采集的不同皱叶类型的大豆叶片进行实时荧光定量PCR检测。依据各基因的ΔCt值进行SSCLD鉴定,并将鉴定结果与田间鉴定结果相比较,以验证分子标记鉴定结果的吻合度。
1. 8 统计分析
利用Excel 2019进行数据整理分析;采用IBM SPSS Statistics 19进行t检验。
2 结果与分析
2. 1 转录组测序分析结果
由转录组测序质控数据(表2)可知,12个样本的测序碱基错误率为0.0226~0.0238,均在0.1000%以下,Q20和Q30分别在99.00%和99.90%以上的碱基数量占总碱基数量均大于95.00%,GC含量为45.60%~46.24%。由表3可知,每个样本93.00%以上的reads均可比对到大豆参考基因组上,表明转录组测序数据质量较好,可用于后续数据分析。
2. 2 DEGs筛选及同源性分析結果
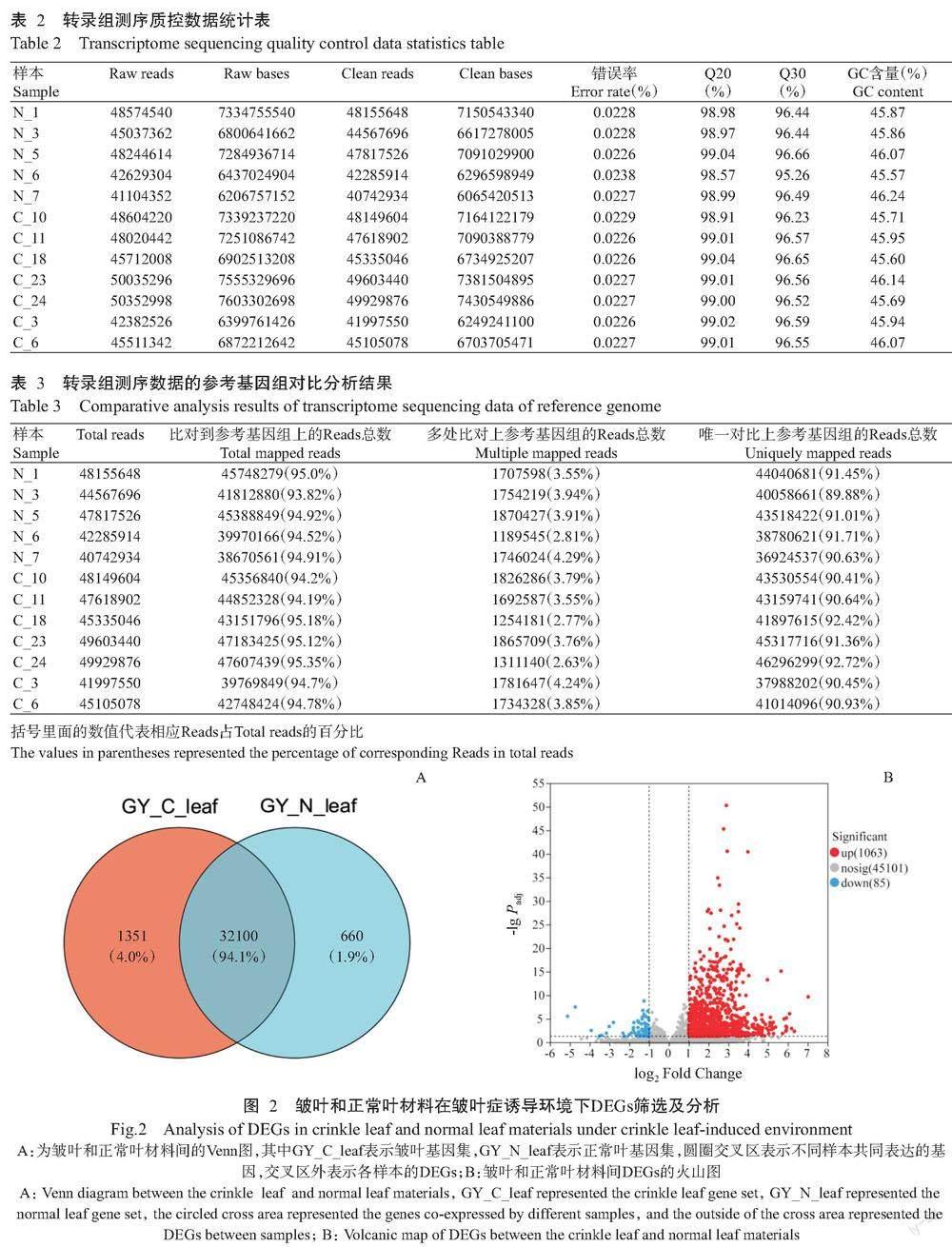
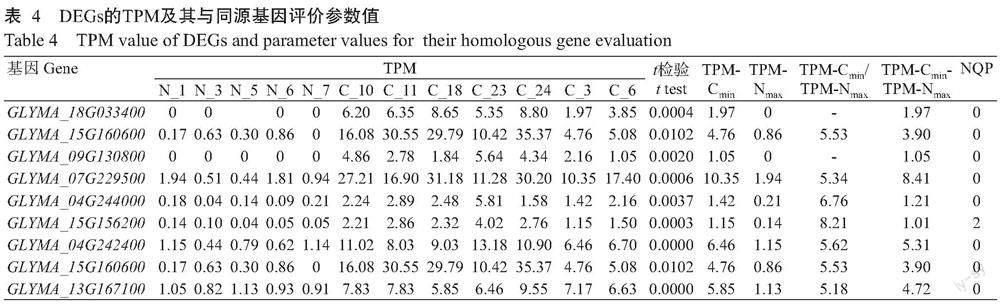
在皱叶症诱导环境下,皱叶材料和正常叶材料叶片共同表达基因数为32100个,占表达基因总数的94.1%,其中在皱叶材料叶片中DEGs数为1351个,占表达基因总数的4.0%;在正常叶材料叶片中DEGs数为660个,占表达基因总数的1.9%(图2-A)。与正常叶材料相比,皱叶材料中有1063个DEGs上调表达,85个DEGs下调表达(图2-B)。其中,上调的DEGs中TPM-Cmin>TPM-Nmax的基因有540个,下调的DEGs中TPM-Cmax<TPM-Nmin的基因有39个。下调的DEGs中无TPM-Nmin/TPM-Cmax≥5.00的基因,上调的DEGs中在正常叶中不表达的共有13个,但其中11个基因的TPM平均值小于1.00,可作为备选基因。最终筛选出9个表达差异较大的DEGs(表4),且均表现为明显表达上调。
提取上述9个DEGs的cDNA序列,并在NCBI数据库中进行BLAST比对,结果显示仅有GLYMA_15G156200基因在大豆基因组中存在2个高度相似基因(表4),且与GLYMA_09G049500基因的Query cover和Percent identity分别为88.00%和92.27%。因此选择余下的8个DEGs用作开发分子标记的候选基因。
2. 3 辅助鉴定SSCLD的分子标记开发
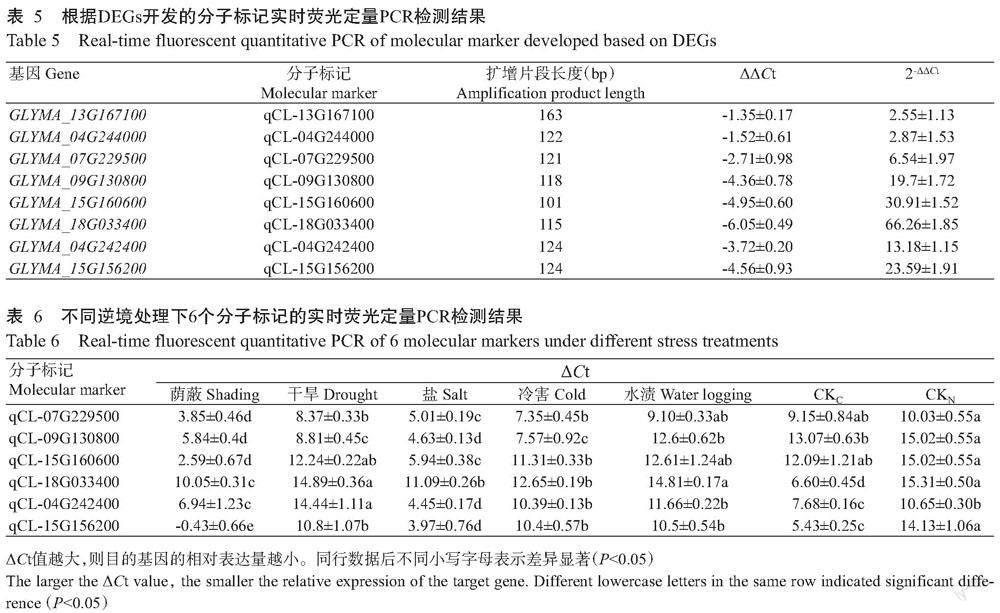
根据筛选出的8个候选基因cDNA序列信息设计引物,以温度梯度PCR筛选引物的最佳退火温度。以皱叶症诱导环境下的皱叶材料(GY_C)和正常叶材料(GY_N)株系叶片cDNA为模板,对8个候选基因进行实时荧光定量PCR检测,结果(表5)显示,与正常叶材料相比,皱叶材料叶片中8个候选基因的表达量均明显增加,其中差异倍数最小的是GLYMA_13G167100基因,仅为2.55倍;差异倍数最大的是GLYMA_18G033400基因,达66.26倍。表达差异较小可能会影响SSCLD的鉴定效果。因此,选择表达差异较大的GLYMA_18G033400、GLYMA_09G130800、GLYMA_15G160600、GLYMA_15G156200、GLYMA_04G242400和GLYMA_07G229500共6个基因开发的分子标记进行后续专一性评价试验。
2. 4 分子标记专一性评价结果
干旱、水渍、冷害、荫蔽和盐胁迫等逆境是生产中常见的非生物逆境胁迫。对皱叶材料进行干旱、水渍、冷害、荫蔽和盐胁迫处理,利用实时荧光定量PCR对表达量差异较大的6个基因开发的分子标记进行专一性评价,结果(表6)显示,qCL-09G130800在荫蔽、干旱、冷害和盐胁迫处理下的叶片中相对表达量较CKC和CKN均显著增加(P<0.05,下同),qCL-15G160600在荫蔽和盐胁迫处理下的叶片中相对表达量较CKN显著增加,qCL-04G242400在盐胁迫处理的叶片中相对表达量较CKC显著增加,qCL-15G156200在荫蔽、和盐胁迫处理的叶片中相对表达量较CKC显著增加,表明这些基因的表达受逆境胁迫影响的程度高于SSCLD,尽管qCL-18G033400在荫蔽、冷害和盐胁迫处理植株叶片中相对表达量显著高于CKN,但均显著低于CKC。因此,选择qCL-18G033400为辅助鉴定SSCLD的候选分子标记。
2. 5 分子标记可靠性验证结果
2021年4—6月,从南宁、贵港和贺州等地采集不同皱叶类型的大豆叶片样品共19份(图3)。其中,从南宁明阳基地采集到14份样品(编号为样1~14),从贵港市良种繁殖示范场采集到2份样品(编号为样15和样16),从贺州市黄姚镇采集1份(样17),从贺州市农业科学院基地采集到2份(编号为样18和样19)。利用GLYMA_18G033400基因开发的分子标记qCL-18G033400对19份皱叶样品进行实时荧光定量PCR检测。根据内参基因Actin11的表达量计算出各样本GLYMA_18G033400基因的相对表达量ΔCt值,同时进行田间皱叶症抗性鉴定,结果如表6所示。当材料的GLYMA_18G033400基因ΔCt值大于10.00时,田间鉴定为高抗性材料(皱叶症级为0级);当材料的GLYMA_18G033400基因ΔCt值小于10.00时,田间鉴定为中抗、感等材料,表明该标准可满足分子标记辅助鉴定SSCLD的要求。
3 讨论
SSCLD在我国南方多个省(区)可导致大豆严重减产(陈文杰等,2022a)。本研究课题组团队发现SSCLD不同以往的大豆皱叶类型,极有可能由土壤中未知微生物所致,建立快速准确的SSCLD鉴定方法可为SSCLD的防治提供技术支持。陈文杰等(2022a)根据大豆叶片皱缩程度建立了SSCLD田间鉴定方法,但该方法需要稳定发病的田间环境,且无法直接鉴定生产上遇到的皱叶是否属于SSCLD,若采用南方皱叶症田间鉴定方法对种子进行鉴定,可能出现假阳性现象,比如对于携带皱叶基因(显性)材料,叶片可能发生非SSCLD类型的皱缩,收集该材料种子进行田间鉴定结果就会出现假阳性。转录组测序技术广泛用于植株分子标记的开发(胡小文等2022;秦英之等,2022;徐晓丹等,2023),尤其是用于没有参考基因组的植物上(伍越等,2021)。为了克服表型鉴定SSCLD的缺点,本研究以遗传背景相近的皱叶材料和正常叶材料为研究对象,通过转录组测序分析筛选其叶片在皱叶症诱导环境中的DEGs,并进一步开发出辅助鉴定SSCLD的分子标记。
本研究筛选出用作开发辅助鉴定皱叶分子标记的候选基因GLYMA_18G033400。该基因在Soybase网站(https://www.soybase.org/)查询结果显示,其序列全长为790 bp,含2个外显子,编码区序列仅为249 bp。再将该基因序列提交至NCBI数据库进行比对,结果显示,该基因与LOC114394687基因的序列一致。LOC114394687基因编码一种lncRNA,但功能未知,在NCBI数据库进行BLAST比对分析,结果显示,僅有一条来自芸豆(Phaseolus vulgaris)的mRNA与其同源。lncRNA虽不编码蛋白,但在表观遗传调控(Gupta et al.,2010)、细胞周期调控(Kitagawa et al.,2013)和细胞分化调控(Yao et al.,2022)等生命活动中发挥重要作用。一些lncRNA是人类肿瘤重要的调控因子,在肿瘤发生和发展中具有表达专一性,故常被作为一种生物标志物用于肿瘤的诊断(王思宇和王宇,2022)。大量研究表明,lncRNA广泛参与调控植物生长发育和响应生物或非生物逆境等生理过程(李宁等,2019),如lncRNASABC1基因通过响应化学信号等介导的早期快速抗性反应激活植物对病原的免疫(Liu et al.,2022)。本研究的实时荧光定量PCR检测结果显示,与正常叶相比,GLYMA_18G033400基因在皱叶中的相对表达量显著性增加,且其表达受遮荫和干旱等生产上常见的非生物逆境胁迫影响较小,推测该基因在SSCLD发生过程中具有一定的表达专一性。GLYMA_18G033400基因是否在其他逆境诱导的叶片中表达量高于皱叶症,目前尚未知需要更多的逆境试验进行验证,但其并不影响GLYMA_18G033400基因作为SSCLD鉴定的指示基因。在涉及大量皱叶性状的材料需鉴定时,可利用该基因开发的分子标记进行初筛。低表达的皱叶类型可断定为非南方皱叶症类型,而对于高表达的皱叶类型可借助田间鉴定方法进行综合判定。目前该分子标记鉴定采集的19份皱叶材料同田间鉴定结果一致性较好,但还需要更多的皱叶材料验证该分子标记鉴定结果的准确性。另外,导致大豆皱叶的诱因还有很多,如病毒、除草剂、金属离子等,分子标记在这些诱因导致的皱叶中GLYMA_18G033400基因是否过表达目前尚未知,还需要进一步收集这些诱因导致的皱叶样品对qCL-18G033400进行验证。
分子标记辅助鉴定SSCLD的方法较田间鉴定方法缩短了鉴定时间,能在较短时间内判断生产上遇到的皱叶症状是否为SSCLD,为今后生产上SSCLD的快速诊治提供技术参考。但目前生产上存在皱叶症状的严重程度不等的情况,目前还缺乏不同皱叶症级与标记qCL-18G033400表达量方面的数据。因此,若要想精准判定叶片皱缩是否为SSCLD需尽快找出SSCLD致病因子,通过检测土壤或植株中致病因子含量情况进行精准判定。
4 结论
根据遗传背景相近的皱叶材料和正常叶材料的转录组数据,从DEGs中筛选出SSCLD表达专一性表达的基因GLYMA_18G033400,利用其开发的分子标记qCL-18G033400可辅助鉴定SSCLD。
参考文献:
车海彦,曹学仁,贺延恒,罗大全. 2020. 海南岛黄瓜病毒病种类鉴定及其发生分布研究[J]. 热带作物学报,41(11):2280-2284. [Che H Y,Cao X R,He Y H,Luo D Q. 2020. Distribution and identification of viruses from cucumber in Hainan Island[J]. Chinese Journal of Tropical Crops,41(11):2280-2284.] doi:10.3969/j.issn.1000-2561. 2020.11.019.
陈文杰,陈渊,韦清源,郭小红,汤复跃,杨萌,叶万典,梁江. 2020. 一种大豆皱叶症发生特性及材料症级鉴定[J]. 大豆科学,39(3):431-441. [Chen W J,Chen Y,Wei Q Y,Guo X H,Tang F Y,Yang M,Ye W D,Liang J. 2020. Study on inducement of soybean crinkle leaf in south of China[J]. Soybean Science,39(3):431-441.] doi:10. 11861/j.issn.1000-9841.2020.03.0431.
陈文杰,陈渊,韦清源,郭小红,汤复跃,赵团结,梁江. 2022a. 南方大豆皱叶发生时叶片形态变化及其对产量性状的影响[J]. 南方农业学报,53(2):460-468. [Chen W J,Chen Y,Wei Q Y,Guo X H,Tang F Y,Zhao T J,Liang J. 2022a. Changes of leaf morphology during the occurrence of wrinkled leaves in southern soybean and its effects on yield traits[J]. Journal of Southern Agriculture,53(2):460-468.] doi:10.3969/j.issn.2095-1191.2022. 02.019.
陈文杰,梁江,宁德娇,韦清源,汤复跃,郭小红,梁俊,陈渊. 2022b. 南方大豆皱叶症诱因分析研究[J]. 大豆科学,41(3):300-307. [Chen W J,Liang J,Ning D J,Wei Q Y,Tang F Y,Guo X H,Liang J,Chen Y. 2022b. Study on inducement of soybean crinkle leaf in Southern of China[J]. Soybean Science,41(3):300-307.] doi:10.11861/j.issn.1000. 9841.2022.030300.
董旭旭,孙威,曹攀,刘晓丹. 2022. 胶体金免疫层析试纸条技术在病毒检测领域的应用研究现状[J]. 生物工程学报,38(9):3243-3254. [Dong X X,Sun W,Cao P,Liu X D. 2022. Colloidal gold immunochromatographic test strip for virus detection:A review[J]. Chinese Journal of Biotechnology,38(9):3243-3254.] doi:10.13345/j.cjb.220052.
盖志佳,张敬涛,刘婧琦,蔡丽君,杜佳兴,孟庆英,谷维,陈磊. 2019. 低温胁迫对大豆幼苗形态生理指标及籽粒产量的影响[J]. 农学学报,9(12):1-4. [Gai Z J,Zhang J T,Liu J Q,Cai L J,Du J X,Meng Q Y,Gu W,Chen L. 2019. Cold stress affects morphological and physiological indexes of soybean seedling and seed yield[J]. Journal of Agriculture,9(12):1-4.] doi:10.11923/j.issn.2095-4050.cjas18100009.
龚明霞,赵虎,王萌,吴星,赵曾菁,何志,黄金梅,孟生德,王日升. 2022. 广西辣椒病毒的sRNA深度测序和RT-PCR鉴定[J]. 园艺学报,49(5):1060-1072.[Gong M X,Zhao H,Wang M,Wu X,Zhao Z J,He Z,Huang J M,Meng S D,Wang R S. 2022.Identification of viruses infecting peppers in Guangxi by small RNA deep sequencing and RT-PCR[J]. Acta Horticulturae Sinica,49(5):1060-1072.] doi:10.16420/j.issn.0513-353x.2021-0673.
桂敏,高雪,贾志强,杜磊,张芮豪,胡华冉,钟秋月,李永忠,刘雅婷,龙洪进. 2022. 云南辣椒正番茄斑萎病毒属病毒的分子鉴定[J]. 植物保护,48(4):286-292. [Gui M,Gao X,Jia Z Q,Du L,Zhang R H,Hu H R,Zhong Q Y,Li Y Z,Liu Y T,Long H J. 2022. Molecular identification of Orthotospovirus on hot pepper in Yunnan[J]. Plant Protection,48(4):286-292.] doi:10.16688/j.zwbh.2021 290.
胡小文,孔冉,刘洋,徐志军,苏俊波. 2022. 利用转录组测序开发甘蔗SNP分子标记[J]. 南方农业学报,53(9):2527-2536. [Hu X W,Kong R,Liu Y,Xu Z J,Su J B. 2022. SNP molecular marker development based on sugarcane transcriptome sequencing[J]. Journal of Southern Agriculture,53(9):2527-2536.] doi:10.3969/j.issn.2095-1191. 2022.09.015.
吉颖,吴淑华,崔晓艳,涂丽琴,高丹娜,程兆榜,周益军,陈新,季英华,郭青云. 2022. 江苏省大豆上一种新粉虱传病毒的分子鉴定及系统进化分析[J]. 江苏农业科学,50(10):30-36. [Ji Y,Wu S H,Cui X Y,Tu L Q,Gao D N,Cheng Z B,Zhou Y J,Chen X,Ji Y H,Guo Q Y. 2022. Molecular identification and phylogenetic analysis of a new whitefly-transmitted virus infecting soybean from Jiangsu Province[J]. Jiangsu Agricultural Sciences,50(10):30-36.] doi:10.15889/j.issn.1002-1302.2022.10.005.
李宁,王柏柯,王娟,黄少勇,戴麒,帕提古麗,高杰,余庆辉. 2019. 植物长链非编码RNA的生物学功能研究进展[J]. 植物生理学报,55(10):1427-1435. [Li N,Wang B K,Wang J,Huang S Y,Dai Q,Pa T G L,Gao J,Yu Q H. 2019. Advances in functional research of long no-coding RNAs in plants[J]. Plant Physiology Journal,55(10):1427-1435.] doi:10.13592/j.cnki.ppj.2019.0235.
刘婷,刘卫国,任梦露,杜勇利,邓榆川,邹俊林,方萍,杨文钰. 2016. 遮荫程度对不同耐荫性大豆品种光合及抗倒程度的影响[J]. 中国农业科学,49(8):1466-1475. [Liu T,Liu W G,Ren M L,Du Y L,Deng Y C,Zou J L,Fang P,Yang W Y. 2016. Effects of shade degrees on photosynthesis and lodging resistance degree of different shade tolerance soybean[J]. Scientia Agricultura Sinica,49(8):1466-1475.] doi:10.3864/j.issn.0578-1752.2016.08.004.
秦英之,車佳航,尹跃,饶书培,安巍,戴国礼,陈金焕. 2022. 基于全长转录组信息的枸杞SSR标记开发[J]. 植物遗传资源学报,23(6):1816-1827. [Qin Y Z,Che J H,Yin Y,Rao S P,An W,Dai G L,Chen J H. 2022. SSR Marker development of Lycium germplasm based on full-length transcriptome information[J]. Journal of Plant Genetic Resources,23(6):1816-1827.] doi:10.13430/j.cnki.jpgr.20220330004.
孙慧敏,赵团结,盖钧镒. 2010. 大豆苗期耐淹性的遗传与QTL分析[J]. 作物学报,36(4):590-595. [Sun H M,Zhao T J,Gai J Y. 2010. Inheritance and QTL analysis of submergence tolerance at seedling stage in soybean [Glycine max (L.) Merr.][J]. Acta Agronomica Sinica,36(4):590-595.] doi:10.3321/j.issn:0496-3490.2008.05.004.
涂丽琴,吴淑华,干射香,崔晓艳,赵文浩,程兆榜,陈新,周益军,季英华,朱月林. 2019. 江苏省蚕豆上菜豆黄花叶病毒的分子鉴定[J]. 江苏农业学报,35(4):804-810. [Tu L Q,Wu S H,Gan S X,Cui X Y,Zhao W H,Cheng Z B,Chen X,Zhou Y J,Ji Y H,Zhu Y L. 2019. Molecular identification of bean yellow mosaic virus infecting Vicia faba from Jiangsu Province[J]. Jiangsu Journal of Agricultural Sciences,35(4):804-810.] doi:10.3969/j.issn. 1000-4440.2019.04.008.
王大刚,李凯,智海剑. 2018. 大豆抗大豆花叶病毒病基因研究进展[J]. 中国农业科学,51(16):3040-3059. [Wang D G,Li K,Zhi H J. 2018. Progresses of resistance on soybean mosaic virus in soybean[J]. Scientia Agricultura Sinica,51(16):3040-3059.] doi:10.3864/j.issn.0578-1752.2018.16.002.
王佳,崔高峰,祝宁,任俊达,尚巧霞. 2021. 北京地区设施草莓8种病毒的酶联免疫吸附测定检测[J]. 微生物学通报,48(3):1048-1056. [Wang J,Cui G F,Zhu N,Ren J D,Shang Q X. 2021. Detection of eight viruses infecting greenhouse,strawberry in Beijing by enzyme-linked immunosorbent assay[J]. Microbiology China,48(3):1048-1056.]doi:10.13344/j.microbiol.china.200334.
王思宇,王宇. 2022. LncRNA UCA1在肿瘤诊断中的研究进展[J]. 生命科学,34(7):806-814. [Wang S Y,Wang Y. 2022. Research progress of lncRNA UCA1 in tumor diagnosis[J]. Chinese Bulletin of Life Sciences,34(7):806-814.] doi:10.13376/j.cbls/2022088.
王兴荣,刘章雄,张彦军,李玥,李永生,苟作旺,祁旭升,邱丽娟. 2021. 大豆种质资源不同生育时期抗旱性鉴定评价[J]. 植物遗传资源学报,22(6):1582-1594. [Wang X R,Liu Z X,Zhang Y J,Li Y,Li Y S,Gou Z W,Qi X S,Qiu L J. 2021. Evaluation on drought resistance of soybean germplasm resources at multiple growth periods[J]. Journal of Plant Genetic Resources,22(6):1582-1594.] doi:10.13430/j.cnki.jpgr.20210430002.
魏瑶,刘燕,图门白拉,赵明敏. 2020. 内蒙古自治区马铃薯病毒病的检测[J]. 江苏农业科学,48(8):111-115. [Wei Y,Liu Y,Tu M B L,Zhao M M. 2020. Detection of potato virus disease in Inner Mogolia Autonomous Region[J]. Jiangsu Agricultural Sciences,48(8):111-115.] doi:10. 15889/j.issn.1002-1302.2020.08.020.
伍越,王甜甜,李澤秀,纪庆柱,毛培利,诸葛绪钦,曹帮华. 2021. 木瓜转录组数据SSR标记的开发及其遗传多样性分析[J]. 植物生理学报,57(4):847-861. [Wu Y,Wang T T,Li Z X,Ji Q Z,Mao P L,Zhuge X Q,Cao B H. 2021. Identification of Chaenomeles EST-SSR markers and phylogenetic analysis[J]. Plant Physiology Journal,57(4):847-861.] doi:10.13592/j.cnki.ppj.2020.0512.
徐晓丹,冷淼,张明媛,柯希望,殷丽华,左豫虎. 2023. 基于转录组的小豆SSR分子标记开发及其应用[J]. 干旱地区农业研究,41(1):13-18. [Xu X D,Leng M,Zhang M Y,Ke X W,Yin L H,Zuo Y H. 2023. Development and application of SSR molecular markers based on transcriptome sequencing of adzuki bean[J]. Agricultural Research in the Arid Areas,41(1):13-18.] doi:10.7606/j.issn. 1000-7601.2023.01.02
严勇亮,张金波,路子峰,赵晓玲,刘海军,赵康,丛花. 2021. 大豆种质资源耐盐性鉴定与评价[J]. 新疆农业科学,58(1):65-71. [Yan Y L,Zhang J B,Lu Z F,Zhao X L,Liu H J,Zhao K,Cong H. 2021. Salt tolerance evaluation of soybean germplasm[J]. Xinjiang Agricultural Sciences,58(1):65-71.] doi:10.6048/j.issn.1001-4330.2021.01.008.
赵小慧,吴茜,陈正强,刘冲,邢锦城,陶小荣. 2021. 山东济宁大豆病毒病的病原鉴定研究[J]. 大豆科学,40(6):829-834. [Zhao X H,Wu X,Chen Z Q,Liu C,Xing J C,Tao X R. 2021. Identification of the viral pathogens infecting soybean in Jining,Shandong Province[J]. Soybean Science,40(6):829-834.] doi:10.11861/j.issn.1000-9841. 2021.06.0829.
Gupta R A,Shah N,Wang K C,Kim J,Horlings H M,Wong D,Tsai M C,Hung T,Argani P,Rinn J,Wang Y L,Brzoska P,Kong B,Li R,West R B,Vijver M J,Sukumar S,Chang H Y. 2010. Long non-coding RNA HOTAIR reprograms chromatinstate to promote cancer metastasis[J]. Nature,464(7291):1071-1076. doi:10.1038/nature08975.
Kim D,Pertea G,Trapnell C,Pimentel H,Kelley R,Salzberg S L. 2013. TopHat2:Accurate alignment of transcriptomes in the presence of insertions,deletions and gene fusions[J]. Genome Biology,14(4):R36. doi:10.1186/gb-2013-14-4-r36.
Kitagawa M,Kitagawa K,Kotake Y,Niida H,Ohhata T. 2013. Cell cycle regulation by long non-coding RNAs[J]. Cellular and Molecular Life Sciences,70:4785-4794. doi:10.1007/s00018-013-1423-0.
Li B,Dewey C N. 2011. RSEM:Accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics,12:323. doi:10.1186/1471-2105-12-323.
Liu N K,Xu Y Z,Qi L,Cao Y X,Yang D C,Liu S S,Wang X K,Mi Y J,Liu Y,Ding C X,Liu Y,Li Y,Yuan Y W,Gao G,Chen J F,Qian W Q,Zhang X M. 2022. A lncRNA fine-tunes salicylic acid biosynthesis to balance plant immunity and growth[J]. Cell Host and Microbe,30(8):1124-1138. doi:10.1016/j.chom.2022.07.001.
Ramon E C,Nicolás B,Claudia N,Irma G L,Patricia R P. 2014. Bean yellow mosaic virus in soybean from Argentina[J]. Journal of Phytopathology,164:322-325. doi:10.1111/jph. 12185.
Samertwanich K,Kertipakorn K,Chiemsombat P,Ikegami M. 2001. Complete nucleotide sequence and genome organization of soybean crinkle leaf virus[J]. Journal of Phytopathology,149(6):333-336. doi:10.1046/j.1439-0434. 2001.00629.x.
Trapnell C,Williams B A,Pertea G,Mortazavi A,Kwan G,Baren M J,Salzberg S L,Wold B J,Pachter L. 2010. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology,28(5):511-515. doi:10.1038/nbt.1621.
Yang X D,Niu L,Zhang W,He H L,Yang J,Xing G J,Guo D G,Zhao Q Q,Zhong X F,Li H Y,Li Q Y,Dong Y S. 2019. Increased multiple virus resistance in transgenic soybean overexpressing the double-strand RNA specific ribonuclease gene PAC1[J]. Transgenic Research,28(1):129-140. doi:10.1007/s11248-018-0108-8.
Yao C,Li C,Liu Z J,Xiao L,Bai H W,Shi B Y. 2022. LNCRNA XIST inhibits miR-377-3p to hinder Th17 cell differentiation through upregulating ETS1[J]. Computational Intelligence and Neuroscience,2022:6545834. doi:10.1155/2022/6545834.
(责任编辑 陈 燕)
收稿日期:2023-03-05
基金项目:国家自然科学基金项目(32060490,32260451);国家现代农业产业技术体系建设专项(CARS-04-CES30);廣西自然科学基金项目(2019GXNSFAA185009)
通讯作者:陈渊(1971-),https://orcid.org/0000-0003-0876-0154,研究员,主要从事大豆栽培和遗传育种研究工作,E-mail:chenyuan 313@163.com
第一作者:陈文杰(1982-),https://orcid.org/0000-0002-3317-4205,副研究员,主要从事大豆遗传育种及耐逆境机制研究工作,E-mail:cenwenji1030@163.com
陈文杰(1982-),副研究员,主要从事大豆遗传育种及耐逆境机制研究工作。主持国家自然科学基金项目“新型大豆皱叶基因CL12的图位克隆及其对产量性状影响”和“大豆皱叶症关键致病因子分析及其诱发的皱叶分子机制”2项、广西自然科学基金项目“大豆皱叶症抗基因鉴定及功能标记开发”1项,作为主要成员参与省部级项目8项;获广西科技进步奖二、三等奖各1项;以第一完成人育成已通过广西农作物品种审定委员会审定的大豆新品种1个,作为主要参与者育成已通过广西农作物品种审定委员会审定的大豆新品种8个;获第一发明人授权发明专利7件,实用新型专利7件;在《中国油料作物学报》《南方农业学报》《大豆科学》等期刊发表科技论文35篇,其中以第一作者发表中文核心期刊论文14篇。
