化学蛋白质组学在天然产物分子靶标鉴定中的应用
2023-10-25周璐祺崔婷茹郝楠赵雨薇赵斌刘颖超
周璐祺 崔婷茹 郝楠 赵雨薇 赵斌 刘颖超
(1.河北农业大学植物保护学院 河北省植物生理与分子病理学重点实验室,保定 071000;2.保定市气象局,保定 071000)
粮食安全是关系到国民经济可持续发展和社会稳定的国家战略,也是国家安全体系的重要内容。绿色农药在保障农产品产量和质量、防治各种农作物病虫草害的发生、保障粮食安全方面起着重要作用。通过农药的合理使用,可挽回世界农作物总产30%-40%的损失[1]。绿色新农药的开发和利用有利于农业增产增收和可持续发展,是各国农药产业发展的主要方向。经过20年的发展,虽然我国在绿色农药的创制和应用上取得了较多原创性成果,但仍然存在较多问题,例如原创性靶标少、新农药品种单一、植物耐药性增强等[2]。近年来,在医学上,天然产物已经逐渐被开发为新型药剂用于治疗各类疾病[3]。在农业上,我国的植物化学家们也基于天然的活性物质开发了农用化学品,但是品种相对较少,例如以白头翁(Pulsatilla chinensis(Bunge)Regel)、虎杖(Polygonum cuspidatum Sieb.et Zucc)、蛇床花(Cnidium monnieri(L.)Cuss)、马蓝(Baphicacanthus cusia(Nees)Bremek)等植物为材料,提取到活性物质并以其为基础开发了原白头翁素A、大黄素甲醚、蛇床子素、喹唑啉酮等4种新型杀菌剂[4];基于苦皮藤(Celastrus angulatus Maxim)开发出了对鳞翅目昆虫具有较好的杀虫效果的杀虫剂苦皮藤素[5]。尽管天然产物具有安全无毒、结构新颖等特点,但其化学结构复杂不能直接作为农药使用。天然产物具有多元的结构,可以以天然产物为先导设计新型衍生物或研究其新型作用靶标,进而加速具有自主知识产权的新型农药创制与开发。
新农药创制可以采取不同策略,常见创制方法有表型筛选和靶向筛选。基于药物靶向筛选策略是目前较为常用的新农药分子设计方法;然而大量、重复使用单一靶标农药,无疑会使“农药残留、有害生物再猖獗及生物抗药性”问题日益凸显,若轮换使用靶标相同或相似的农药,也会增加此类风险。近年来有研究表明,基于表型筛选发现新药的成功率高于靶向筛选[6],基于表型筛选获得的生物活性化合物是以相关病虫草害为基础进行筛选,并不限于单一的基因或者蛋白质。因此表型筛选近年来重新受到重视,但表型筛选的最大问题在于得到的化合物靶点未知[7],尽管在新农药创制的早期阶段不一定需要了解药物靶标,但是对候选化合物的靶点研究对全面解析候选化合物的作用模式是非常有必要的[8]。同时一个新靶标的价值往往高于一个新产品,例如以鱼尼丁为探针发现细胞膜上一类独特的作用靶标——鱼尼丁受体(ryanodine receptors,RyRs),化学家们基于鱼尼丁受体开发出目前常用的邻苯二甲酰类和邻甲酰氨基苯甲酰胺类杀虫剂[9]。新靶标的发掘作为绿色农药创制的重要途径之一,已成为国内外研究的重点和热点。蛋白质是大多数药物的主要靶点,小分子化合物与细胞内的蛋白靶点相结合是药物发挥药理活性的基础,然而监测蛋白活性及功能的方法在药物发现过程中仍未得到系统性的应用[10]。同时,药剂在发挥活性时往往不是针对单一蛋白,而是与多个蛋白质相互作用[11]。这种多靶点的作用模式使得确定天然产物的单一靶点并对其进行特异性研究十分困难。因此迫切需要一种能够全面识别天然产物靶点的方法。
以小分子天然产物为起点筛选靶标蛋白常用技术手段,包括化学蛋白质组学、化学基因组学和生物物理学等,其中化学蛋白质组学可以直接分析小分子化合物的靶标蛋白,是一种捕获和鉴定小分子化合物蛋白质靶标的有效方法,已经成为蛋白质组与药物靶点研究和新药研究的重要手段,及在蛋白质水平上鉴定小分子化合物与细胞内蛋白质相互作用的主要方法之一[12-13]。在本综述中,通过药物靶标发现实例,总结了基于化学蛋白质组学开发出的药物靶标鉴定方法,包括标记法和非标记法,并详细探讨了它们的原理、实验流程、优缺点及适用范围。由于医药领域的发展要先于农药,因此本文以医药靶标开发方法为主,旨在通过阐述基于化学蛋白质组学发现药物作用靶标最新方法的进展,为天然产物靶点及新农药创制研究提供参考。
1 基于标记的化学蛋白质组学靶标识别方法
生物体中小分子化合物与蛋白质相互作用关系是药物结构改造及成药的关键,分析小分子药物与靶点相互作用的信号和代谢规律变得日益重要,一方面通过活性分子的靶点信息可以对化合物进行针对性的改造从而提高其作用效果,另一方面可以确定活性分子对目标靶标的选择性,有效的预测其潜在的毒副作用,降低研发风险。化学蛋白质组学是化学生物学研究的重要手段之一,有别于以往对蛋白质进行定性或者定量鉴定的蛋白质组学技术,化学蛋白质组学旨在通过一系列功能各异的化学探针,结合蛋白质组学技术,解决复杂体系如细胞裂解液、活细胞、组织中的蛋白质如何与小分子药物相互作用的问题,目前已被应用于小分子药物靶点发现、药物先导化合物的筛选等研究中[14]。其一般流程为将引入亲和基质或报告基团的化学探针与蛋白质组进行孵育,利用亲和纯化等方法富集与探针结合的蛋白,再通过质谱等其他辅助技术来分析。常用的基于化学蛋白质组学的小分子药物靶点研究方法有以化合物为中心的化学蛋白质组学(compoundcentric chemical proteomics, CCCP)和以活性探针为基础的蛋白质谱分析(activity-based protein profiling,ABPP),工作流程如图1所示[15]。
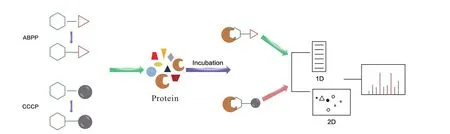
图1 CCCP和ABPP化学蛋白质组比较Fig.1 Comparison of CCCP and ABPP chemical proteome
1.1 以化合物为中心的化学蛋白质组学靶标鉴定方法
CCCP是最早用于确定靶标蛋白的方法,早在20世纪90年代,Harding等[16]就通过亲和层析方法筛选到天然免疫抑制剂 FK506 的结合蛋白——FKBP12(FK506 binding protein 12)。CCCP将药物分子进行修饰后引入标签分子,然后固定到基质上如琼脂糖凝珠或其他树脂,富集可以与药物分子结合的蛋白质,然后通过质谱技术进行鉴定。这是鉴定化合物靶标较为经典的方法,具有快速、大量富集靶标蛋白的优点。该技术主要用于研究具有生物活性的小分子化合物与蛋白质的相互作用,更有利于识别目标蛋白质以外的蛋白质,为新药开发提供理论依据。
Bach等[17]发现(R)-roscovitine除可作为周期蛋白依赖性激酶(cyclin-dependent kinases, CDKs)的专性抑制剂外,还可以抑制吡哆醛激酶的活性。酪蛋白激酶2(casein kinase 2, CK2)是一种临床上常见的治疗癌症的靶点,Gyenis等[18]利用固定在Sepharose磁珠上的ATP/CK2抑制剂TBB,从细胞裂解液中捕获和识别结合蛋白,证明CK2可以与TBB相互作用,同时还识别到一些潜在的目标蛋白,如羰基磷酸合成酶I(carbamoyl-phosphate synthase I,CPSM)、真核细胞生长因子1α(eukaryotic elongation factor 1-alpha 1, EEF1α1)等,并为CK2激酶抑制剂以及抗癌药物的开发奠定了基础。Faiella等[19]对Hardwickiic acid(HAA)进行修饰并固定在基质载体上与细胞裂解液进行孵育发现,HAA可以与Hsp27结合,为Hsp27化学抑制剂的开发提供了方向[19]。苦皮藤素作为化学杀虫剂可以影响昆虫的消化系统,Lu等[20]采用亲和层析的方法,从昆虫中肠中鉴定到11种可以作为苦皮藤素的潜在靶标蛋白的蛋白质,并成功预测了苦皮藤素与V-ATPase的结合位点。
尽管利用亲和色谱的方法鉴定小分子化合物靶标较为经典,但其对细胞材料要求较高,同时亲和层析实验在体外进行无法反映生理条件下药物与活细胞中蛋白质的相互作用。在此基础上,Hu等[21]开发了水溶性纳米聚合物药物载体,并利用其在活细胞中筛选药物靶点。此外,Bantscheff等[22]开发出kinobeads技术,成功解决了小分子药物无法进行化学修饰的问题。
1.2 以活性探针为基础的蛋白质谱靶标鉴定方法
ABPP是由美国的Cravatt课题组最先提出的蛋白质组学技术[23],是目前应用较为广泛的化学蛋白质组学技术。其在CCCP技术的基础上改进,是一种更为简便且适用性更为广泛的鉴定方法。与CCCP不同,ABPP的核心是将基于活性的探针(activitybased probes, ABPs)用于靶向功能相关的蛋白质组,并且这些探针可以共价修饰从丝氨酸水解酶到糖苷酶等酶类的活性位点[24]。因此,ABPP技术的关键步骤在于活性探针的设计及合成。一般来说,探针分子应以活性化合物为基础进行设计,同时探针分子可以与靶标蛋白共价结合,但在探针合成的过程中引入的标记物会影响小分子化合物的活性,点击化学(click chemistry)及光亲和标记技术(photoaffinity labeling technique)的引入进一步开拓了ABPP技术的适用范围[25],具体流程如图2所示。

图2 基于亲和蛋白质组分析策略Fig.2 Analysis strategies based on affinity proteomic
ABPP技术最初用于某一类蛋白质的活性监测,如半胱天冬酶、丝氨酸水解酶等[26]。除此之外,ABPP还可以作为工具来研究某一类化合物的具体作用机制,确定其有效官能团或者骨架,为开发更有效的药物提供基础[27]。近年来,利用ABPP鉴定小分子化合物在全蛋白组水平的作用靶标也得到了越来越多的应用,该技术以天然产物分子骨架中特定官能团的活性/功能为基础,选择相应的活性探针,利用竞争性策略筛选靶标蛋白[28],主要通过如下手段来实现。
1.2.1 生物素修饰 生物素修饰是一种被广泛应用的亲和纯化方法,生物素能与链霉抗生素蛋白(streptavidin)和亲和素(neutr avidin)特异性结合,是目前已知蛋白-配体作用力中最强的一种,因此链霉抗生素蛋白或亲和素常被用于与生物素结合来富集、纯化和识别活性小分子在细胞内的靶点。该方法的优点是生物素修饰的小分子化合物可以在进行亲和纯化之前,与细胞蛋白质组甚至与细胞膜内的靶蛋白充分接触。生物素富集方式主要为生物素与链霉亲和素不可逆结合,因此会对ABPP工作流程的复杂程度和质谱能检测到的蛋白数量产生较大影响,因而被不断优化与开发,如2019年,Zanon等[29]开发了一种脱硫生物素(desthiobiotin, DTB,结构式见图3)标签可被轻松洗脱,排除了内源生物素化的干扰,减少了肽段的损失,通过合成带有DTB标签的探针分子与金黄色葡萄球菌蛋白质组进行孵育,结果表明,带有DTB标签的探针分子操作起来较为简便,同时增加了金黄色葡萄球菌蛋白质组中半胱氨酸的覆盖率。
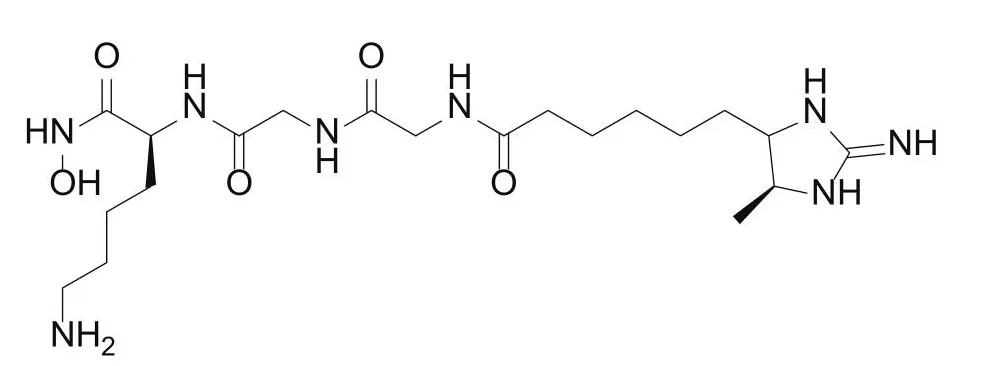
图3 DTB 标签的分子结构Fig.3 Molecular structure of DTB label
Eriocalyxin B是从中草药枇杷素中提取的抗肿瘤有效成分,Kong等[30]采用生物素富集的方式对Eriocalyxin B的靶标蛋白进行富集,成功鉴定到P50蛋白为其靶标蛋白。小白菊内酯具有抗炎活性,为确定其在体内的直接作用靶点,Kwok等[31]对小白菊内酯结构进行改造以合成亲和素探针,试验发现小白菊内脂可特异性结合并抑制IκB kinase β(IKKβ),导致核因子κB(nuclear factor-κB, NF-κB)活化的丧失,为开发新的抗炎药物提供了方向。此外,表1列举了近5年通过生物素富集进行小分子化合物靶标鉴定及作用机制探究的实例。
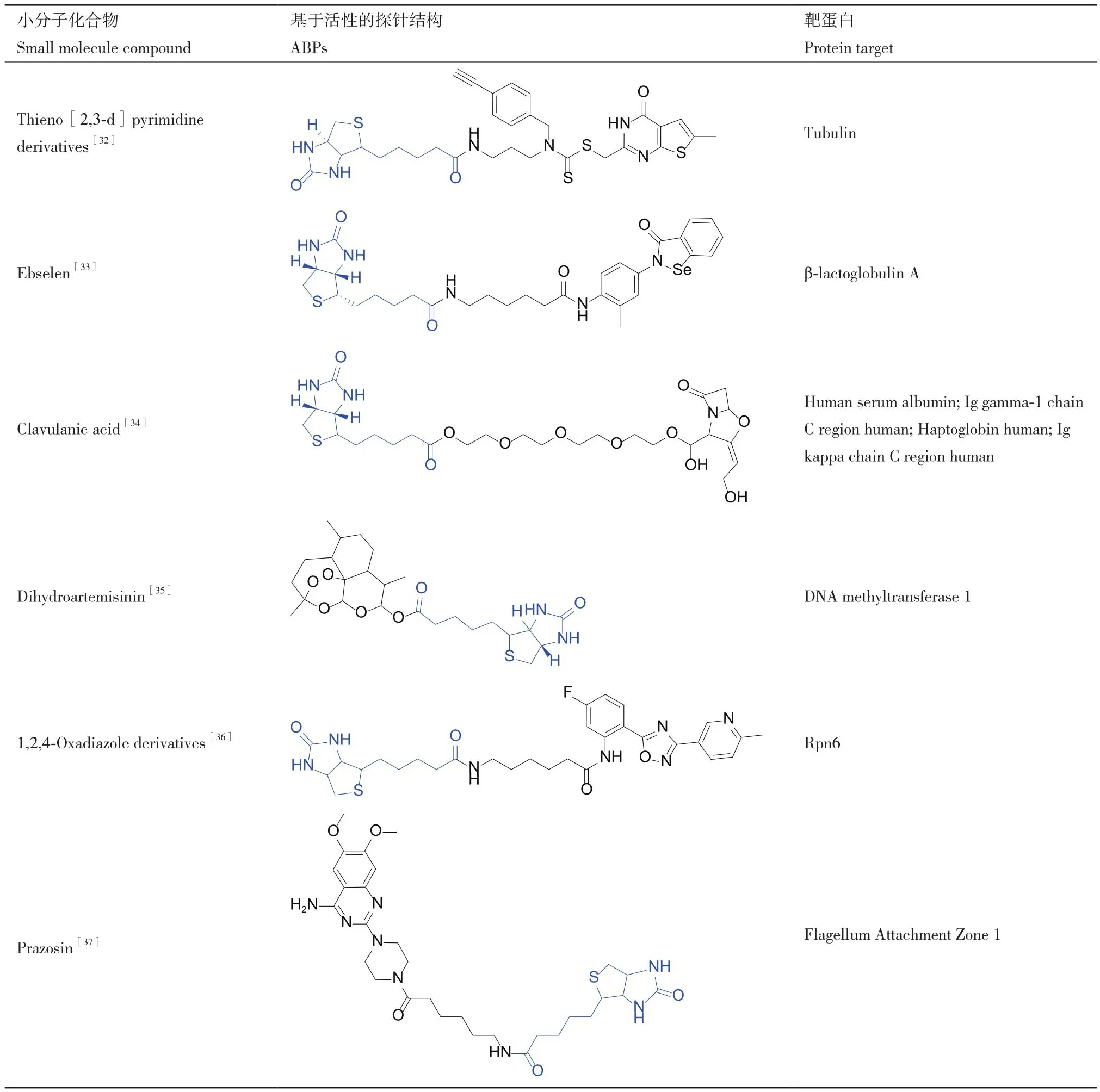
表1 生物素探针用于小分子化合物的靶点鉴定实例Table 1 Examples of biotin probes used for the target identification of small molecular compounds
1.2.2 生物正交化学反应 生物素标记的探针分子虽然被广泛应用, 但由于生物素体积较大,一般会影响小分子化合物的原始活性,造成活性下降或者消失,不利于靶标蛋白的鉴定。随着生物正交概念的出现及其反应类型的不断扩充,生物正交反应被引入到探针分子设计中。原有的生物素标签被替换为结构简单的生物正交基团——叠氮或炔基,后续可通过 Cu(I)催化的叠氮化物-炔烃环加成(Cu(I)-catalyzed azide-alkyne cycloaddition, CuAAC)反应连接上报告基团[38]。在试验过程中,活性探针通过对蛋白质组进行靶向标记,而后再凭借其末端手柄与报告基团共价交联,从而达到可视化呈现与富集下拉等目的。该设计可以减小探针的空间位阻,优化探针对于靶标蛋白的标记效果,同时简化合成步骤,降低整个实验的复杂程度。近年来,在天然产物骨架中引入生物正交反应基团是应用最为广泛的靶点鉴定策略之一。许多天然产物的作用靶标在应用点击化学合成探针后被解析,如胆固醇、姜黄素及青蒿素等[39-41]。甲磺酰菌唑作为高杀菌活性的候选药物,Chen等[42]采用ABPP策略系统地研究了其作用机制,利用生物正交化学反应设计亲和探针成功鉴定到二氢硫辛酸琥珀酰转移酶(dihydrolipoamide S-succinyltransferase, DLST)为其作用靶标,为DLST作为杀菌剂靶标及其抑制剂的创制打下了良好基础。近5年基于CC-ABPP鉴定小分子化合物靶标的实例如表2所示,该方法已经得到广泛应用。
1.2.3 光亲和基团探针 上述所有化学探针均具有活性基团,可以共价修饰靶蛋白中的氨基酸残基,从而在点击反应和富集过程中使探针与靶标之间稳定结合,但不是所有化合物都以共价修饰的方式与靶标蛋白结合,并且由于磁珠等外部因素的影响,易造成假阳性的结果。对于这种情况,研究人员利用合成的光敏性小分子化合物作为探针开发了光亲和标记法,形成具有位点特异性的不可逆共价结合,在药物靶点的研究中发挥巨大作用。光亲和标记结合蛋白质组学技术是近年来发展的在全细胞蛋白质组水平上鉴定药物分子的作用靶标最有效的方法之一[49]。
茉莉酸(jasmonates, JA)是一种调控植物生长发育的植物激素,内源性活性JA分子与F-box蛋白COI1和JAZ阻遏物形成复合物,以COI1依赖的方式通过26S蛋白酶体途径诱导JAZ降解。为了揭示COI1能否与Jas直接结合,Yan等[50]设计了一系列光亲和探针以验证COI1与Jas直接结合,为进一步阐明茉莉素的作用机制打下基础。除此之外,通过光亲和探针进行靶标识别的代表性天然产物还有万古霉素[51]和环孢菌素[52]等。近5年通过光亲和探针鉴定小分子化合物的靶标实例如表3所示。
以上不同的探针合成方法都在小分子化合物靶标鉴定过程中起着至关重要的作用,理论上亲和磁珠、生物素及光亲和基团标记的探针主要作用于细胞裂解液内,用来富集与小分子化合物作用的靶标蛋白,但不能获得较为全面的靶标蛋白。利用生物正交化学反应合成的探针可作用于活细胞内,获取的靶标蛋白较为全面,但同时也会存在非特异性结合造成假阳性。当同一化合物在不同官能团设计合成探针,或者设计不同类型的探针,最后得到的标记蛋白可能会有所不同[57-59]。因此在引入报告基因时应尽可能保证小分子化合物的活性不会发生改变,同时需要在实验过程中设置未加入探针的阴性对照。
2 非标记化学蛋白质组学靶标识别方法
虽然基于标记的化学蛋白质组学靶标识别方法已经完善并广泛应用,但是这些方法仍然存在局限性。基于标记法的靶标鉴定方法需要对化合物进行修饰,这往往会导致化合物活性改变甚至是丧失活性,同时部分小分子化合物结构复杂,没有合适的修饰位点,这些都成为基于标记的化学蛋白质组学靶标识别鉴定的干扰因素。随着药物靶标识别方法的不断发展,人们对小分子化合物结合靶标蛋白后蛋白酶水解、化学变性和热变性的认识逐渐加深,非标记化学蛋白质组学靶标识别方法应运而生。这些方法不需要对小分子化合物的结构进行修饰,并且可以有效地筛选混合化合物中的目标蛋白,较为快速的确定药物的作用机制,有利于促进天然产物药物的开发及应用,可对ABPP方法进行必要补充。图4列举出了常见的基于非标记化学蛋白质组学识别小分子化合物靶标的示意图。
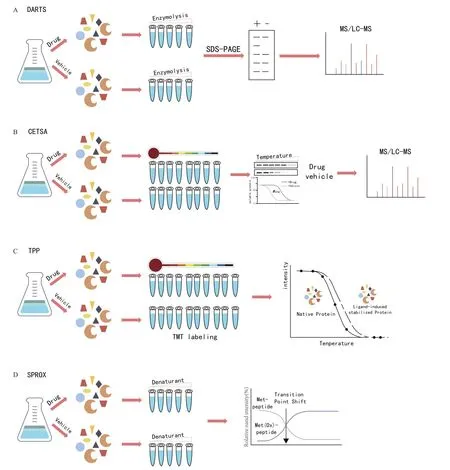
图4 非标记法识别药物靶标蛋白策略Fig.4 Schematics of label-free target identification methods
2.1 药物亲和力反应靶标稳定性
药物亲和反应的靶点稳定性(drug affinity responsive target stability, DARTS)技术的概念最初由Lomenick等[60]在2009年提出并进行的相关研究。该研究组推测药物与靶蛋白结合后,靶标蛋白会增强抗蛋白酶水解的能力,而在结合前后稳定性发生变化的蛋白质可以通过电泳检测到,然后通过质谱分析来确定小分子的直接靶点。该课题组首先通过雷帕霉素和FK506及其已知靶蛋白FKBP12验证了该方法的可行性,然后通过DARTS技术鉴定了白藜芦醇的作用靶标eIF4A。由于DARTS 技术操作简单、耗时短,得到了广泛的应用。Zhang等[61]运用DARTS的策略发现Src激酶可以作为苦参碱的靶标,这与亲和层析的结果一致。Kim等[62]通过DARTS和MSI的系统结合,鉴定和验证VEGFR2激酶作为Voacangine的靶标。Zhu等[63]采用 DARTS 技术筛选出 349 个与人参皂苷相关蛋白靶点,并最终确定人参皂苷可能通过靶向呼吸链复合物I和调节线粒体功能来缓解轻度认知障碍。除此之外,利用该技术还找到了新型杀菌剂YZK-C22的靶标蛋白丙酮酸激酶[64]、帕金森潜在治疗药物Andrographolide的靶蛋白DRP1(dynamin-related protein 1)[65]、结肠癌治疗药物Azelastine的靶蛋白ARF1(ADP-ribosylation factor 1)[66]。DARTS试验具有不需要对蛋白质进行修饰且不要求蛋白质的纯度,同时也不要求化合物的形式,既可以是单一的化合物,也可以是复杂的天然产物等优点[67],这为确定天然产物的作用靶标提供了有利条件。但由于DARTS试验后期主要依赖于SDS-PAGE和凝胶染色观察,对蛋白丰度较低的蛋白鉴定有限[60],同时DARTS试验选择的裂解液和蛋白酶类型也会影响天然产物靶标的鉴定[68],这些因素也会限制DARTS技术的准确性。
2.2 细胞热转移分析
蛋白质热稳定性分析(cellular thermal shift assay, CETSA)2013年由Molina等[69]首次提出,CETSA基于传统的热移位测定(thermal shift assay,TSA),该测定可检测由配体结合诱导的蛋白质热稳定性变化。这两种方法最主要的区别是TSA是针对单个纯化的重组蛋白或分离的蛋白质结构域进行的,而CETSA是用全细胞或细胞裂解物进行的。CETSA最早通过PCR和蛋白免疫印迹(western blot)显示靶标结合情况,但这种方法鉴定出的蛋白数量有限而且工作量大,随着质谱技术的发展,Savitski等[70]将CETSA与质谱结合起来,建立起完整的蛋白质测定方法。辣椒素对癌细胞具有抑制活性,有证据表明辣椒素可以抑制与肿瘤相关的NADH氧化酶(tumor-associated NADH oxidase, tNOX),Islam等[71]利用CETSA技术发现辣椒素可以与tNOX结合并使tNOX降解。Dziekan等[72]利用CETSA结合质谱技术,研究了奎宁和甲氟喹两种抗疟疾药物的靶标,证实了嘌呤核苷磷酸化酶(purine nucleoside phosphorylase, PfPNP)是这两种药物的共同靶标。Destruxin A(DA)是一种环肽霉菌毒素,因其良好的杀虫活性具有成为新型杀虫剂的潜力,有研究表明DA参与转录和蛋白质合成的调节且精氨酸tRNA合成酶(BmArgRS)、Lamin-C蛋白(BmLamin-C)和ATP依赖性RNA解旋酶PRP1(BmPRP1)为其候选结合蛋白,Wang等[73]通过CETSA技术验证DA与BmArgRS和BmLamin-C结合,为DA靶蛋白的发现及新型杀虫剂的创制提供了新的方向。
CETSA技术操作简单,受其他因素干扰较小,可直接在活细胞或整体组织水平进行研究,是目前较为常用的靶标识别方法之一。但由于某些靶标不会因与配体相互作用而稳定,或者在较高温度下相互作用丢失,从而掩盖了结合事件,可能出现假阴性结果[74]。为了克服 CETSA 的缺点,增加其在靶点和脱靶识别以及生物标志物发现上的应用,Savitski等[70]在 2014 年开发了基于CETSA的 TPP技术。TPP技术将CETSA技术与多重质谱技术结合,用于药物的靶标鉴定。利用TMT-10标签进行高通量筛选及鉴定,能够同时监测10个温度点,对数据进行标准化后绘制熔解曲线以发现稳定性差异蛋白。凭借高通量质谱分析的优势,TPP方法被广泛地用于测定药物或其他小分子诱导的蛋白质结构变化中。Li等[75]利用TPP技术发现Sp1可作为咔唑生物碱Murrayafoline A(MA)的直接靶标,并阻断IKKβ/NF-κB和p38/JNK MAPKs信号通路,为药物靶向Sp1用于神经炎症治疗以及新型药物开发提供了新方向。虽然TPP技术具有稳定性好、可以同时鉴别多种蛋白质等优点,但其具有耗时长、对膜蛋白检测有限等不足,仍需进一步改进。
2.3 氧化蛋白稳定性
SPROX与DARTS试验类似,是基于蛋白质折叠热力学变化原理的无标记靶标识别方法,将细胞裂解产物用药物分子处理后,与未经药物处理的对照组同时用不同浓度的氯化胍(蛋白变性剂)处理氧化,蛋白质被消化成多肽,对多肽片段中被氧化的残基进行质谱分析。Hatstat等[76]通过SPROX等技术鉴定N-芳基苯并咪唑化合物NAB2的靶标,并鉴定到一个全新的靶标Rab1a(一种GTP酶),这一发现增加了对NAB2减轻α-突触核蛋白毒性的机制的理解。此外,SPROX技术还可以与SILAC技术和iTRAQ技术相结合,Ogburn等[77]将SPROX 技术和SILAC技术结合,共鉴定出他莫昔芬(tamoxifen, TAM)和N-去甲基他莫昔芬(N-desmethyl tamoxifen, NDT)的结合蛋白163种和200种,其中Y-box结合蛋白1(Y-box binding protein 1, YBX1)可作为TAM的直接作用靶标。Geer Wallace等[78]将SPROX技术与iTRAQ技术相结合,鉴定出抗癌药物Manassantin A的靶点蛋白,为阐明其作用机制提供了新的理论支撑。虽然 SPROX 技术具有较好的应用潜力,但也存在一些不足:首先,由于甲硫氨酸氧化是测量参数,SPROX技术只能用于分析含有甲硫氨酸的肽段;其次,不同的甲硫氨酸残基可能表现出相同的氧化速率[79]。同时在实验过程中需要添加一定的氧化剂,因此SPORX并不适用于活细胞测定。
2.4 靶标结合可及性变化谱
靶标结合可及性变化谱(target-responsive accessibility profiling, TRAP)通过在蛋白质组学水平上监测配体诱导的赖氨酸可及性变化,来识别细胞环境中药物分子的结合蛋白。该方法通过对反应性赖氨酸的可及性变化的全局分析,来测量由于配体结合而在蛋白质靶标中诱导的空间位阻。简单地说,在药物分子存在时表现出显著丰度变化的小分子肽段被称为靶标反应肽[80]。Zhu等[81]通过TRAP技术筛选到雷公藤红素的结合蛋白CAP1,雷公藤红素通过CAP1抑制巨噬细胞cAMP-PKA-NF-κB信号通路,从而改善高脂饮食诱导的小鼠代谢综合征。环黄芪醇具有抗病毒、抗衰老、抗炎等功能,Deng等[82]通过TRAP技术寻找环黄芪醇的作用靶点,经筛选发现组织蛋白酶B(cathepsin B, CTSB)为CAG直接靶点。
2.5 色谱共洗脱
色谱共洗脱(target identification by chromatographic co-elution, TICC)是一种在非变性条件下,利用液相色谱技术检测小分子化合物靶蛋白的方法[83]。与靶标蛋白结合后,小分子化合物在液相色谱中的保留时间将与结合蛋白的保留时间一致。随后,通过质谱检测与小分子化合物同时洗脱的蛋白质就可以获取关于候选靶标蛋白的信息。利用该策略,Emili课题组鉴定了抗真菌天然产物 4513-0042的靶标蛋白 Erg6p[83]。TICC主要用于研究真核系统中的药物-靶点相互作用。2020年,Schäkermann等[84]用TICC技术鉴定抗生素靶点,证明了TICC适用于抗生素研究。通过合理的色谱保留时间,大量潜在的抗生素靶标(临床相关的抗生素靶标以及必需蛋白质)被覆盖,蛋白质复合物得以完整保存。该方法不需要药物或蛋白质的固定化或衍生化,适用于多种天然产物和合成化合物。同时该方法可以检测较低丰度的蛋白,但该方法对蛋白质稳定性要求较高,同时检测结果可能会受其他共洗脱蛋白的影响。
3 总结与展望
我国天然产物资源丰富,但是由于天然产物结构复杂且可能会有“多成分、多靶点”的情况,通过对其靶点鉴定及作用机制解析,可以优化并指导药物研究[6,85]。早期农药靶标与作用机制的研究往往采用生理生化的方法,从药物对生物表型、营养物质、激素分布和各类氧化还原酶的影响方面进行研究,现在样品制备方法与检测仪器都有了很大的改善,新型药物靶标与作用机制的研究已经更加深入精准[86]。本文综述了几种靶标鉴定的方法,化学修饰法是最早应用于靶标鉴定的方法,然而天然产物结构复杂、可修饰位点有限,同时结合小分子标签后可能会改变化合物活性,因此并不一定适用于天然产物的靶点鉴定。基于化学蛋白质组学的直接亲和富集的方法是小分子靶点识别的主流方法,非标记的化学蛋白质组学方法也为寻找小分子化学物的靶标提供了新的选择。此外,基于表型筛选的鉴定策略,针对基于化学蛋白质组学靶标鉴定方法的缺陷,提供了很好的补充和辅助。
当前,我国在新农药创制方面仍面临着许多挑战,如农药品种和剂型老化、原创靶标少、对现有农药抗性加剧、新剂型短缺等[2]。开发高效、低毒、选择性好、安全系数高的绿色农药已成为农药研发的重要目标。在新农药创制过程中,新靶标的发现具有十分重要的意义。新靶标一经发现,不仅会成为一系列农药创制的突破口,还将延缓抗性问题,为病虫草害的防治提供帮助。同基于表型筛选的方法进行新农药创制相比,基于靶标的药物创制由于其靶标已知,会耗时更短、更简便、花费更少[87],同时相较于基于表型筛选的药物,对作物更加安全,在农药研发过程中的重要性不言而喻。但无论哪种方法,得到的靶标蛋白都不是确切的,需要通过多种方式进行验证。随着化学蛋白质技术的发展,越来越多的技术被开发出来用于鉴定化合物蛋白质,其中大多数应用于医药上,对于农药创制而言,可以借鉴其中的鉴定技术,通过上述各种化学生物学研究策略和技术手段识别天然产物复杂体系的直接靶标,从根本上阐明其发挥生物学功能的基础,这对于新农药的开发和创制具有重要意义。
