咖啡酸苯乙酯对HepG2细胞氧化应激和脂质代谢的调节作用
2023-10-17陈瑞达林萌慧蔡成岗赵敏洁蔡海莺
刘 畅,常 超,陈瑞达,林萌慧,孙 蓉,蔡成岗,赵敏洁,蔡海莺,,3,*
(1.浙江科技学院生物与化学工程学院,浙江省农业生物资源生化制造协同创新中心,浙江省农产品化学与生物加工技术重点实验室,浙江 杭州 310023;2.浙江大学生物系统工程与食品科学学院,浙江 杭州 310058;3.美欣达集团有限公司,浙江 湖州 313002)
随着居民膳食结构的变化,高脂膳食摄入的不断增加成为日益显著的现象,过量摄入的高脂膳食会导致机体物质和能量代谢失衡[1],引起体质量增加、脂肪积累、氧化应激提高以及系统性炎症[2-3],并且伴随着肥胖、非酒精性脂肪肝(nonalcoholic fatty liver disease,NAFLD)、代谢综合征等多种疾病的产生[4-5]。引起人们广泛关注的脂代谢紊乱也和高脂膳食的过量摄入关系密切[6],当大量的能量被吸收后,未被消耗的能量会在脂肪组织中储存[7],脂肪积累在现有的脂肪细胞中,从而增大细胞体积,然而脂肪细胞储存脂肪的能力有限,多余的脂肪无法被脂肪组织吸收,从而引起血液、肝脏等的功能紊乱,而脂肪组织的扩大会逐渐导致肥胖[8-9]。咖啡酸苯乙酯(caffeic acid phenethyl ester,CAPE)是来源于蜂胶中的天然活性物质[10],具有许多生物学特性,如抗氧化、免疫调节、抗肿瘤[11-13]等。Sun Lulu等[14]的研究表明,CAPE具有改善脂代谢紊乱的作用,其能够调节不同组织脂代谢的多个信号通路,改善高脂膳食脂肪合成调控的一系列关键基因的表达,抑制组织中的脂肪细胞分化,减少脂肪的积累。迟乐[15]研究发现,CAPE通过调节过氧化物酶体増殖物激活受体-γ(peroxisome proliferator activated receptor-γ,PPAR-γ)、CCAAT/增强子结合蛋白α(enhancer binding protein α,CEBPα)和转化生长因子-β(transforming growth factor β,TGF-β)信号通路影响3T3-L1细胞生成脂肪,抑制脂质合成相关转录因子和下游相关因子的表达,显著降低细胞中甘油三酯(triglyceride,TG)含量和甘油-3-磷酸脱氢酶的活性,具有潜在的抗肥胖作用。有研究显示,CAPE可通过抑制糖尿病小鼠和人肝脏肿瘤细胞(HepG2)模型的应激,活化蛋白激酶(c-Jun N-terminal kinase,JNK)和核转录因子-κB(nuclear factor-κB,NF-κB)炎症信号通路,改善胰岛素抵抗[16]。目前这些研究都只集中于CAPE调节脂代谢的生化指标测定以及信号通路的验证,对于其直接作用的位点尚不明确。本研究用CAPE处理油酸诱导的HepG2细胞高脂肪模型,通过转录组测序,对2.5 μmol/L CAPE及高脂肪组的细胞进行测序,拟从转录组水平分析CAPE处理后HepG2细胞的基因表达差异,进而对基因进行富集分析,同时筛选出与脂代谢相关的基因,以期揭示CAPE调节脂代谢的潜在机理,为改善由高脂膳食引起的脂代谢紊乱提供理论参考。
1 材料与方法
1.1 材料与试剂
CAPE、油酸、油红O 上海阿拉丁生化科技股份有限公司;MEM(minimum essential medium)培养基、HepG2细胞 武汉普诺赛生命科技有限公司;TRIzol试剂南京诺唯赞生物科技股份有限公司;CCK-8细胞活力试剂盒、TG试剂盒、总胆固醇(total cholesterol,T-CHO)试剂盒、总蛋白定量试剂盒(BCA法) 南京建成生物工程研究所。
1.2 仪器与设备
超净工作台、CO2细胞培养箱、Sorvall™ ST 8小型台式离心机 美国赛默飞世尔科技有限公司;CKX53倒置显微镜 日本Olympus公司;SpectraMax iD3多功能酶标仪 北京众力挽生物科技有限公司。
1.3 方法
1.3.1 细胞处理
将复苏后的HepG2细胞培养至细胞密度达80%~90%,进行传代培养。HepG2细胞培养在10%(质量分数,下同)胎牛血清混合1%青霉素-链霉素试液的MEM完全培养基中,置于37 ℃、5% CO2培养箱中,常规换液、传代,当细胞处于对数生长期时取出进行实验。参考文献[17]的结果,使用0.2 mmol/L的油酸诱导HepG2细胞形成高脂肪模型的效果最佳,因此将细胞分为正常组(NCD)、高脂肪组(HFD)、高脂肪+处理组(包括HFD+7.5 μmol/L CAPE(CAPE7.5)、HFD+5 μmol/L CAPE(CAPE5)、HFD+2.5 μmol/L CAPE(CAPE2.5)、HFD+2 μmol/L CAPE(CAPE2)、HFD+1 μmol/L CAPE(CAPE1)),每组设置6 个平行对照,对处理后的细胞进行染色观察,在冰水浴条件下超声破碎细胞(300 W超声3~5 s/次,间隔20 s)后进行理化指标检测,选出降脂效果最优组别进行转录组水平分析。
1.3.2 指标检测
1.3.2.1 CCK-8细胞活力检测
细胞按照正常组(NCD)、高脂肪组(HFD)、高脂肪+处理组(包括HFD+10 μmol/L CAPE、HFD+7.5 μmol/L CAPE、HFD+5 μmol/L CAPE、HFD+2.5 μmol/L CAPE、HFD+2 μmol/L CAPE、HFD+1 μmol/L CAPE)分组,使用CCK-8试剂盒检测各处理组细胞活力。
1.3.2.2 油红O染色观察及吸光度测定
正常组、高脂组及高脂肪+处理组细胞弃掉旧培养液并用磷酸盐缓冲液(phosphate buffered saline,PBS)润洗2 次后,加入200 μL质量分数4%多聚甲醛溶液,将孔板转移到4 ℃冰箱内固定20 min,弃废液,用PBS润洗2 次,24 孔板中每孔加入200 μL 油红O染色10 min。待染色观察样品采用60%(体积分数,下同)异丙醇或70%乙醇溶液快速润洗,吸去废液,每孔加入1 mL PBS后在显微镜下观察拍照;待测吸光度样品使用60%异丙醇或70%乙醇溶液[18]萃取油红O,室温放置20 min,将萃取出的染液以500 μL/孔转移至24 孔培养板中,测定其在490 nm波长处吸光度[19]。
1.3.2.3 细胞内TG和T-CHO水平测定
分别收集各处理组细胞并制备成匀浆液。参照相应试剂盒说明书,测定各组HepG2细胞内的TG、T-CHO水平,并通过定量细胞中的蛋白进行标准化处理。
1.3.2.4 计算机预测靶点
通过S w i s s T a r g e t P r e d i c t i o n(h t t p://swisstargetprediction.ch/)数据库[20]检索CAPE的作用靶蛋白及对应基因,通过GeneCards(https://www.genecards.org/)数据库[21]检索脂代谢相关靶点基因,将结果导入STRING(https://string-db.org/)数据库构建共有靶点基因的蛋白相互作用关系网络[22]。使用DAVID(https://david.ncifcrf.gov)数据库[23]对共有的靶点基因进行京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析。以核心靶点基因编码蛋白为受体、CAPE为配体,采用AutoDock4软件进行分子对接,模拟CAPE调节脂代谢靶点。
1.3.2.5 细胞转录组样本处理及转录组测序分析
根据实验结果,对调节脂代谢指标效果较显著的HFD+2.5 μmol/L CAPE组进行转录组水平分析。取对数生长期[24]的HepG2细胞,以5×105个/孔接种于6 孔细胞板中,每个条件设置3 组平行,即HFD-1、HFD-2、HFD-3(HFD组)和CAPE-1、CAPE-2、CAPE-3(HFD+2.5 μmol/L CAPE组)。待细胞密度达到80%左右[25],加入0.2 mmol/L油酸将细胞诱导成高脂肪模型。培养24 h后,处理组弃去旧液,加入0.2 mmol/L油酸+2.5 μmol/L CAPE混合培养液。孵育24 h后,用移液枪吸去孔板中的细胞培养基,每孔加入质量分数0.25%胰酶1 mL,消化细胞3~5 min,吸取MEM完全培养基5 mL终止细胞消化,收集细胞后转入10 mL离心管中,4 ℃、200×g离心5 min,弃上清液,再加入3 mL PBS重悬,重复离心步骤,细胞沉淀中加入5×106个/mL TRIzol试剂[26],吹打混匀,1 mL/管分装入1.5 mL RNase-free离心管中,液氮速冻0.5 h,包埋在干冰中送样。细胞样品RNA质量检测及转录组分析由上海美吉生物医药科技有限公司完成。转录组测序步骤:利用Illumina测序平台对基因序列进行高通量转录组测序;通过Fastp软件对原始测序序列5’→3’的碱基质量及测序数据中A、T、G、C碱基含量进行评估,将与分析标准不符合的读数(reads)删除,保留对后续分析有帮助的高质量读数(clean reads)。
1.3.2.6 差异表达基因功能注释及富集分析
通过Hisat2软件进行基因序列比对分析,综合蛋白质直系同源簇(clusters of orthologous groups of proteins,COG)、非冗余蛋白库(non-redundant protein sequence database,NR)、基因本体(gene ontology,GO)、KEGG、蛋白质序列数据库(Swiss-Prot protein sequence database,Swiss-Prot)、蛋白质家族数据库(protein families database,Pfam)对所表达的基因进行注释;利用DESeq2、DEGseq和edgeR软件进行基因表达量差异分析。差异表达基因(differentially expressed genes,DEGs)筛选标准:|log2FC|≥1(FC:实验组基因表达量/对照组基因表达量)且P≤0.05。采用美吉生信云分析平台(https://cloud.majorbio.com/)对基因集中的基因/转录本进行KEGG通路富集分析[27],当P<0.05时认为该通路富集显著。
1.4 数据统计分析
采用Origin 2019软件对数据进行分析与处理,并用GraphPad prism6软件作图,实验数据以平均值±标准差表示,数据间比较采用单因素方差分析(ANOVA),以P<0.05表示差异显著。
2 结果与分析
2.1 CAPE对油酸诱导HepG2细胞脂质代谢的影响
根据文献[28-29]报道,CAPE在高于20 μmol/L时具有一定细胞毒性,CCK-8细胞活力检测结果显示,10 μmol/L CAPE显著降低细胞活力,而CAPE浓度为7.5、5、2.5、2、1 μmol/L时,HepG2细胞的活力均保持在98%以上,因此,确定调节脂代谢的CAPE浓度为1~7.5 μmol/L。采用油红O染色观察各组细胞脂滴积累情况,并于490 nm波长处测定其吸光度。和NCD组相比,HFD组油红O染色效果变化显著(图1),说明油酸诱导高脂肪模型建模成功。根据染色效果及吸光度结果分析,在CAPE浓度为2.5 μmol/L时,其对脂质积累的抑制效果最为显著。对改善脂肪积累效果最佳的处理组和高脂肪组(即2.5 μmol/L CAPE组和HFD组)进行转录组分析,并对2 组细胞进行油红O染色以确定处理效果,结果如图2A、B所示。和HFD组相比,CAPE 2.5组细胞脂肪积累水平显著降低。进一步测定破碎细胞中TG及T-CHO水平,经过蛋白校准后的细胞TG和T-CHO水平变化如图2C、D所示。与HFD组对比,CAPE 2.5组细胞中TG水平有明显的下降趋势,但CAPE处理对于胆固醇代谢调节效果不显著,这进一步证明CAPE干预具有明显缓解高脂细胞模型中脂滴积累的效果。

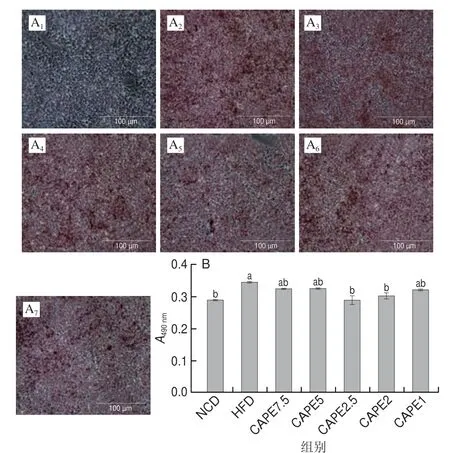
图1 经CAPE处理的HepG2细胞染色观察及吸光度Fig.1 Observation and absorbance of stained HepG2 cells treated with CAPE
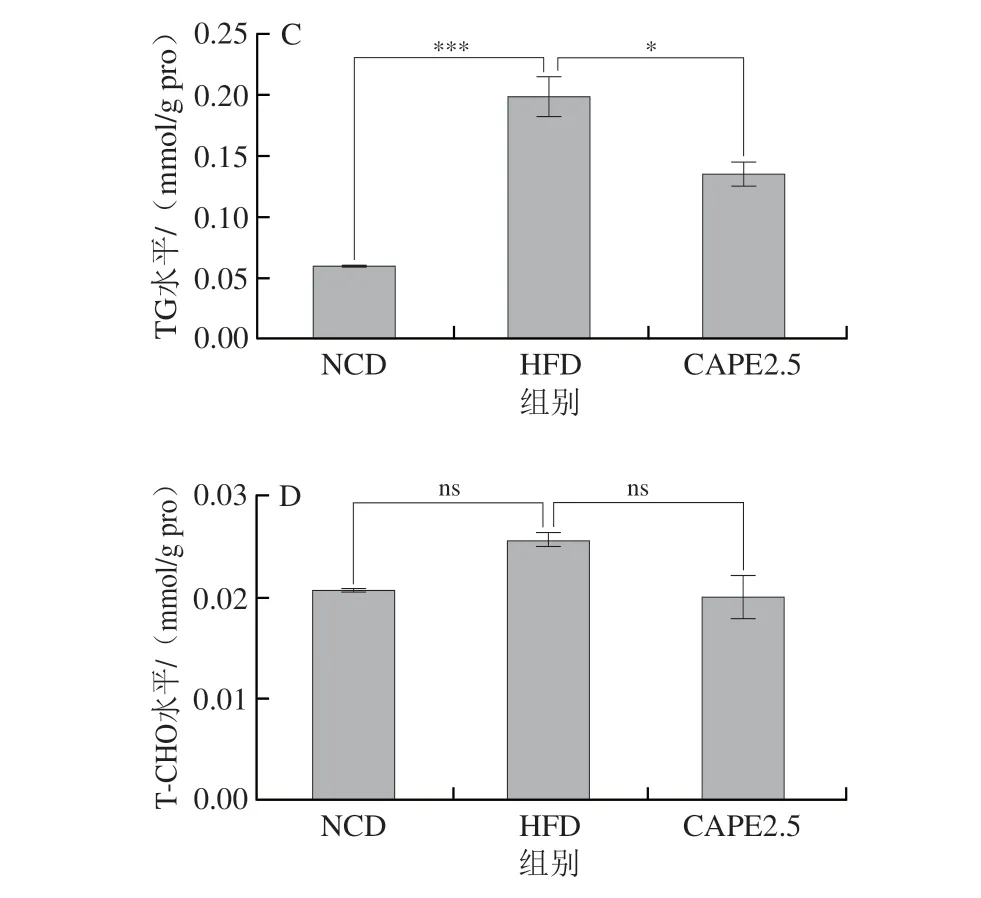
图2 经2.5 μmol/L CAPE处理HepG2细胞的染色及指标检测Fig.2 Observation and characterization of stained HepG2 cells treated with 2.5 μmol/L CAPE
2.2 经CAPE处理的HepG2细胞转录组测序及DEGs分析
对CAPE处理的HepG2细胞与高脂肪组细胞进行转录组测序及DEGs分析,CAPE组和HFD组经过过滤后的序列数量(即clean reads数目)分别有47 575 973 条和47 867 835 条,所占比例分别为96.07%和96.03%,根据经CAPE处理的HepG2细胞中基因表达的变化情况,共筛选出3 270 个DEGs。其中,表达上调的DEGs有1 351 个,表达下调的DEGs有1 919 个,DEGs表达模式聚类见图3。
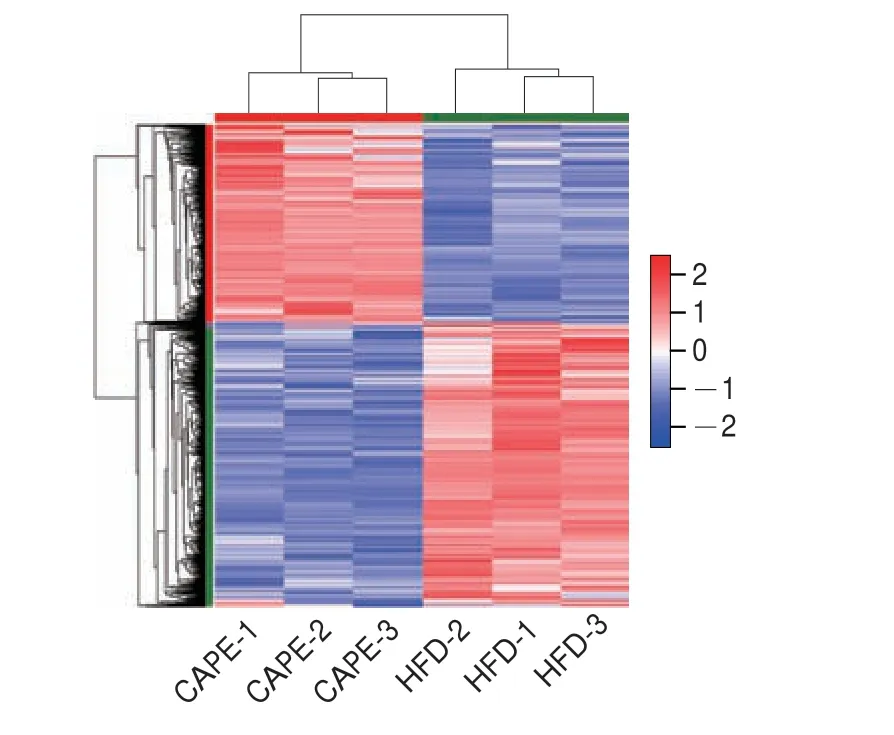
图3 DEGs表达模式聚类Fig.3 Clustering analysis of DEGs expression pattern
2.3 经CAPE处理的HepG2细胞DEGs KEGG通路功能注释及富集分析
为进一步明确DEGs的功能,对HFD组及CAPE组之间的DEGs进行KEGG功能注释分析。如图4所示,DEGs分别注释到代谢、遗传信息的处理、环境信息处理、细胞过程、有机系统和人类疾病中。经CAPE处理的HepG2高脂肪模型细胞的DEGs在脂代谢上有相关注释,说明脂代谢相关基因在CAPE的影响下产生差异性表达。
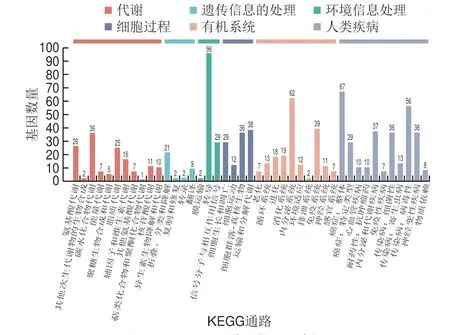
图4 KEGG通路功能注释分析Fig.4 Functional annotation analysis of KEGG pathways
前15 条显著富集的KEGG通路如表1所示,表中比例表示相应KEGG通路在目标基因集中所占比例,分子为基因集中富集到该KEGG通路的基因或转录本数目,分母为该基因集中具有KEGG注释的基因或转录本总数目,将P<0.05定义为该功能显著富集,可以发现显著富集的通路主要集中在代谢和信号转导上。对DEGs进行KEGG通路富集分析,发现DEGs主要参与果糖和甘露糖代谢(fructose and mannose metabolism)、糖酵解/糖异生(glycolysis/gluconeogenesis)、HIF-1信号通路(HIF-1 signaling pathway)、脂肪酸降解(fatty acid degradation)等。进一步分析KEGG通路中与脂质代谢和氧化应激相关的主要富集信号通路基因转录水平发现,在HIF-1α信号通路上DEGs显著富集,推测CAPE通过HIF-1α通路改善高脂诱导的HepG2细胞氧化应激,从而改善脂代谢紊乱和氧化应激压力;另外,多个脂肪酸降解通路相关关键基因表达量差异显著,脂肪酸代谢上游调控基因PPARα表达也发生上调,而PPARα信号通路与脂肪酸分解代谢[30-31]关系密切。与GeneCards数据库(https://www.genecards.org/)[21]靶点预测结果一致,CAPE对肝脏细胞具有多个作用靶点,其通过与靶点的相互作用调节脂代谢相关的多个代谢通路。Sun Lulu等[14]所进行的小鼠实验结果显示,CAPE能够调节不同组织脂代谢的多个信号通路以及脂肪合成的一系列关键基因的表达,改善高脂膳食小鼠脂代谢紊乱,说明CAPE对脂代谢调节具有多靶点作用。
2.4 CAPE调节HepG2细胞HIF-1α通路改善氧化应激
转录组DEGs分析显示,CAPE处理能上调高脂诱导细胞的HIF-1α基因转录表达水平(提高了0.326 倍),并且显著调节HIF-1α下游厌氧呼吸等通路基因PDK1、HK1、PFKP、PFKFB3、LDHA、ALDOC、ENO1、PGK1、EGLN3的表达(表2),表明CAPE处理能调节HIF-1α通路。另外,CAPE组细胞RTK、MEK、elf4E相关基因表达相较于高脂肪组分别上升了1.5、1.37、1.2 倍,且4E-BP1表达量下降了42%,表明CAPE可能通过MEK-ERK-4E-BP1-elf4E通路上调HIF-1α的表达(图5)。计算机辅助预测发现,mTOR和MAPK相关的蛋白激酶为CAPE的潜在作用靶点,表明CAPE分子能通过与潜在靶点相互作用,影响HIF-1α蛋白翻译表达。进一步分析显示,CAPE组中参与HIF-1α泛素化降解通路的基因PHD表达量降低,表明CAPE处理能抑制泛素化介导的HIF-1α蛋白降解,提高HIF-1α蛋白积累水平。因此,CAPE处理能调节HIF-1α的翻译和泛素化降解,促进机体无氧呼吸的进程,缓解高脂处理细胞的氧化应激压力。氧化应激在脂代谢紊乱及相关疾病中的重要作用备受瞩目[32]。研究表明,机体氧化应激的增加常伴随脂质的异常堆积加剧以及脂质从头合成的显著改变[33]。另外,活性氧和炎症诱导的氧化应激能够导致脂质过氧化、线粒体和过氧化物酶体的脂肪酸氧化功能损伤,并促进细胞因子释放,进一步加剧炎症和肝细胞纤维化,这是酒精性脂肪肝和其他肝脏疾病的主要发展机制[34]。
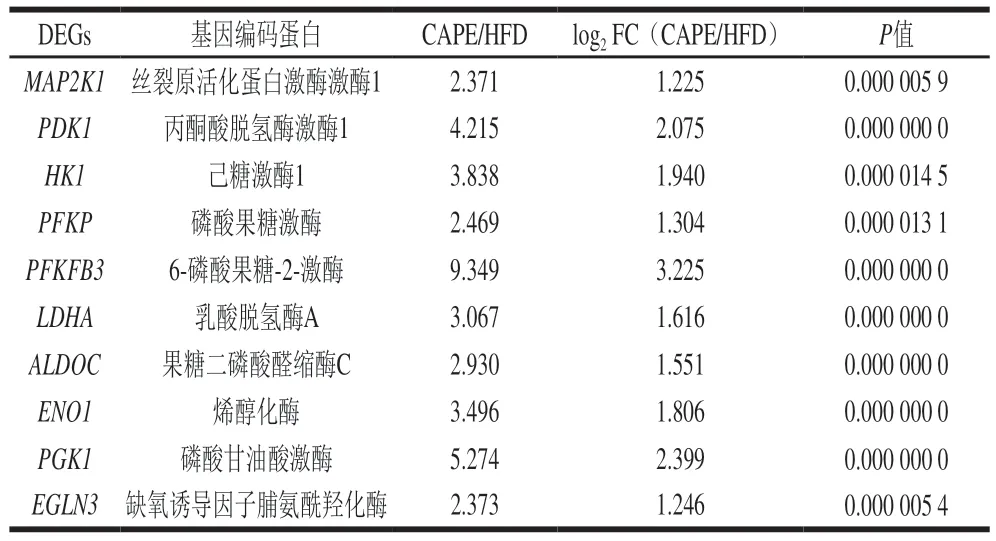
表2 HIF-1α通路DEGs表达Table 2 Expression of DEGs associated with HIF-1α signaling pathway

图5 CAPE调节HIF-1α通路的潜在机制分析Fig.5 Analysis of the underlying mechanism of CAPE regulation of HIF-1α signaling pathway
2.5 CAPE调节HepG2细胞脂肪酸分解代谢通路
经KEGG信号通路基因差异表达分析发现,CAPE能改善高脂HepG2细胞的脂肪酸分解代谢和PPARα信号通路相关多个基因的表达(表3)。PPARα通路参与调节脂肪酸转运、脂肪酸氧化[35]、脂肪酸降解及胆固醇代谢等重要脂代谢通路。如图6所示,相较于高脂肪组,CAPE组细胞PPARα下游与脂肪酸氧化代谢相关的蛋白表达量明显上升,如肉碱棕榈酰转移酶I(carnitine palmotoyltransferase I,CPT1)和肉碱棕榈酰转移酶II(carnitine palmotoyltransferase II,CPT2),其中调控CPT1、CPT2、PPARα表达的CPT1A、CPT2、PPARα基因分别上调了1.039、0.755、0.661 倍,调控脂肪酸结合蛋白5表达的FABP5基因上调了1.598 倍,表明CAPE处理促进了细胞内脂肪酸的活化进程。另外,CAPE组高脂诱导细胞脂肪酸β氧化进程相关酶基因表达上调,在脂肪酸β氧化的氧化、水合、脱氢、硫解[36]4 个过程中,调控酰基辅酶A脱氢酶(acyl-coenzyme A dehydrogenase,ACADM)合成的基因ACADM表达量上升了0.38 倍,编码长链酰基辅酶A脱氢酶(acyl coenzyme a dehydrogenase very long chain,ACADVL)合成的基因ACADVL表达量上升了0.39 倍;调控烯醇辅酶A水合酶S1(enoylcoenzyme A hydratase S1,ECHS1)和烯酰辅酶A 水合酶(hydroxyacyl-coenzyme A dehydrogenase,HADHA)合成的基因ECHS1及HADHA表达量分别升高了0.2 倍和0.31 倍;调控L-β-羟脂酰CoA脱氢酶(hydroxyacylcoenzyme A dehydrogenase,HADH)合成基因HADH表达量提高了1.42 倍;编码脂酰CoA硫解酶(cetyl-coenzyme A acyltransferase,ACAA)合成的基因ACAA1和ACAA2表达量分别没有变化和提高了0.47 倍,促进了长链脂肪酸的氧化分解。由此可知,CAPE处理可能通过PPARα通路和脂肪酸β氧化通路改善高脂诱导HepG2细胞内的脂代谢。
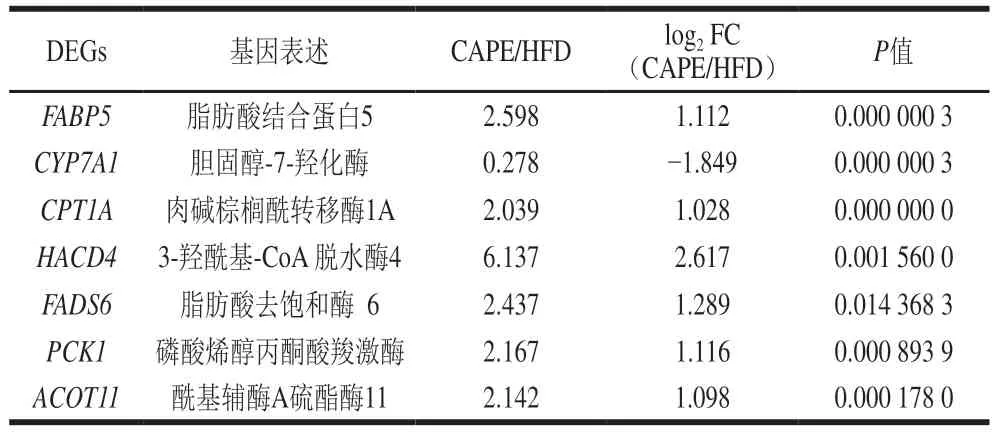
表3 PPARα通路DEGs表达Table 3 Expression of DEGs associated with PPARα signaling pathway

图6 CAPE调节脂肪酸分解的潜在通路分析Fig.6 Analysis of the underlying pathways of CAPE regulation of fatty acid breakdown
对CAPE调节脂代谢作用具体机制的研究表明,可能存在多种作用靶点和调节通路以及组织和细胞特异性[37]。迟乐[15]对3T3-L1脂肪细胞研究发现,CAPE通过调节PPAR-γ、CEBPα和TGF-β信号通路影响脂肪合成,抑制脂质合成相关转录因子和下游相关因子的表达,发挥抗肥胖的作用。Nie Jiarui等[16]研究发现,CAPE可通过抑制糖尿病小鼠和HepG2细胞模型的JNK和NF-κB炎症信号通路改善胰岛素抵抗和糖脂代谢。最近有研究表明,CAPE能显著抑制小鼠肠道菌群胆盐水解酶(bile salt hydrolase,BSH)活性,提高胆汁酸中牛磺β-鼠胆酸含量,从而选择性抑制胆汁酸法尼醇X受体(farnesoid X receptor,FXR),抑制高脂膳食引起的神经酰胺含量升高和脂肪生成,同时改善胰高血糖素样肽-1(glucagonlike peptide-1,GLP-1)分泌,改善小鼠NAFLD[38]。另外,金铭等[39]利用25-羟固醇(25-hydroxycholesterol,25-OHC)通过14-3-3η信号蛋白研究蛋白组磷酸化修饰作用诱导肝脏上皮L02细胞的脂代谢紊乱,发现CAPE可通过泛素化依赖途径促进14-3-3η信号蛋白降解,进而逆转25-OHC诱导的肝脏细胞脂代谢紊乱。有别于小鼠实验以及脂肪细胞体外实验研究,本研究采用HepG2细胞体外实验和转录组测序高通量筛选方法,进一步分析CAPE对脂肪酸诱导的高脂肪模型细胞的直接作用靶点。研究发现,CAPE对肝细胞同样具有多重靶点,与脂代谢相关的作用通路包括调节HIF-1α通路(缓解高脂细胞氧化应激)和脂肪酸分解代谢通路。CAPE通过调节HIF-1α的翻译和泛素化降解,促进机体无氧呼吸的进程,缓解高脂处理细胞的氧化应激压力,改善细胞代谢微环境和脂代谢紊乱;CAPE处理可能通过调节PPARα通路和脂肪酸β氧化通路多个关键基因表达从而改善高脂诱导HepG2细胞内的脂代谢。
3 结 论
CAPE对细胞脂代谢的改善机制和关键靶基因的研究并不完善,本研究结果显示,CAPE可以显著影响油酸诱导的HepG2高脂肪模型细胞的基因表达谱,表明CAPE调节脂代谢的靶点具有多重性。KEGG信号通路富集分析结果表明,CAPE的主要调节通路包括HIF-1α通路和脂肪酸降解通路,CAPE处理促进高脂细胞模型中HIF-1α的表达,并抑制了HIF-1α蛋白的泛素化降解,通过对下游调节基因表达的调控,缓解肝细胞内的氧化应激并改善细胞微环境。此外,受CAPE影响的高脂细胞模型PPARα通路被激活,与脂肪酸转运和氧化代谢相关的CPT1、CPT2等蛋白表达量升高,表明CAPE也可通过PPARα通路调节细胞内脂肪酸氧化分解,改善脂代谢。本研究为CAPE调节脂代谢的分子机制研究提供了一定的理论依据,为膳食干预高脂膳食引起的脂代谢紊乱提供参考。
