花生KNOX 基因家族的全基因组鉴定及组织表达分析
2023-09-13宋晓峰鲁成凯董晓娜高晓东孙熙文付春
宋晓峰鲁成凯董晓娜高晓东孙熙文付春
(潍坊市农业科学院,山东潍坊 261071)
同源异型盒基因家族(homeobox gene family)编码一类含有同源异型盒结构域的转录调控因子。 同源异形盒基因首先在果蝇突变体研究中发现并命名[1-2],随后发现该基因家族在动物、植物、真菌中广泛存在[3-7]。 同源异型盒基因的典型特征是含有一段高度保守的DNA 序列,长度约180 bp,其编码的60个氨基酸残基在三维空间上形成一个三螺旋区结构域。 根据蛋白质的结构特征,通常将植物同源异形盒蛋白分为14 类,其中KNOX(knotted1-like homeobox)和BELL 属于非典型同源异型盒蛋白,其同源异型盒区的螺旋Ⅰ和螺旋Ⅱ之间多3个氨基酸残基(Pro-Tyr-Pro),从而组成TALE(three amino-acid loop extension)同源异型盒蛋白超家族[8]。
KNOX基因广泛存在于植物中[3],其编码的蛋白质具有四个结构域,分别为KNOX1、KNOX2、ELK 和Homeobox KN[9]。 植物中第一个KNOX基因KNOTTED-1(Kn1)在玉米中发现,起控制细胞分化作用[10]。 随后在玉米、水稻、拟南芥、小麦、棉花、豆科植物等中发现大量KNOX基因[11-20]。 研究者根据基因家族成员所编码蛋白的结构域序列相似性及表达模式,将玉米中KNOX基因家族分为两个亚家族——Class Ⅰ和Class Ⅱ[21]。 在表达模式上,Ⅰ类KNOX基因亚家族成员主要在分生组织中特异性表达,通过参与调节细胞分化,进而影响组织器官的形态建成[22-23]及控制籽粒大小[24];Ⅱ类KNOX基因亚家族成员在各组织器官中普遍表达[21]。 拟南芥中Ⅰ类KNOX基因有 4个, 包括STM(At1g62360)、KNAT1(At4g08150)、KNAT2(At1g70510)、KNAT6(At1g23380),其中,STM对茎尖分生组织的形成以及功能维持起重要作用[25],KNAT1能够激活细胞分裂素的合成[12];Ⅱ类KNOX基因也有4个,即KNAT3(At5g25220)、KNAT4(At5g11060)、KNAT5(At4g32040)、KNAT7(At1g62990),由于突变体表型缺乏,对其功能研究相对较少。
我国是世界上最大的花生生产国,花生单产是世界花生单产的2.31 倍[26]。 花生是我国重要的油料作物,总产居油料作物之首。 鉴于花生基因组庞大,遗传背景相对复杂,其分子生物学研究相对滞后于其他豆科植物,如大豆、蒺藜苜蓿等。但随着栽培种花生基因组序列信息的公布,这种局面正在改变。 本研究在全基因组水平上对花生KNOX基因家族成员进行鉴定,并对其序列特征、进化关系和表达模式进行系统分析,以期为解析花生KNOX基因的重要作用提供参考。
1 材料与方法
1.1 AhKNOX 全基因组鉴定
栽培种花生(Arachis hypogaeaL.)参考基因组、蛋白序列及注释文件来自花生数据库(https://www.peanutbase.org/)。 拟南芥(Arabidopsis thalianaL.)KNOX基因家族成员数据下载自TAIR 网站(http:/ /www.arabidopsis.org/)。 以拟南芥的8 条KNOX 蛋白序列(STM、KNAT1 ~KNAT7)为种子序列,利用TBtools v1.108[27]软件的Blast 命令(E-value<e-5),在全基因组水平检索花生蛋白数据库中候选KNOX 蛋白成员,并删除冗余序列。 为保证候选成员的真实性,利用NCBI-CDD 和Pfam 在线工具进一步鉴定,去除不包含结构域的序列。
利用ProtParam 网站预测每个KNOX 成员的蛋白理化性质,具体包括氨基酸(amino acid,AA)数、分子量、等电点和亲水性,并在Softberry 网站上利用ProtComp 9.0 工具预测其亚细胞定位。
1.2 AhKNOX 基因结构与染色体定位分析
利用TBtools v1.108 的VisualizeGene Structure(Basic)工具对花生KNOX家族成员进行基因结构分析,Gene Location Visualize From GTF/GFF工具进行染色体定位分析。
1.3 AhKNOX 基因家族成员蛋白保守基序与结构域分析
利用MEME 工具对花生KNOX 成员的保守基序进行在线预测,设置基序数目为10,其他参数为默认值;蛋白保守基序和CDD 预测的结构域利用TBtools v1.108 的Gene Structure View(Advanced)工具绘制。
1.4 AhKNOX 基因家族成员系统发育分析
利用软件MEGA 5[28]的“ClustalW”程序对花生与拟南芥的KNOX基因家族成员进行多重序列比对,参数选择默认。 并选择邻接法(Neighborjoining,NJ)构建系统发育树,相关参数设置如下:计算距离的替代模型(Substitution Model)选择泊松校验(Poisson correction),Bootstrap 1 000 次。导出.nwk 文件利用FigTree v1.4.4 软件进行美化并参考拟南芥KNOX基因家族的分类系统,划分亚家族和亚组。
1.5 AhKNOX 启动子顺式作用元件分析
根据花生基因组数据,提取AhKNOX基因转录起始位点上游2 000 bp 的序列作为启动子区,并递交至PlantCARE 数据库,检索并筛选潜在的顺式作用元件,并利用TBtools v1.108 的Basic Biosequence View 工具进行可视化。
1.6 AhKNOX 全生育期及非生物胁迫下表达模式分析
从NCBI 下载花生22 种不同组织的RNA-seq数据(PRJNA291488)[29],分析AhKNOX基因的时空表达模式。 基于非生物胁迫处理下的RNA-seq 数据(PRJNA553073),分析AhKNOX基因逆境胁迫下的表达模式。 具体处理如下:分别用1% NaCl、20%PEG6000、20 mg/L ABA、1% H2O2、125 mg/L 茉莉酸甲酯(methyl jasmonate,MJ)、高温(HT,37 ℃)以及低温(LT,4 ℃)处理幼苗24 h。 RNA-seq 数据通过Hisat2、Samtools 和Cufflinks 分析得到FPKM 值。 将FPKM 值转化为log2(FPKM),使用TBtools v1.108 制作表达谱的热图。
1.7 AhKNOX 在模拟干旱胁迫下的RT-qPCR验证
根据转录组数据分析筛选2个显著响应PEG胁迫的AhKNOX基因进行RT-qPCR 分析。 以潍花8 号为试验对象,2023年2月1日催芽,2月4日定植于水培盒利用Hoagland 溶液在人工气候箱(上海一恒,MGC-300H)中培养,2月14日发育至三叶期时开始处理。 用 18% (W/V)PEG6000 做胁迫处理,处理3 d 后换回Hoagland营养液复水培养2 d;对照组用不含PEG6000 的Hoagland 溶液培养。 分别于处理0、1、2、3、4、5 d采集根,液氮速冻后用于总RNA 的提取。 利用MolPure®Plant RNA Kit 试剂盒(19291ES50)提取RNA,用Thermo Scientific Fermentas 反转录试剂盒(K1622)获得cDNA。 RT-qPCR 分析采用SYBR®Green real-time PCR Master Mix (TOYOBO)在ABI 7500 FAST 平台上进行,以Ahactin11为内参基因,利用NCBI 的Primer-BLAST 进行引物设计(表1),每个样品3 次重复。
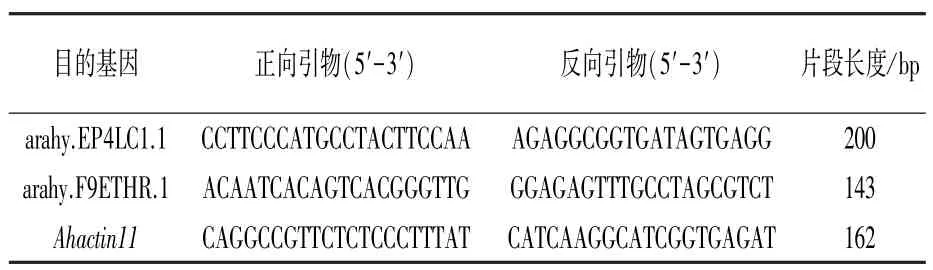
表1 RT-qPCR 引物信息
2 结果与分析
2.1 AhKNOX 全基因组鉴定
经过比对搜索,在花生基因组数据库中共鉴定到26个AhKNOX基因(表2)。 理化性质预测结果显示,AhKNOX基因编码的氨基酸长度差异较大,最短为139 aa(arahy.ZTJV1A.1),最长为478 aa(arahy.TRGC7G.1),分子量对应为最小16 116.39 Da、最大52 987.90 Da。 除arahy.ZTJV1A.1 编码的蛋白质等电点达到8.63 外,其余KNOX 蛋白等电点均低于7,为酸性蛋白。 所有编码的蛋白均为亲水性蛋白,亚细胞预测分析表明所有编码蛋白均定位于细胞核,符合转录因子细胞核定位的特征。
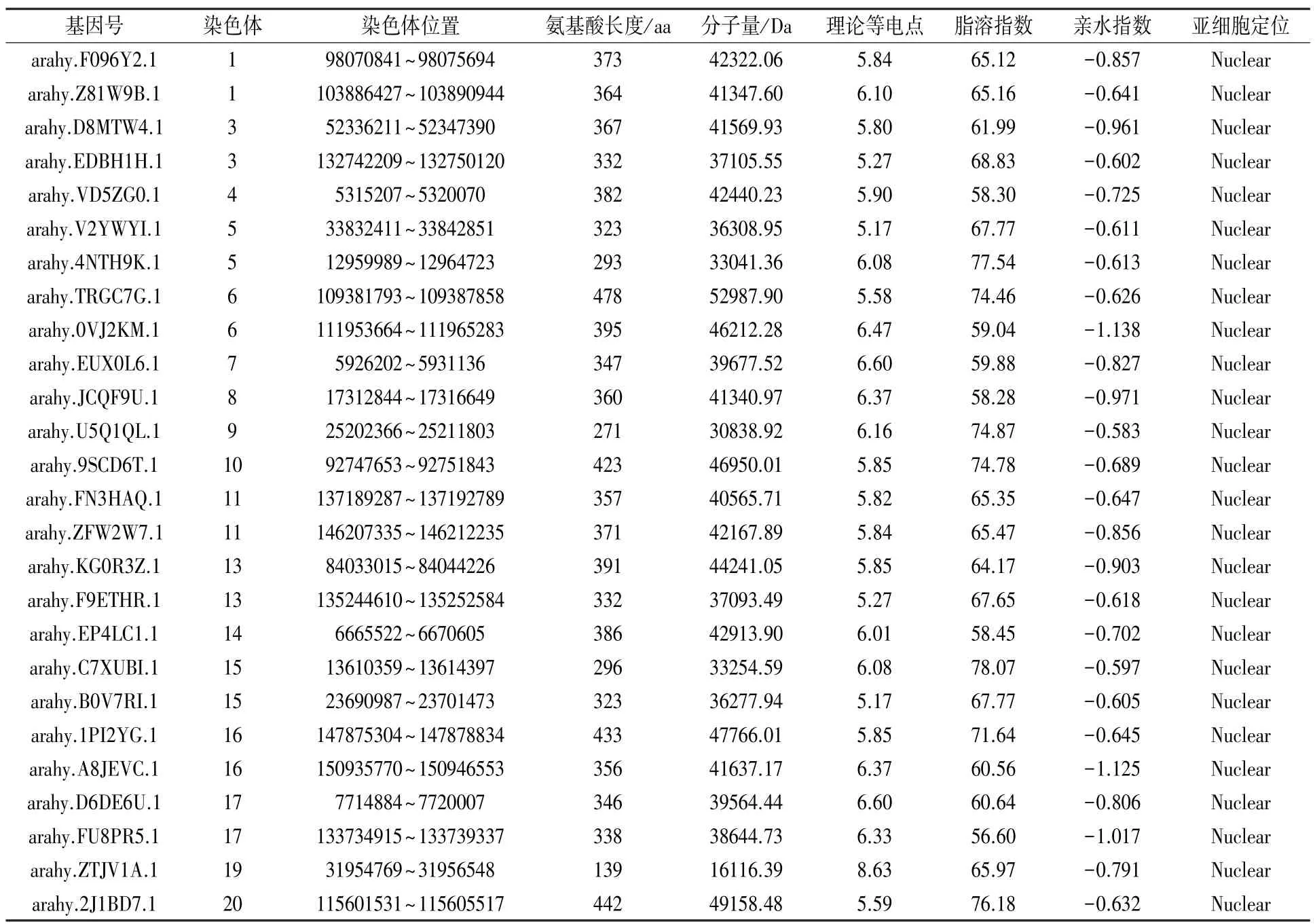
表2 花生KNOX 基因家族信息
2.2 AhKNOX 基因结构与染色体定位分析
对AhKNOX在染色体上的具体分布进行可视化分析,结果显示除2、12、18 号染色体外,Ah-KNOX在其余17 条染色体上均有分布(图1A)。AhKNOX基因结构分析发现,各成员基因含有的外显子数目范围为2 ~8个,其中,arahy.ZTJV1A.1的外显子最少,为2个;arahy.TRGC7G.1 含有的外显子最多,达到8个(图1B)。
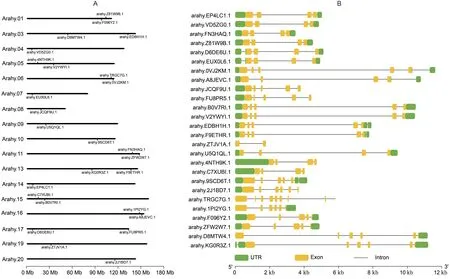
图1 花生KNOX 基因在染色体上的分布及基因结构
2.3 AhKNOX 系统进化分析
利用拟南芥、花生的KNOX 氨基酸序列构建进化树,KNOX基因家族蛋白可以分为Class Ⅰ和Class Ⅱ两个亚家族,每个亚家族又可分为A 和B 两个亚组(图2)。 其中12个花生KNOX 蛋白与拟南芥STM、KNAT1 归为Class ⅠA 组;4个花生KNOX 蛋白与拟南芥KNAT2、KNAT6 归为Class ⅠB 组;2个花生KNOX 蛋白与拟南芥KNAT7 归为Class ⅡA 组;8个花生KNOX 蛋白与拟南芥KNAT3、KNAT4、KNAT5 归为Class ⅡB组。 所有花生KNOX 蛋白进化树末端的分支均有2个成员,且一个来自染色体1 ~10(四倍体A基因组),另一个来自染色体11~20(四倍体B 基因组),两者为直系同源基因关系。
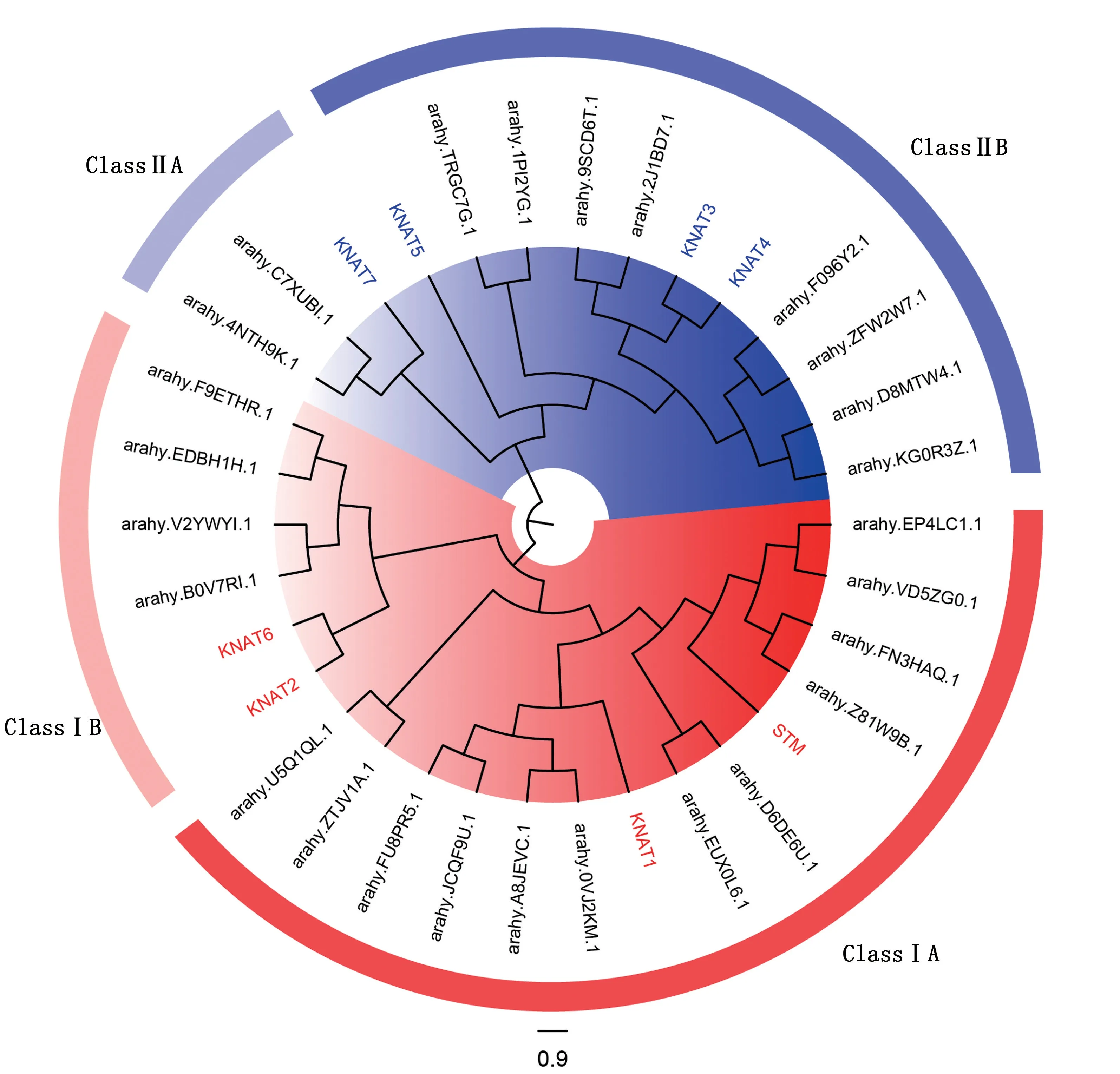
图2 花生与拟南芥KNOX 基因家族系统进化分析
2.4 AhKNOX 基因家族成员蛋白保守基序与结构域分析
利用MEME 在线工具对AhKNOX 进行保守基序分析,结果显示除arahy.ZTJV1A.1 外,其他KNOX 蛋白家族成员都含有Motif3;除arahy.JCQF9U.1、arahy.FU8PR5.1 外,其他KNOX 蛋白家族成员都含有Motif1;Motif5、Motif9 只在Class Ⅰ亚家族中存在,Motif4 只在Class Ⅱ亚家族中存在。 这可能说明不同分支的KNOX基因在花生中发挥不同功能。
利用CDD 工具检测保守结构域,结果显示,26 条花生KNOX蛋白有20 条同时具有4个典型的结构域,即KNOX1、KNOX2、ELK 和Homeobox_KN;有5 条具有3个结构域,其中arahy.D6DE6U.1、arahy.EUX0L6.1、arahy.4NTH9K.1、arahy.C7XUBI.1 缺少ELK 结构域,arahy.FU8PR5.1 缺少Homeobox_KN 结构域;仅arahy.ZTJV1A.1 只含有Homeobox_KN 和ELK 两个结构域(图3)。
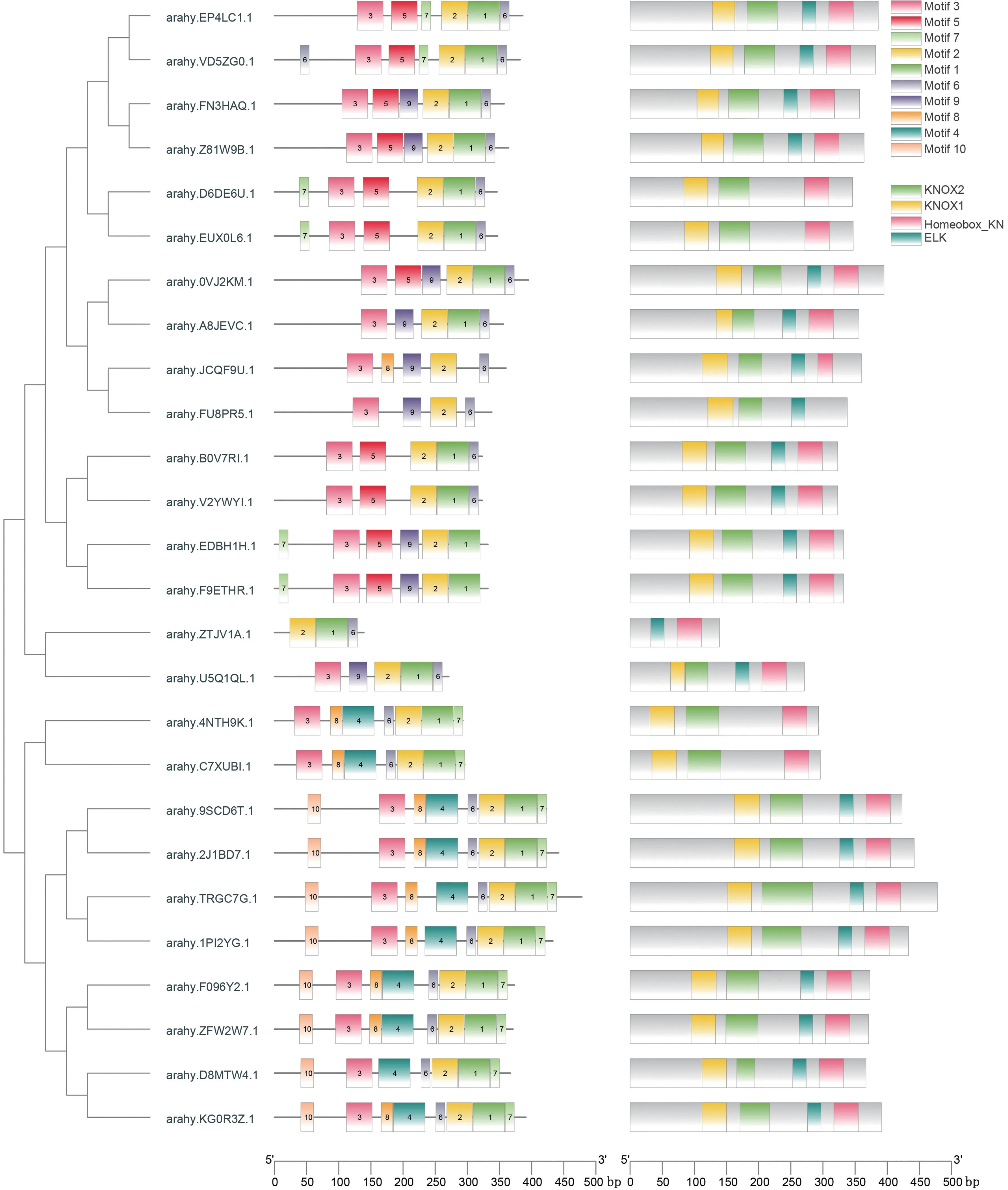
图3 花生KNOX 基因家族成员蛋白保守基序与结构域
2.5 AhKNOX 启动子顺式作用元件分析
为了更好地研究AhKNOX表达调控机制,提取AhKNOX家族成员上游2 000 bp 的基因组序列并预测可能的顺式作用元件。 结果显示各成员的启动子区域均含有大量的顺式作用元件,而且数量与种类存在明显差异(图4)。 26 条AhKNOX基因共预测到51 种元件,主要涉及植物生长发育及光、胁迫、激素响应四大类。 其中,光响应相关元件最多,包含Box 4、G-box、GT1-motif 等22种;其次为植物生长发育相关元件,包括种子特异性元件(RY-element)、胚乳表达元件(GCN4_motif)、O2-site(玉米醇溶蛋白代谢调节元件)、分生组织表达元件(CAT-box)等15 种元件,说明Ah-KNOX基因广泛参与植物生长发育调控。 激素响应相关元件包括脱落酸响应(ABRE)、茉莉酸甲酯响应(CGTCA-motif、TGACG-motif)、水杨酸响应(TCA-element)、生长素响应(TGA-element、AuxRR-core)、赤霉素响应(P-box、GARE-motif)等8 种元件。 逆境胁迫相关元件包括ARE(厌氧诱导)、TC-rich repeats(防御与胁迫压力)、MBS(干旱诱导)、LTR(低温响应)、GC-motif(缺氧诱导)和WUN-motif(创伤响应)6 种元件,推测部分AhKNOX基因参与逆境胁迫响应。
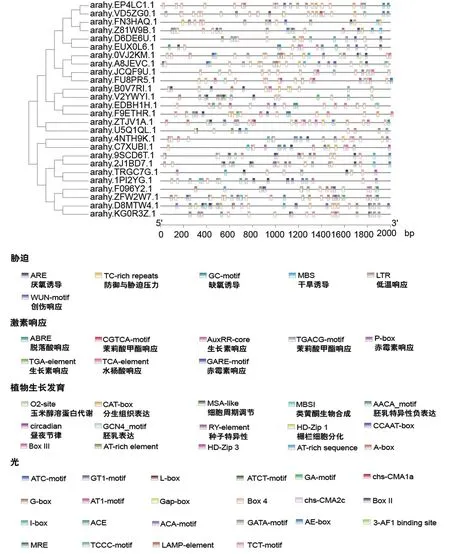
图4 花生KNOX 启动子顺式作用元件分析
2.6 AhKNOX 全生育期及非生物胁迫下表达模式分析
利用22个花生组织转录组测序数据,构建了25个AhKNOX基因的FPKM 值热图,如图5A 所示,而arahy.0VJ2KM.1 未检测到表达。 与玉米研究成果相似[17],AhKNOX基因的表达模式与其蛋白质进化分类呈现出较强一致性。 Class Ⅰ类基因主要在茎尖、果针等组织特异性高表达。 同一个基因在营养茎尖、生殖茎尖表达丰度趋于一致;入土前果针、入土后果针表达丰度趋于一致;但arahy.D6DE6U.1、arahy.EUX0L6.1、arahy.EP4LC1.1、arahy.VD5ZG0.1 在茎尖、果针表达丰度差异较大,推测这4个基因在花生果针与茎尖分生组织的形成以及功能维持上出现了功能分化。 arahy.B0V7RI.1、arahy.V2YWYI.1 在花器官中表达丰度较高,arahy.U5Q1QL.1、arahy.Z81W9B.1、arahy.EDBH1H.1、arahy.F9ETHR.1 在花生荚果发育初期表达丰度较高。 Class Ⅱ类基因在不同组织及发育时期均有表达。
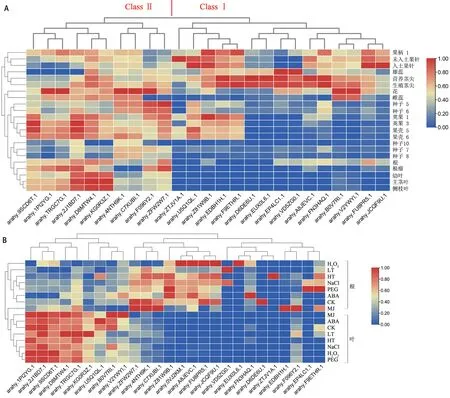
图5 花生KNOX 基因表达热图
为探究不同非生物胁迫处理下AhKNOX基因的表达模式,利用NaCl、PEG6000(模拟干旱)、低温、高温、过氧化氢、ABA、茉莉酸甲酯处理下花生幼苗期叶片和根组织转录组测序数据,采用TBtools 构建相对表达量的热图。 结果显示部分基因受非生物胁迫影响,表达量较对照变化较大(图5B)。 其中一些基因参与特定胁迫下的应答,如arahy.VD5ZG0.1 在盐和低温胁迫下的根中表达大幅上调,arahy.EP4LC1.1、arahy.F9ETHR.1 在干旱胁迫下的根中表达大幅上调,arahy.EUX0L6.1在H2O2处理下的根中表达大幅上调,arahy.ZTJV1A.1 在高温胁迫下的根中表达大幅下调,推测部分AhKNOX基因参与花生抗逆响应。
2.7 PEG6000 处理下AhKNOX 表达分析
基于转录组数据筛选得到两个能够特异性响应PEG 处理的AhKNOX基因(arahy.EP4LC1.1 和arahy.F9ETHR.1),为探究其具体响应规律,用18%的PEG6000 处理3 d 后复水2 d,通过RTqPCR 分析其表达模式。 结果(图6)显示,arahy.EP4LC1.1 和arahy.F9ETHR.1 均特异性响应PEG胁迫,但响应模式不同。 arahy.EP4LC1.1 随着处理时间的延长表达量呈逐渐降低趋势,复水2 d能够恢复到正常水平;arahy.F9ETHR.1 的表达在处理1 d 后下调,处理2 d 上调至2.5 倍,处理3 d表达量又下降但仍显著高于对照,复水2 d 后仍显著低于正常表达水平。
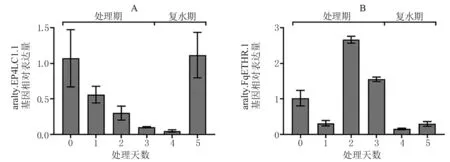
图6 arahy.EP4LC1.1(A)和arahy.F9ETHR.1(B)在PEG6000 处理下的表达分析
3 讨论与结论
KNOX基因家族作为一类转录因子,广泛参与植物生长发育调控。 随着高通量测序技术的发展,KNOX 转录因子家族已在多种植物中鉴定出来,但目前尚未见对花生KNOX基因的研究报道。本研究第一次在花生全基因组水平上对KNOX基因家族进行了鉴定,并对其基因结构、蛋白质保守结构域、系统进化、基因表达模式进行分析,可为进一步研究AhKNOX基因的功能奠定基础。
本研究共鉴定出26个花生KNOX基因家族成员,数量与同为豆科植物的大豆(27个)相近[30]、远多于模式植物拟南芥(8个)。 扩充了的AhKNOX基因家族,可以满足花生特有器官发育、复杂环境适应等其他功能的细化需求。 对花生KNOX 蛋白保守结构域分析发现,除6个Ah-KNOX 蛋白外,其余AhKNOX 蛋白均含有4个典型结构域;arahy.ZTJV1A.1 仅含有Homeobox_KN结构域和ELK 结构域,arahy.FU8PR5.1 缺少Homeobox_KN 结构域,但两者在果针入土前后有较高的表达。 果针为花生属特异性组织器官,推测这两个基因在果针发育中发挥一定的功能,具体机制有待进一步研究。
AhKNOX在花生22个组织中的表达丰度分析显示,Class Ⅰ类基因主要在茎尖、果针等组织中表达,其中4个与拟南芥STM基因直系同源的基因在茎尖和果针处表达丰度差异较大。 拟南芥中STM在茎尖分生组织的形成以及功能维持中发挥作用,进一步研究这4个KNOX基因在花生茎、果针发育过程中的功能分化,将有助于理解花生果针发育的特殊性。
植物KNOX基因不仅调控生长发育,还可以响应不同非生物胁迫[31-32]。 本研究对AhKNOX家族成员的启动子顺式作用元件进行分析,发现AhKNOX基因除了有保守的光响应元件外,还包含多种不同的逆境胁迫相关元件,推测该家族基因在花生响应植物逆境胁迫中存在功能分化。 利用7 种非生物胁迫处理的花生转录组数据对Ah-KNOX基因家族表达模式进行进一步分析,发现有5个基因参与某特定胁迫下的应答,如arahy.EP4LC1.1 和arahy.F9ETHR.1 特异性响应干旱胁迫。 RT-qPCR 验证结果显示这两个基因在PEG处理及复水后表达均发生显著变化。 这些基因在花生应对干旱胁迫中的具体作用有待进一步研究。
