基于微卫星标记的塔里木盆地5 个地区南疆沙蜥种群遗传多样性及遗传结构分析
2023-09-13段嘉宝农静娴李孟纯杨天乐
段嘉宝,农静娴,李孟纯,杨天乐,李 铀,2,3*
( 1. 西北民族大学生命科学与工程学院,甘肃 兰州 730100 ;2. 西北民族大学生物医学研究中心 甘肃省动物细胞技术创新中心,甘肃 兰州 730030 ;3. 西北民族大学生物医学研究中心 生物工程与技术国家民委重点实验室,甘肃 兰州 730030 )
塔里木盆地深居亚欧大陆内部,位于青藏高原北部、帕米尔高原东部和天山南部,是中国面积最大的内陆盆地。塔里木盆地东西长1 500 km,南北宽约600 km,面积约530 000 km2,四周海拔高度在800~1 300 m 之间。盆地降水量少且蒸发量高,属于典型的大陆性干旱气候[1-2]。盆地因其独特的地质地貌及环境属性,孕育了丰富独特的生物多样性资源。南疆沙蜥(Phrynocephalus forsythii)隶属于鬣蜥科(Agamidae)中的沙蜥属(Phrynocephalus),是塔里木盆地特有爬行物种之一,是栖息于稀疏灌丛或戈壁滩的卵胎生蜥蜴。与同域分布的叶城沙蜥相比,南疆沙蜥更喜欢较为湿润的栖息地[3]。
微卫星标记(simple sequence repeat,SSR)是基于DNA长度多态性的分子标记技术,可直接反映核酸水平的遗传变异,广泛应用于野生动物遗传多样性研究[4-6]。张沼等[7]利用8对微卫星分子标记对赛罕乌拉国家级自然保护区的马鹿粪便样品进行遗传多样性分析,表明该地区马鹿种群的遗传多样性处于中上水平。张于光等[8]基于SSR对三江源和祁连山国家公园雪豹进行种群遗传多样性研究发现,野外生存雪豹种群遗传多样性相对较低,但祁连山国家公园雪豹种群遗传多样性相对较高,该研究成果对野生动物科学化保护提供了参考。目前学者多关注南疆沙蜥的系统进化关系及进化历史的研究,群体遗传结构的研究也在不断完善[3,9]。本研究利用选取的4个微卫星位点,对塔里木盆地5个区域的南疆沙蜥的遗传多样性和遗传结构进行探究,以期为塔里木地区南疆沙蜥的遗传资源保护以及荒漠化地区其他物种的保护提供参考。
1 材料与方法
1.1 试验材料
试验研究对象为塔里木盆地5个地区(民丰、策勒、于田、和田、皮山)采集共计74个南疆沙蜥样本,取肌肉与肝脏组织保存于无水乙醇与生理盐水(体积比1∶1)组织保存液中,-20 ℃保存,用于后续分子试验。塔里木盆地5个区域的南疆沙蜥样本采样点见表1。

表1 塔里木盆地5个区域的南疆沙蜥样本采样点Tab.1 Sampling point of P. forsythii samples in five regions of Tarim Basin
1.2 基因组DNA的提取
使用SteadyPure 通用型基因组DNA 提取试剂盒提取样本的基因组DNA,并用1.4%琼脂糖凝胶电泳及分光光度计检测DNA完整性及纯度,保存-20 ℃。
1.3 引物设计及PCR扩增
参照已发表的沙蜥文献中选取其中4个微卫星位点进行引物设计[10-11],引物信息见表2。

表2 南疆沙蜥多态性微卫星位点信息Tab.2 Information of polymorphic microsatellite loci for P. forsythii
PCR 反应体系(12 μL):5×buffer (Mg2+free)2.4 μL、TakapaHs 酶0.06 μL、tag F 0.09 μL、tag R 0.09 μL、Primers特异性引物2.4 μL、无菌水4.96 μL、DNA样本含量2 μL。
PCR 扩增反应条件:95 ℃预变性10 min;92 ℃变性60 s,50 ℃退火90 s,72 ℃延伸60 s,5 个循环;92 ℃变性30 s,63 ℃退火90 s,72 ℃延伸60 s,20 个循环;92 ℃变性15 s,54 ℃退火60 s,72 ℃延伸60 s,40 个循环;72 ℃延伸10 min,25 ℃ 10 min。
扩增产物进行琼脂糖凝胶电泳,并在凝胶成像仪上观察结果。成功扩增的合格PCR 产物送至苏州金唯智生物科技有限公司进行测序分析,得到原始数据FASTA文件。
1.4 数据统计与分析
使用Genemarker[12]对测序仪得到的原始数据进行基因分型,分型后的数据使用MicroChecker 2.2.3[13]检测是否存在无效等位基因。使用GenAlex V 6.5[14]分析等位基因数(number of allele,Na)、有效等位基因数(effective number of allele,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、近交系数(Fis)、Shannon 指数(I)、群体间Nei's 遗传距离和遗传相似度、群体间的遗传分化指数(Fst)、基因流(Nm)和分子方差分析(AMOVA)。使用Cervus3.0[15]计算各位点的多态信息含量(PIC)。使用MEGA X[16]绘制基于群体间的Nei's 遗传距离的UPGMA(unweighted pair-group method with arithmetic mean,UPGMA)聚类树。主成分判别分析(discriminant analysis of principal components,DAPC)使用R中的“adegenet”包[17]。
2 结果与分析
2.1 遗传多样性(见表3、表4)

表3 4个微卫星位点的遗传多样性Tab.3 Genetic parameters of four microsatellite loci

表4 5个南疆沙蜥种群在4个微卫星位点的遗传多样性参数Tab.4 Genetic diversity parameters at four microsatellite loci in five populations of P. forsythii
MicroChecker 检验微卫星基因分型数据,结果显示,4个位点在5个群体均未检出大量等位基因缺失或无效等位基因,因此选取的4个微卫星位点的基因分型数据可用于后续分析。4个多态性微卫星位点在5个群体内共检测到98个等位基因。
由表3可知,各位点的平均Ho介于0.025~0.737之间,平均值为0.464。He 最低为0.250(Phr30),最高为0.886(Pvms20)。PIC介于0.375~0.950之间,平均值为0.788,其中PIC≥0.50 的高度多态性位点有3 个,分别为Phr79、Pvms20、Pvms38;0.25≤PIC≤0.50 的中度多态性位点有1 个,为Phr30。Fis和群体间的Fst分别介于0.327~0.900 和0.068~0.426。Nm 最低为0.337(Phr30),最高为3.418(Pvms20)。
由表4 可知,在5 个南疆沙蜥群体中,Na 和Ne 最多的为PS 群体,分别为11.500 和7.882,较少的为YT 和HT 群体,说明南疆沙蜥的5个群体的等位基因数存在一定差异。Na、Ho、He、PIC 等参数反映群体遗传多样性水平。根据5个群体的基础遗传参数表明,MF群体表现出比其他群体较高的杂合性且具有丰富的遗传多样性。
2.2 群体遗传分化水平(见表5、表6)

表5 南疆沙蜥种群遗传变异的分子方差分析Tab.5 Analysis of molecular variance of population genetic variation of P. forsythii

表6 各群体间的遗传分化指数Tab.6 Coefficient of population differentiation among populations
由表5 可知,5 个南疆沙蜥群体的遗传变异主要来自群体内(92%),群体间的遗传变异仅有8%,表明南疆沙蜥群体之间遗传分化程度较小。
由表6可知,各群体间的Fst介于0.043(HT-PS)~0.191(MF-YT)之间,表明群体间存在中等程度的遗传分化(Fst<0.15)。此外,HT 群体与PS 群体的群体分化程度最小,为0.043,表明两群体间较其他群体最有可能存在基因交流现象。
2.3 群体遗传结构(见图1、图2)
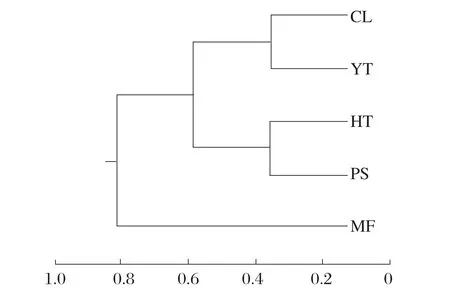
图1 基于Nei's遗传距离构建的5个南疆沙蜥群体UPGMA聚类树Fig.1 UPGMA clustering tree of five populations of P. forsythii based on Nei's genetic distance

图2 南疆沙蜥的DAPC聚类图Fig.2 DAPC cluster diagram of P. forsythii
由图1 可知,5 个南疆沙蜥群体分为3 个聚类。其中CL和YT群体聚为一类,HT和PS群体聚为一类,MF群体单独聚为一类。
由图2 可知,MF 群体明显区别于其他群体,CL 与YT群体混杂在一起,HT 与PS 群体混杂一起。DAPC 聚类结果与UPGMA聚类树结果基本一致。
3 讨论
3.1 南疆沙蜥的群体遗传多样性
遗传多样性对物种的生存和适应性具有重要影响,是物种进化演变的遗传基础。种群的遗传多样性水平越高,表明该群体适应环境变化的能力越强;反之,遗传多样性低下或下降很可能会导致物种的环境适应力和抵挡疾病的能力降低、雌雄性别比例失衡,甚至物种灭绝[18-19]。南疆沙蜥作为荒漠化地区的优势物种,研究其遗传多样性对评估群体丰富度及其遗传资源科学化保护尤为重要。本研究中,5 个南疆沙蜥群体在4 个微卫星位点上均检测到Na 高于Ne,说明等位基因存在差异,分布不均匀,可能受到群体间潜在的近交现象影响。杂合性作为评估遗传多样性的重要遗传参数,能够较好地反映群体遗传变异程度。本研究中,5个南疆沙蜥群体的Ho均低于He,表明遗传多样性水平较低,可能导致南疆沙蜥群体规模下降等潜在风险,但由于采集的样本量不够,有待进一步研究。
3.2 南疆沙蜥的遗传分化
Fst的取值范围0~1,最大值为1 表明两个群体完全分化,最小值为0 表明群体间无分化。0<Fst<0.05 时,说明群体间遗传分化很小,可忽略不计;0.05<Fst<0.15 时,表明群体间存在中等程度的遗传分化;0.15<Fst<0.15 及Fst>0.25时,表明群体间普遍存在较大及以上的遗传分化[20-21]。本研究中,除CL-YT、CL-PS 及HT-PS 外,其余两群体间均存在中等及以上的遗传分化程度。总体来说,5 个南疆沙蜥的群体分化仍处于中等的遗传分化水平,其群体间的差异较大。
3.3 南疆沙蜥群体遗传结构
除了基于Nei's遗传距离构建的UPGMA聚类树外,基于DAPC 也是分析聚类关系的常用方法之一[22]。本研究中,UPGMA 聚类树与DAPC 聚类图中的聚类关系基本一致,均显示MF群体明显单独聚为一类。MF群体与其他群体无混杂,可能与存在人为干扰等影响导致的基因交流屏障有关。在全球气候变暖的背景下,对于变温动物尤其是爬行动物的影响尤为显著。因此,探究环境温度对南疆沙蜥遗传结构及适应性遗传变异的影响,对栖息在塔里木盆地及少部分高原地区的南疆沙蜥而言,是否存在更大生存风险挑战有待后续进一步研究加以验证。
4 结论
本研究利用4个微卫星位点探究塔里木盆地5个区域的南疆沙蜥群体的遗传多样性及种群结构,发现5个南疆沙蜥群体在4 个微卫星位点的Ho、He 为0.464、0.692。多态信息含量均值为0.788,Fis及Fst均值分别为0.433、0.180。基于DAPC聚类分析结果显示,MF群体单独聚类为一支,与UPGMA 聚类树结果一致。结果表明,5 个南疆沙蜥群体的遗传多样性水平低下,群体间存在较大的遗传分化。
南疆沙蜥作为塔里木盆地内的爬行优势物种,群体遗传多样性仍处于较低水平,群体分化水平受到人为因素和地质事件等多重影响,因而亟须加强对南疆沙蜥群体的保护。本试验通过对塔里木盆地部分区域南疆沙蜥遗传多样性及种群结构进行研究,为南疆沙蜥的科学化保护提供参考意见,也为塔克拉玛干沙漠等荒漠化地区物种保护提供一定参考。
