贵州栽培型地方茶树叶片气孔性状全基因组关联分析
2023-08-22白鼎臣赵支飞牛素贞陈正武
白鼎臣,赵支飞,龚 雪,刘 源,牛素贞,4,*,陈正武
(1.贵州大学 茶学院,贵州 贵阳 550025; 2.贵阳人文科技学院 文化旅游与区域发展学院,贵州 贵阳 550025; 3.贵阳市农业技术推广站,贵州 贵阳 550081; 4.贵州大学 农业生物工程研究院,贵州 贵阳 550025; 5.贵州省农业科学院 茶叶研究所,贵州 贵阳 550025)
茶(CamelliasinensisL. O. Ktze.)隶属山茶科(Theaceae)、山茶属(CamelliaL.)、茶种(Camelliasinensis),是世界三大饮料之一,具有重要的文化价值和经济价值[1]。茶叶提取物富含多种次生代谢物质,如茶多酚、氨基酸、咖啡碱、多糖类等,具有抗氧化、提神醒脑、利尿、降血糖、提高免疫力等作用[2-6]。茶树起源于中国西南,在60多个国家广为栽培,在农业经济中占有举足轻重的地位[7]。种质资源是决定育种计划成败的作物遗传改良的宝贵物质基础[8]。茶树育种效率低下、优良种质资源缺乏等问题,已经成为未来全球茶产业面临的重大挑战[9]。
分子标记技术作为茶树遗传研究的有力工具,茶叶科技工作者通过RAPD(random amplified polymorphic DNA)[10],SSR(simple sequence repeats)[11],EST-SSR(expressed sequence tags-simple sequence repeats)[12-13]和AFLPs(amplified fragment length polymorphism)等[14]分子标记技术进行了大量的研究,基于不同分子标记技术构建了茶树遗传连锁图谱[15-17],它为广泛的遗传学和基因组研究提供了可靠的依据,例如数量性状图谱、分子标记辅助育种以及比较基因组研究。随着二代测序技术的逐渐成熟,快速、经济、高效的简化基因组测序技术(GBS)在许多植物遗传学和基因组研究中广泛应用,如小麦[18]、玉米[19]、辣椒[20]、松树[21]。近年来利用GBS技术对茶树开展研究也逐步增多,基于GBS测序技术,Nisha等[22]构建了茶树单倍型图谱。王松琳[23]开展了QTL定位研究,研究茶树黄化品种白鸡冠的色素和游离氨基酸的特异性状。贵州茶树种质资源科研工作者还先后在贵州开展了大量利用GBS技术的研究工作,郭燕等[24]分析了古贵州茶树的遗传关系,乔大河等[25]构建了贵州省主要栽培茶树品种遗传图谱并分析了这些茶树品种的遗传结构,郭灿等[26]揭示了贵州地方茶组植物的亲缘关系,牛素贞等[27-28]分析了贵州415份茶树材料的遗传多样、连锁不平衡、群体结构并构建了核心种质。这些研究促进了人们对贵州茶树种质资源的概况及其分布特征的认识。
贵州保存了大量栽培型地方茶树种质资源。但因经济发展和土地利用相对缓慢,地理条件独特,气候特点多样,且雨量充沛,加之茶树异花授粉等原因,导致其种质资源的生态适应性好,遗传多样性广,在产量、抗性、品质、特异等方面具有较高的研究和利用潜力。本项研究利用GBS技术,以贵州栽培型地方茶树种质资源为材料,对其叶片气孔性状进行全基因组关联分析。以期能从这些具有复杂遗传背景和丰富生态类型的乡土栽培型茶树资源中,筛选出适应能力强的优良茶树品种,促进贵州茶产业品种结构调整。
1 材料与方法
1.1 材料
以253份贵州栽培型地方茶树种质资源作为研究材料,参考Niu等[28]的研究,所有材料均经过扦插育苗后,种植于贵州大学南校区苗圃地,统一水肥管理。
1.2 方法
1.2.1 DNA提取与GBS测序
采集253份栽培型地方茶树种质资源嫩芽并经过液氮处理,放入-80 ℃超低温冰箱中冷藏备用。DNA提取和GBS测序参考Nachman等[29]的研究。
1.2.2 序列比对与SNP的挖掘
在BWA v. 0.7.10软件上与舒茶早参考基因组进行比对(http://tpia.teaplant.org/)[1],利用GATK v. 3.7.0软件完成SNPs统计。SNP过滤参考前人的研究方法[30-33]。完成过滤后,253份贵州栽培型地方茶树种质资源材料共得到112 072个SNP位点,并用于后续分析。
1.2.3 气孔性状分析
参考杨克彤等[34]的取样方法,选择晴朗无大风天气,于2022年2月至3月上旬09:00—11:00,在贵州大学南校区茶园进行茶树叶片采集,并对茶树叶片进行编号。简单清理叶背后,避开叶面主脉,用无色透明的亮甲油均匀涂抹,待其自然干后取回实验室,用刀片在涂抹的甲油上划出方形,揭去涂抹层,做成临时玻片,在Leica Mmil-MC170显微下观察(目镜10倍,物镜10倍、40倍)。基于其配套的LAS v. 4.12软件,在显微镜放大100倍视野下取3个镜头,在放大400倍视野下取3个镜头。于放大100倍视野下记录单位面积气孔数量,用于计算气孔密度(SD),在放大400倍视野下用LAS v. 4.12软件分析模块测量气孔长度(SL)、气孔宽度(SW)及单个气孔的面积(SA),气孔周长(SP)基于公式[35]:
(1)
式(1)中,SP为气孔周长的值,单位为 μm;SL为气孔长度的值,单位为 μm;SW为气孔宽度,单位为 μm。
1.2.4 气孔性状全基因组关联分析
一般线性模型(general linear model,GLM)是在TASSELV.5.2.72(https://tassel.bitbucket.io)全基因组关联分析的基础上形成的[36]。将主成分分析(PCA)中的Components参数设置为3,P矩阵是通过计算分析得到的。作为一般线性模型的协变量,使用P矩阵和群体结构分析Q值矩阵。设置亲缘关系(Kinship)分析选择Centered IBS,Max Alleles设置为3,从而得到K矩阵。混合线性模型(mixed linear model,MLM)和压缩混合线性模型(compressed mixed linear model,cMLM)是将P矩阵和K矩阵联合起来作为协变量,或将Q矩阵和K矩阵作为协变量。因此,用于5个茶树叶片气孔性状全基因组关联分析的共有以下6种:1)GBS测序数据+性状数据+P矩阵;2)GLMQ模型,GBS测序数据+性状数据+Q矩阵;3)MLMP模型,基于MLM模型,GBS测序数据+性状数据+P矩阵+K矩阵;4)MLMQ模型,基于MLM模型,GBS测序数据+性状数据+Q矩阵+K矩阵;5)cMLMP模型,基于cMLM模型,GBS测序数据+性状数据+P矩阵+K矩阵;6)cMLMQ模型,基于cMLM模型,GBS测序数据+性状数据+Q矩阵+K矩阵。根据关联分析结果,确定与栽培型茶树叶片SL、SW、SD、SA、SP性状紧密相关的SNP位点,以P=1×10-4为阈值。
2 结果与分析
2.1 SNP挖掘及群体结构
经过比对过滤后共挖掘到112 072个高质量SNP位点用于全基因组关联分析,关联分析Q值参考Zhao等[37]的群体结构分析系结果。
2.2 气孔形态性状
对栽培型茶树种质资源进行气孔性状分析,结果显示,SL变幅为25.49~44.80 μm,变异系数为0.73%;SW变幅为15.69~39.60 μm,变异系数为1.13%;SD变幅为202.78~633.33 mm-2,变异系数为1.47%;SA变幅为292.96~1 169.37 μm2,变异系数为1.64%;SP变幅为67.45~124.85 μm,变异系数为0.82%(表1、表2)。对上述叶片气孔性状进行相关性分析发现,SL、SW、SA和SP两两之间呈显著正相关,而以上4个性状与SD均呈显著负相关(图1-A)。当偏度Z-score与峰度Z-score检验值在±1.96区间内时,该组数据服从正态分布[38]。

表1 贵州栽培型地方茶树种质资源茶树5个气孔性状统计表

表2 贵州栽培型地方茶树种质资源茶树5个气孔性状调查描述统计
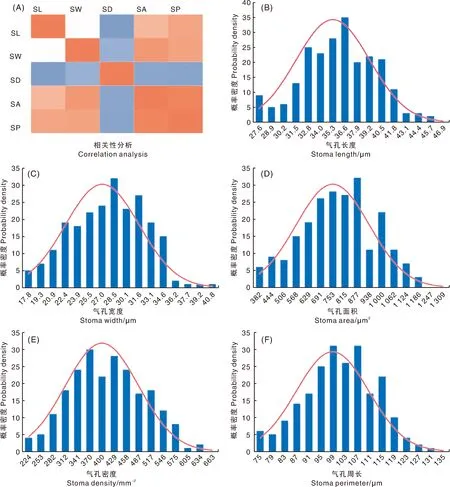
SL,气孔长度; SW,气孔宽度; SD,气孔密度; SA,气孔面积; SP,气孔周长。
经检验,SL、SW、SD、SA和SP的偏度Z-score检验值分别为-1.17、-0.74、0.90、0.60和-1.51,峰度Z-score检验值分别为-0.47、-1.58、-1.49、-1.47和-0.75。进一步可视化5个气孔性状数据的正态分布图,结果显示,SL、SW、SD、SA和SP均服从正态分布,且与Z-score验证结果一致(图1)。综上所述,所调查的贵州栽培型地方茶树种质资源叶片的SL、SW、SD、SA和SP 数据均能用于进一步全基因组关联分析。
2.3 关联分析
2.3.1 气孔长度关联分析
基于6种模型,利用SNP数据和SL统计数据对栽培型茶树叶片SL制作关联分析Q-Q图。结果见图2,选择GLMQ模型进行茶树叶片SL全基因组关联分析优选模型,曼哈顿图以-lg10-4作为阈值(下同),曼哈顿图中显示共关联到了15个SNP位点。
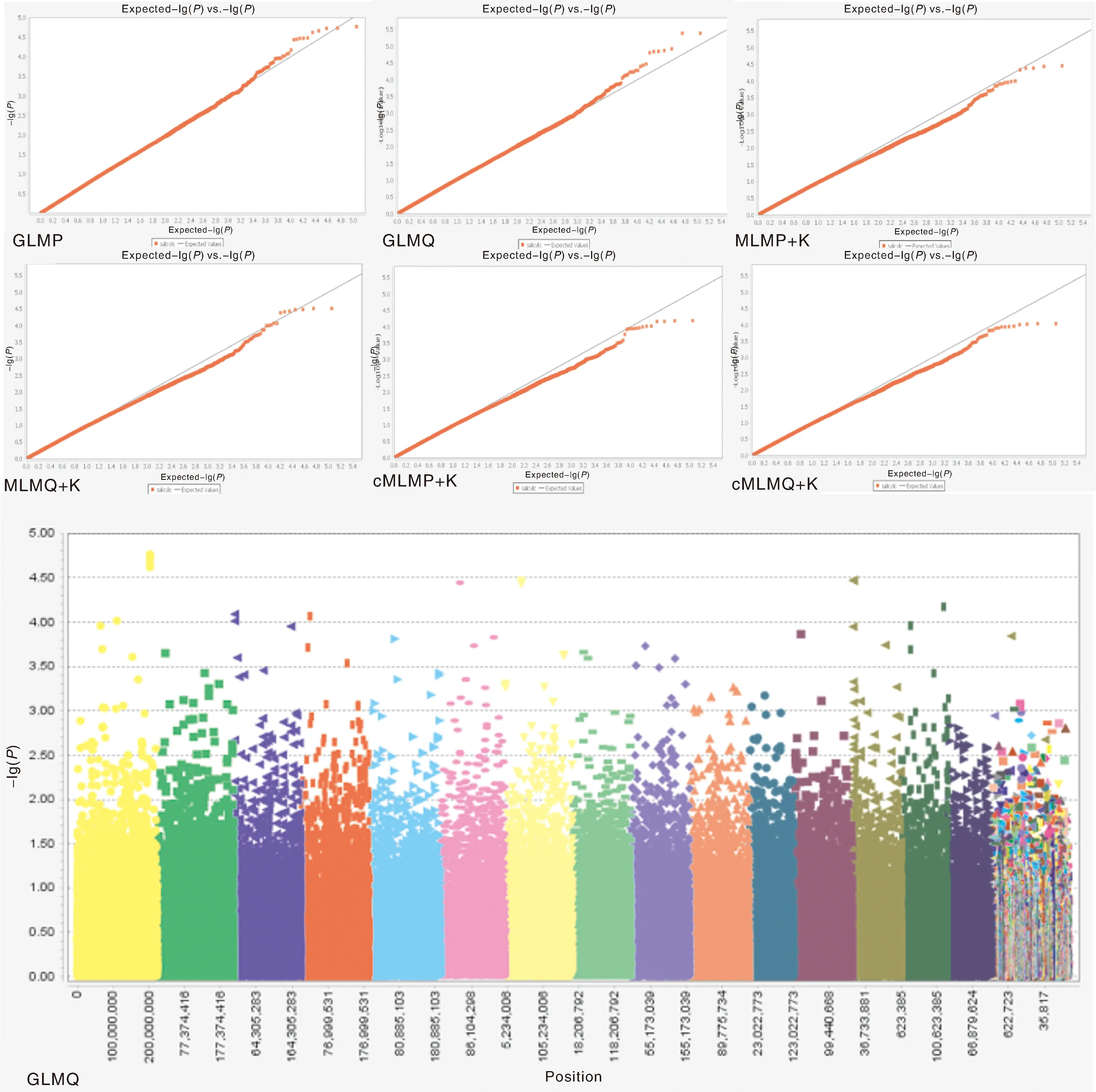
图2 基于6种模型叶片SL全基因组关联分析Q-Q图和曼哈顿图
2.3.2 气孔宽度关联分析
基于6种模型制作叶片SW全基因组关联分析Q-Q图结果显示(图3),选择GLMP模型作为进行叶片SW全基因组关联分析优选模型,曼哈顿图中显示总计关联到了4个SNP位点。
图3 基于6种模型叶片SW全基因组关联分析Q-Q图和曼哈顿图
2.3.3 气孔面积关联分析
基于6种模型制作叶片SA全基因组关联分析Q-Q图结果显示(图4),选择GLMQ模型关联进行茶树叶片SA全基因组关联分析优选模型,曼哈顿图中显示共关联到了16个与SA相关的SNP位点。

图4 基于6种模型叶片SA全基因组关联分析Q-Q图和最优模型曼哈顿图
2.3.4 气孔密度关联分析
基于6种模型制作叶片SD全基因组关联分析Q-Q图,结果(图5)显示,选择GLMQ模型进行茶树叶片SD全基因组关联分析优选模型,曼哈顿图中显示共关联到的SNP位点有8个。
图5 基于6种模型叶片SD全基因组关联分析Q-Q图和曼哈顿图
2.3.5 气孔周长关联分析
基于6种模型制作SP全基因组关联分析Q-Q图,结果显示(图6),选择GLMQ模型作为茶树进行SP全基因组关联分析优选模型,曼哈顿图中显示总计关联到了16个SNP位点。
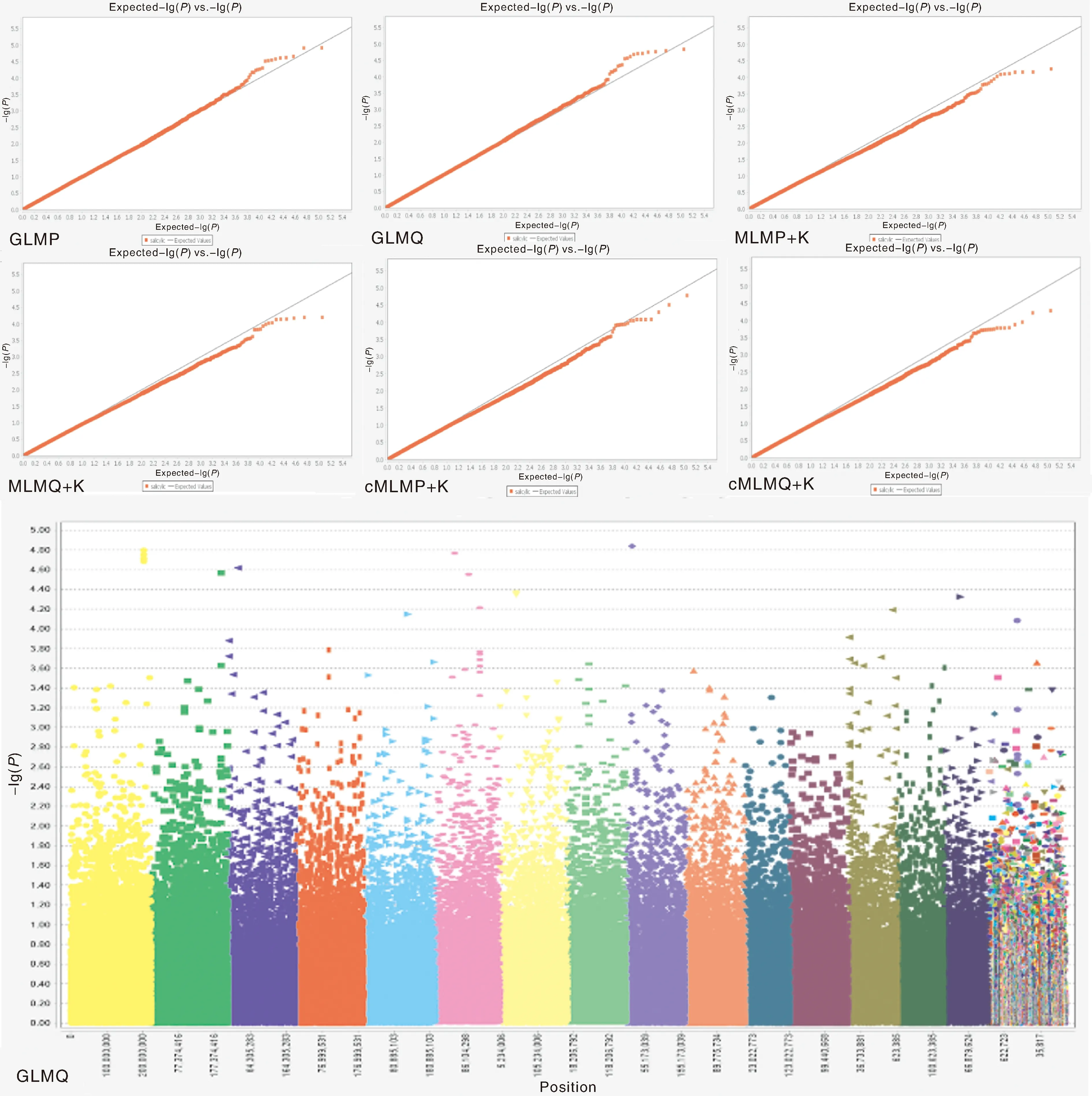
图6 基于6种模型叶片SP全基因组关联分析Q-Q图和曼哈顿
根据以上5个气孔性状在6种模型关联分析输出的Q-Q图和曼哈顿图,选取GLMP模型作为SW全基因组关联分析优选模型,选取GLMQ模型SL、SA、SD和SP全基因组关联分析的优选模型。结果发现,5个气孔表型性状共检测到45个SNP,SL关联15个SNP位点、SW关联4个SNP位点、SA关联16个SNP位点、SD关联8个SNP位点、SP关联16个SNP位点。其中SL和SW共同关联的位点有1个,SL和SA共同关联的位点有3个,SL和SP共同关联的位点有5个,SW和SA共同关联的位点有2个,SW和SP共同关联的位点有2个,SA和SP共同关联的位点有6个。
2.4 候选基因预测
在检测到的45个SNP上下游各50 kb范围内检索基因,并借助数据库(TPIA和NCBI)对检索到基因进行功能注释。结果发现,选取GLMQ模型作为SL关联分析优选模型。在15个与SL显著相关的SNP位点上下游各50 kb范围内,共检测到6个基因(表3),ID号分别为CSS0006464、CSS0014538、CSS0049436、CSS0014397、CSS0006487和CSS0038141。

表3 SL候选基因及功能注释
选取GLMP模型作为SW关联分析优选模型。在4个与SW显著相关的SNP位点上下游各50 kb范围内共检测到1个基因,编号为CSS0005692.1,其基因位点S6_151022344,其关联位点为S6_151013307,可能与TIR基因家族同源,介导植物抗性,也可能与富含亮氨酸重复序列(LRR)同源,LRR通常参与蛋白质的相互作用。
取GLMQ模型作为SA关联分析优选模型。在9个与SA显著相关的SNP位点上下游各50 kb范围内,共检测到7个基因(表4),ID号分别为CSS0006464.1、CSS0032323.1、CSS0018568.1、CSS0008651.1、CSS0003026.1、CSS0049436.1和CSS0004296.1。
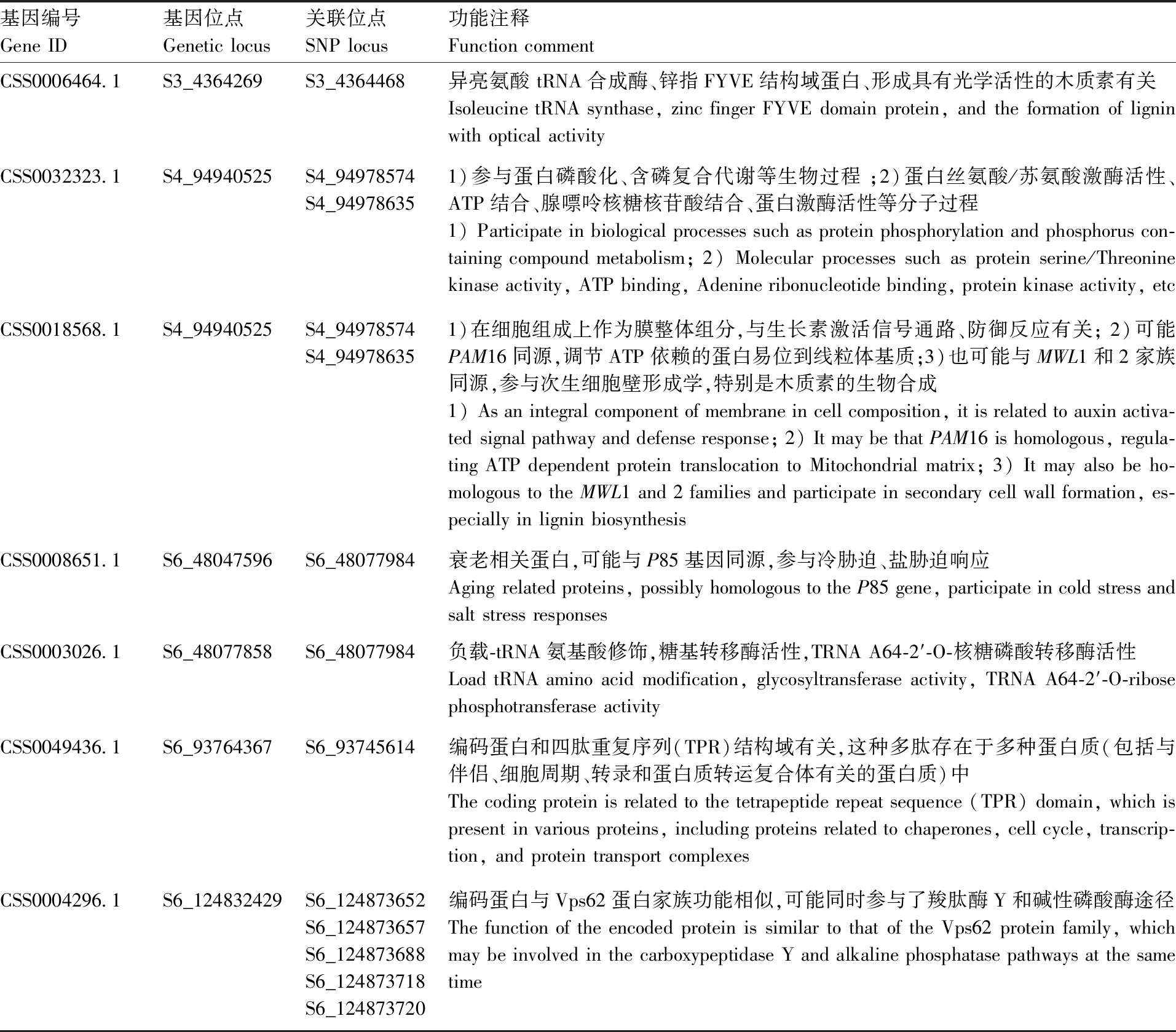
表4 SA候选基因及功能注释
选取GLMQ模型作为SD关联分析优选模型。在3个与SD显著相关的SNP位点上下游各50 kb范围内共检测到2个基因,分别为CSS0028538.1和CSS0010060.1。2个基因位点分别为S3_131677226与S12_52331200,关联位点分别为S3_131660065与S12_52353615,其功能分别与DNA修复、对X射线的反应和与1-酰基-SN-甘油-3-磷酸酰基转移酶4相关。
选取GLMQ模型作为SP关联分析优选模型。在14个与SP显著相关的SNP位点上下游各50 kb范围内共检测到了4个基因(表5),分别为CSS0004296.1、CSS0049436.1、CSS0014397.1和CSS0030051.1。

表5 SP候选基因及功能注释
3 讨论
气孔是植物与外界进行气体和水分交换的通道,气孔在碳同化、呼吸、蒸腾作用等气体代谢中起到关键作用,在植物生长过程中对水分、温度的胁迫及物质生产有着重要影响[39-40]。茶树气孔器是由两个保卫细胞围绕形成的孔隙,植物气孔的形成与成熟需要经过复杂的发育过程,受转录因子、蛋白激酶和各种功能基因编码蛋白等协同控制,同时也受其他环境因素的影响[41]。郭昱[42]对谷子核心种质全基因组进行关联分析,预测了一个候选基因Si4G14140,该基因与下表皮气孔密度有关。陈宏伟[43]以抽穗期相近的水稻种质资源451份为试材,利用4.8 M高品质SNP对水稻剑叶正、背面气孔密度、长度和宽度进行全基因组关联分析(GWAS),预测到影响水稻剑叶气孔密度、宽度QTL的7个候选基因。孙鹏娅[44]对500余份番茄进行了气孔性状全基因组关联分析,鉴定出调控番茄叶片气孔数量的关键基因SIGMDH和SIALMT15,并对其功能进行了验证。
本研究对栽培型茶树气孔测量分析表明,SL、SW、SA、SP和SD均服从正态分布。SL变幅为25.49~44.80 μm,变异系数为0.73%;SW变幅为15.69~39.60 μm,变异系数为1.13%。与之前调查结果的SL变幅(25.56~39.13 μm)、SW变幅(27.78~38.89 μm)相近[45]。相关性分析显示,SL、SW、SA、SP与SD均为负相关,与以往关于水稻[46]、玉米[47]的研究结果相符。全基因组关联分析发现,共有59个SNP位点与5个气孔表型性状相关,并且关联在5个气孔性状的优选模型基础上。这59个SNP位点的上、下游50 kb范围内共检测出20个基因,与SL相关的有6个基因,与SW相关的有1个基因,与SA相关的有7个基因,与SD相关的有2个基因,与SP相关的有4个基因。
光在气孔发育过程中起着重要作用,光敏色素和隐花色素对环境的感知影响气孔的发育过程[48]。与SL性状相关的6个基因中,CSS0006464.1、CSS0014538.1和CSS0005692.1与SL关系较为直接。CSS0006464.1能调控光学活性的木质素生成,该基因可能影响气孔的发育。生长素在植物生长发育过程中可与F-BOX蛋白TIR1结合降解Aux/IAA(AUXIN/indole-3-acetic acid)转录抑制因子,最终影响气孔发育[48]。CSS0014538.1可能与F-BOX蛋白有关,以此调节气孔发育。在植物体能量代谢时,存在于线粒体电子传递链的一部分细胞色素C1,可能与CSS0014538.1基因同源的CYT1基因有关[49]。B-Box锌指蛋白能在转录、翻译水平上调控基因表达[50]。但CSS0014397.1基因与COL16基因同源,编码蛋白与B-BOX锌指蛋白相关,从而调节气孔发育。与SW性状有关的CSS0005692.1基因可能与TIR家族同源,介导植物抗性。同时CSS0005692.1基因也可能与富含亮氨酸重复序列(LRR)同源,参与气孔发育时蛋白质之间的互作[51]。与SA性状相关的7个基因中,CSS0032323.1参与蛋白质磷酸化、含磷复合代谢等生物过程,尤其在代谢过程中可能影响气孔发育的分子过程,如蛋白质丝氨酸/苏氨酸激酶活性、ATP结合、腺苷核糖核苷酸结合等。CSS0018568.1编码蛋白在细胞组成上作为膜整体组分,与生长素激活信号通路和防御反应有关,也可能与调节ATP依赖的蛋白易位到线粒体基质的PAM16同源[52]。生长素通过载体蛋白TIR1/AFB和受体AUXIN/IAA-ARFs信号通路来调控STOMAGEN的表达,再经STOMAGEN-类LRR受体蛋白激酶ERf-MAPKs级联反应激活SPCH转录因子的信号通路,启动气孔发育的进程[53]。CSS0018568.1基因还可能参与次生细胞壁形成,尤其是木质素的生物合成,与MWL1和2家族同源[54]。CSS0018568.1对气孔发育可能有重要的影响,包括细胞组成、激素反应和能量代谢等。与SD性状相关的2个基因中,CSS0010060.1与1-酰基-SN-甘油-3-磷酸酰基转移酶4有关,甘油-3-磷酸酰基转移酶(GPAT)通过调控叶片和茎的角质合成,影响植株的抗旱性和抗病性,造成气孔结构异常,导致气孔增大[55]。也许CSS0010060.1基因对气孔形态大小有调节作用。与SP性状相关联的4个基因中,CSS0014397.1基因与锌脂蛋白B-BOX有关,并可能在转录和翻译水平调控气孔发育。
