玉米转录因子ZmEREB211对非生物逆境胁迫的应答
2023-07-20悦曼芳邹华文吴忠义
艾 蓉 张 春 悦曼芳 邹华文 吴忠义,*
玉米转录因子对非生物逆境胁迫的应答
艾 蓉1,2,**张 春2,**悦曼芳2邹华文1,*吴忠义2,*
1长江大学农学院, 湖北荆州 434025;2北京市农林科学院 / 生物技术研究所 / 农业基因资源与生物技术北京市重点实验室, 北京 100097
AP2/ERF (APETALA2/ethylene-responsive factor)转录因子是植物中最大的转录因子家族之一, 在调控植物生长发育和响应逆境胁迫等方面起重要作用。探究玉米(L.) AP2/ERF家族基因功能将为玉米新种质创制提供重要的基因资源。本研究克隆获得了(Gene ID: 103647485)基因, 利用生物信息学分析、实时荧光定量PCR等技术对该基因的基本特性、组织表达特性及响应逆境胁迫表达模式等进行了分析; 对转基因拟南芥株系进行了相应逆境胁迫处理和表型鉴定。结果显示: 该基因只包含1个外显子, cDNA全长为792 bp, 编码263个氨基酸; ZmEREB211蛋白分子量为27.9 kD, 理论等电点为6.01, 具有AP2家族所特有的保守结构域;基因在玉米根系中的表达量最高, 且在幼根中的表达量高于成熟根中的表达量; 同时该基因在脱水、高盐、干旱和低温等处理条件下均有不同程度的诱导表达。在分别含有不同浓度梯度的NaCl、甘露醇(mannitol)和茉莉酸(jasmonic acid, JA)的1/2 MS培养基上, 转基因拟南芥株系的根长显著长于野生型。在干旱和高盐处理下, 盆栽转基因拟南芥株系较野生型株系表现出更强的耐受性, 且苗期的绿叶数显著多于野生型, 过氧化物酶(POD)活性和叶绿素含量均显著高于野生型。研究表明可能参与调控玉米根系生长发育, 对高盐、干旱、渗透等逆境胁迫及JA激素处理均能起到正向的调控作用。本研究为进一步解析在玉米中的生物学功能提供了重要的参考依据。
玉米;; 转录因子; 异源表达; 根系; 逆境胁迫
植物在生长过程中会遇到多种环境胁迫, 干旱、涝渍、高温、盐碱、低温等逆境均会影响植物正常生长, 为了生存, 植物已建立起多种响应机制来调节体内的生理活动[1-2]。转录因子(transcription factor, TF)又称为反式作用因子, 在植物生长发育和响应逆境胁迫等过程中具有重要调控作用[3]。从蛋白质结构方面分析, 典型的植物转录因子由4个部分组成: DNA结合区(DNA binding domain)、转录调控区(transcription regulation domain)、寡聚化位点区(oligomerization site)和核定位信号区(nuclear localization signal)[4]。DNA结合区由一段60~100个氨基酸序列组成。转录调控区有转录激活的结构域和转录抑制的结构域2类。寡聚化位点区所含有的氨基酸序列具有高度保守的特点, 为转录因子相互作用提供一个功能区域。核定位信号区是一个短肽序列, 赖氨酸和精氨酸残基较多, 与受体蛋白相结合, 在亲核蛋白的帮助下引导整个蛋白通过核孔进入细胞核[5-6]。转录因子通过调控下游逆境相关基因的表达从而改善植物对逆境胁迫的抵抗能力, 因此, 转录因子在植物抗逆研究中有着至关重要的作用。植物中约有58个转录因子家族, 其中有6个主要的转录因子家族: AP2/ERF、bHLH、MYB、NAC、WRKY和bZIP, 被发现参与植物生长发育调控和生物与非生物应激响应, 作为植物防御的主要调节因子, 这些转录因子的表达和活性受到各种转录和转录后调控以及翻译后修饰的影响[7-8]。
AP2/ERF家族转录因子是参与植物非生物胁迫反应的最大转录因子家族之一, 该家族转录因子的特征是含有1~2段约60个氨基酸残基组成的AP2/ERF结构域, 该结构域由3条β-折叠(负责DNA结合)和1个α-螺旋基序组成[9]。基于AP2结构域外保守基序的变异和存在, 确定了几个具有不同DNA亲和力的亚家族, 将其分为AP2 (APETALA2)、ERF (ethylene-responsive factor)、DREB (dehydration- responsive element-binding protein)、RAV (related to ABI3/VP1)和Soloist五个亚家族[10]。AP2亚家族含有2个AP2/ERF结构域; ERF和DREB亚家族含有1个AP2/ERF结构域; RAV亚家族含有1个AP2/ERF结构域和1个B3结构域; Soloist亚家族含有1个AP2/ERF结构域, 每个亚家族成员识别不同的顺式作用元件, 并拥有其他分子功能所需的额外特定保守基序, 如蛋白间的相互作用和磷酸化[11]。ERF亚家族蛋白可以与一个被称为乙烯响应(ERE)元件的保守AGCCGCC序列结合, 该序列不仅参与调节植物非生物胁迫反应中的基因表达, 而且与植物抗性基因的调节密切相关[12], 而DREB亚家族蛋白与顺式作用的DRE/CRT元件相互作用, 该元件具有应激诱导基因启动子中a/GCCGAC的核心基序, 以调节基因表达, 并对植物激素如乙烯、ABA等作出反应[13-14]。Nakano等[15]对拟南芥进行全基因组分析, 鉴定出147个AP2/ERF转录因子, 其中, AP2亚家族基因18个, DREB与ERF亚家族基因122个, RAV亚家族基因6个。异源表达拟南芥表现出更高的盐胁迫耐受性, 该基因参与ABA信号转导、促使脯氨酸生物合成和活性氧清除基因的表达增强; 在棉花中, 沉默导致棉花对盐胁迫的敏感性增强[16]。Tang等[17]发现在拟南芥中异源表达增强了拟南芥对盐和寒冷的耐受性。Xie等[18]发现油菜素类固醇信号负调控拟南芥AP2/ERF转录因子基因, 通过激活干旱反应基因和促进脱落酸介导的气孔关闭正调控干旱响应。大豆基因和受到盐、干旱和茉莉酸等多种胁迫处理的诱导, 异源表达或转基因烟草也表现出对盐胁迫更高的耐受性[19-20]。在水稻中, 异源表达增加了其对盐胁迫的敏感性[21]。可以调控干旱响应基因表达, 过表达显著提高水稻的耐旱性, 并具有更高的光合效率[22]。枸橘和是2个受冷胁迫诱导的AP2/ERF家族转录因子, 对抵御冷胁迫具有重要调控作用[23]。玉米(L)作为单子叶植物功能基因组学研究的重要模式植物之一, 对植物分子生物学的研究具有重要意义。然而,基因在玉米响应非生物胁迫中的分子机制研究仍十分有限。在玉米中, Zhang等[24]对基因进行了全面的分析, 包括它们的系统发育关系、基因结构、保守基序、顺式元件、染色体分布、基因复制、进化机制和可能的调控网络分析, 为进一步探究基因在玉米生长发育和适应非生物胁迫中的功能和调控机制奠定了基础。Hao等[25]研究发现基因启动子区含有与植物生长发育、激素和非生物胁迫相关的元件, 可能参与植物发育以及对盐和干旱胁迫的响应。Wang等[26]发现过表达能够显著增加叶绿素含量和抗氧化酶活性, 并调节激素信号和胁迫应答基因的表达, 从而增强玉米幼苗的耐旱性。综上,基因家族陆续在棉花、拟南芥、大豆、水稻、柑橘等植物中被报道出来, 但在玉米中开展功能研究的还比较少。实验室前期通过转录组测序(RNA-seq)鉴定了玉米根系生长发育的关键调控基因, 我们对玉米生长发育过程中的4个关键时期(六叶期(V6)、十二叶期(V12)、抽雄期(VT)和乳熟期(R3))进行了根系表型鉴定和转录组测序分析, 筛选到26个在相邻时期间均显著差异表达的转录因子基因[27], 其中包含一个AP2/ERF家族成员基因。为了验证该基因对玉米根系生长发育的调控机制及是否参与对非生物逆境胁迫的响应, 本研究克隆了玉米基因, 研究了该基因的表达模式, 以及在干旱、高盐、脱水、低温等逆境胁迫下的表达情况, 将该基因在拟南芥中进行异源表达, 利用转基因拟南芥株系进行相关抗逆研究, 旨在深入了解AP2/ERF蛋白家族的生理功能, 为玉米抗逆遗传改良和新种质创制提供新思路和新的基因资源。
1 材料与方法
1.1 试验材料与试剂
玉米自交系B73 (本实验室保存及繁种); 野生型拟南芥(Col-0); 大肠杆菌Trans1 T1购自北京全式金生物技术有限公司; 农杆菌GV3101购于北京酷莱博科技有限公司; RNA提取试剂盒、逆转录试剂盒购于北京擎科生物技术有限公司; 过氧化物酶活性检测试剂盒、叶绿素含量检测试剂盒等购于北京索莱宝科技有限公司; 限制性内切酶等购自北京百灵克生物科技有限责任公司; 2×Phanta Max Master Mix等高保真酶购于南京诺唯赞生物科技股份有限公司。
1.2 植物种植与胁迫处理
将B73玉米种子播种于大田, 待成苗后, 分别取三叶一心时期的幼叶、幼根, 抽雄期的根、茎、叶、雄穗与花丝, 授粉1周后的幼胚, 用于组织差异表达分析。对于逆境胁迫处理, 挑选饱满的自交系B73种子进行发芽, 出苗后挑选长势一致的玉米幼苗移入水培桶中。分别设置对照组、渗透处理组、高盐处理组、脱水处理组和冷处理组, 每组设置6个水培桶, 每桶放置3株玉米幼苗, 各设3个生物学重复。B73幼苗正常生长至三叶一心时期分别进行渗透(0.2 mol L–1甘露醇)、高盐(0.2 mol L–1NaCl)、脱水(玉米幼苗整株取出, 吸水纸擦干表面水分, 室温放置, 自然脱水)、冷(4℃)处理。分别取各处理0、1、2、5、10和24 h的地上部和根, 液氮速冻后置于–80℃冻存。对于激素胁迫处理, 将B73玉米种子播种于花盆中, 对照组与激素处理组每组各设3盆, 每盆播种20粒。观察玉米幼苗生长至三叶一心时期, 分别喷洒0.1 mmol L–1浓度的茉莉酸(jasmonic acid, JA)、脱落酸(abscisic acid, ABA)、赤霉素(gibberellins, GA)、2,4-二氯苯氧乙酸(2,4-dichlorophenoxyacetic acid, 2,4-D)溶液, 使溶液均匀覆盖整个玉米幼苗后包裹保鲜膜, 分别取各处理的0、1、3、5和10 h后的叶片, 液氮速冻后置于–80℃冻存。
1.3 玉米总RNA提取及ZmEREB211基因克隆
按照植物总RNA提取试剂盒说明提取玉米根组织的总RNA, 逆转录得到cDNA。设计引物pZmEREB211-F和pZmEREB211-R, 以cDNA为模板进行PCR扩增, 用2×Phanta Max Master Mix高保真酶进行扩增, 反应程序为: 95℃预变性3 min; 95℃变性15 s, 60℃退火15 s, 72℃延伸30 s, 35个循环; 72℃终延伸5 min, 获得全长。
1.4 ZmEREB211生物信息学分析
通过ProtParam工具(http://web.expasy.org/ protparam/)分析编码氨基酸的数目, 蛋白相对分子量、等电点等理化性质; 应用SPOMA (https://npsa-prabi.ibcp.fr/cgibin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)预测蛋白质二级结构; 应用SWISS-MODEL (https://www.swissmodel.expasy. org/)预测其空间结构; 应用TMHMM2.0 (http://www. cbs.dtu.dk/services/TMHMM-2.0/)对氨基酸序列进行跨膜分析; 通过NCBI CDD (https://www.ncbi.nlm. nih.gov/Structure/cdd/docs/cdd_search.html)分析该基因的保守结构域。使用PlantCARE (http:// bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站进行启动子区顺式作用元件分析。

表1 本实验中所用的引物
1.5 ZmEREB211基因的表达模式分析
按照植物总RNA提取试剂盒说明提取玉米叶片RNA, 逆转录后得到的cDNA。以cDNA作为模板进行实时荧光定量PCR (qPCR)分析。设计特异引物pZmEREB211RT-F、pZmEREB211RT-R及内参基因特异引物pGAPDHRT-F、pGAPDHRT-R用于组织差异表达、胁迫处理下基因的表达量分析; 使用BIO-RAD CFX96 Real-Time System qPCR仪进行实时荧光扩增。每组实验设3个生物学样本, 每个生物学样本各设3个技术重复, 数据为3个生物学重复平均值±标准误。应用2–∆∆Ct方法分析基因的表达模式。组织特异性表达分析以花丝中表达量记为1。逆境处理表达量分析以对照(0 h)在地上部和根中的表达量计为1。
1.6 植物异源表达载体pYBA1132: ZmEREB211的构建及拟南芥遗传转化
1.6.1 异源表达载体构建 参照王莉等[28]的方法构建异源表达载体, 设计引物pZmEREB211-F、pZmEREB211-R扩增获得基因的编码区片段。扩增产物回收后无缝克隆至pYBA1132- bar-EGFP表达载体中获得重组质粒, 将重组质粒转化大肠杆菌后, 挑取阳性单克隆提质粒后进行酶切验证, 酶切正确的质粒送生工生物工程(上海)股份有限公司测序。测序正确的质粒转入农杆菌菌株GV3101中, 挑取单克隆进行菌液PCR鉴定。
1.6.2 拟南芥遗传转化 培养含有pYBA1132: ZmEREB211重组质粒的农杆菌菌株, 利用浸花法[29]侵染拟南芥花序。收取拟南芥种子, 干燥后置于4℃春化备用。将种子撒播于基质中, 喷施浓度为0.5‰的草铵膦筛选后获得T1代转基因拟南芥单株。提取T1代株系叶片基因组DNA为模板, 利用引物pZmEREB211-F、pZmEREB211-R进行PCR检测, 对转基因拟南芥株系进行单株收种并加代直至获得T4代纯合植株。
1.7 转基因拟南芥株系的表型分析
1.7.1 转基因拟南芥在1/2 MS培养基中不同逆境和激素处理的表型分析 将野生型和转基因拟南芥种子用0.3% NaClO (有效氯)消毒10 min后, 点播于1/2 MS培养基上, 竖直放置于拟南芥温室中培养7 d, 挑选根长和生长状态一致的野生型和转基因拟南芥幼苗, 分别移栽到含不同浓度NaCl、甘露醇、JA和ABA的1/2 MS固体培养基上[30]。NaCl浓度梯度为0、0.10、0.15和0.18 mol L–1; 甘露醇浓度梯度为0、0.15、0.20和0.30 mol L–1; JA浓度梯度为0、50、75和100 μmol L–1; ABA浓度梯度为0、10、25和50 μmol L–1 [31]。野生型和转基因拟南芥各5株, 各设置3个生物学重复, 竖直放置于拟南芥温室中培养, 7 d后拍照并用ImageJ软件统计根长。
1.7.2 土壤中转基因拟南芥植株的表型分析及生理指标的测定 选取1/2 MS培养基上生长8~10 d且生长状态一致的转基因拟南芥及野生型对照植株移栽至土壤中, 分别设置对照组、干旱组、盐处理组, 每组4盒, 每盒移植4株幼苗, 各组设3个生物学重复、3个技术重复。幼苗在品氏基质(PINDSTRUP)中生长3周后, 对照组进行正常浇水处理; 对干旱组进行自然干旱处理4周后和覆水处理24 h后分别观察表型及取样; 对盐处理组浇灌300 mmol L–1NaCl进行胁迫处理1周后观察表型并统计绿叶率。表型观察后, 根据试剂盒说明书测定植株的过氧化物酶活性和叶绿素含量。
2 结果与分析
2.1 ZmEREB211基因序列及其生物信息学分析
基因仅含有一个外显子, 编码序列全长为792 bp, 编码263个氨基酸。通过保守结构域分析发现, 该基因含有AP2家族所特有的保守结构域(图1-A)。生物信息学分析表明其蛋白分子量为27.9 kD, 理论等电点为6.01。二级结构分析表明,中不规则卷曲所占比例最大为52.85%, 其次是α螺旋35.74%, 应用TMHMM2.0分析发现该蛋白不具跨膜结构(图1-B)。空间结构预测结果如图1-C所示。对基因启动子上游2 kb序列进行在线分析, 发现该序列含有与生长发育、激素响应、光反应等功能相关的顺式作用元件MSA-like、GCN4-motif、ABRE、AuxRR-core、TGACC-motif、G-box等(图1-D)。
2.2 ZmEREB211在玉米中的组织差异表达分析
分别提取玉米三叶期的幼根、幼叶, 抽雄期根、茎、叶、雄穗与花丝以及授粉1周后的幼胚等组织的总RNA, 逆转录获得cDNA后进行qPCR分析。结果如图2所示,在玉米不同组织中的表达量存在较大的差异, 将抽雄期花丝中的表达量记为1,在玉米幼根中的表达量最高, 约为抽雄期花丝中表达量的7.2倍; 其次是抽雄期的根和茎, 分别为5.7倍和1.4倍; 其他组织的表达量均低于花丝, 雄穗和幼胚中的表达量最低。结果表明基因主要在玉米根系中表达, 且在幼根中的表达量最高, 推测该基因可能参与调控玉米根系生长发育。
2.3 逆境胁迫下ZmEREB211基因的表达分析
以胁迫处理0 h后的表达量记为1。在脱水处理下,在地上部的表达量下调, 直至处理24 h后的表达量上调, 约为对照的3.3倍; 其在根中的表达量迅速下调, 且一直处于受抑制状态(图3-A)。在盐处理下,在地上部的表达量下调; 在盐处理2 h后,在地上部的表达量上调, 约为对照的1.5倍, 随后下调, 直至处理24 h后, 在地上部的表达量上调至最大, 约为对照的36.8倍;在根中的表达量一直处于下调状态(图3-B)。在甘露醇处理下,在地上部的表达量呈现先下调后上调又下调的变化趋势, 其中处理5 h后,在地上部的表达量达到最高, 约为对照的19.0倍; 在根中表达量下调, 随后上调, 在甘露醇处理5 h后上调至最大, 约为对照的0.8倍(图3-C)。在4℃冷处理下,在地上部的表达量下调, 在处理24 h后,在地上部的表达量上调, 约为对照的2.2倍; 在根中的表达量呈现先下调后上调又下调的变化趋势(图3-D)。由此推测可能参与响应脱水、高盐、渗透以及低温等胁迫。
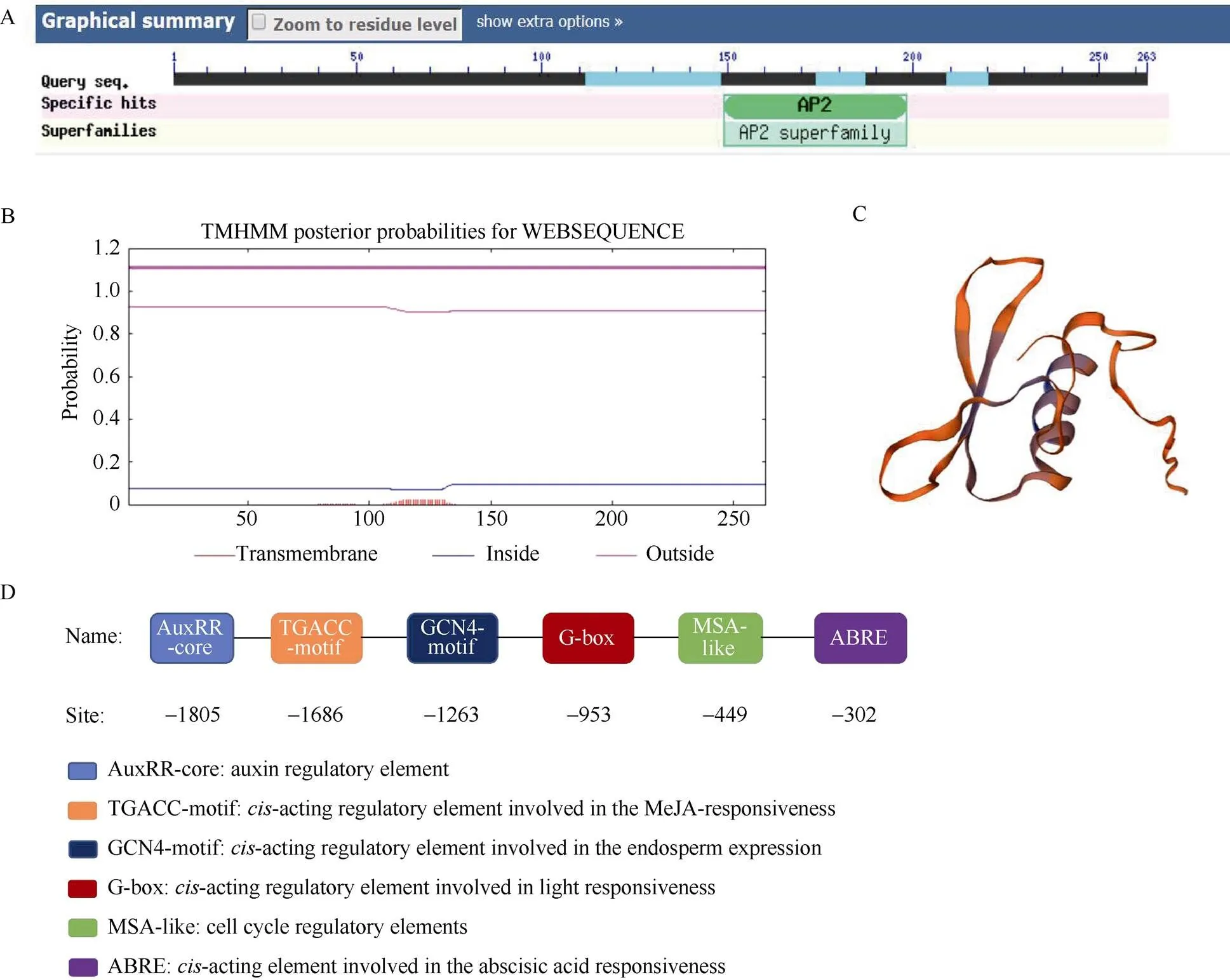
图1 ZmEREB211生物信息学分析
A: 保守结构域分析; B: 跨膜结构预测; C: 蛋白空间结构预测; D: 启动子区顺式作用元件分析。
A: the conserved domain analysis; B: the transmembrane structure prediction; C: protein tertiary structure prediction; D: the analysis of cis-acting elements in promoter region.

图2 ZmEREB211在玉米不同组织中的相对表达量
不同小写字母表示差异显著(< 0.05)。
Different lowercase letters are significantly different at the 0.05 probability level.
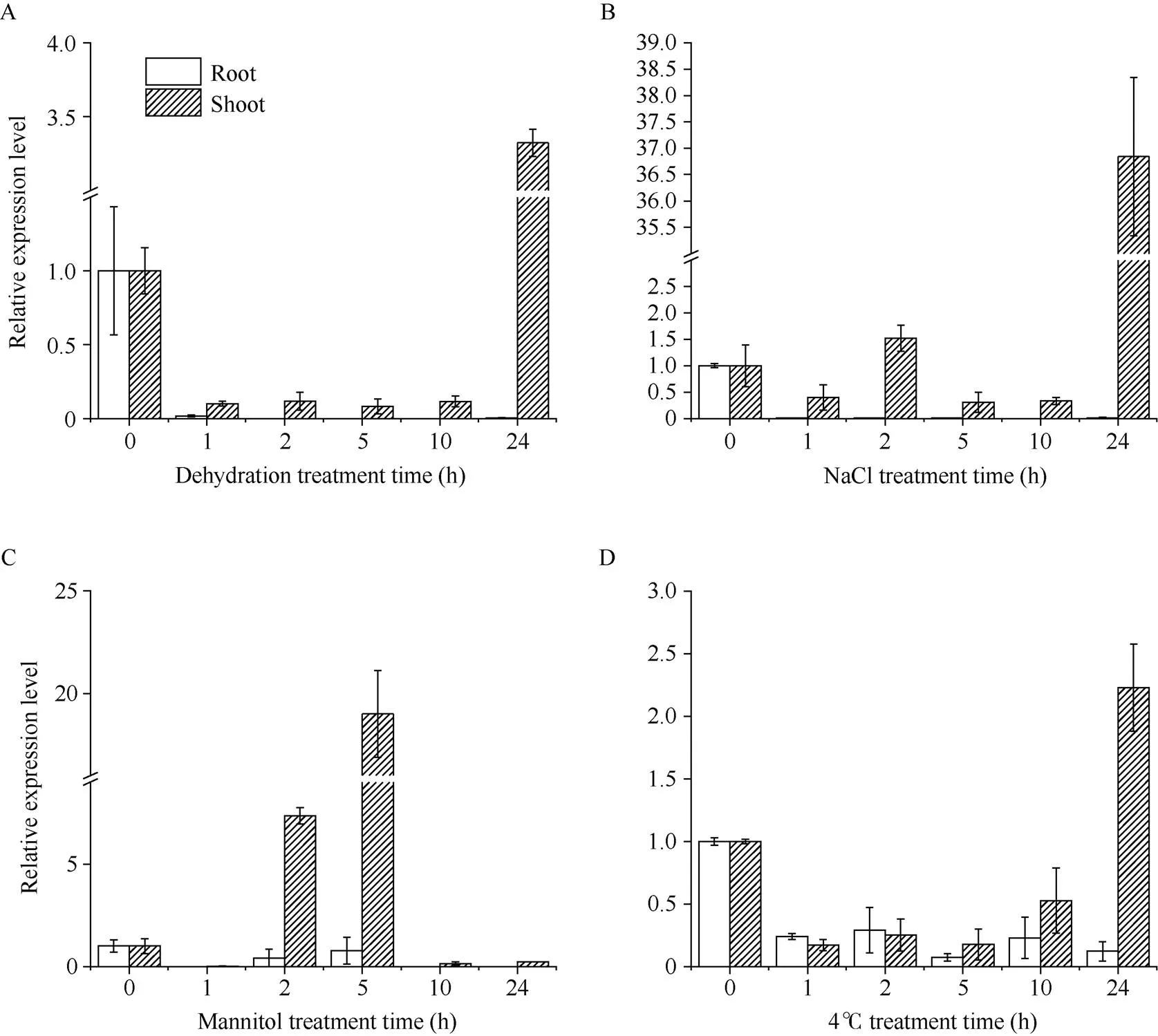
图3 ZmEREB211在不同非生物胁迫下的相对表达量
A~D:分别在脱水、高盐、渗透、低温处理后的表达量。
A–D: the relative expression level ofgenes after dehydration, high salt, osmotic, and low temperature treatments.
2.4 ZmEREB211基因在激素胁迫处理条件下的表达分析
如图4所示, 将0 h的表达量记为1, 在4种不同激素的处理条件下,的表达量呈现出不同的变化趋势。在JA处理1 h后的表达量下调, 随后在处理3 h后上调并达到最高, 约为对照的5.5倍。在ABA处理1 h后的表达量上调, 约为对照的2.3倍,随后一直下调, 至处理10 h后表达量仅为对照的0.03倍。在2,4-D处理1~5 h后,的表达量不断下降, 在处理10 h后上调, 约为对照的4.8倍。在GA处理下,先呈现连续上调表达趋势, 直至处理3 h后表达量最高, 约为对照的8.9倍, 随后下调。上述结果表明,的表达均不同程度地受到激素胁迫的影响。推测可能参与JA、ABA、2,4-D和GA等激素的诱导响应。

图4 ZmEREB211在玉米不同激素处理中的相对表达量
*:< 0.05; **:< 0.01.
2.5 转基因拟南芥株系PCR和qPCR检测
提取草铵膦抗性植株叶片DNA进行PCR检测, 结果如图5-A所示, 有6个转基因拟南芥株系含有目的基因, 依次编号为L-1、L-2、L-3、L-4、L-5和L-6, 阴性对照和野生型均无目的条带。对6个株系进行qPCR检测, 结果如图5-B所示, 发现L-3、L-4、L-5株系的表达量较高, 后续的抗逆性分析选用这3个株系进行。
2.6 转ZmEREB211基因拟南芥株系在1/2 MS培养基上胁迫处理分析
2.6.1 转基因拟南芥株系的耐盐胁迫分析 在0 mol L–1NaCl浓度下, 野生型和转基因拟南芥均能正常生长, 主根长度无明显差异。在0.1 mol L–1NaCl浓度下, 野生型和转基因拟南芥均能正常生长, 其中有2个转基因株系的根长长于野生型拟南芥的根长(图6-E)。在0.15 mol L–1NaCl浓度下, 与野生型拟南芥相比, 3个转基因拟南芥株系均表现出更高的耐受性, 主根更长, 且差异显著(<0.05) (图6-E)。在0.18 mol L–1NaCl浓度下, 野生型和转基因拟南芥均不能正常生长, 植株萎蔫, 叶片发黄, 主根不生长。由此推测基因对一定浓度的盐胁迫起到重要的耐受调控作用。

图5 T4代拟南芥PCR检测和qPCR检测
A: T4代转基因拟南芥PCR检测; B: T4代转基因拟南芥qPCR检测。M: DL2000 marker; N: 阴性对照; W: 水对照; WT: 野生型拟南芥; L-1~L-6: T4代转基因拟南芥株系; *:< 0.05; **:< 0.01。
A: PCR detection of T4generation; B: qPCR detection of T4generation; M: DL2000 marker; N: negative control; W: water control; WT: wild-type; L-1–L-6: T4 transgeniclines; *:< 0.05; **:< 0.01.
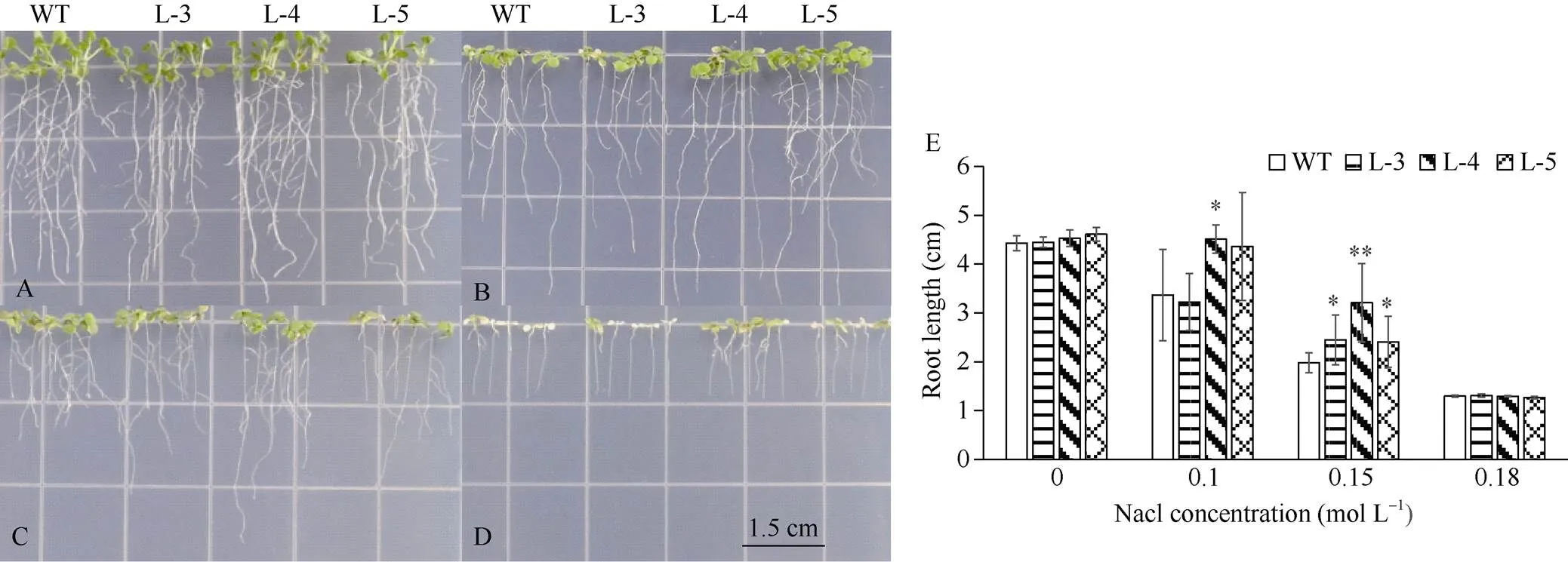
图6 不同盐浓度下转基因拟南芥株系根长比较
A~D分别为0、0.1、0.15和0.18 mol L–1盐处理的生长情况; E: 不同盐浓度下拟南芥株系平均主根长度; WT: 野生型拟南芥; L-3、L-4、L-5: 转拟南芥株系; 标尺为1.5 cm; *:< 0.05; **:< 0.01。
A–D indicate the seedling growth under the NaCl concentrations of 0, 0.1, 0.15, and 0.18 mol L–1, respectively. E: the average main root length ofunder different salt concentrations; WT: wild type; L-3, L-4, and L-5:transgeniclines; Bar: 1.5 cm; *:< 0.05; **:< 0.01.
2.6.2 转基因拟南芥株系的耐渗透胁迫分析 在0 mol L–1甘露醇浓度下, 野生型和转基因拟南芥植株均能正常生长, 主根长度无明显差异。在0.15 mol L–1甘露醇浓度下, 野生型和转基因拟南芥均能正常生长, 转基因拟南芥与野生型拟南芥的主根长无显著差异(图7-E)。在0.2 mol L–1甘露醇浓度下, 转基因拟南芥的根长比野生型拟南芥长, 且其中2个转基因株系的根长较对照的根长差异呈极显著水平(<0.01) (图7-E)。在0.3 mol L–1甘露醇浓度下, 拟南芥叶片出现萎蔫现象, 但转基因拟南芥株系均比野生型生长状态更好, 主根长无显著差异(图7-E)。推测基因可能参与渗透胁迫响应。
2.6.3 转基因拟南芥株系在JA和ABA处理下的分析 在0 mol L–1JA浓度下, 野生型和转基因拟南芥均能正常生长, 主根长度无明显差异。在50 µmol L–1和75 µmol L–1JA浓度下, 野生型和转基因拟南芥均生长迟缓, 侧根数减少, 野生型和转基因拟南芥主根长无明显差别。在100 µmol L–1JA浓度下, 野生型拟南芥株系的生长受到进一步的抑制, 转基因拟南芥与野生型拟南芥主根长度有明显差异, 其中, L-3株系的根长较对照差异呈显著水平(<0.05), L-4、L-5株系的根长较对照差异均呈极显著水平(<0.01) (图8-B)。推测基因对较高浓度的JA激素具有一定的耐受性, 可能参与JA信号转导途径。

图7 不同甘露醇浓度下转基因拟南芥株系根长比较
A~D 分别为0、0.15、0.2和0.3 mol L–1甘露醇处理的生长情况; E: 不同甘露醇浓度下拟南芥株系平均主根长度。缩写同图6; 标尺为1.5 cm; *:< 0.05; **:< 0.01。
A–D indicate the seedling growth under the mannitol concentrations of 0, 0.15, 0.2, and 0.3 mol L–1, respectively. E: the average main root length ofunder different mannitol concentrations. Abbreviations are the same as those given in Fig. 6; Bar: 1.5 cm; *:< 0.05; **:< 0.01.
在0 mol L–1ABA浓度下, 野生型和转基因拟南芥均能正常生长, 主根长度无明显差异。在10 µmol L–1ABA浓度下, 野生型和转基因拟南芥叶片均呈黄色, 野生型和转基因拟南芥株系主根长度无明显差别。在25 µmol L–1和50 µmol L–1ABA浓度下, 野生型和转基因拟南芥叶片均呈黄色, 转基因拟南芥株系主根长度较野生型短, 且L-3和L-4株系的根长较对照根长差异均为极显著水平(<0.01) (图8-D)。推测基因的表达受ABA激素的调控, 且表现为负向调控作用。
2.7 转ZmEREB211拟南芥株系在土壤中的抗逆性分析
2.7.1 转拟南芥株系在土壤中的耐旱性分析 如图9-A所示, 在正常培养条件下, 野生型和转基因拟南芥株系均生长良好, 无明显表型差异。经过干旱处理4周后, 野生型和转基因拟南芥均出现萎蔫现象, 但野生型拟南芥叶片萎蔫失水程度比转基因拟南芥更加严重, 另外转基因拟南芥的绿叶率(图9-B)、过氧化物酶活性(图9-D)和叶绿素含量(图9-E)均显著高于野生型拟南芥。干旱处理实验组拟南芥进行复水处理24 h后, 野生型拟南芥未恢复绿叶状态, 复水前后转基因拟南芥株系的绿叶率(图9-B, C)均显著高于野生型拟南芥株系。推测可能通过调节过氧化物酶活性响应干旱胁迫。

图8 不同JA和ABA浓度下转基因拟南芥株系根长比较
A: 0、50、75和100 µmol L–1JA处理下的生长情况; B: 不同JA浓度下拟南芥株系平均主根长度; C: 0、10、25和50 µmol L–1ABA处理下的生长情况; D: 不同ABA浓度下拟南芥株系平均主根长度。缩写同图6; 标尺为1.5 cm; *:< 0.05; **:< 0.01。
A: growth under 0, 50, 75, and 100 μmol L–1JA treatment; B: the average main root length ofunder different JA concentrations; C: growth under 0, 10, 25, and 50 μmol L–1ABA treatment; D: the average main root length ofunder different ABA concentrations. Abbreviations are the same as those given in Fig. 6; Bar: 1.5 cm; *:< 0.05; **:< 0.01.

图9 土壤中干旱处理下拟南芥株系表型分析
A: 拟南芥表型; B: 干旱处理后绿叶率; C: 复水处理后绿叶率; D: POD活性的测定; E: 叶绿素含量测定。缩写同图6; *:< 0.05; **:< 0.01。
A: the phenotype of; B: the rate of green leaves after drought treatment; C: the rate of green leaves after re-water treatment; D: the determination of POD activity; E: the determination of chlorophyll content. Abbreviations are the same as those given in Fig. 6; *:< 0.05; **:< 0.01.
2.7.2 转拟南芥株系在土壤中的耐盐性分析 如图10-A所示, 在正常培养条件下, 野生型和转基因拟南芥株系生长良好, 无明显表型差异。经过300 mmol L–1NaCl处理7 d后, 野生型拟南芥叶片严重萎蔫发黄, 而转基因拟南芥含有更多的绿叶(图10-B), 表现出更健康的生长状态。另外, 转基因拟南芥的过氧化物酶活性(图10-C)和叶绿素含量(图10-D)均显著高于野生型拟南芥。推测可能通过调节过氧化物酶活性响应盐胁迫。
3 讨论
干旱、高盐等非生物逆境胁迫严重影响植物的生长发育和生产力, AP2/ERF家族是植物界中最大的转录因子家族之一, 在植物响应各种生物和非生物胁迫中发挥着重要作用[32]。本研究克隆了基因, 具有AP2/ERF转录因子家族所特有的保守结构域。目前, 其它植物中有许多AP2/ERF家族响应逆境胁迫的报道, 在拟南芥中异源表达羊草可以提高转基因拟南芥的抗旱和抗盐能力[33]。在乙烯和高盐诱导下, 异源表达火龙果基因的拟南芥植株对盐胁迫的耐受性显著增强, 同时也表明参与调控乙烯介导的盐胁迫耐受过程[34]。这些研究表明, 不同物种中的AP2/ERF转录因子对不同逆境的响应存在差异。在玉米不同时期不同组织中表达量不同, 在根中的表达量最高, 且在幼根中的表达量比成熟根中的表达量更高, 说明该基因可能参与调控玉米根系生长发育。有研究表明在拟南芥中的同源基因()通过介导JA信号转导和生长素生物合成, 调控拟南芥侧根形成[35]。为了进一步探索该基因在响应玉米非生物胁迫中的作用, 我们对转基因拟南芥株系进行了抗逆分析, 发现在分别含有0.1 mol L–1NaCl和0.2 mol L–1甘露醇的1/2 MS培养基中, 转基因拟南芥根长长于野生型, 且差异显著(<0.05)。土壤中生长的转基因拟南芥, 在干旱和含有300 mmol L–1NaCl的高盐处理下, 较野生型拟南芥生长状态更好, 绿叶率更高且差异显著(<0.05)。在这2种胁迫处理条件下, 转基因拟南芥株系具有更高的POD活性, 进一步说明在渗透、高盐和干旱胁迫中可能是一个积极的胁迫响应因子。基因起始密码子上游2 kb区域内具有与JA激素相关的顺式作用元件, 并且在含有100 µmol L–1JA的1/2 MS培养基上, 转基因拟南芥比野生型拟南芥的根更长, 说明基因响应JA处理, 可能参与JA信号转导。由此我们推测,基因可能是通过JA信号通路来参与干旱和盐的胁迫应答。

图10 土壤中高盐处理下拟南芥株系表型分析
A: 拟南芥表型; B: 高盐处理后绿叶率; C: POD活性的测定; D: 叶绿素含量的测定。缩写同图6; *:< 0.05; **:< 0.01。
A: the phenotype of; B: the rate of green leaves after high salt treatment; C: the determination of POD activity; D: the determination of chlorophyll content. Abbreviations are the same as those given in Fig. 6; *:< 0.05; **:< 0.01.
4 结论
本研究克隆了基因, 对其进行了生物信息学分析、组织差异表达分析和转基因拟南芥株系表型鉴定及分析。基因编码AP2/ERF家族转录因子, 该基因在玉米多个组织器官中均有表达, 但主要在根系中表达, 且在幼根中表达量最高, 受脱水、高盐、甘露醇、冷胁迫处理以及JA、ABA等激素胁迫的诱导表达。转基因拟南芥在高盐、渗透和JA处理下的生长状态比野生型拟南芥更好; 在干旱和高盐处理下, 土壤中生长的转基因植株相较于对照表现出更强的耐受性, 且POD活性和叶绿素含量均显著升高。由此表明,对玉米根系生长发育可能具有显著的调控作用, 在应对高盐、干旱、渗透胁迫以及激素处理等过程中起到积极的响应作用。本研究初步解析了基因的生物学功能, 为进一步探究其在玉米中的生物学功能奠定了基础, 同时为今后挖掘和利用该类转录因子提供了重要参考。
[1] 仇焕广, 李新海, 余嘉玲. 中国玉米产业: 发展趋势与政策建议. 农业经济问题, 2021, (7): 4–16. Qiu H G, Li X H, Yu J L. China’s corn industry development trends and policy recommendations.2021, (7): 4–16 (in Chinese).
[2] Osmond C B, Austin M P, Berry J A, Billings W D, Boyer J, Dacey J W H, Nobel P S, Smith S D, Winner W E. Stress physiology and the distribution of plants: the survival of plants in any ecosystem depends on their physiological reactions to various stresses of the environment., 1987, 37: 38–48.
[3] 张雪莹, 刘欣. 转录因子与叶片发育的研究进展. 植物生理学报, 2022, 58(1): 91–100. Zhang X Y, Liu X. Research progress of transcription factors and leaf development., 2022, 58(1): 91–100 (in Chinese with English abstract).
[4] 葛宝宇, 林轶, 侯和胜. ERF类转录因子的结构与功能. 安徽农学通报, 2007, (20): 32–35. Ge B Y, Lin Y, Hou H S. Structure and function of ERF transcription factors., 2007, (20): 32–35 (in Chinese with English abstract).
[5] 刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用. 科学通报, 2000, 45: 1465–1474.Liu Q, Zhang G Y, Chen S Y. Structure and regulation of plant transcription factors., 2000, 45: 1465–1474 (in Chinese).
[6] 张艳馥, 沙伟. 转录因子概述. 生物学教学, 2009, 34(10): 7–8.Zhang Y F, Sha W. Overview of transcription factors., 2009, 34(10): 7–8 (in Chinese).
[7] 悦曼芳, 张春, 吴忠义. 植物转录因子AP2/ERF家族蛋白结构和功能的研究进展. 生物技术通报, 2022, 38(12): 11–26. Yue M F, Zhang C, Wu Z Y. Advances in the structure and function of plant transcription factor AP2/ERF family proteins., 2022, 38(12): 11–26 (in Chinese with English abstract).
[8] Ng D W K, Abeysinghe J K, Kamali M. Regulating the regulators: the control of transcription factors in plant defense signaling., 2018, 19: 3737.
[9] Allen M D, Yamasaki K, Ohme-Takagi M, Tateno M, Suzuki M. A novel mode of DNA recognition by a beta-sheet revealed by the solution structure of the GCC-box binding domain in complex with DNA., 1998, 17: 5484–5496.
[10] Licausi F, Ohme-Takagi M, Perata P. APETALA2/Ethylene Responsive Factor () transcription factors: mediators of stress responses and developmental programs., 2013, 199: 639–649.
[11] Sakuma Y, Liu Q, Dubouzet J G, Abe H, Shinozaki K, Yamaguchi-Shinozaki K. DNA-binding specificity of thedomain ofDREBs, transcription factors involved in dehydration-and cold-inducible gene expression., 2002, 290: 998–1009.
[12] Ohme-Takagi M, Shinshi H. Ethylene-inducible DNA binding proteins that interact with an ethylene-responsive element., 1995, 7: 173–182.
[13] Jiang C, Iu B, Singh J. Requirement of a CCGAC-acting element for cold induction of thegene from winter., 1996, 30: 679–684.
[14] Yamaguchi-Shinozaki K, Shinozaki K. A novel-acting element in angene is involved in responsiveness to drought, low-temperature, or high-salt stress., 1994, 6: 251–264.
[15] Nakano T, Suzuki K, Fujimura T, Shinshi H. Genome-wide analysis of thegene family inand rice., 2006, 140: 411–432.
[16] Lu L L, Qanmber G, Li J, Pu M L, Chen G Q, Li S D, Liu L, Qin W Q, Ma S Y, Wang Y, Chen Q J, Liu Z. Identification and characterization of the ERF subfamily B3 group revealedimproves salt tolerance in upland cotton., 2021, 12: 705883.
[17] Tang M J, Sun J W, Liu Y, Chen F, Shen S H. Isolation and functional characterization of thegene, a putative AP2/EREBP domain-containing transcription factor, in the woody oil plant, 2007, 63: 419–428.
[18] Xie Z L, Nolan T, Jiang H, Tang B Y, Zhang M C, Li Z H, Yin Y H. The AP2/ERF transcription factormodulates brassinosteroid-regulated plant growth and drought responses in., 2019, 31: 1788–1806.
[19] Zhang G Y, Chen M, Chen X P, Xu Z S, Li L C, Guo J M, Ma Y Z. Isolation and characterization of a novel EAR-motif-containing genefrom soybean (L)., 2010, 37: 809–818.
[20] Zhai Y, Wang Y, Li Y J, Lei T T, Yan F, Su L T, Li X W, Zhao Y, Sun X, Li J W, Wang Q Y. Isolation and molecular characterization of, a soybean ethylene-response factor that increases salt stress tolerance in tobacco., 2013, 513: 174–183.
[21] Tang Y H, Qin S S, Guo Y L, Chen Y B, Wu P Z, Chen Y P, Li M R, Jiang H W, Wu G J. Genome-wide analysis of thegene family in physic nut and overexpression of thegene in rice increased its sensitivity to salinity stress., 2016, 11: e0150879.
[22] Jung S E, Bang S W, Kim S H, Seo J S, Yoon H B, Kim Y S, Kim J K. Overexpression of, a vascular tissue-specific transcription factor gene, confers drought tolerance in rice., 2021, 22: 7656.
[23] Şahin-Çevik M, Moore G A. Two AP2 domain containing genes isolated from the cold-hardy citrus relativeare induced in response to cold., 2006, 33: 863–875.
[24] Zhang J, Liao J Y, Ling Q Q, Xi Y, Qian Y X. Genome-wide identification and expression profiling analysis of maizesuperfamily genes reveal essential roles in abiotic stress tolerance., 2022, 23: 125.
[25] Hao L D, Shi S B, Guo H B, Li M, Hu P, Wei Y D, Feng Y F. Genome-wide identification and expression profiles of ERF subfamily transcription factors in., 2020, 8: e9551.
[26] Wang Z Y, Zhao X, Ren Z Z, Abou-Elwafa SF, Pu X Y, Zhu Y F, Dou D D, Su H H, Cheng H Y, Liu Z X, Chen Y H, Wang E, Shao R X, Ku L X.directly regulates hormone signaling and stress-responsive gene expression to influence drought tolerance in maize seedlings., 2022, 45: 312–328.
[27] Zhang C, Li X L, Wang Z P, Zhang Z B, Wu Z Y. Identifying key regulatory genes of maize root growth and development by RNA sequencing., 2020, 112: 5157–5169.
[28] 王莉, 王作平, 张中保, 白玲, 吴忠义. 玉米早期籽粒中强表达启动子的筛选. 作物杂志, 2020, (4): 114–120. Wang L, Wang Z P, Zhang Z B, Bai L, Wu Z Y. Screening of strong expression promoters in early kernels of maize., 2020, (4): 114–120 (in Chinese with English abstract).
[29] Clough S J, Bent A F. Floral dip: a simplified method formediated transformation of., 1998, 16: 735–743.
[30] 杨梦婷, 张春, 王作平, 邹华文, 吴忠义. 玉米基因的克隆及功能研究. 作物学报, 2020, 46: 2008–2016. Yang M T, Zhang C, Wang Z P, Zou H W, Wu Z Y. Cloning and functional analysis ofgene in maize., 2020, 46: 2008–2016 (in Chinese with English abstract).
[31] 悦曼芳, 张春, 郑登俞, 邹华文, 吴忠义. 玉米转录因子对非生物逆境胁迫的应答. 作物学报, 2022, 48: 3004–3017. Yue M F, Zhang C, Zheng D Y, Zou H W, Wu Z Y. Response of maize transcriptional factorto abiotic stress., 2022, 48: 3004–3017 (in Chinese with English abstract).
[32] Wang Y Q, Xia D N, Li W Q, Cao X Y, Ma F, Wang Q Q, Zhan X Q, Hu T X. Overexpression of a tomato AP2/ERF transcription factor Sincreases sensitivity to salt and drought stresses., 2022, 304: 111332.
[33] Peng X J, Ma X Y, Fan W H, Su M, Cheng L Q, Alam I, Lee B H, Qi D M, Shen S H, Liu G S. Improved drought and salt tolerance ofby transgenic expression of a novelgene from., 2011, 30: 1493–1502.
[34] Qu Y J, Nong Q D, Jian S G, Lu H F, Zhang M Y, Xia K F. An AP2/ERF gene,, from pitaya () positively regulates salt tolerance., 2020, 21: 4586.
[35] Cai X T, Xu P, Zhao P X, Liu R, Yu L H, Xiang C B.mediates cross-talk between jasmonic acid and auxin biosynthesis during lateral root formation., 2014, 5: 5833.
Response of maize transcriptional factorto abiotic stress
AI Rong1,2,**, ZHANG Chun2,**, YUE Man-Fang2, ZOU Hua-Wen1,*, and WU Zhong-Yi2,*
1College of Agriculture, Yangtze University, Jingzhou 434025, Hubei, China;2Institute of Biotechnology, Beijing Academy of Agriculture and Forestry Sciences, Beijing Key Laboratory of Agricultural Gene Resources and Biotechnology, Beijing 100097, China
AP2/ERF (APETALA2/ethylene-responsive factor) transcription factor is one of the largest transcription factor families in plants, which plays an important role in regulating plant growth and development and responding to various stresses. Exploring the function of AP2/ERF family genes in maize (L) will provide the important genetic resources for the creation of new maize germplasm. In this study,(Gene ID: 103647485) gene was cloned and its basic characteristics, tissue expression characteristics, and the relative expression patterns in response to stress were analyzed by bioinformatics and qRT-PCR. The transgeniclines were subjected to corresponding stress treatment and phenotypic identification. The results showed that the gene contained only one exon and the full-length cDNA was 792 bp which encoding 263 amino acids. The ZmEREB211 protein had a molecular weight of 27.9 kD and a theoretical isoelectric point of 6.01. It had a conserved domain unique to the AP2/ERF family. The relative expression level ofgene was the highest in maize root system and the relative expression level in young roots was higher than mature roots. At the same time, the gene had different degrees of induced expression under dehydration, high salt, drought, and low temperature treatment conditions. On 1/2 MS medium containing different concentrations of NaCl, mannitol, and jasmonic acid (JA), root length oftransgeniclines was significantly longer than wild type. Under drought and high salt treatments, transgeniclines had stronger tolerance than wild type, and the number of green leaves at seedling stage was significantly higher than wild type and the peroxidase (POD) activity and chlorophyll content were significantly higher than wild type.may be involved in the regulation of root growth and development in maize, and can play a positive regulatory role in high salt, drought, osmotic stress, and JA hormone treatments. This study provides an important reference for further analysis of the biological function ofin maize.
maize;; transcription factor; heterologous expression; root system; adversity stress
2022-10-21;
2023-02-21;
2023-03-01.
10.3724/SP.J.1006.2023.23071
通信作者(Corresponding authors): 邹华文, E-mail: zouhuawen@yangtzeu.edu.cn; 吴忠义, E-mail: zwu22@126.com
**同等贡献(Contributed equally to this work)
艾蓉, E-mail: airong202203@163.com; 张春, E-mail: spring2007318@163.com
本研究由国家自然科学基金项目(32001430, 32171952, 31971839)和北京市农林科学院科技创新能力建设专项(KJCX20220402)资助。
This study was supported by the National Natural Science Foundation of China (32001430, 32171952, 31971839) and the Beijing Academy of Agricultural and Forestry Sciences (KJCX20220402).
URL: https://kns.cnki.net/kcms/detail//11.1809.S.20230228.1533.008.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
