青海湖裸鲤和花斑裸鲤线粒体基因组比较及其系统进化分析
2023-07-08保长虹李昭楠关却多杰李长忠尹格玛贺彩霞金文杰周叶吉陈艳霞
保长虹,李昭楠,关却多杰,李长忠,尹格玛,贺彩霞,金文杰,周叶吉,陈艳霞
(1.青海大学 生态环境工程学院,青海 西宁 810016;2.青海省共和县农牧业综合服务中心,青海 海南 813000)
青海湖裸鲤(Gymnocyprisprzewalskii)和花斑裸鲤(Gymnocypriseckloni)两者同属鲤形目(Cypriniformes)、鲤科(Gyprinidae)、裂腹鱼亚科(Schizothoracinae)、裸鲤属(Gymnocypris),是青藏高原特有的土著鱼类和水生生物资源,具有重要的生态价值和经济价值。其中,青海湖裸鲤主要分布在全国最大的咸水湖—青海湖及其支流中,鱼体长,稍侧扁且体表无鳞,仅在肛门和臀鳍两侧及肩带部位有稀疏的特化鳞片,鱼体背部呈灰褐色或黄色,腹部则为灰白色或浅黄色,体侧有不规则的褐色块斑,也有个别鱼体呈浅黄色[1]。曾经一度由于捕捞强度过大和自然灾害,导致青海湖裸鲤资源量急剧下降[2]。90年代末青海湖裸鲤资源量下降到0.34万t,破坏了青海湖裸鲤群体的自身平衡能力[3],2004年青海湖裸鲤在《中国物种红色名录》中列为濒危物种。因此,青海省采取了6次封湖育鱼并持续开展了青海湖裸鲤人工增殖放流工作。2014年资源量达到5.05万t[4],截至2019年已累计放流青海湖裸鲤1龄鱼种1.56亿尾[5],2021年底青海湖裸鲤资源蕴藏量达到10.86万t[6]。
花斑裸鲤主要分布在黄河上游、扎陵湖、鄂陵湖和奈齐河水系,生长速度缓慢,肉质较好,体稍长,侧扁,头呈锥形,口亚下位,口裂较大,下颌没有锐利角质,唇较薄,下唇侧叶狭窄,唇后沟呈不连续状,身体几乎完全裸露,身体背部呈暗褐色或青灰色,腹部为浅黄色或银灰色,体侧常有云状斑点或条状斑纹[7]。2009年起,花斑裸鲤人工繁育在青海省渔业技术推广中心获得成功,随后开展了人工增殖放流,目前年增殖放流量达到100万尾,有力促进了花斑裸鲤野生资源的恢复。
青海湖裸鲤和花斑裸鲤经过长期的进化,形成了适应青藏高原高海拔、低温、低氧、强紫外线辐射等极端环境的形态特征和生存能力[8-9],逐渐成了一种理想的适应性进化研究模型[8]。
动物线粒体基因组具有典型的母系遗传、编码区较保守、控制区进化速度快、突变率高以及独立的复制单位等遗传特性,是分子进化研究中十分有用的材料。绝大多数后生动物的线粒体基因组是一个大小为14~20 kb的双链闭合环状DNA分子,共编码37个基因,包括22个转运RNA(tRNA)基因、13个蛋白编码基因(PCGs)和2个核糖体RNA(rRNA)基因[10]。此外,线粒体基因组中还有1段长的非编码区,称为控制区(Control region,CR)或富集区(AT-rich)[11]。
目前,线粒体基因组数据广泛用于硬骨鱼类不同分类阶元的系统进化关系研究[12]。然而,裸鲤属中基于线粒体基因组数据的比较研究很少。本研究以青海湖裸鲤和花斑裸鲤为研究对象,测定二者线粒体基因组全序列,对其线粒体全基因组序列的结构特征进行分析,并结合GenBank数据库中已公布的36种鲤科物种线粒体基因组全序列,构建系统发育树,为进一步研究两者的遗传进化和分类提供依据。
1 材料和方法
1.1 试验材料
本试验青海湖裸鲤样品来源于青海湖裸鲤救护中心,花斑裸鲤样品来源于青海省渔业技术推广中心。取二者肌肉组织样本于-80 ℃保存,用于基因组DNA的提取。
1.2 基因组DNA提取
取约50 mg的青海湖裸鲤和花斑裸鲤肌肉组织,应用TIANamp Genomic DNA Kit血液/细胞/组织基因组DNA提取试剂盒(DP201101X,TIANGEN)提取二者肌肉组织的基因组DNA,通过紫外分光光度计对DNA纯度和浓度进行测定,再经1%的琼脂糖凝胶电泳检测其完整性。
1.3 基因组测序
样品基因组DNA检测合格后,用机械打断的方法(超声波)将DNA片段化,然后对片段化的DNA进行片段纯化、末端修复、3′端加A、连接测序接头,再用琼脂糖凝胶电泳进行片段大小选择,进行PCR扩增富集,构建测序文库,建好的文库进行纯化去接头污染,再进行文库质检,质检合格的文库用Illumina Novaseq平台进行测序。
1.4 序列组装与分析
为降低序列组装的复杂度,使用bowtie2 v2.2.4软件[13]very-sensitive-local模式比对线粒体基因组数据库,将比对上的测序序列作为线粒体基因组测序序列(mtDNA序列)。采用SPAdes[14]软件组装线粒体基因组,组装不依赖参考基因组。使用参考序列AB239595.1.gbk(https://www.ncbi.nlm.nih.gov/nuccore/)进行组装完成后的质控。使用Mitos2[15](http://mitos2.bioinf.uni-leipzig.de)对组装好的序列进行注释,将Mitos2注释结果与近缘物种比较,校正标准后获取最终的注释结果。使用OGDRAW[16](https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)制作线粒体基因组图谱。使用软件CGVIEW[17](http://stothard.afns.ualberta.ca/cgview_server/,默认参数),针对近源物种进行线粒体基因组结构的比较分析。
1.5 系统进化分析
从NCBI上下载36个鲤科鱼类的线粒体全基因组序列,应用全基因组做进化树分析,将环形序列设置相同起点,物种间序列用MAFFT软件[18](v7.427,--auto模式)进行多序列比对,将比对好的数据用RAxML v8.2.10(https://cme.h-its.org/exelixis/software.html)软件[19](选用GTRGAMMA模型,rapid Bootstrap分析,Bootstrap=1 000),构建最大似然进化树。
2 结果与分析
2.1 线粒体基因组基本特征
经测序分析,青海湖裸鲤线粒体基因组大小为16 720 bp,共注释37个基因,其中蛋白质编码基因13个,tRNA基因22个,rRNA基因2个(图1)。花斑裸鲤线粒体基因组大小为16 760 bp,共注释37个基因,其中蛋白质编码基因13个,tRNA基因22个,rRNA基因2个(图2)。
分析青海湖裸鲤和花斑裸鲤核苷酸组成,青海湖裸鲤A+T含量占整个线粒体基因组的55.97%,蛋白编码基因(Primordial germ cells,PCGs)、tRNA和rRNA的A+T含量分别占整个线粒体基因组的56.42%,54.80%和53.74%(表1)。

表1 青海湖裸鲤线粒体基因组核苷酸组成Tab.1 Nucleotide composition of Gymnocypris przewalskii mitochondrial genome
花斑裸鲤A+T含量占整个线粒体基因组的55.85%,PCGs(蛋白编码基因)、tRNAs和rRNAs的A+T含量分别占整个线粒体基因组的56.41%,54.67%和53.67%(表2)。

表2 花斑裸鲤线粒体基因组核苷酸组成Tab.2 Nucleotide composition of Gymnocypris eckloni mitochondrial genome
以往的研究报道中,碱基组成偏度在转录和复制过程中起着重要作用[20],青海湖裸鲤线粒体基因组的AT偏度(0.025)与花斑裸鲤相同,表明腺嘌呤(As)含量等于胸腺嘧啶(Ts)含量。青海湖裸鲤线粒体基因组GC的负偏度(-0.175)低于花斑裸鲤GC负偏度(-0.173),表明胞嘧啶(Cs)的含量高于鸟嘌呤(Gs)的含量(表1-2)。
青海湖裸鲤线粒体基因组中发现2个基因区域的碱基与邻近基因重叠,分别是nad5和nad6,重叠区大小为7 bp。基因组中重叠的片段在鱼类中一般只有7~10 bp,而在哺乳动物中一般可达40~46 bp[21]。青海湖裸鲤线粒体基因组共有17个基因间隔区,核苷酸长度分布为1~124 bp,其中trnT和trnP之间的间隔最长,为124 bp(表3)。

表3 青海湖裸鲤线粒体基因组特征Tab.3 Mitochondrial genome characteristics of Gymnocypris przewalskii
花斑裸鲤线粒体基因组中同样发现2个基因区域的碱基与邻近基因重叠,分别是nad5和nad6,重叠区大小为7 bp。花斑裸鲤线粒体基因组共有17个基因间隔区,核苷酸长度分布为1~164 bp,其中trnT和trnP之间的间隔最长,为164 bp(表4)。
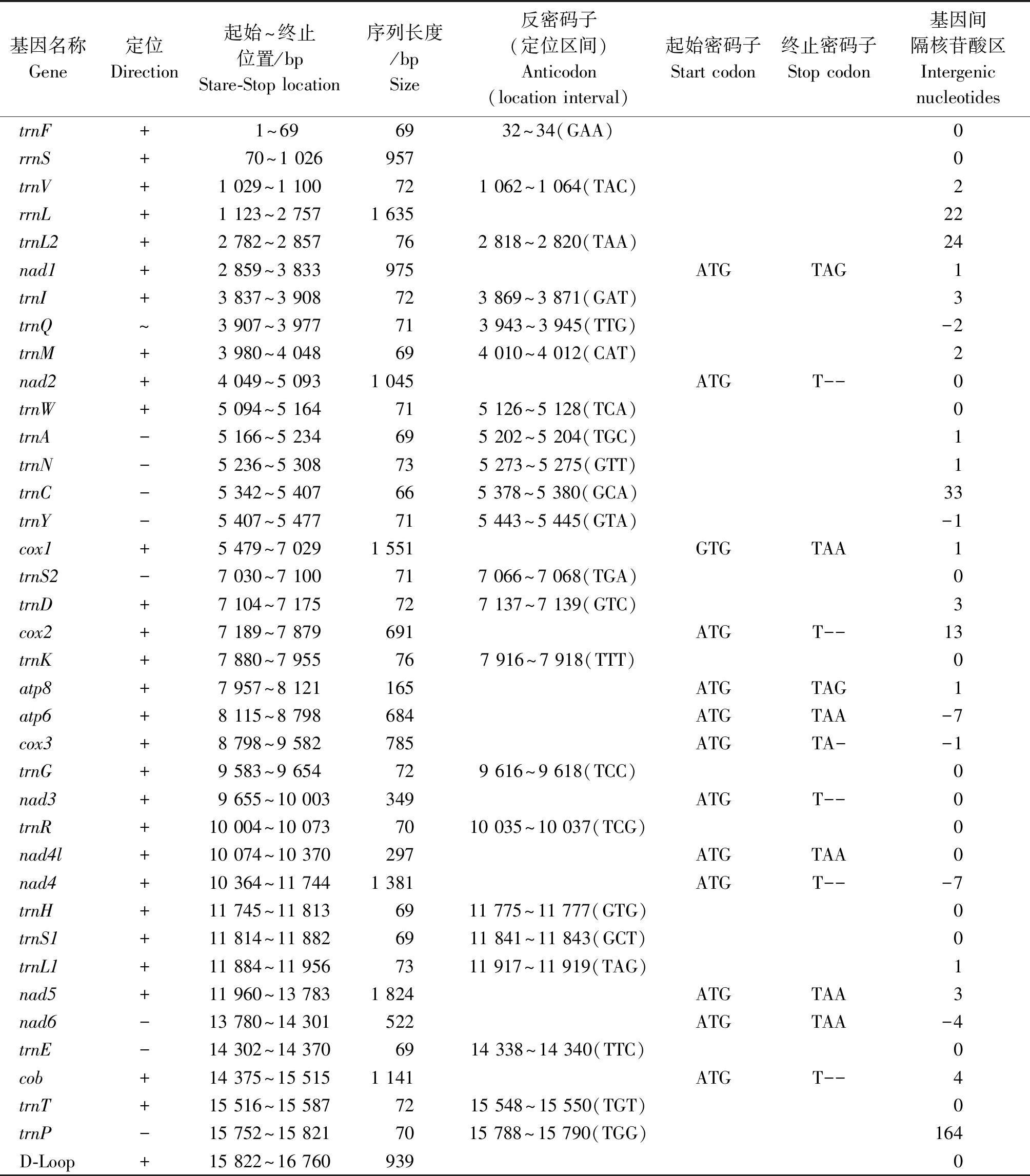
表4 花斑裸鲤线粒体基因组特征Tab.4 Mitochondrial genome characteristics of Gymnocypris eckloni
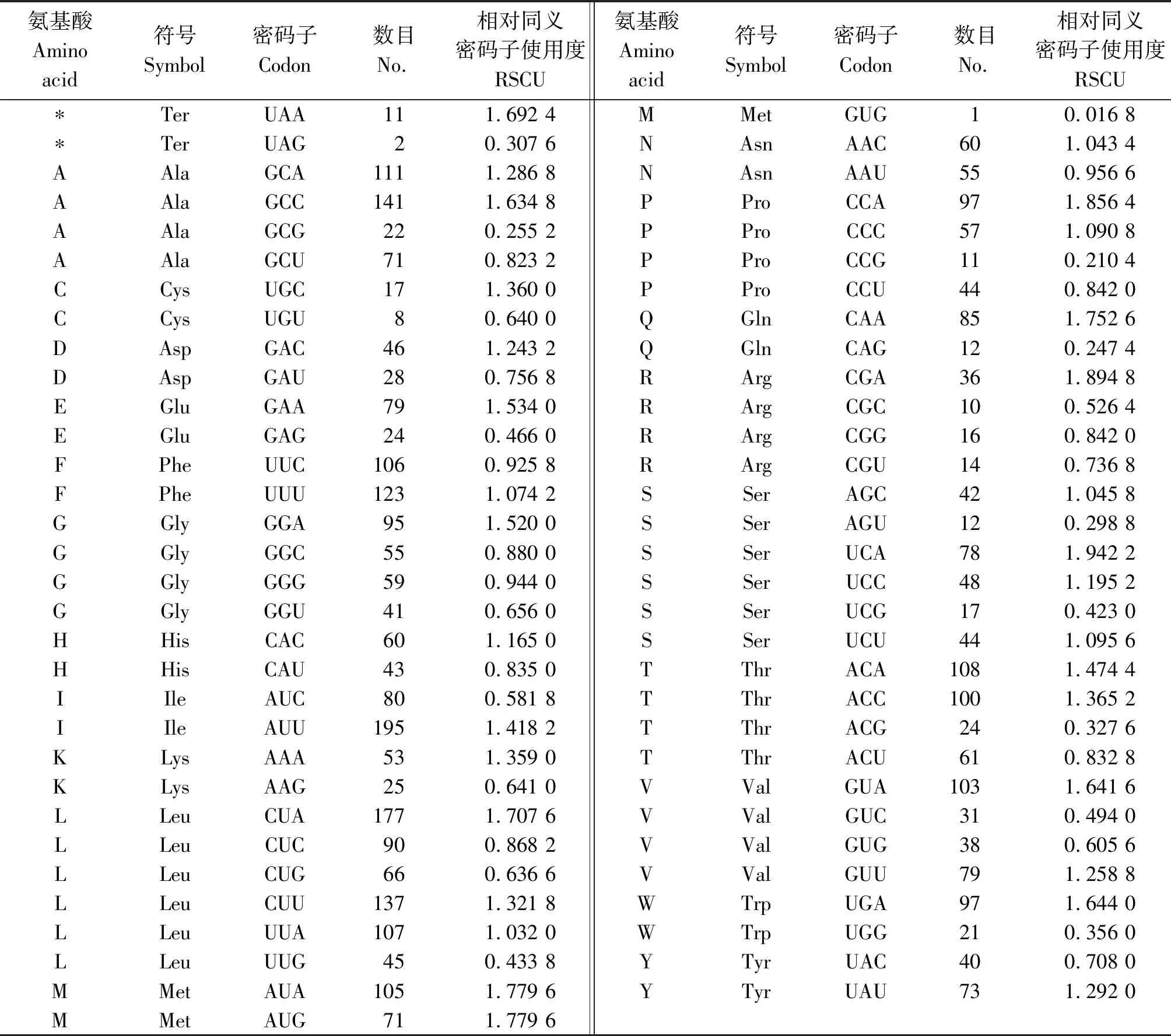
表5 青海湖裸鲤蛋白编码基因密码子的使用Tab.5 Codon usage of protein-coding gene of Gymnocypris przewalskii

表6 花斑裸鲤蛋白编码基因密码子的使用Tab.6 Codon usage of protein-coding gene of Gymnocypris eckloni
虽然基因组整体上排列紧凑,但是在青海湖裸鲤和花斑裸鲤线粒体基因组中均发现17个基因间隔区,核苷酸长度分别为239,279 bp,最长的间隔长达124,164 bp,最短的基因间隔为1 bp,存在于多个位置(表3-4)。
从青海湖裸鲤的线粒体全基因组中获得13个蛋白质编码基因,长度为11 410 bp,A+T含量占56.42%。除COX1使用GTG作为起始密码子外,其他PCGs均是以ATG作为起始密码子。这在鲤科鱼类和其他脊椎动物线粒体基因组中都很常见[22]。蛋白编码基因cox1、atp6、nad4l、nad5、nad6使用TAA作为终止密码子,nad1和atp8使用TAG作为终止密码子,cox3由不完全终止密码子TA编码,而nad2、cox2、nad3、nad4和cob由不完全终止密码子T编码(表3)。花斑裸鲤的线粒体全基因组包含13个蛋白编码基因,长度为11 410 bp,A+T含量占56.41%。除COX1使用GTG作为起始密码子外,其他PCGs均是以ATG作为起始密码子,这与青海湖裸鲤线粒体基因组特征一致。13个PCGs中,12个(nadl、nad2、coxl、cox2、atp6、atp8、cox3、nad3、nad4l、nad4、nad5、cob)位于重链上,1个(nad6)位于轻链上。蛋白编码基因cox1、atp6、nad4l、nad5、nad6使用TAA作为终止密码子,nad1和atp8使用TAG作为终止密码子,cox3由不完全终止密码子TA编码,而nad2、cox2、nad3、nad4和cob由不完全终止密码子T编码(表4)。青海湖裸鲤和花斑裸鲤蛋白编码基因相似性高达90%,说明亲缘关系最亲。
2.2 转移RNA(tRNA)和核糖体RNA(rRNA)
在蛋白质合成过程中,tRNA在适配分子中起着至关重要的作用[23]。青海湖裸鲤的tRNA长度为1 562 bp,A+T含量为54.80%,其中A为28.23%,T为26.57%,G为23.82%,C为21.38%,其AT偏度为0.030,GC偏度为0.054。其线粒体基因组rRNA长度为2 592 bp,A+T含量为53.74%,该物种的As(33.02%)高于Ts(20.72%),导致AT偏度为0.229。同样,Gs(22.34%)低于Cs(23.92%),导致GC负偏度为0.034(表1)。在青海湖裸鲤线粒体基因组中预测有22个tRNA分子,其长度约为70~80 bp,具有典型的三叶草结构(图3)。

图3 青海湖裸鲤线粒体基因组tRNA修饰示意图Fig.3 Schematic diagram of mitochondrial genome tRNA modification in Gymnocypris przewalskii
花斑裸鲤的tRNA长度为1 562 bp,A+T含量为54.67%,其中A为28.10%,T为26.57%,G为23.94%,C为21.38%,其AT偏度为0.028,GC偏度为0.056。花斑裸鲤线粒体基因组rRNA长度为2 592 bp,A+T含量为53.67%,花斑裸鲤的As(32.99%)高于Ts(20.68%),导致AT偏度为0.229,Gs(22.38%)低于Cs(23.96%),导致GC负偏度为0.034(表2)。在花斑裸鲤线粒体基因组中预测有22个tRNA分子,其长度约为70~80 bp,具有典型的三叶草结构(图4)。tRNA分子转录后加工修饰会导致其稳定性下降[24],会出现tRNA结构发生改变的情况。

图4 花斑裸鲤线粒体基因组tRNA修饰示意图Fig.4 Schematic diagram of tRNA modification in the mitochondrial genome of Gymnocypris eckloni
密码子-反密码子相互作用受修饰位置的影响较大,修饰位置通常在摆动位置附近。这一特性在真核生物中得到了很好的保存,直接影响着翻译效率、转移和维持的调控[25]。tRNA的稳定性取决于中心tRNA结构发生的变化,这可能会导致tRNA降解和差异。
2.3 控制区和密码子使用偏倚
测序发现青海湖裸鲤和花斑裸鲤线粒体基因组分别有一个控制区信息(表1-2)。D-Loop控制区是线粒体基因组中进化速率最快的区域,并且其序列变异程度也最高[26]。本研究比较分析了两者的控制区序列,结果发现,青海湖裸鲤和花斑裸鲤的控制区长度同为939 bp,青海湖裸鲤的控制区A+T含量(63.79%)低于花斑裸鲤控制区A+T含量(63.90%)。已有研究表明,线粒体DNA是研究包括硬骨鱼在内的各种鱼类的重要材料[27],它们以控制区(CR)作为研究种内变异的标志,控制区在硬骨鱼[28]、人类[29]和鸟类[30]等许多脊椎动物中都有变化。青海湖裸鲤和花斑裸鲤线粒体基因组氨基酸利用的相对同义密码子使用(Relative Synonymous Codon Usage, RSCU)情况见表5-6。
相对同义密码子使用(RSCU)分析表明,青海湖裸鲤和花斑裸鲤线粒体基因组以Ile、Leu、Ala、Phe、Met、Val和Pro氨基酸密码子出现频率最高,Glu和Cys氨基酸密码子较少。疏水氨基酸密码子在脊椎动物线粒体基因组中的使用相对高于亲水氨基酸密码子[31]。这表明接近CR的基因组区域被大量利用,表现出较高的翻译效率,可以在脊椎动物线粒体基因组中被有效翻译[32]。
本研究测定了青海湖裸鲤和花斑裸鲤的线粒体基因组全序列,其中青海湖裸鲤线粒体基因组全长为16 720 bp,包括13个蛋白编码基因,22个tRNA基因,2个rRNA基因和1个D-Loop控制区,而花斑裸鲤全长为16 760 bp,包括13个蛋白编码基因,22个tRNA基因,2个rRNA基因和1个D-Loop控制区。青海湖裸鲤和花斑裸鲤与其余裸鲤属物种线粒体基因组基因排列顺序一致[33-34]。松潘裸鲤(Gymnocyprispotanini)线粒体序列长度为16 680 bp,线粒体基因组包含13个蛋白编码基因、2个核糖体RNA、22个转运RNA和1个非编码控制[33]。兰格湖裸鲤(Gymnocyprischui)整个线粒体序列长度为16 864 bp,包含2个控制区(D-Loop区)、2个rRNA基因(12S和16S rRNA)、13个蛋白编码基因和22个tRNA基因[34]。
2.4 系统发育分析
mtDNA具有遗传速度快的特点,是很好的遗传材料,mtDNA的系统发育研究被广泛用于概述物种之间的关系[35]。然而,当研究小区域时,从mtDNA获得的数据和信息都很少,所以分类学被认为是理解生物多样性和进化行为的基础[36]。系统发育分析则用来比较和研究一个家族内部的异同点[37]。目前有3种预测系统发育的方法,即最大简约法、最大似然法和基于距离的系统发育方法[36]。在这3种方法中,最大似然(ML)方法获得了最接近的匹配,并被脊椎动物基因组分析广泛接受。
根据线粒体全基因组数据构建包含青海湖裸鲤和花斑裸鲤在内的38种鲤科鱼类的系统发育树(图5)。结果显示,青海湖裸鲤、花斑裸鲤、松潘裸鲤(Gymnocyprispotanini)、硬刺松潘裸鲤(Gymnocyprisfirmispinatus)、拉孜裸鲤(Gymnocyprisscleracanthu)、兰格湖裸鲤(Gymnocyprischui)和高原裸鲤(Gymnocypriswaddellii)为同一支,值得注意的是,裸鲤属(Gymnocypris)物种并不是聚为一支形成单系群,而是分为2支,另一支由光倒刺鲃(Spinibarbushollandi)、大理裂腹鱼(Schizothoraxtaliensis)和新疆扁吻鱼(Aspiorhynchuslaticeps)3个物种组成。裸鲤属广泛分布在高原的中心腹地地区,阿尔金山以北还有怒江水系不分布,与青海湖裸鲤同为裂腹鱼亚科的大理裂腹鱼(Schizothoraxtaliensis)有着亲缘关系[38]。代贵应和肖海[39]整理了三大类群裂腹鱼物种的地理分布,原始类群分布在海拔1 250~2 500 m的水体中,包含裂腹鱼属(Schizothorax)、扁吻鱼属(Aspiorhynchus)和裂鲤属(Schizocypris),裂腹鱼属广泛分布除青藏高原的边缘海拔较低区域(黄河以外),扁吻鱼属分布在塔里木河水系以及雅鲁藏布江中游水域,裂鲤属仅分布在国外(阿富汗、巴基斯坦河)。光倒刺鲃(Spinibarbushollandi)属鲤科,鲃亚科(Barbinae),倒刺鲃属(Spinibarbus),主要分布在长江中游的干支流中。在研究青藏高原的鱼类区系的过程中,首次在青藏高原地区柴达木盆地发现了较为完整的渐新世鲃亚科化石[40]。在青藏高原隆升的过程中,由于不适应高原寒冷气候,鲃亚科逐步退出青藏高原,而原始等级的裂腹鱼亚科、鲃亚科鱼类在那时分布到青藏高原中部地区,一部分甚至分布到目前环境条件不允许鱼类生存的地区[41]。由于海拔、气候、温度等的自然因素及人为干扰的影响,致使裸鲤属并未聚为一支形成单系群,而是分为2支,为相关系统发育分析的研究提供线索。

图5 鲤科鱼类线粒体基因组系统进化树Fig.5 Phylogenetic tree of mitochondrial genome of Cyprinid fishes
3 结论与讨论
本研究对青海湖裸鲤和花斑裸鲤线粒体基因组结构特征、碱基组成、密码子偏好性及蛋白编码基因等做了系统分析。结果表明,青海湖裸鲤的线粒体基因组全序列全长为16 720 bp,共注释37个基因(13个蛋白质编码基因,22个tRNA基因,2个rRNA基因),其碱基组成为A:28.68%、T:27.29%、G:18.16%、C:25.87%,GC含量约为44.03%,呈现出明显的AT偏好性;花斑裸鲤的线粒体基因组全序列全长为16 760 bp,共注释37个基因(13个蛋白质编码基因,22个tRNA基因,2个rRNA基因),其碱基组成为A:28.63%、T:27.22%、G:18.26%,C:25.88%,GC含量约为44.15%,具有明显的AT偏好性,这与脊椎动物线粒体基因组碱基组成相似[42]。其中,青海湖裸鲤和花斑裸鲤中的G碱基含量与其他硬骨鱼类如斑鱚(Sillagoaeolus)(18.75%)、西里伯斯青鳉(Oryziascelebensis)(17.60%)[43]等含量相似,表现出显著的抗鸟嘌呤现象[21]。在青海湖裸鲤和花斑裸鲤线粒体基因组中,13个蛋白编码基因除了nad6外,其余均在重链上。对于青海湖裸鲤和花斑裸鲤密码子偏好性分析显示,ATG密码子使用频率最高。本研究通过对青海湖裸鲤和花斑裸鲤线粒体基因组的37个基因的起止位置、长度及蛋白编码基因等特征分析显示与之前相关研究人员得出的结论一致[44-46]。
线粒体全基因组测序中mtDNA易发生突变且不易修复,并且高通量测序获得的数据间接得到mtDNA信息的方法存在一个重要问题即序列污染[47],造成生物多样性、种群遗传、物种系统进化关系和线粒体疾病的错误研究和推断。本研究对青海湖裸鲤和花斑裸鲤的整个线粒体全基因组进行测序,揭示了近缘种的遗传特性及其分化,结合形态学与生物信息学未将青海湖裸鲤与花斑裸鲤准确区分,将以其他方式对青海湖裸鲤和花斑裸鲤进行鉴定。
近年来,DNA条形码技术作为一种分子鉴定方法逐渐被发展起来[48]。在动植物的物种鉴定过程中,DNA条形码技术主要是利用一段长度约为650 bp的细胞色素C氧化酶Ⅰ(Cytochrome coxidase subunit Ⅰ,COI)基因序列对物种进行鉴定区分,逐步实现鉴定过程的自动化和标准化,从而减少物种鉴定对传统形态学分类经验方法的依赖。COI基因,作为一种重要的线粒体基因,具有母系遗传、结构简单、进化速度适中、多态性较高及易被通用引物扩增等特点[49],常用于物种鉴定、分析亲缘关系较近的种、亚种以及地理种群间的系统发育关系等研究[50]。目前,DNA条形码技术已被广泛应用于淡水鱼类[51]和海水鱼类[52]的种类识别,识别率可达90%以上[53]。此外,DNA条形码技术在发现物种、评价物种的遗传多样性等方面也显示出明显的优越性[54]。Wong等[55]利用DNA条形码技术,从北美洲东北部市场和餐厅中抽取的96种鱼和海鲜制品检测出包括大西洋鳕鱼(Gadusmorhua)、狭鳕(Theragrachalcogramma)和深水鳕鱼(Merlucciusparadoxus)3种类型的鳕鱼成分。Lakra等[56]使用COI基因对印度洋115种鱼类系统进化进行了分析,发现COI基因在NJ进化树中所形成的类群传统的分类学特征相符,且很好地揭示了该类群之间的亲缘关系。产久林等[57]利用COI及16S rRNA的基因片段,对拉萨市羊卓雍措的5种高原裂腹鱼进行了物种鉴定,结果显示,COI基因能有效鉴定高原裂腹鱼不同鱼种。Mat Jaafar等[58]对分布在印度马来群岛水域内652种假定的鲹科家族物种的723个个体进行了长度为36 bp的COI片段测序,比较物种内部和物种之间的线粒体DNACOI片段的变异性,以评估COI片段对物种鉴定的适用性,发现所有的物种在系统发育树中形成了单系簇,表明DNA条形码技术在鱼类种类识别中具有较高的应用价值。本研究基于线粒体基因组发现青海湖裸鲤和花斑裸鲤的COI基因存在碱基差异,后续将以此设计特异性引物,通过DNA条形码技术对二者进行鉴定。
本研究基于线粒体基因组的系统发育分析未将青海湖裸鲤和花斑裸鲤准确区分,但为重新建立更清晰的鲤科鱼类分类体系奠定了基础,并为进一步的分类研究提供了新的方向。
