小麦TaPSKR1基因的克隆与表达分析
2023-07-08张沛沛景凡丽马靖福杨德龙
张沛沛,陈 涛,景凡丽,,刘 媛,马靖福,田 甜,王 鹏,杨德龙,
(1.干旱生境作物学国家重点实验室,甘肃 兰州 730070;2.甘肃农业大学 生命科学技术学院,甘肃 兰州 730070)
植物磺肽素(Phytosulfokine,PSK)是一类小肽类植物内源激素,为仅有5个氨基酸组成的磺化五肽,在植物生长发育、抗逆和抗病反应等方面发挥重要作用[1-3]。已有的研究结果表明,PSK与细胞膜上的磺化肽激素受体PSKR(PSK receptor)结合来发挥功能。PSKR是一类胞外区富含亮氨酸重复序列LRRs(Luecine-rich repeat)型的跨膜类受体蛋白激酶(Receptor-like protein kinases,RLKs)。从结构上看,拟南芥PSKR由胞外的配体识别结构域、LRR结构域、跨膜结构域和胞内激酶结构域组成。PSK配体与PSKR胞外结构域结合后,激活胞内蛋白激酶域,进一步通过磷酸化下游调节因子来发挥调控作用[4]。目前已有研究表明,PSK-PSKR介导的信号转导途径在响应非生物胁迫、调控植物生长和发育等方面发挥重要作用,如调控根形态、子叶伸长、细胞增殖、体细胞胚的形成、对病原菌侵染的抗性以及花粉的萌发与受精等[5-10]。
目前,PSKR家族个别成员在拟南芥、水稻和其他物种中相继被克隆和研究。在拟南芥中PSKR包含2个旁系同源基因,分别为AtPSKR1和AtPSKR2[11]。在拟南芥中,AtPSKR1除了与共受体BAK1结合外,还可以与H+-ATPases的2个成员AHA1 和AHA2结合形成复合体,该复合体进而与环核苷酸门控离子通道(Cyclic nucleotide-gated channel 17,CNGC17)结合,从而通过细胞增殖促进了子叶和根的伸长[12]。过表达AtPSKR1的拟南芥植株还表现出更高的愈伤组织分化能力,同时植株的衰老显著延迟。在水稻中已经鉴定了15个PSKR家族成员[13],并对少数几个成员的功能进行了较深入的研究,如OsPSKR15、LRK1和LRK2基因。在水稻中,已发现OsPSKR15的激酶结构域可以磷酸化ABA受体AtPYL9和OsPYL11,从而正向调控ABA信号转导途径,过表达OsPSKR15可显著增强拟南芥植株的抗旱性[14]。之前研究人员在水稻中克隆了一个富含亮氨酸重复序列的类受体激酶(Leucine-rich repeat receptor kinases,LRKs)基因簇,该基因簇是一个与提高产量相关的QTL,包含8个LRKs基因(LRK1~LRK8)。Nagar等[13]最近报道,水稻LRK1~LRK8基因所编码的蛋白均属于PSKR家族成员。在水稻中LRK1基因的过量表达通过提高细胞的增殖能力从而提高了谷粒数和籽粒千粒质量,使水稻的单株产量增加了27.09%[15]。Kang等[16]对水稻PSKR家族成员研究发现,过表达LRK2基因可显著提高转基因植株的侧根数、分蘖数,进而提高了水稻的抗旱性。系统进化分析表明,水稻LRK2与拟南芥AtPSKR1同源性最高。迄今为止,对PSKR家族基因功能的研究主要集中在模式植物拟南芥和水稻中,关于PSKR受体激酶对小麦产量及抗逆性的影响还未进行研究。
小麦是重要的粮食作物之一,提高小麦的产量直接关系到我国粮食安全的重大问题。鉴于植物PSKR1在调控细胞增殖及提高植物抗逆性中具有重要作用。本研究对象是TaPSKRs基因家族成员中与拟南芥AtPSKR1、AtPSKR2以及水稻LRK1/2同源性最高的TaPSKR1基因。采用同源克隆的方法,从普通小麦品种晋麦47根组织中克隆TaPSKR1的3个部分同源基因的cDNA序列,命名为TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D,利用生物信息学对其基因和编码的氨基酸序列进行了分析,同时研究了TaPSKR1基因在不同组织器官及非生物胁迫下的表达模式,以期为进一步研究小麦TaPSKR1的生物学功能奠定基础。
1 材料和方法
1.1 植物材料的培养与处理
供试小麦品种为晋麦47。
选取籽粒饱满的种子经1%次氯酸钠消毒5 min,无菌水冲洗3次后,置于铺有滤纸的培养皿中黑暗培养48 h(25 ℃),待种子露白后置于光照培养箱(光暗周期16 h/8 h,温度25 ℃/20 ℃)中进行水培培养。7 d后换用1/2 Hoagland营养液继续培养,每2 d更换一次营养液,待幼苗生长至两叶一心时,分别对小麦幼苗进行20% PEG-6000和 200 mmol/L NaCl处理。胁迫处理0,3,6,12,24 h后采集对照组和处理组幼苗的叶片和根,迅速置于液氮中速冻后,-80 ℃冰箱保存备用。同时未处理的幼苗转移至4 ℃春化柜中春化30 d左右后,移载到装满营养土的花盆中,在温室中继续培养,于小麦的孕穗期采集植株的幼穗、根、茎、叶片以及灌浆期10 d的籽粒,用于后续基因的特异表达分析。
1.2 试验方法
1.2.1 总RNA的提取和cDNA合成 利用天根生化科技(北京)有限公司的RNAprep pure植物RNA提取试剂盒,提取晋麦47根、茎、叶片、幼穗和籽粒以及逆境胁迫处理后的根和叶片总RNA,使用Dnase Ⅰ去除gDNA的污染。单链cDNA的合成采用TOYOBO公司的反转录试剂盒(ReverTra Ace qPCR RT Master Mix with gDNA Remover)。
1.2.2 引物设计及TaPSKR1基因的克隆 在拟南芥(http://www.arabidopsis.org)数据库获得AtPSKR1(基因ID At2g02220)和AtPSKR2(基因ID At5g53890)的蛋白序列,同时根据Kang等[16]研究报道的OsPSKR8/LRK2(基因ID LOC_Os02g05970)基因序列在水稻数据库(http://rice.plantbiology.msu.edu/)获得其蛋白序列。利用水稻和拟南芥的PSKR基因序列在WheatOmics 1.0[17]数据库进行同源搜索,获得小麦TaPSKR1的3个部分同源基因的cDNA序列,分别定位在6A、6B和6D染色体上,基因ID分别是TraesCS6A02G124300、TraesCS6B02G152300、TraesCS6D02G114600,使用Primer Premier 5.0 设计基因特异性引物(表1)。以晋麦47根组织的cDNA为模板进行PCR扩增。PCR扩增程序为:98 ℃预变性2 min;98 ℃变性10 s,59 ℃退火30 s,72 ℃延伸3.5 min,共35个循环。PCR扩增产物经1%琼脂糖凝胶电泳检测,使用琼脂糖凝胶回收试剂盒DP219(天根,北京)进行目标产物的回收,并与克隆载体pEASY-Blunt Cloning vector(全式金,北京)连接,之后转化大肠杆菌感受态细胞DH5α,经蓝白斑筛选和菌液PCR鉴定后送至西安擎科生物技术有限公司进行测序。

表1 基因克隆和荧光定量的引物序列Tab.1 The primers for gene cloning and fluorescence quantification
1.2.3 小麦TaPSKR1基因的生物信息学分析 利用ExPASy在线工具(https://web.expasy.org/compute_pi/)计算小麦TaPSKR1蛋白的等电点(Isoelectric point,pI)和分子量(Molecular weight,MW)。采用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对TaPSKR1的启动子中的顺式作用元件进行分析,进一步使用GSDS 2.0 在线软件(http://gsds.cbi.pku.edu.cn/)对调控元件的分布进行绘制。使用DNAMAN软件对TaPSKR1的氨基酸序列进行多序列比对和保守性分析。在NCBI 网站(http:/ /www.ncbi.nlm.nih.gov/)利用BlastP程序搜索不同物种的PSKR蛋白,利用MEGA 6.0 软件中NJ(Neighbor Joining)邻接法,Bootstrap迭代数设置为1 000,构建系统进化树。利用TargetP 2.0在线软件(http://www.cbs.dtu.dk/services/SignalP/)分析蛋白质是否含有信号肽。通过在线分析软件TMHMM-2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)分析目的蛋白的跨膜结构域。利用在线工具ProtComp 9.0(http://www.softberry.com/berry.phtml?topic=protcomppl&group=programs&subgroup=proloc)对编码蛋白的亚细胞定位进行分析。利用SMART在线网站(http://smart.embl-heidelberg.de/)分析目的蛋白的保守结构域。蛋白的二级结构预测使用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行分析。使用SWISS-MODEL(http://swissmodel.expasy.org)程序对蛋白质的三级结构进行预测。利用在线数据库STRING(https://string-db.org/)预测TaPSKR1-6A的互作蛋白。
1.2.4TaPSKR1基因的表达模式分析 利用实时定量检测TaPSKR1的3个部分同源基因在不同组织和不同胁迫下的表达模式。qRT-PCR所用特异引物序列详见表1。实时荧光定量使用KOD SYBR qPCR Mix(TOYOBO,日本)试剂在 QuanStudioTM5(Foster City,CA,美国)实时荧光定量PCR仪上进行。qRT-PCR采用20 μL反应体系:2×KOD SYBR qPCR Mix 10 μL、上下游引物(10 μmol/L)各0.4 μL,cDNA模板1 μL,ddH2O 8.2 μL。扩增条件:98 ℃ 变性20 s;98 ℃ 变性10 s,60 ℃退火10 s,68 ℃延伸30 s,共40个循环。每个试验3次生物学重复,以小麦TaACTIN作为内参基因,基因表达量采用2-ΔΔCt进行计算。
2 结果与分析
2.1 TaPSKR1基因全长cDNA克隆及序列分析
以晋麦47生长至三叶期的根组织cDNA为模板,使用特异性引物对TaPSKR1进行扩增,扩增产物经1%琼脂糖凝胶电泳检测,获得3 000 bp左右的特异性条带(图1),与预期结果基本一致。测序结果发现,TaPSKR1的3条cDNA序列位于6A、6B和6D染色体上,将其分别命名为TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D,其编码序列CDS全长各自为 3 153,3 132,3 156 bp,分别编码1 050,1 043,1 051个氨基酸。利用DNAMAN对3个蛋白的氨基酸序列进行比对,结果表明,TaPSKR1-6B和TaPSKR1-6D两者同源性达到94%,3个蛋白间的同源性在91%左右,氨基酸序列较保守,都包含ATP结合位点、钙调蛋白结合位点和鸟苷酸环化酶催化中心(图2)。Protparam预测TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D的分子式分别为C5100H8171N1369O1544S43、C5064H8070N1368O1524S43和C5098H8143N1379O1539S46,相对分子量为114.75,113.88,114.85 ku,理论等电点(pI)分别为 5.91,6.08,6.09,属于弱酸性蛋白。基因结构分析表明,TaPSKR1的3个部分同源基因仅包含一个外显子。

M.D5000;1.TaPSKR1-6A;2.TaPSKR1-6B;3.TaPSKR1-6D。

红色方框的氨基酸表示可能的ATP结合位点,蓝色方框氨基酸代表推测的钙调蛋白结合位点,黑色下划线序列表示可能的鸟苷酸环化酶催化中心。The ATP binding site is shown in red box,the calmodulin binding site is framed in blue,the black underlined amino acids represent the potential guanosine cyclase catalytic center.
2.2 TaPSKR1蛋白的二级结构和三级结构预测
利用SOPMA对TaPSKR1的3个同源蛋白进行二级结构预测,结果显示,3个TaPSKR1蛋白二级结构组分相似,主要由α螺旋、β折叠、延伸链和无规则卷曲组成。其中,无规则卷曲占总蛋白44.58%~48.14%,α螺旋占35.20%~37.97%,β折叠和延伸链占少部分(表2)。由于TaPSKR1蛋白同源性较高,故利用在线软件SWISS-MOEDL对3个小麦TaPSKR1蛋白进行三级结构预测,以65%序列覆盖度的磺肽素受体蛋白(Phytosulfokine receptor)4z63.1.A为参考建立模型,结果显示,3个TaPSKR1蛋白主要由α螺旋和无规则卷曲组成,其三维结构模型相似,但是在无规则卷曲处稍有差别(图3)。

表2 TaPSKR1蛋白的二级结构预测Tab.2 Predicted secondary structure of TaPSKR1 proteins %

图3 TaPSKR1-6A(A)、TaPSKR1-6B(B)和TaPSKR1-6D(C)蛋白的三级结构预测Fig.3 Prediction of the tertiary structure of TaPSKR1-6A(A),TaPSKR1-6B(B)and TaPSKR1-6D(C)
2.3 TaPSKR1基因启动子顺式作用元件分析
利用在线数据库PlantCARE对TaPSKR1起始密码子上游2.0 kb的启动子序列的顺式作用元件进行分析,结果显示,TaPSKR1基因启动子序列包含激素响应元件、逆境胁迫相关的作用元件、光响应元件以及与生长发育相关的元件。其中激素响应元件只包含的脱落酸ABA响应元件(ABRE、ABRE4、ABRE3a、AAGAA-motif),而不含有其他激素响应元件。植物逆境有关的响应元件主要是干旱胁迫响应元件(MBS、MYC、MYB、DRE1、W-box)。此外,还有较多的光响应有关的元件(Sp1、G-box)以及参与植物生长发育的响应元件(CCGTCC-motif、CAT-box)(图4)。

图 4 TaPSKR1启动子顺式作用元件预测Fig.4 Predicted cis-acting elements in TaPSKR1 promoters
2.4 系统进化树分析
为进一步了解TaPSKR蛋白的进化关系,使用MEGA 6.0对小麦(Triticumaestivum)与乌拉尔图小麦(Triticumurartu)、拟斯贝尔托山羊草(Aegilopsspeltoides)、粗山羊草(Aegilopstauschii)、二穗短柄草(Brachypodiumdistachyon)、拟南芥(Arabidopsisthaliana)、大麦(Hordeumvulgare)、玉米(Zeamays)、高粱(Sorghumbicolor)、谷子(Setariaitalica)、油菜(Brasiccarapa)、陆地棉(Gossypiumhirsutum)、玉米(Zeamays)的PSKR氨基酸序列的系统发育进行分析。进化分析表明,TaPSKR1与乌拉尔图小麦、拟斯卑尔托山羊草、粗山羊草和水稻分为一组。TaPSKR1-6A与乌拉尔图小麦TuPSKR1-6A,TaPSKR1-6B与拟斯卑尔托山羊草AsPSKR1-6B,TaPSKR1-6D与粗山羊草AetPSKR1-6D的氨基酸序列相似度分别为100%,94%和100%(图5)。
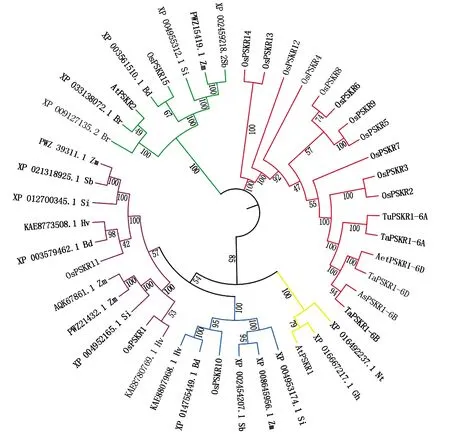
Ta.小麦;At.拟南芥;Os.水稻;Zm.玉米;Tu.乌拉尔图小麦;As.拟斯卑尔托山羊草;Aet.粗山羊草;Hv.大麦;Bd.二穗短柄草;Si.谷子;Sb.高粱;Nt.烟草;Gh.陆地棉;Br.油菜。Ta.Triticum aestivum;At.Arabidopsis thaliana;Os.Oryza sativa;Zm.Zea mays;Tu.Triticum urartu;As. Aegilops speltoides;Aet.Aegilops tauschii;Hv.Hordeum vulgare;Bd.Brachypodium distachyon;Si. Setaria italica;Sb.Sorghum bicolor;Nt.Nicotiana tabacum;Gh.Gossypium hirsutum;Br.Brasicca rapa.
2.5 信号肽、功能结构域和亚细胞定位分析
通过TMHMM-2.0在线工具(http://www.cbs.dtu.dk/services/TMHMM-2.0/)分析发现,3个基因编码的蛋白均含有一个sec信号肽(可能性是95%以上),推测小麦TaPSKR1蛋白可能是一种分泌蛋白或跨膜蛋白。利用SMART在线分析(图6),预测到3个小麦TaPSKR1蛋白均含有跨膜结构域、8个LRR结构域和一个激酶结构域。其中TaPSKR1-6A和TaPSKR1-6B含有2个跨膜结构域,TaPSKR1-6A蛋白的第13—32位、以及第684—706位含有跨膜结构域,TaPSKR1-6B蛋白的第13—35位、以及第684—706位含有跨膜结构域,而TaPSKR1-6D只含有一个跨膜结构域(685—707位)。使用ProtComp 9.0在线预测3个蛋白的亚细胞定位,结果显示TaPSKR1蛋白主要定位于细胞膜上。

图6 小麦PSKR1蛋白结构域的分布Fig.6 Domain organization of TaPSKR1 proteins
2.6 TaPSKR1 蛋白互作网络
在线软件STRING进行蛋白互作预测发现,10个蛋白与TaPSKR1-6A互作, Traes_2AS_8E399F637.1、Traes_2AS_82E3A46B8.1、Traes_2BS_AE4EF2B88.1、Traes_2BS_50B4F489A.1、Traes_2DS_1A29CC291.1和Traes_2DS_7A6AC6376.1编码丝氨酸/苏氨酸蛋白激酶;Traes_1AL_2312E9D23.1编码肌球蛋白(Myosin-17);Traes_4DS_AE1B1438E.1和Traes_4DS_AE1B1438E1.1编码人类Ski互作蛋白,与水稻OsSKIPa同源;Traes_3B_16817E311.1编码剪切因子3a亚基(图7)。

图7 TaPSKR1与其他小麦蛋白聚类互作网络图Fig.7 The protein-protein interaction network between TaPSKR1 and other wheat proteins
2.7 TaPSKR1基因特异表达分析
为进一步挖掘TaPSKR1基因的潜在功能,本研究以三叶期的根、茎、叶片和发育中期的穗和种子的cDNA为模板,利用实时荧光定量PCR分析TaPSKR1的3个部分同源基因在不同组织中的表达模式。结果表明,TaPSKR1在不同组织中表达水平存在显著差异,3个部分同源基因在小麦根、茎、叶片、穗和种子中均有表达,但是在根中的表达量显著高于其他组织,叶片中次之,在茎和穗中的表达最低。TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D在根中的表达量分别是穗的97.0,26.7,96.2倍,显示出极强的组织特异性(图8)。

不同小写字母表示0.05水平差异显著。图9同。Different lowercase letters indicate significant differences at 0.05 level.The same as Fig.9.
2.8 NaCl和PEG胁迫下TaPSKR1的表达特性
为了进一步研究TaPSKR1在非生物胁迫处理下的表达模式,利用qRT-PCR方法分析PEG和NaCl处理后小麦叶片和根中基因的表达情况。结果表明,小麦叶和根中TaPSKR1的3个部分同源基因响应2种胁迫处理,但是响应程度有所不同(图9)。在PEG胁迫处理下,叶片中TaPSKR1的3个部分同源基因在0~6 h内表达量未见显著变化,之后呈逐步上调的表达趋势,特别在胁迫处理48 h后,TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D表达急剧上调,分别是对照的34,18,23倍。而在根中,TaPSKR1的3个部分同源基因表达量受诱导程度比叶片中要弱很多。其中,TaPSKR1-6B在胁迫处理3 h后显著上调表达,为对照的2.2倍,之后又逐渐下降。TaPSKR1-6A在处理3 h基因表达量最高,之后总体呈下降趋势。TaPSKR1-6D的表达量在胁迫处理的12,48 h显著高于对照,分别是对照的1.3,1.5倍。
在NaCl胁迫条件下,叶片中TaPSKR1的3个部分同源基因随着处理时间增加表达量呈不断上升的趋势,直到12 h,TaPSKR1-6A和TaPSKR1-6B基因相对表达量已剧烈增加了约40倍。TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D均在处理48 h基因表达量最高,分别是对照的128,164,87倍。在根部,TaPSKR1的3个部分同源基因受盐胁迫诱导均上调表达,其中TaPSKR1-6A在处理6 h表达量最高,上调倍数是18倍,而TaPSKR1-6B和TaPSKR1-6D在48 h表达量达到最高,分别是未处理前的22,14倍以上。总体来看,叶片中TaPSKR1对盐胁迫的响应比根中更加剧烈。
3 结论与讨论
在植物中,PSK介导的信号转导途径主要通过细胞增殖控制器官的生长。PSK被细胞膜外的受体激酶PSKR所感知。PSKR胞外区含有数量不等的LRR结构域,属于LRR-RLK X家族[18]。目前,通过结构生物学研究发现,PSK可以与第17,18位LRR之间的岛区结合,诱导其构象发生改变产生能够与共受体SERK1、SERK2和BAK1结合的新界面并形成异源二聚体,通过LRR二聚化使胞内激酶结构域磷酸化从而激活受体PSKR[19]。PSKR不仅通过促进细胞增大和分裂调控植物生长,还参与了植物对逆境胁迫响应。本研究通过同源克隆的方法从小麦品种晋麦47中克隆了TaPSKR1的3个同源基因,将其命名为TaPSKR1-6A、TaPSKR1-6B和TaPSKR1-6D,其编码的氨基酸长度接近,理化性质相似。保守结构域分析发现,小麦TaPSKR1具有信号肽、8个LRR胞外结构域、跨膜结构域和胞内激酶结构域,属于LRR-RLK超家族。氨基酸序列比对分析发现,3个TaPSKR1蛋白N端氨基酸一致性差异较大,但是在C端都具有ATP结合位点、钙调蛋白结合位点和鸟苷酸环化酶催化中心。这些结合位点与已报道的拟南芥AtPSKR1结构域特征相一致[20-21]。研究发现,拟南芥AtPSKR1具有自我磷酸化和对下游底物的磷酸化活性[22]。此外,AtPSKR1的激酶结构域还具有鸟苷酸环化酶催化中心,可以使细胞内的GTP转化为cGMP[23]。AtPSKR1还包含钙调蛋白结合位点,该位点突变显著抑制根和地上部位的生长[4]。系统进化分析发现,TaPSKR1与水稻OsPSKRs所有家族成员聚为一类,表明它们亲缘关系相对较近。
Nagar等[13]分析了水稻OsPSKRs家族成员的组织表达模式,发现多数OsPSKRs家族成员在根中表达量较高,只有少数家族成员在叶片、穗和胚中表达量较高,猜测大多数OsPSKRs参与调节了根组织的形态建成。Kutschmar等[24]研究表明,拟南芥中AtPSKR1基因在幼苗的根和叶片中均有表达,但是在叶片中表达量较低,进一步研究发现,AtPSKR1在主根和侧根的根冠组织中高表达,在根的成熟区和伸长区表达量较低。另外,拟南芥AtPSKR2同样在根尖中高表达。在本研究中,TaPSKR1在小麦根组织中表达量极高,叶片中次之,穗中最少,说明TaPSKR1基因在调控根的生长发育过程中发挥重要作用。
受体激酶在植物对逆境适应中起着重要的调节作用。拟南芥胞质受体激酶(Receptor-like cytoplasmic kinase,RLCK)家族成员CARK1正向调控ABA信号转导,过表达该基因可显著提高拟南芥抗旱性[25]。水稻中的OsRLCK241编码LRR型RLK,在干旱和盐胁迫下强诱导表达,其过量表达的转基因水稻表现出对干旱和盐胁迫更高的耐受性[26]。Kang等[16]的研究表明,过量表达水稻OsPSKRs家族成员LRK2基因可显著提高水稻的耐旱性。OsPSKR15过表达后显著提高了水稻的抗旱性。本研究中对启动子元件分析表明,TaPSKR1启动子含有较多的干旱胁迫响应元件,预示TaPSKR1可能参与非生物胁迫的表达调控。实时定量结果显示,在干旱胁迫下,小麦根中TaPSKR1的3个部分同源基因表达小幅波动,而叶片中该基因在胁迫后期急剧上调表达,说明相较于根,叶片中的TaPSKR1基因对干旱胁迫产生更积极的响应。在盐胁迫下,TaPSKR1在根和叶片中均显著上调表达,但是上调程度和趋势有所不同。小麦根中TaPSKR1在处理6,48 h 显著高于其他时间点。与根相比,叶片中TaPSKR1在盐胁迫后期上调剧烈,其中,TaPSKR1-6A和TaPSKR1-6B表达上调至 100 倍以上,其上调幅度显著高于根组织。以上结果表明,在小麦不同组织部位,TaPSKR1对非生物胁迫表现出不同的响应程度,暗示TaPSKR1基因在干旱和盐胁迫中具有潜在应答功能。本研究结果为后续小麦TaPSKR1基因功能研究提供一定的理论基础。
