表面增强拉曼光谱法对侵袭性真菌病相关病原体的鉴别
2023-06-25王梦凡黄嘉维刘春龙
王梦凡,黄嘉维,刘春龙, 3,洪 杰,齐 崴, 4
表面增强拉曼光谱法对侵袭性真菌病相关病原体的鉴别
王梦凡1, 2,黄嘉维1,刘春龙1, 3,洪 杰1,齐 崴1, 4
(1. 天津大学化工学院,天津 300350;2. 天津大学生命科学学院,天津 300072;3. 丹娜(天津)生物科技股份有限公司,天津 300457;4. 天津化学化工协同创新中心,天津 300072)

表面增强拉曼光谱;侵袭性真菌病;主成分分析;正交偏最小二乘判别分析
侵袭性真菌病(invasive fungal disease,IFD)是危害人类生命安全的一类重要疾病,在过去的几十年中,由于放射治疗和移植病例的增加以及抗生素和抗肿瘤药物的滥用,导致免疫缺陷病例的数量急剧增加,这些病例更容易受到真菌的感染并转变为侵袭性真菌疾病(IFD)[1-2].有研究估计,侵袭性真菌病每年在全球造成150多万人死亡,这与结核病造成的死亡数相近,比疟疾高3倍[3-4].
在临床上,引起侵袭性真菌病最主要的病原体是白假丝酵母菌()、新生隐球菌()和烟曲霉菌()[5].白假丝酵母菌是一种能够在健康人体的黏膜表面和皮肤感染的病原体,当人体免疫系统缺乏相应的识别时,它会进一步侵染人体并导致严重的疾病.据调查,有40%的侵袭性真菌病死亡病例是由白假丝酵母菌造成的[2].新生隐球菌是一种机会致病菌,最初引起肺部感染之后可传播到其他器官,最为严重时会侵入中枢神经系统而导致脑膜炎.每年隐球菌影响超百万人,在拉丁美洲和撒哈拉以南的非洲有着高达70%的死亡率[6].烟曲霉菌是人曲霉菌病的主要致病种类,多年来,烟曲霉菌一般被认为是一种较弱的病原体,主要引起过敏形式的疾病.然而,近年来烟曲霉菌感染的流行率和疾病的严重程度急剧上升,这与免疫抑制患者数量的增加直接相关[7].
由于治疗方案因病原体种类而异,快速准确地识别这些IFD相关病原体对患者的诊断和后续治疗至关重要[8].目前,基于病原体培养和形态学的检测仍然是真菌病原鉴定的主要方法.非培养的方法,如G试验(检测(1-3)-β-D-葡聚糖)、GM试验(检测半乳甘露聚糖)结合酶联免疫法(ELISA)或化学发光法(CLIA)的方法越来越受到人们的关注[9].但这些方法都存在特异性低、灵敏度差以及交叉反应等问题.近年来被广泛关注的PCR技术[10]也有对引物的准确性以及严格的实验环境的要求.
表面增强拉曼光谱(surface-enhanced Raman spectroscopy,SERS)是基于金属纳米结构与入射/散射辐射场之间的共振效应,由于其有着高灵敏度、快速响应的优点,在食品控制[11-13]、环境监测[14]和化学物检测[15]等领域受到越来越多的关注.对于一种待测分子所产生的拉曼位移信号是唯一的,所以SERS技术在检测微生物表面化学指纹特征上具有独特优势.通过结合数学统计方法,SERS技术已成为多种微生物物种鉴定和表型分析的有力工具[16-20].例如Dina等[16]利用无标签SERS方法,结合模糊主成分分析、线性判别分析实现了包括烟曲霉菌、烟曲霉菌复合体和微小根毛霉3种真菌.Mabbott等[21]利用DNA功能化的银纳米粒子作为探针来检测4种真菌的核酸扩增产物,并将得到的光谱进行主成分分析,从而实现了对真菌的区分.
在本研究中,SERS技术被用于检测和鉴别白假丝酵母菌、新生隐球菌以及烟曲霉菌这3种IFD的主要病原体.笔者在真菌表面原位生长了银纳米粒子并收集其拉曼信号,从而获得了3种真菌在633nm激发波长下的拉曼光谱.随后利用主成分分析(PCA)与正交偏最小二乘法判别分析(OPLS-DA)两种数学分析方法对3种真菌的拉曼指纹谱进行分析,从而完成了对3种真菌的鉴别和区分.本研究有望为侵袭性真菌病相关病原体的检测及临床疾病诊断提供一种新思路.
1 材料与方法
1.1 试剂和仪器
硝酸银(AgNO3,99%),购自上海阿拉丁生化科技股份有限公司;盐酸羟胺(H2NOH·HCl,99%),购自天津市光复科技发展有限公司;白假丝酵母菌、新生隐球菌和烟曲霉菌,由丹娜(天津)生物技术有限公司提供.其他化学试剂均从商业渠道购买.
紫外-可见分光光度计(TU-1900),北京普析通用仪器有限责任公司;纳米粒度及ZETA电位仪(Nano ZS),英国马尔文仪器有限公司;多功能扫描探针显微镜(NTEGRA Spectra),俄罗斯NT-MDT公司;场发射透射电子显微镜(JEM-2100F),日本电子公司.
1.2 真菌培养
白假丝酵母菌和新生隐球菌的复苏是先将其从保存的斜面上划线接种至沙氏葡萄糖琼脂平板上,于30℃下生长3d.之后用无菌接种环取平板上的单菌落接种至500mL的YM肉汤培养基中,在30℃下以200r/min转速培养,直至OD600值达到0.5~0.6.烟曲霉菌的复苏是将烟曲霉冻存菌株取20μL加至沙氏葡萄糖琼脂平板上,用无菌涂布器涂抹均匀后在30℃下培养66h.之后将单个菌落接种于1000mL无菌高压灭菌的沙氏葡萄糖液体培养液中,在37℃下以200r/min转速振荡培养48h.此时培养基内有大量微小菌体产生,取100mL菌液加入1000mL灭菌沙氏葡萄糖液体培养基液中,在37℃下200r/min培养72h,直至产生真菌球.
1.3 真菌检测样品的制备
为去除菌液中培养基对实验结果的影响,先对菌液进行清洗.分别取200μL隐球菌、念珠菌的菌液和一个曲霉菌球加生理盐水至总体积为500μL,5000离心去除上清后再分别加入500μL生理盐水,5000离心去除上清,重复2次.之后分别加入200μL生理盐水重悬,振荡1min,完成对菌体的 清洗.
本研究采用原位生长的方式在菌体表面直接合成银纳米粒子.实验前预先配制所需的还原剂,还原剂由0.1mol/L的盐酸羟胺和氢氧化钠溶液按17∶33的体积比配制[22].在清洗过的菌液中加入200μL 0.01mol/L硝酸银溶液并静置1min,使银离子紧密地吸附在菌体表面.待溶液颜色变白后,各加入1.8mL还原剂并静置3min,使吸附在菌体表面的银离子充分被还原而形成银纳米粒子.
1.4 拉曼光谱检测
分别取5μL制备好的新生隐球菌和白假丝酵母菌检测样品以及一个包覆了银粒子的烟曲霉真菌球于载玻片上,干燥后对其收集拉曼光谱.拉曼光谱仪选择633nm激发波长,光谱扫描范围400~1800cm-1,积分时间为10s,积分次数为5,显微系统放大倍数为50.
1.5 数据处理
拉曼光谱数据使用OriginPro 2016软件(版本为b9.3.226)进行归一化处理.使用R(版本4.0.2)和R studio(版本1.3.1056.0)对光谱数据进行主成分分析.使用SIMCA软件(版本13.0.0.0)对数据进行正交偏最小二乘判别分析.
2 结果讨论
2.1 检测样品的表征
拉曼光谱虽然可以通过将微生物置于在激光下,收集丰富的微生物成分化学信息,但较差的信噪比仍然是统计分析的主要挑战.而SERS技术由于纳米金属的局域表面等离子体共振(local surface plasmon resonance,LSPR)效应可以产生很强的拉曼信号,因此将纳米金属附着在微生物表面,可以产生微生物表面成分的强烈信号.在此,通过原位生长将银纳米粒子包覆在真菌表面,采用透射电镜(TEM)对银纳米粒子包覆后真菌的形态进行了表征.酵母菌(白假丝酵母菌和新生隐球菌)的生物学显著特征是它能够以多种形态生长,从单细胞芽殖酵母到具有平行侧壁的假菌丝[23-24].如图1(a)和(b)所示,由于白假丝酵母菌和新生隐球菌的培养温度较低(30℃),因此呈现出出芽的形态.图中也可以看出银纳米粒子更容易在真菌的交界处形成,而银纳米粒子在白假丝酵母菌(图1(a))上的分布比在新生隐球菌(图1(b))上的更均匀,粒径更小.图1(c)为烟曲霉菌丝顶端被银纳米粒子覆盖的放大图像.此外,还发现银纳米粒子更容易集中在分生组织中,如芽胞壁或菌丝顶端,推测这可能与出芽过程中细胞壁多糖的变化有关[25].虽然银纳米粒子在单个真菌表面的分布不是很均匀,但由于真菌的大小与激光光斑的直径相差不多(约1μm),因此吸附的银纳米粒子仍然可以为SERS检测提供增强的电磁场.

图1 银纳米粒子包覆下3种真菌的TEM图
Zeta电位分析也证明了银纳米粒子附着在了真菌表面.由于真菌细胞壁上甘露糖侧链的磷酸基团以及其他脂多糖[26],真菌通常带负电荷.因此在原位生长了银纳米粒子后,这些真菌的表面电位都一定程度上被中和(图2).这也证明了银粒子成功附着在真菌表面,可以达到增强拉曼信号的效果.

图2 银纳米粒子包覆3种真菌前后的Zeta电位图
2.2 拉曼光谱分析
通过对银纳米粒子包覆的真菌进行拉曼检测,收集样品在633nm激发波长下的SERS光谱.对于每一种真菌,均制备了多组样品,共收集到20组SERS光谱数据(图3),得到的光谱数据反映了真菌细胞壁和代谢产物的组成,具有统计学分析区分不同真菌种类的潜力.之后,20组光谱经过平滑处理、基线校正和归一化,最后取平均得到该真菌的平均拉曼指纹光谱图(图4).对光谱中的一些主要特征峰进行了峰位归属(表1),从表中可以看出由于不同种类真菌的细胞壁主要组成相同,白假丝酵母菌、新生隐球菌和烟曲霉菌有一些相似的特征峰,这些特征峰分别代表着鸟嘌呤和酪氨酸(640~682cm-1)、碳水化合物中的C—C拉伸和C—N拉伸(1022~1060cm-1)、苯丙氨酸、半乳甘露聚糖和C—C键(1116~1126cm-1)、鸟嘌呤、腺嘌呤、蛋白质的酰胺Ⅲ和蛋白质中的C—H弯曲键(1315~1345cm-1)、蛋白质和脂质的CH2弯曲键(1440~1475cm-1)、腺嘌呤、鸟嘌呤和苯丙氨酸的C=C弯曲键(1570~1595cm-1)、酰胺Ⅰ和C=C键(1612~1680cm-1).

图3 3种真菌的拉曼光谱图

图4 3种真菌的平均拉曼指纹光谱图
Fig.4 Averaged SERS fingerprint spectra of three fungi
表1 真菌拉曼光谱峰位归属
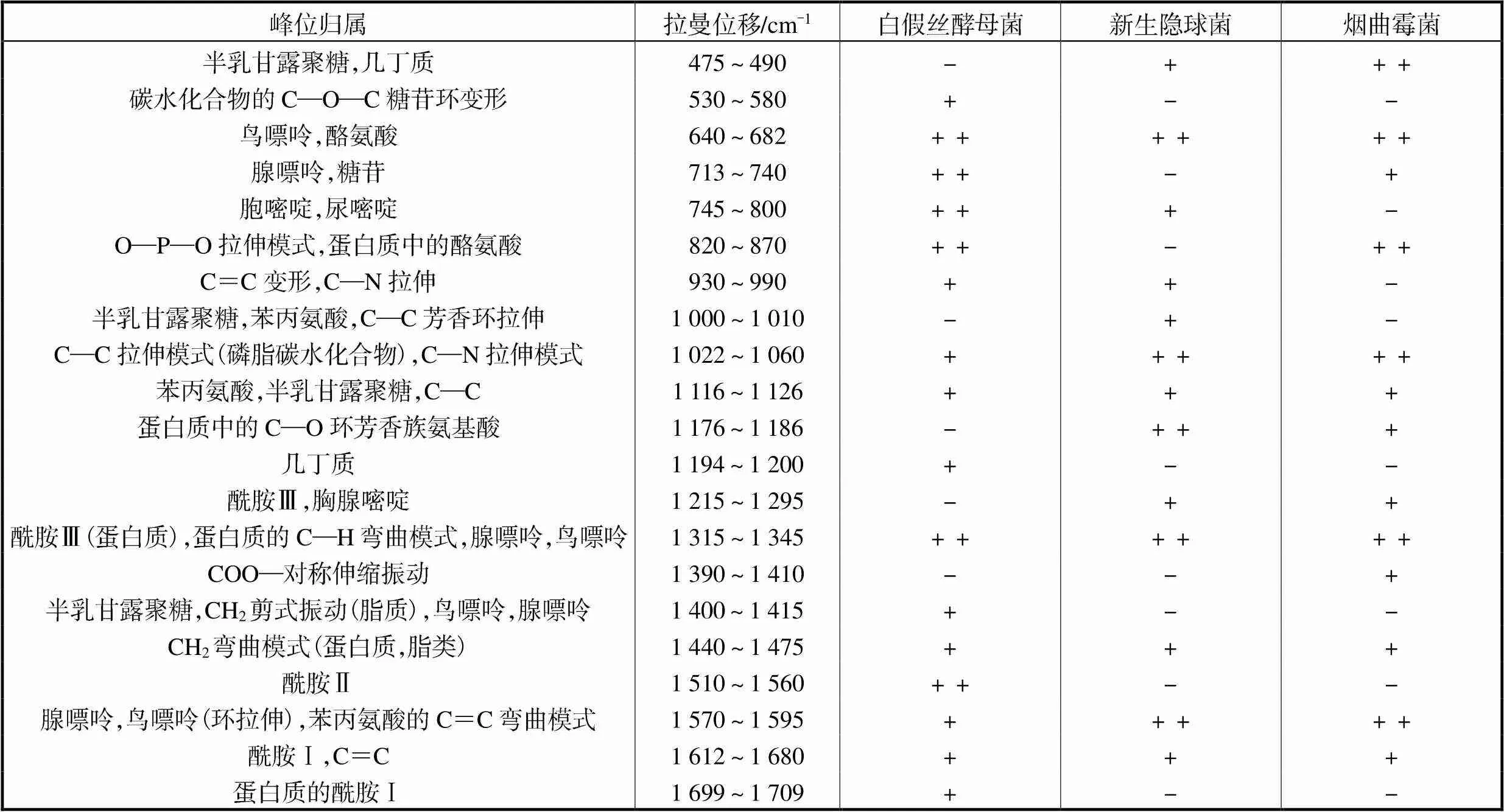
Tab.1 Tentative band assignments of the Raman spectra of fungi
注:++表示特征峰信号强度大;+表示出现特征峰;-表示无特征峰;所有峰位归属信息均来自文献[11, 16-17, 27-38].
此外,每种真菌也有其独特的拉曼峰,说明这些真菌之间存在差异.白假丝酵母菌特有的拉曼特征峰分别代表了碳水化合物的C—O—C糖苷环变形(536cm-1)、几丁质(1194cm-1)、鸟嘌呤、腺嘌呤、半乳甘露聚糖和脂质的CH2剪式振动(1413cm-1)、蛋白质的酰胺Ⅱ(1539cm-1)和酰胺Ⅰ(1699cm-1).新生隐球菌在1002cm-1处有着其特有的拉曼特征峰,归属于半乳甘露聚糖、苯丙氨酸和C—C芳香环拉伸.烟曲霉菌特有的特征峰分别归属于几丁质和半乳甘露聚糖(492cm-1)、COO—对称拉伸(1408cm-1)、腺嘌呤、鸟嘌呤(环拉伸)和C=C弯曲模式(1590cm-1).每种真菌病原体都有其特定的指纹图谱,收集的光谱中所有的特征峰及其分配列于表1中.
2.3 光谱数据的主成分分析
主成分分析是一种主要用于描述不同数据之间的微小差异的数理统计方法.主成分分析可以降低多变量数据的维数,使大量的变量可以转换成少量的新变量,而这些新变量就被称为主成分.各主成分用方差来表示包含的原始数据的信息,方差数值越大表示主成分包含的原始数据的信息越多,一般来说主成分累计方差贡献率大于80%时可以认为包含了原始数据的大部分信息.为了避免光谱采集时强度差异引起的干扰,这里对采集的原始光谱进行了归一化处理,之后将3种真菌的60组SERS光谱进行主成分分析.图5(a)为分析后的主成分得分图,图中可以看出主成分分析的前2个主成分(PC1和PC2)的方差累计贡献率达到了91.7%,不同种的真菌被区分在了3个不同的区域,不同种真菌的分值点周围有一个95%置信椭圆,大部分的数据点都落在了置信椭圆中.由此可以表明第一、第二主成分包含了原始光谱数据的大部分信息,并且利用主成分分析对3种隐球菌的区分效果较好且可信度高.
图5(b)为第一主成分的载荷图,载荷图显示了影响第一主成分的最重要的变量.如图所示,拉曼位移处于667cm-1、731cm-1、792cm-1、853cm-1、1328cm-1、1538cm-1和1584cm-1的光谱信息对第一主成分在负方向有着重要的贡献,而拉曼位移处于581cm-1、675cm-1、780cm-1、1326cm-1、1457cm-1和1547cm-1的光谱信息对第二主成分有着重要的贡献(图5(c)).处于这些拉曼位移处的特征峰正是图4中显示的3种真菌最有差异的特征峰,这也表明主成分分析可以通过降维的方法排除光谱中大量信息的干扰,准确识别出3种真菌指纹光谱的细微差别并完成区分.
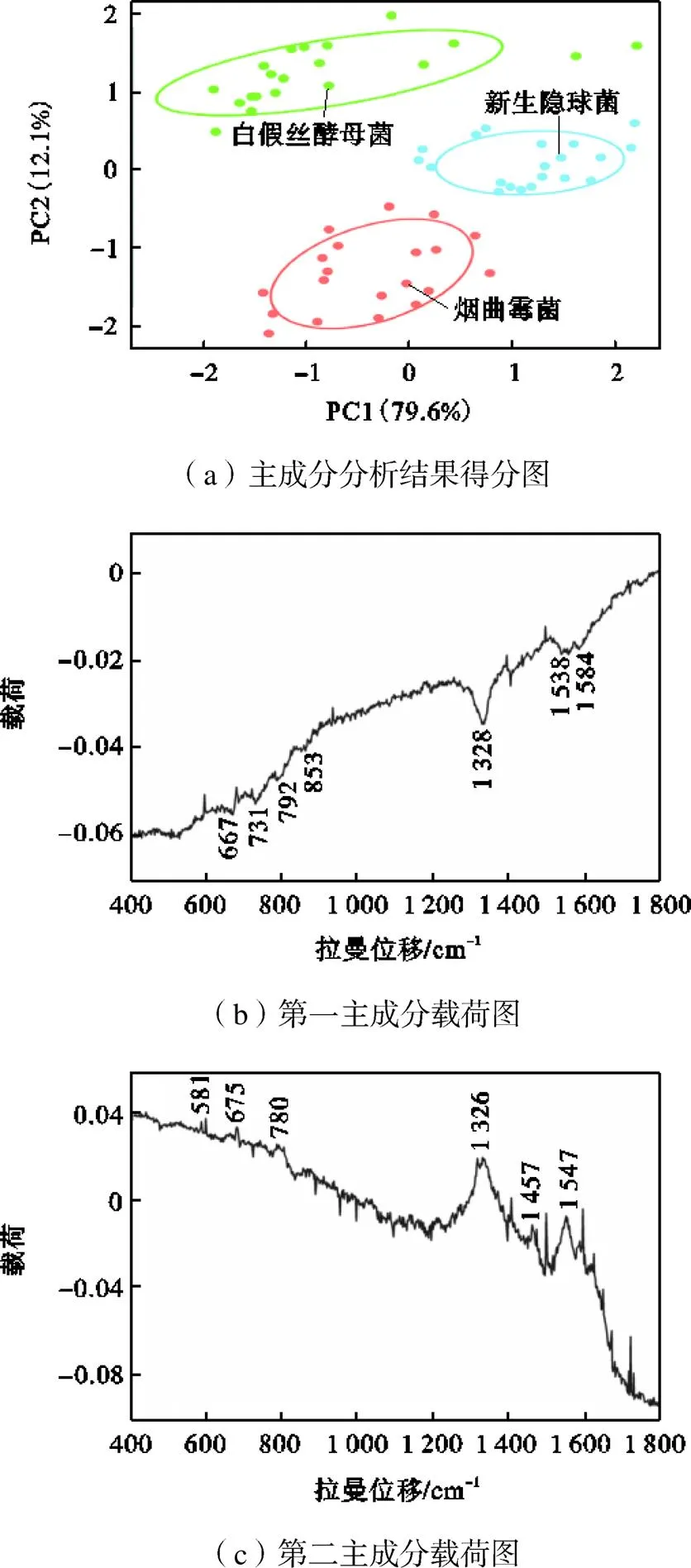
图5 主成分分析结果得分图与第一主成分、第二主成分的载荷图
2.4 正交偏最小二乘法处理光谱数据



图6 3种真菌SERS光谱的OPLS-DA得分图
表2 OPLS-DA法的模型参数

Tab.2 Parameters of OPLS-DA
注:模型1用于鉴别白假丝酵母菌和烟曲霉菌;模型2用于鉴别白假丝酵母菌和新生隐球菌;模型3用于鉴别新生隐球菌和烟曲霉菌.
3 结 语
近年来,侵袭性真菌感染对人类健康造成的危害越来越受到重视,因此对相关病原体的检测和鉴别提出了更高的需求,SERS光谱分析具有非标记的指纹特征优势,在微生物的高效检测与鉴别方面展示出良好的应用潜力.真菌作为一种复杂的微生物,其拉曼光谱提供的信息量大且复杂,本研究通过在白假丝酵母菌、新生隐球菌和烟曲霉菌表面原位生长银纳米粒子,获得了这3种侵袭性真菌的拉曼指纹光谱.根据指纹谱中特征峰的峰位归属分析了3种真菌表面的主要化学成分及其异同;随后分别利用主成分分析和正交偏最小二乘判别分析对3种真菌的SERS光谱进行统计分析,实现了对3种真菌的区分.该工作不仅为侵袭性真菌病相关病原体的鉴别、鉴定、耐药性研究以及疾病的临床诊断提供了新方法,同时也为真菌“拉曼组学”的研究提供了原始数据.
[1] Taylor D L,Hollingsworth T N,McFarland J W,et al. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning[J]. Ecological Monographs,2014,84(1):3-20.
[2] Pfaller M A,Diekema D J. Epidemiology of invasive mycoses in North America[J]. Crit Rev Microbiol,2010,36(1):1-53.
[3] Firacative C. Invasive fungal disease in humans:Are we aware of the real impact?[J]. Memórias do Instituto Oswaldo Cruz,2020,115:e200430.
[4] Bongomin F,Gago S,Oladele R O,et al. Global and multi-national prevalence of fungal diseases-estimate precision[J]. Journal of Fungi,2017,3(4):57.
[5] Robbins N,Wright G D,Cowen L E,Antifungal drugs:The current armamentarium and development of new agents[J]. Microbiology Spectrum,2016,4(5):FUNK-0002-2016.
[6] Brown G D,Denning D W,Gow N A,et al. Hidden killers:Human fungal infections[J]. Sci Transl Med,2012,4(165):165rv13.
[7] Latge J P. Aspergillus fumigatus and aspergillosis[J]. Clinical Microbiology Reviews,1999,12(2):310-350.
[8] Taccone F S,van den Abeele A M,Bulpa P,et al. Epidemiology of invasive aspergillosis in critically ill patients:Clinical presentation,underlying conditions,and outcomes[J]. Critical Care,2015,19:7-15.
[9] Ivanova A E,Shutova A S,Gannesen A V,et al. Determination of the mycelium and antigens of a number of micromycetes in soil extracts via enzyme-linked immunosorbent assay[J]. Applied Biochemistry and Microbiology,2020,56(1):72-77.
[10] Arya M,Shergill I S,Williamson M,et al. Basic principles of real-time quantitative pcr[J]. Expert Review of Molecular Diagnostics,2005,5(2):209-219.
[11] Huayhongthong S,Khuntayaporn P,Thirapanmethee K,et al. Raman spectroscopic analysis of food-borne microorganisms[J]. LWT,2019,114:108419.
[12] Fu F Y,Yang B B,Hu X M,et al. Biomimetic synthesis of 3D Au-decorated chitosan nanocomposite for sensitive and reliable sers detection[J]. Chemical Engineering Journal,2020,392:123693.
[13] Zhang H,Cui Q Q,Xu L L,et al. Blue laser-induced photochemical synthesis of CuAg nanoalloys on h-BN supports with enhanced sers activity for trace-detection of residual pesticides on tomatoes[J]. Journal of Alloys and Compounds,2020,825:153996.
[14] Hao B Q,Bu X F,Wu J W,et al. Determination of Hg2+in water based on acriflavine functionalized AgNPs by SERS[J]. Microchemical Journal,2020,155:104736.
[15] Fu Z W,Shen Z D,Fan Q Z,et al. Preparation of multi-functional magnetic-plasmonic nanocomposite for adsorption and detection of thiram using sers[J]. Journal of Hazardous Materials,2020,392:122356.
[16] Dina N E,Gherman A M R,Chis V,et al. Charac-terization of clinically relevant fungi via sers fingerprint-ing assisted by novel chemometric models[J]. Anal Chem,2018,90(4):2484-2492.
[17] Witkowska E,Jagielski T,Kaminska A. Genus- and species-level identification of dermatophyte fungi by surface-enhanced Raman spectroscopy[J]. Spectro-chimica Acta Part A:Molecular and Biomolecular Spectroscopy,2018,192:285-290.
[18] Zhou H B,Yang D T,Ivleva N P,et al. Label-free in situ discrimination of live and dead bacteria by surface-enhanced Raman scattering[J]. Analytical Chemistry,2015,87(13):6553-6561.
[19] Hu S,Gu F,Chen M,et al. A novel method for identifying and distinguishing cryptococcus neoformans and cryptococcus gattii by surface-enhanced Raman scattering using positively charged silver nanoparticles [J]. Sci Rep,2020,10(1):12480.
[20] Cui L,Zhang D,Yang K,et al. Perspective on surface-enhanced Raman spectroscopic investigation of microbial world[J]. Anal Chem,2019,91(24):15345-15354.
[21] Mabbott S,Thompson D,Sirimuthu N,et al. From synthetic DNA to PCR product:Detection of fungal infections using sers[J]. Faraday Discussions,2016,187:461-472.
[22] Zhou H,Yang D,Ivleva N P,et al. SERS detection of bacteria in water by in situ coating with Ag nanoparticles[J]. Analytical Chemistry,2014,86(3):1525-1533.
[23] Sudbery P,Gow N,Berman J. The distinct morphogenic states of candida albicans[J]. Trends in Microbiology,2004,12(7):317-324.
[24] Edwards M R,Gordon M A,Lapa E W,et al. Micromorphology of cryptococcus neoformans[J]. Journal of Bacteriology,1967,94(3):766-777.
[25] Reid I D,Bartnicki-Garcia S. Cell-wall composition and structure of yeast cells and conjugation tubes of tremella mesenterica[J]. Journal of General Microbiology,1976,96(1):35-50.
[26] Lipke P N,Ovalle R. Cell wall architecture in yeast:New structure and new challenges[J]. Journal of Bacteriology,1998,180(15):3735-3740.
[27] Witkowska E,Jagielski T,Kamińska A,et al. Detection and identification of human fungal pathogens using surface-enhanced Raman spectroscopy and principal component analysis[J]. Analytical Methods,2016,8(48):8427-8434.
[28] Lemma T,Saliniemi A,Hynninen V,et al. SERS detection of cell surface and intracellular components of microorganisms using nano-aggregated Ag substrate[J]. Vibrational Spectroscopy,2016,83:36-45.
[29] Liu S,Li H,Hassan M M,et al. Amplification of Raman spectra by gold nanorods combined with chemometrics for rapid classification of four pseudomonas[J]. Int J Food Microbiol,2019,304:58-67.
[30] Lemma T,Wang J,Arstila K,et al. Identifying yeasts using surface enhanced Raman spectroscopy[J]. Spectrochimica Acta Part A:Molecular and Biomolecu-lar Spectroscopy,2019,218:299-307.
[31] Schuster K C,Urlaub E,Gapes J R. Single-cell analysis of bacteria by Raman microscopy:Spectral information on the chemical composition of cells and on the heterogeneity in a culture[J]. Journal of Microbi-ological Methods,2000,42(1):29-38.
[32] Ivleva N P,Wagner M,Szkola A,et al. Label-free in situ SERS imaging of biofilms[J]. Journal of Physical Chemistry B,2010,114(31):10184-10194.
[33] Sayin I,Kahraman M,Sahin F,et al. Characterization of yeast species using surface-enhanced Raman scattering[J]. Applied Spectroscopy,2009,63(11):1276-1282.
[34] Venkatakrishna K,Kurien J,Pai K M,et al. Optical pathology of oral tissue:A Raman spectroscopy diag-nostic method[J]. Current Science,2001,80(5):665-669.
[35] Walter A,Marz A,Schumacher W,et al. Towards a fast,high specific and reliable discrimination of bacteria on strain level by means of SERS in a microfluidic device[J]. Lab Chip,2011,11(6):1013-1021.
[36] Kalachyova Y,Erzina M,Postnikov P,et al. Flexible SERS substrate for portable Raman analysis of biosamples[J]. Applied Surface Science,2018,458:95-99.
[37] De Gelder J,de Gussem K,Vandenabeele P,et al. Reference database of Raman spectra of biological molecules[J]. Journal of Raman Spectroscopy,2007,38(9):1133-1147.
[38] Szeghalmi A,Kaminskyj S,Rosch P,et al. Time fluctuations and imaging in the SERS spectra of fungal hypha grown on nanostructured substrates[J]. Journal of Physical Chemistry B,2007,111(44):12916-12924.
Identification of Invasive Fungal DiseaseRelated Pathogens Using Surface-Enhanced Raman Spectroscopy
Wang Mengfan1, 2,Huang Jiawei1,Liu Chunlong1, 3,Hong Jie1,Qi Wei1, 4
(1. School of Chemical Engineering and Technology,Tianjin University,Tianjin 300350,China;2. School of Life Sciences,Tianjin University,Tianjin 300072,China;3. Dynamiker Biotechnology(Tianjin)Co.,Ltd.,Tianjin 300457,China;4. Collaborative Innovation Center of Chemical Science and Engineering(Tianjin),Tianjin 300072,China)

surface-enhanced Raman spectroscopy;invasive fungal disease;principal components analysis (PCA);orthogonal partial least-squares discriminant analysis(OPLS-DA)
10.11784/tdxbz202205011
Q5-33
A
0493-2137(2023)09-0927-08
2022-05-08;
2022-06-24.
王梦凡(1983— ),女,博士,教授.Email:m_bigm@tju.edu.cn
王梦凡,mwang@tju.edu.cn.
国家自然科学基金资助项目(21621004,22178260);天津市科学技术局重大专项资助项目(20ZXGBSY00040).
the National Natural Science Foundation of China(No. 21621004,No. 22178260),the Major Project of Tianjin Science and Technology Bureau(No. 20ZXGBSY00040).
(责任编辑:田 军)
