原发性干燥综合征中铁死亡相关生物标志物的生物信息学挖掘与诊断模型构建
2023-06-19燕红梅吴舒婷巫燕琴董光富
燕红梅, 吴舒婷, 巫燕琴, 董光富
(1. 华南理工大学 医学院, 广东 广州, 510006;2. 南方医科大学附属广东省人民医院/广东省医学科学院 风湿免疫科, 广东 广州, 510080;3. 汕头大学 医学院, 广东 汕头, 515041; 4. 南方医科大学, 广东 广州, 510515)
原发性干燥综合征(pSS)是一种慢性自身免疫性疾病,主要临床表现为口干和眼干,可伴发全身性损害[1]。目前, pSS的确切病因尚不明确,且症状特异性差,易误诊和延迟诊断,导致患者预后不佳。铁死亡是一种新的程序性细胞死亡方式,在多种自身免疫性疾病的发病机制中起着关键作用。铁死亡的特征是抗氧化剂谷胱甘肽消耗和谷胱甘肽过氧化物酶4水平降低,导致脂质过氧化物积累,从而引起广泛的组织细胞死亡。异常死亡细胞释放的组分具有潜在的免疫刺激效应,并可引发自身免疫反应[2]。研究[3-4]表明,铁死亡参与了包括类风湿性关节炎(RA)和系统性红斑狼疮(SLE)在内的多种自身免疫性疾病的发生和发展。pSS作为自身免疫性疾病,其特点之一是持续的慢性炎症,研究[5]表明, EB病毒感染等多种感染参与了pSS的发病和进展。铁死亡是一种特殊类型的细胞死亡方式,可将慢性炎症与氧化应激联系起来[6]。当细胞被感染时,铁死亡可以加剧炎症反应,从而引起器官损害[7]。此外,氧化应激也是铁死亡的主要特征之一,故铁死亡可能在pSS的发病中扮演重要角色。本研究基于多种生物信息学手段识别与pSS相关的铁死亡相关基因,通过机器学习算法去除冗余基因并构建诊断模型,分析铁死亡相关基因在pSS发病过程中的可能作用,以期为pSS的诊断和治疗提供新的视角。
1 资料与方法
1.1 数据来源
本研究从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中下载包含外周血单个核细胞(PBMC)样本的5个RNAseq数据集进行分析,包括GSE51092(n=222)、GSE66795(n=164)、GSE84844(n=60)、GSE48378(n=27)和GSE208260(n=57)数据集,其中前2个为训练集,后3个为验证集。另从GEO数据库下载1个单细胞数据集GSE157278, 包括5个来自pSS患者的样本和5个来自健康供体的正常样本。
1.2 铁死亡相关基因收集
FerrDb是用于管理和鉴定铁死亡相关标志物、调控因子以及铁死亡关联疾病的数据库,本研究从FerrDb数据库中获取铁死亡相关基因集,去除重复基因后,最终获得484个基因。
1.3 数据处理和差异表达基因(DEGs)筛选
应用R软件Combat包去除GSE51092数据集和GSE66795数据集的批次效应后进行合并,通过R软件limma包进行差异分析,将调整后P值(P.adj)<0.05且|差异倍数(FC)|>1.3的基因作为DEGs。
1.4 基因本体论(GO)和京都基因与基因组百科全书(KEGG)富集分析
应用R软件clusterProfiler包和org.Hs.eg.db包对pSS相关DEGs进行GO和KEGG富集分析,并应用ggplot2包将数据可视化。
1.5 加权基因共表达网络分析(WGCNA)
基于R软件WGCNA包构建pSS患者的加权基因共表达网络,首先剔除离群的基因和样本,确定软阈值为4, 并构建无尺度网络和拓扑重叠矩阵,然后将表达高度相关的基因划分至同一基因模块中,最后根据不同模块与pSS的关联性筛选出相关性最高的基因模块。
1.6 诊断模型的构建和验证
机器学习是人工智能的分支,本研究基于分类算法和回归分析算法构建预测模型。最小绝对值收敛和选择算子(LASSO)是一种正则化模型,通常与线性分类器算法支持向量机(SVM)同时使用。LASSO通过对高维数据集中无关变量的系数进行惩罚或缩减,最终获得1个变量较少的模型,具有从高度相关的变量中选择相关特征的能力[8]。SVM是一种监督学习算法,通过建立2个类别之间的决策边界,可从1个或多个特征向量中获得预测标签,结合多个机器学习方法能进一步提高模型的预测能力。
从DEGs、铁死亡相关基因和WGCNA红色模块中筛选出关键基因,通过LASSO和SVM-递归特征消除(RFE)这2种机器学习算法去除冗余基因。绘制受试者工作特征(ROC)曲线,评估关键基因的诊断效能,将曲线下面积(AUC)大于0.8的关键基因重新输入LASSO模型中构建最终的诊断模型。将GSE84844、GSE48378、GSE208260数据集作为验证队列,基于ROC曲线评估诊断模型的诊断性能。
1.7 免疫浸润分析
采用CIBERSORT算法,估计22种不同免疫细胞亚型的浸润情况。从CIBERSORT网站(http://cibersort.stanford.edu/)获取LM22文件,通过R软件对结果进行可视化处理,应用ggcorrplot包可视化表示关键基因与免疫细胞的相关性。
1.8 单细胞RNA测序的质控、降维和注释
使用R软件Seurat包进行单细胞测序数据分析,过滤表达少于3个基因或少于200个基因的细胞,并将表达超过2 500个基因或线粒体基因超过10%的细胞排除。通过主成分分析(PCA)对单细胞测序数据进行降维处理,应用harmony软件包合并单细胞样本,通过FindNeighbors和FindClusters(Dim=30, Resolution=1.9)进行细胞聚类,然后用统一流形逼近和投影(UMAP)图展示聚类结果。应用FindAllMarkers函数确定每个聚类的标记基因。将主成分进行聚类和UMAP表示,最终确定聚类。基于既往研究报道的细胞标记基因,对每个聚类进行手动注释,最终注释出细胞类型。
1.9 不同PBMC细胞类型中关键基因的表达情况
通过ggplot2和ggalluvial包绘制每个样本中不同细胞类型的百分比,通过plot1cell包展示PBMC中各种细胞类型关键基因的表达情况。
1.10 统计学分析
应用R软件(版本4.2.1)进行统计学分析,采用Wilcoxon秩和检验或学生t检验分析2组间的差异,采用Spearman秩相关检验确定变量之间的相关性。所有统计检验采用双侧检验,P<0.05为差异有统计学意义。
2 结 果
2.1 pSS中DEGs的鉴定及其GO和KEGG富集分析
本研究将2个大样本数据集(GSE51092、GSE66795数据集)作为训练集,应用R软件Combat包消除两者的批次效应后进行合并。去除批次效应后, UMAP图显示2个数据集的样本交织在一起,提示批次效应已成功消除,见图1A、图1B。合并后的数据集中共鉴定出265个DEGs(P.adj<0.05且|FC|>1.3), 其中179个上调, 86个下调。
应用R软件对265个DEGs进行GO和KEGG富集分析,结果见图1C、图1D。GO富集分析包括生物学过程(BP)、细胞成分(CC)和分子功能(MF), DEGs的BP包括防御病毒反应、防御共生反应、对病毒的反应等, CC主要富集于ISGF3复合物、含有胶原的细胞外基质和TAP复合物等, MF主要包括双链R结合、D+蛋白质ADP核糖转移酶活性、MHC I类b蛋白质结合和D+ ADP核糖转移酶活性; KEGG富集分析显示, DEGs参与多种信号通路,包括流感A病毒、丙型肝炎病毒、Epstein-Barr(EB)病毒感染等。
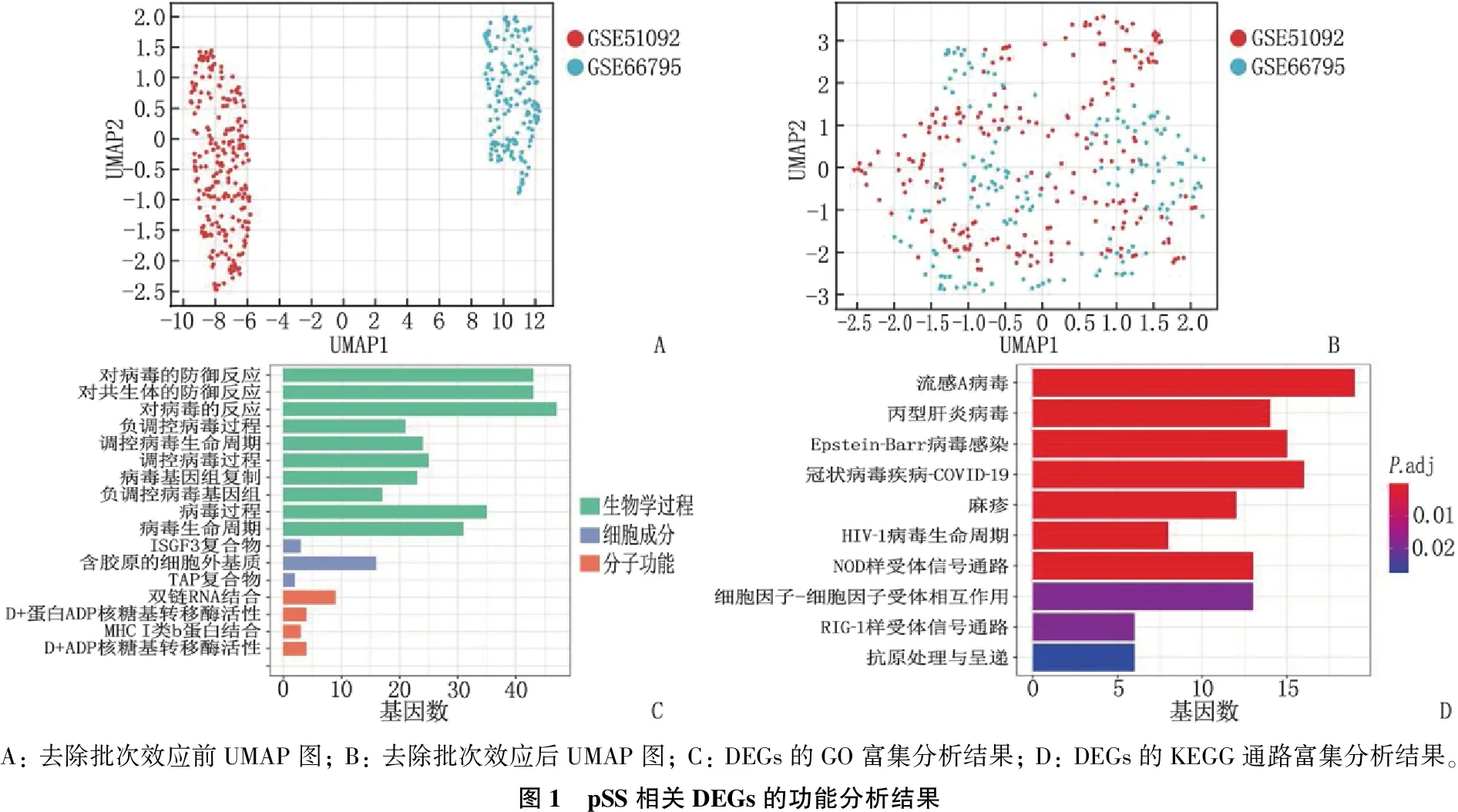
A: 去除批次效应前UMAP图; B: 去除批次效应后UMAP图; C: DEGs的GO富集分析结果; D: DEGs的KEGG通路富集分析结果。图1 pSS相关DEGs的功能分析结果
2.2 WGCNA鉴定与pSS相关的模块
当软阈值确定为β=4,R2=0.86, 网络符合无标度网络的分布,见图2A、图2B。应用混合动态剪切树法识别相似的模块并进行合并,最终获得27个基因模块,见图2C。模块-临床特征相关性分析结果显示,红色模块与pSS的相关性最高(r=0.44,P<0.01), 见图2D。红色模块内基因成员与疾病状态高度相关(r=0.82), 见图2E。将红色模块基因、铁死亡相关基因、DEGs取交集,共得到8个关键基因,见图2F。

A、B: 基于比例独立性、平均连接分析选择β=4作为软阈值构建网络; C: 基因树显示不同基因共表达模块(各模块以不同的唯一颜色表示);D: 热图显示基因模块与pSS的相关性(红色模块与pSS显著相关); E: 红色模块内基因成员身份与基因重要性的相关性分析散点图; F: 红色模块基因、铁死亡相关基因、DEGs取交集的韦恩图。图2 基于WGCNA鉴定与pSS相关的模块
2.3 与铁死亡相关的关键基因的鉴定及pSS诊断模型建立
为了去除冗余基因,应用LASSO模型筛选出4个与铁死亡相关的诊断pSS的关键基因,见图3A; 为了简化诊断模型并提高诊断模型的准确性,基于SVM-RFE算法对取交集得到的8个关键基因进行二次筛选,见图3B。2种机器学习方法筛选出4个共同的基因,即TRIM21、PARP9、PARP12和PARP14。绘制ROC曲线评估这4个基因的诊断价值(图3C~图3F), 将AUC>0.8的关键基因PARP9、PARP12、PARP14作为pSS的生物标志物。将3个关键基因输入LASSO模型中,得到最终模型方程,即风险评分=-0.523 290 132 2+0.000 010 735 4×PARP12+0.115 903 498 1×PARP9+0.027 443 175 8×PARP14。
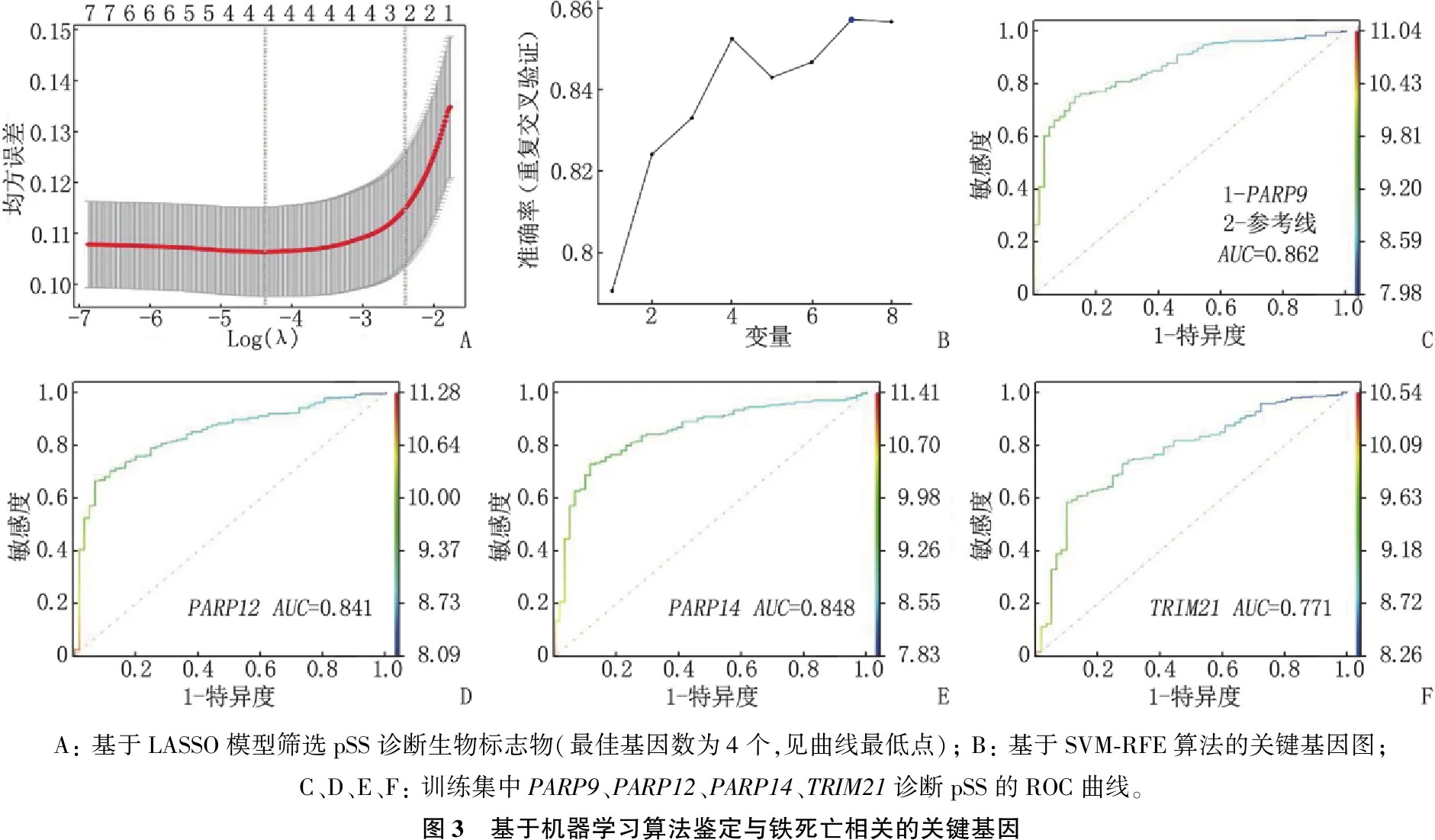
A: 基于LASSO模型筛选pSS诊断生物标志物(最佳基因数为4个,见曲线最低点); B: 基于SVM-RFE算法的关键基因图; C、D、E、F: 训练集中PARP9、PARP12、PARP14、TRIM21诊断pSS的ROC曲线。图3 基于机器学习算法鉴定与铁死亡相关的关键基因
2.4 诊断模型的验证结果
为了验证诊断模型的准确性,本研究将GSE84844、GSE208260、GSE48378数据集作为验证集。3个验证集中, 3个关键基因的表达情况见图4A~图4C; 3个关键基因在pSS患者(pSS组)和健康个体(对照组)中的表达水平存在差异,见图4D~图4F。ROC曲线显示,诊断模型在GSE84844、GSE208260、GSE48378数据集中的AUC分别为0.848、0.853、1.000, 见图4G~图4I, 表明诊断模型具有区分pSS患者和健康个体的能力。
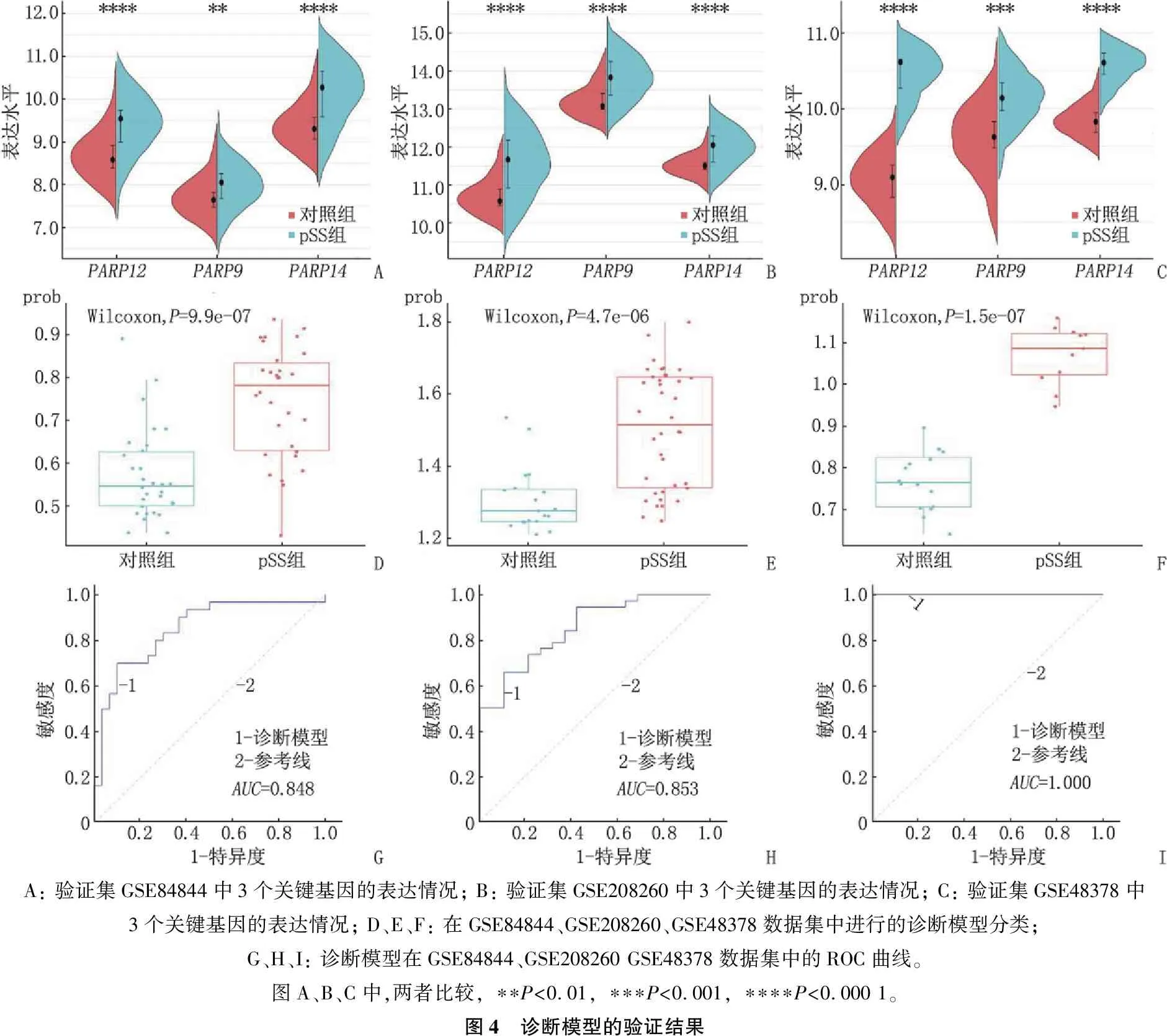
A: 验证集GSE84844中3个关键基因的表达情况; B: 验证集GSE208260中3个关键基因的表达情况; C: 验证集GSE48378中3个关键基因的表达情况; D、E、F: 在GSE84844、GSE208260、GSE48378数据集中进行的诊断模型分类; G、H、I: 诊断模型在GSE84844、GSE208260 GSE48378数据集中的ROC曲线。图A、B、C中,两者比较, ∗∗P<0.01, ∗∗∗P<0.001, ∗∗∗∗P<0.000 1。图4 诊断模型的验证结果
2.5 免疫细胞浸润分析
本研究采用直方图显示每个样本免疫细胞分布的总体情况,结果见图5A。箱线图表明,相较于对照组, pSS组患者的活化树突状细胞(DC)、幼稚B细胞、活化的CD4+记忆型T细胞水平较高,而静止期CD4+记忆型T细胞和记忆型B细胞水平较低,见图5B。22种免疫细胞之间的相关性分析表明, CD8+T细胞与中性粒细胞呈负相关(r=-0.46), 调节性T细胞(Tregs)与记忆型B细胞呈正相关(r=0.39), 见图5C。进一步分析3个关键基因与22种免疫细胞的相关性,结果显示, 3个关键基因(PARP12、PARP14、PARP9)均与活化DC呈正相关(r=0.76、0.75、0.72), 与M0型巨噬细胞呈负相关(r=-0.24、-0.22、-0.17), 见图5D。

A: 每个样本中22种免疫细胞的分布直方图; B: 健康人和pSS患者之间22种免疫细胞的相关性分析结果(两者比较, ∗∗P<0.01, ∗∗∗∗P<0.000 1, ns P>0.05); C: pSS组和非pSS组的免疫浸润箱线图; D: 3个关键基因与免疫细胞及免疫功能的相关性分析结果。图5 免疫细胞浸润分析
2.6 3个关键基因在pSS患者PBMC中的表达
为了进一步探讨3个关键基因在pSS患者PBMC中的表达情况,本研究基于pSS单细胞数据集进一步分析。该数据集共有10个样本,即5个pSS患者的样本(pSS组)和5个健康供体的对照样本(正常组),包含21 176个基因和54 152个细胞,其中pSS组包含28 938个细胞,正常组包含25 214个细胞。应用Seurat包中的UMAP方法将这些细胞分为31个聚类,根据既往文献报道的细胞标志物,手动注释出9种细胞类型,见图6A、图6B。正常组和pSS组各细胞类型中3个关键基因的表达情况见图6C~图6E, 结果显示,PARP9和PARP14在pSS组的巨噬细胞、常规DC(cDC)、CD14+单核细胞中高度表达,PARP12在巨噬细胞、cDC、CD14+单核细胞中的表达水平未发生变化,但pSS组表达这3个基因的细胞比例增加。

A: 点图显示每个聚类的细胞标记基因的平均表达水平; B: 来自5个pSS患者和5个健康供体的单细胞UMAP图; C、D、E: 各种细胞类型中3个中心基因的表达。图6 pSS单细胞RNA测序谱
3 讨 论
pSS的症状及诊断指标特异性差,常与其他自身免疫性疾病(如SLE和RA)重叠[9], 易导致诊断延迟和误诊,故寻找新的特异性生物标志物有助于pSS的诊断和治疗。本研究共鉴定出265个DEGs, GO分析结果表明其与响应病毒感染密切相关。既往研究[10]表明,病毒可通过各种机制引起自身免疫性疾病,包括旁观者活化、分子模拟、表位扩散和B细胞免疫活化等。KEGG通路分析同样表明, DEGs富集于与病毒相关的通路,包括流感病毒A和EB病毒感染。流感病毒A可通过其NS1mRNA的另类阅读框架触发强烈的CD8+T细胞反应或刺激浆细胞样树突状细胞产生α干扰素(IFN-α)而诱导自身免疫性。另有研究[11]表明, EB病毒也参与了pSS的发病机制。因此,研究人员或有必要更深入地探讨病毒引起的自身免疫性在pSS中的作用。
本研究运用多种生物信息学方法识别pSS中与铁死亡相关的关键基因,并分析了3个与铁死亡相关的关键基因(PARP9、PARP12和PARP14)对pSS发病的诊断价值。铁死亡是一种新的细胞死亡方式[12], 研究[4, 13-14]发现,铁死亡在多种自身免疫性疾病(如SLE、RA和癌症)的发生和进展中具有重要作用。既往研究[15]表明,干扰PARP表达可以调节溶质载体家族7成员11(SLC7A11), 并参与乳腺癌易感基因(BRCA)介导的卵巢癌中的铁死亡过程。在RA中,PARP9是一种具有不同甲基化位点的基因,可影响T淋巴细胞的转录表达,表明PARP9在RA的发病机制中具有重要性[16]。JIANG Z H等[17]发现,在SLE患者的多种免疫细胞中,PARP12表现出低甲基化水平。另有研究[18]表明,基于PARP14、ATP10A和MX1这3个基因构建的评分模型在抗Ro 60阳性的自身免疫性疾病患者中的评分始终高于抗Ro 60阴性的患者。以上研究表明,PARP9、PARP12和PARP14在自身免疫性疾病中可能发挥重要作用,然而PARP9、PARP12和PARP14在pSS发展中的作用尚不明确。
本研究免疫浸润分析结果表明, pSS患者存在活化DC上调, B细胞与T细胞亚群比例失调。进一步观察pSS患者PBMCs的单细胞数据集,本研究发现DC、B细胞、单核细胞和巨噬细胞中PARP9和PARP14上调,表达PARP12的细胞在B细胞和巨噬细胞中的比例略微增加。DC在维持免疫稳态和自身耐受性方面发挥着重要作用,研究[19]已证实cDC破坏CD4+T细胞自身耐受性,导致自身抗体产生、脾肿大和Th1、Th17效应细胞的增加。一项研究[20]表明,在pSS患者中, IFN-α可高度诱导DC中PARP9的表达,且DC的抗原摄取、处理和呈递功能均发生改变,这增加了Ⅰ型IFN的产生并诱导CD4+T细胞扩增,促进了pSS的发展。此外,在RA中,PARP9的甲基化与T细胞转录表达的增加相关[16]。在白细胞介素(IL)-4存在的情况下,PARP14介导信号转导与转录激活因子6(Stat6)与其靶基因结合,促进B细胞失调,这是pSS的病理标志之一[21]。同时,PARP14通过与Stat6相互作用调控细胞因子IL-4、IL-5、IL-13的表达,从而促进Th9细胞的发生发展和Th2细胞的分化[22]。这些研究表明,PARP家族可能通过异常调控免疫细胞参与pSS的发病。
本研究通过差异基因分析和WGCNA鉴定出与pSS相关的铁死亡基因,并应用机器学习算法去除冗余基因,筛选出3个可靠的生物标志物(PARP9、PARP12和PARP14)用于构建诊断模型; 在3个验证集中,该诊断模型均表现出高诊断效能,其AUC分别为0.848、0.853和1.000; 免疫浸润分析发现, pSS患者活化DC水平较高, T细胞与B细胞比例失衡, 3个关键基因与活化DC均呈正相关; 基于单细胞数据集验证了3个关键基因在免疫细胞中的表达情况。综上所述,本研究基于生物信息学手段识别出pSS中与铁死亡相关的3个关键基因并构建pSS诊断模型,或可为pSS的诊断提供新工具。
