烤烟品种中川208特异性指纹图谱的构建及分析
2023-06-11贾振国王亚男罗成刚张玉耿锐梅陈志强杨爱国刘国祥任民
贾振国 王亚男 罗成刚 张玉 耿锐梅 陈志强 杨爱国 刘国祥 任民


摘 要:烤烟品种中川208是经杂交选育而成的雄性不育一代杂交种,目前主要在华中和西南地区推广种植。为了加强对烟草品种的保护和利用,本研究利用中川208及其双亲的重测序数据进行SNP鉴定,并以5161份种质资源的重测序数据为遗传背景,对SNP位点进行筛选,构建了中川208的特异性指纹图谱。结果表明,该指纹图谱由285个SNP位点构成,经可靠性检验和实际测序验证,该指纹图谱对测序错误和位点缺失的容忍度极高,在兼顾可靠性和成本的条件下,可利用2X或3X的WGS测序对待测样品进行测序和检验。综上,本研究构建的烤烟品种中川208特异性指纹图谱,可靠性高,实用性强,为中川208的推广与保护提供了技术支撑,同时为烟草杂交种特异性指纹图谱的构建提供了方法参考。
关键词:中川208;指纹图谱;单核苷酸多态性;种质资源;重测序
中图分类号:S572.03 文献标识码:A 文章编号:1007-5119(2023)02-0001-06
Abstract: Flue-cured tobacco variety Zhongchuan208 is a male sterile first-generation hybrid, which is mainly cultivated in central China and Southwest China. In order to enhance the protection and utilization of tobacco varieties, SNP identification was conducted using resequencing data of Zhongchuan208 and its parents, and SNP loci were screened using resequencing data of 5161 germplasm resources as genetic background, and the specific fingerprint of Zhongchuan208 was constructed. The results showed that the fingerprint was composed of 285 SNP loci. After reliability test and actual sequencing verification, the fingerprint had a high tolerance for sequencing errors and locus missing. Under the condition of both reliability and cost, the test samples could be sequenced and tested by 2X or 3X WGS sequencing. In conclusion, the specific fingerprint of flue-cured tobacco variety Zhongchuan208 was successfully constructed in this study. The fingerprint has high reliability and strong practicability, which provides technical support for the promotion and protection of Zhongchuan208, and also provides method reference for the construction of specific fingerprint of tobacco hybrid.
Keywords: Zhongchuan208; fingerprint; single nucleotide polymorphisms; germplasm resources; resequencing
烤烟品种中川208是以雄性不育同型系MS中烟103为母本,烤烟新品系T137为父本通过杂交选育而成的杂种一代品种。中川208综合评价良好,质量性状、经济性状和抗逆性兼顾,目前主要在西南和华中烟区种植[1],是近年来选育的优良品种。中川208易感青枯病、赤星病和CMV,感染病害后会产生植株形态的改变;不同的氮素营养水平也会引起中川208植物学性状和农艺性状的改变[2]。因此,依靠植株形态鉴定中川208,需要特定的试验条件,对环境与人员要求高,难以准确简便地開展,严重影响中川208的推广和保护。利用指纹图谱对植物品种进行鉴定相较于传统的形态学鉴定具有鉴定周期短、准确性高等优点,且具有不受环境影响、全生育期均可进行鉴定的优势。因此,构建中川208的指纹图谱对该品种的推广和保护具有重要意义。
烟草指纹图谱构建主要利用分子标记进行。目前使用较广泛的是简单重复序列(simple sequencerepeats, SSR)分子标记。李显富等[3]在2015年利用SSR分子标记对烤烟K326的突变体品系“片片黄”进行指纹图谱构建;陈婷婷等[4]在2016年利用SSR分子标记对云烟87突变体品系进行指纹图谱构建;刘国祥等[5]在2017年利用SSR分子标记对33份晒烟种质资源进行指纹图谱构建;陈芳等[6]在2019年利用SSR分子标记构建80份烟草种质资源的指纹图谱;钱雨清[7]在2022年利用SSR分子标记对160份烟草种质资源进行指纹图谱的构建。但是,由于SSR分子标记存在检测成本高,多态性低的特点,近年来逐渐被单核苷酸多态性(single nucleotide polymorphism, SNP)分子标记所取代。王琰琰[8]在2021年利用SNP分子标记构建216份雪茄烟种质资源的指纹图谱;余世洲等[9]在2023年利用SNP分子标记构建可以用于烟草核心种质区分的指纹图谱。以上研究表明,烟草品种指纹图谱构建已经成为当前品种鉴定和保护的重要基础性工作。
目前构建的烟草品种指纹图谱多为通用性指纹图谱,多个品种共用一套SSR或SNP位点,开发与应用相对简便,可以有效区分供试品种与对照品种,但在鉴别供试品种是否为某个特定目标品种时,容易因为个别位点的分型问题而导致鉴定结论错误,不利于特定品种的鉴别。因此,针对特定品种的鉴别需要更加有效的特异性指纹图谱。目前特异性指纹图谱的研究集中于纯系品种,关于烟草杂交种特异性指纹图谱的构建缺乏报道。本研究利用SNP分子标记,对烟草杂交种中川208进行特异性指纹图谱构建,并对该套指纹图谱进行可靠性验证和使用成本分析,以期为烤烟品种中川208的推广和保护提供依据,并为后期大范围开发烟草杂交种特异性指纹图谱,建立烟草杂交种特异性指纹图谱数据库提供方法参考。
1 材料与方法
1.1 供试材料
本研究所采用的供试材料为中川208、MS中烟103和T136,种子由国家烟草种质资源中期库(青岛)提供,2021年9月播种于中国农业科学院烟草研究所人工气候室。待植株生长至大十字期,采集生长状态良好且无病虫害单株的叶片,置于?80 ℃超低温冰箱中保存,用于DNA提取及后续测序。以5161份种质资源的GBS(Genotyping-by- sequencing)简化基因组测序信息作为遗传背景,该数据由国家烟草种质资源中期库提供[10]。
1.2 DNA提取及文库构建
将获取的叶片样品用干冰包埋,寄至北京诺禾致源科技股份有限公司,进行供试材料基因组DNA的提取。将样品基因组DNA用于文库构建,测序方法为GBS简化基因组重测序,由北京诺禾致源科技股份有限公司的Illumina HiSeq PE150测序平台进行[11-16]。
1.3 指纹图谱SNP位点筛选
利用本研究获得的中川208及其双亲GBS简化基因组测序数据及前期已有的5161份种质资源GBS简化基因组测序信息筛选中川208特异性指纹图谱位点:(1)利用GATK 4.1[17]对中川208及其双亲(MS中烟103和T136)进行SNP位点分析,筛选中川208及其双亲的分型结果均不是缺失的SNP位点。(2)对上述位点进行特异性分析和发掘,筛选MS中烟103和T136中纯合且有差异,并在中川208中杂合的位点。(3)进一步分析上述位点对5161份烟草种质资源的群体SNP分型情况,剔除在种质资源群体上质量值(QUAL)小于1000、缺失率高于30%、杂合率高于30%的位点及非二态性位点,得到候选SNP位点。(4)基于5161份烟草种质资源的群体SNP分型数据,计算候选SNP位点的群体多样性指数(pi)。以1 Mb为统计窗口长度,将候选SNP进行分组(bin),选取每个bin内群体多样性指数(pi)最小的SNP作为中川208特异性指纹图谱SNP。群体多样性指数(pi)利用VCFtools[18]进行计算。
1.4 数据可视化及分析程序开发
本研究利用R语言对数据进行可视化。中川208特异性指纹图谱分析程序开发利用python 3.8计算机语言进行。
1.5 指纹图谱分析
利用正交设计的方法,设置位点数(检测到的中川208特异性指纹图谱位点数)及碱基错误率2种因素,探究其对检测正确率的影响。位点数设计10、20、30直到200个SNP(依次增加10个位点),共计20个水平;碱基错误率设计0%、10%、20%、 30%、40%、50%、60%,共计7个水平。2种因素共组成140种组合。在140种组合中对中川208特异性指纹图谱SNP位点进行10次随机抽样,共组成1400种不同的基因型数据。将不同的基因型数据输入分析程序进行检验,统计在不同位点数及不同碱基错误率下检测的正确率。实际测序验证采用WGS(Whole Genome Sequencing)全基因组重测序,测序深度为5 X,由北京诺禾致源科技股份有限公司的Illumina HiSeq PE150测序平台[11-16]进行,并从测序数据中随机分出1~4 X覆盖度的测序数据。
2 结 果
2.1 建库测序数据分析
对中川208及其双亲MS中烟103、T136进行GBS简化基因组重测序,根据得到的测序结果,对测序产出数据、酶捕获率进行统计,并以烟草品种第3代测序组装物理图谱作为参考基因组,将获取的高质量测序数据与参考基因组进行序列比对,得到定位比例、平均测序深度及覆盖度,结果如表1所示,3份材料有效数据率均达到99.99%,GC含量接近40%,表明测序数据质量高,无污染;酶捕获率大于99%,表明酶切效率高;平均测序深度在6.63~7.18之间,覆盖度在3.53%~4.12%之间,表明测序数据均一性及与参考基因组的相似性较好,可满足本研究要求。
2.2 中川208特异性指纹图谱构建
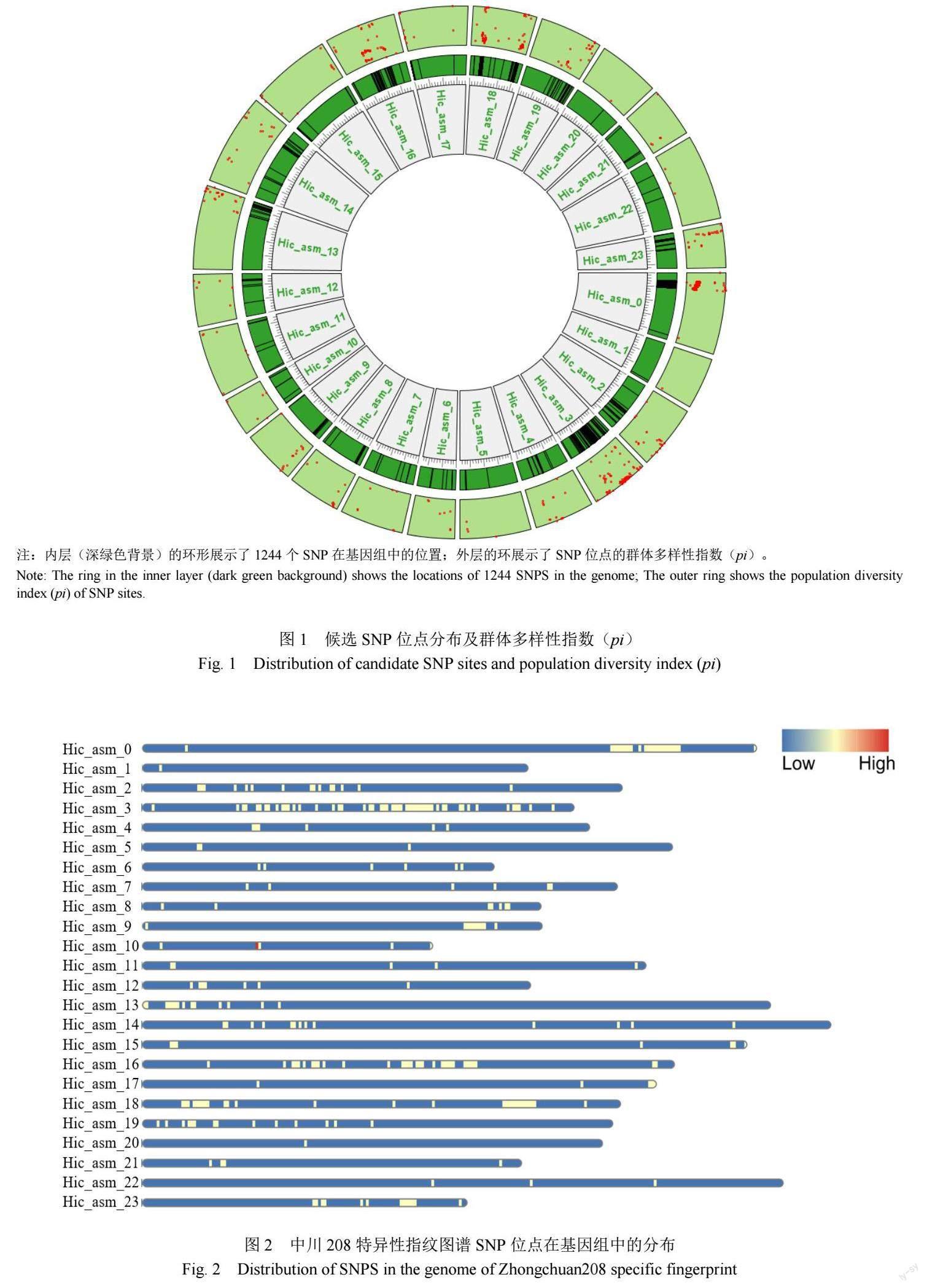
将中川208及其双亲(MS中烟103和T136)GBS重测序数据中检测到的42 465个分型结果不是缺失的SNP位点,依据双亲纯合且有差异,子代杂合的原则进行筛选,共筛选到1541处位点(不含位于Scaffold上的位点)。基于5161份种质资源的分型情况,剔除质量值(QUAL)小于1000的位点6处,缺失率高于30%的位点21处,杂合率高于30%的位点67处,非二态性位点203处,保留1244处候选SNP位点。基于5161份烟草种质资源的群体SNP分型数据,计算1244处候选SNP位点的群体多样性指数(pi)(图1),并以1 Mb为统计窗口长度,将候选SNP分为285个组(bin),选取每个bin内pi值最小的SNP作为中川208特异性指纹图谱SNP。由此得到构成中川208特异性指纹图谱的285个高质量SNP位点,位点的详细信息见文件http://47.104.13.144: 8686/down/xDn15XZ5FCkR.xlsx。构成中川208特異性指纹图谱的285个SNP位点分布于24条染色体上,其中Hic_asm_3染色体上SNP位点最多,有51个,各染色体上的SNP分布情况见图2。
2.3 指纹图谱分析程序开发
为方便中川208特异性指纹图谱的应用,本研究利用python3.8计算机语言,开发中川208特异性指纹图谱分析程序,源代码托管于https://gitee.com/renm79/zc208fp,使用时将样品测序数据整理为4列,其中第一列为位点编号,第二列为染色体编号,第三列为位置,第四列为该位点的基因型,基因型信息采用国际通用的简并碱基代码。利用该程序可方便地计算用户输入的遗传信息与中川208指纹图谱信息间的遗传差异,并以烟草种质资源群体为参照,判断待检材料是否为中川208,操作显示页面见图3,使用时可根据遗传距离进行判断,当检测样品与中川208(ZC208)遗传距离为0时,则可说明该样品为中川208,反之则不是。
2.4 指纹图谱可靠性检验及实际测序验证
在实际测序过程中,供试样品基因型会在一定程度上存在位点缺失及测序错误,本研究利用正交设计的方法针对这2种情况对中川208特异性指纹图谱的可靠性进行检验,并由此获取1400种基因型数据(具体方法见1.5)。将获取的1400种基因型数据依次输入指纹图谱分析程序进行分析,统计检验的正确率,部分试验结果如图4所示。该结果表明,在碱基错误率高达60%的情况下,只要检测位点达到任意80个以上时,检测准确率均可达到100%,而在错误碱基的比例不超过10%的情况下,即使只检测到285个位点中的任意10个位点,也能实现准确检测。
为进一步对中川208特异性指纹图谱的准确率进行检测,降低指纹图谱使用成本,本文对中川208进行5X覆盖度的全基因组重测序,测序检测到的指纹图谱位点与前期开发时的测序分型结果一致。从测序数据中随机分出了1X、2X、3X、4X覆盖度的测序数据。1~5X覆盖度测序数据输入分析程序后的检测结果及成本见表2。表2表明,检测到的指纹图谱位点数随着测序深度增加而增加,1~5X覆蓋度检测的指纹图谱位点数均可正确识别中川208,在综合考虑可靠性和成本的情况下,可在实际使用过程中采用2X或3X的WGS测序获取样品的测序信息。
3 讨 论
大量研究表明,构建指纹图谱可以有效鉴别烟草品种,对烟草品种的推广和保护有重要作用[3-9]。中川208作为近年来选育的优良雄性不育杂交一代品种[1],构建中川208的指纹图谱,是该品种推广和利用的重要基础性工作。
SNP标记作为第3代分子标记,与高通量检测方法相结合,具有成本低、周期短、准确性高等特点[19]。本研究基于SNP分子标记进行烟草品种指纹图谱构建,与利用SSR分子标记构建的烟草指纹图谱技术[3-7]相比,入选分子标记数量显著增加,构建的指纹图谱SNP位点分布于全基因组,使指纹图谱的特异性和分辨力显著提升。在使用方面,本研究构建的SNP指纹图谱直接利用高通量测序技术进行检测,避免了SSR指纹图谱需要电泳检测的繁琐步骤,使指纹图谱的检测效率显著提升。在参考群体方面,本研究以5161份烟草种质资源为对照群体,对照群体数量远高于现有的烟草指纹图谱,筛选的指纹图谱位点能够更加有效地揭示烟草品种的遗传特异性。此外,可靠性检验表明,本研究开发的中川208特异性指纹图谱对测序错误和位点缺失的容忍度极高,相较于烟草通用性SNP指纹图谱[8-9],不会因为1个或几个SNP位点的分型错误导致检测结果改变。针对烟草SNP指纹图谱使用成本的问题,本研究利用WGS全基因组重测序技术,对中川208进行5X覆盖度的全基因组重测序,并从测序数据中随机分离出1~4X覆盖度的测序数据,发现在兼顾可靠性和成本的前提下,2X或3X的测序深度是最佳的测序深度。
烟草杂交种可以融合双亲的优良特性,表现出超亲杂种优势,能兼顾较多的重要性状,是烟草品种选育的重要方法。近年来除中川208以外,不断有优良的烟草杂交种被选育[20-22],特异性指纹图谱可以用于烟草杂交种的准确鉴别,是烟草杂交种推广和保护的重要基础性工作。本研究首次利用SNP分子标记对烟草杂交种进行特异性指纹图谱构建,构建的指纹图谱可靠性高且经济、简便,该方法有望广泛应用到烟草杂交种特异性指纹图谱的构建过程中。
4 结 论
本研究通过高通量测序技术,基于SNP分子标记构建了由285个SNP位点组成的中川208特异性指纹图谱。通过可靠性检验和实际测序验证,表明该指纹图谱具有较高的可靠性和较强的实用性,可以应用于中川208的品种真实性鉴定。本研究构建烟草杂交种特异性指纹图谱的方法切实可行,可以为其他烟草杂交种特异性指纹图谱的构建提供方法参考。
参考文献
[1]张玉,刘杨,王元英,等. 烤烟新品种中川208的选育及特征特性[J]. 中国烟草科学,2019,40(5):1-7.
ZHANG Y, LIU Y, WANG Y Y, et al. Breeding and characterization of a new flue-cured tobacco variety Zhongchuan208[J]. Chinese Tobacco Science, 2019, 40(5): 1-7.
[2]罗政刚,高阳,李伟,等. 烤烟品种中川208优质高效栽培配套技术研究[J]. 种子科技,2023,41(4):27-30,141.
LUO Z G, GAO Y, LI W, et al. Study on high quality and efficient cultivation technology of flue-cured tobacco variety Zhongchuan208[J]. Seed Science & Technology, 2023, 41(4): 27-30, 141.
[3]李显富,陈婷婷,安雪琴,等. 烤烟K326突变体新品系‘片片黄的性状表现及其DNA指纹图谱构建[J]. 西南农业学报,2015,28(3):1300-1305.
LI X F, CHEN T T, AN X Q, et al. Characteristics and DNA fingerprint of new flue-cured tobacco strain ‘Pianpianhuang bred from natural mutant of variety K326 by system selection method[J]. Southwest China Journal of Agricultural Sciences, 2015, 28(3): 1300-1305.
[4]陈婷婷,李显富,贾春雷,等. 云烟87突变体新品系的特征特性及其DNA指纹图谱鉴定[J]. 浙江大学学报(农业与生命科学版), 2016,42(2):179-189.
CHEN T T, LI X F, JIA C L, et al. Characteristics of new flue-cured tobacco strains developed from Yunyan87 mutants and their DNA fingerprint[J]. Journal of Zhejiang University (Agric. & Life Sci.), 2016, 42(2): 179-189.
[5]刘国祥,邹昆晏,任民,等. 33份晒烟种质资源SSR标记的指纹图谱构建[J]. 中国烟草学报,2017,23(5):57-64.
LIU G X, ZOU K Y, REN M, et al. Construction of SSR markers fingerprints of 33 sun-cured tobacco germplasm resources[J]. Acta Tabacaria Sinica, 2017, 23(5): 57-64.
[6]陳芳,徐世晓,李晓辉,等. 基于SSR标记的80份烟草种质指纹图谱的构建及遗传多样性分析[J]. 作物杂志,2019,188(1):22-31.
CHEN F, XU S X, LI X H, et al. Construction of molecular fingerprinting and analysis of genetic diversity for 80 tobacco (Nicotiana tabacum) germplasms based on SSR markers[J]. Crops, 2019, 188(1): 22-31.
[7]钱雨清. 基于SSR标记的烟草DNA指纹图谱的构建与遗传分析[D]. 泰安:山东农业大学,2021.
QIAN Y Q. Fingerprints construction and genetic analysis of Nicotiana tabacum based on SSR markers[D]. Taian: Shandong Agricultural University, 2021.
[8]王琰琰. 雪茄烟种质资源SNP指纹图谱构建及群体遗传分析[D]. 北京:中国农业科学院,2021.
WANG Y Y. Construction of a SNP fingerprinting database and population genetic analysis of cigar tobacco germplasm resources in China[D]. Beijing: Chinese Academy of Agricultural Sciences, 2021.
[9]余世洲,曹领改,王世泽,等. 烟草种质基因分型核心SNP标记的开发[J]. 生物技术通报,2023,39(3):89-100.
YV S Z, CAO L G, WANG S Z, et al. Development core SNP markers for tobacco germplasm genotyping[J]. Biotechnology Bulletin, 2023, 39(3): 89-100.
[10]任民,王志德,牟建民,等. 我国烟草种质资源的种类与分布概况[J]. 中国烟草科学,2009,30(S1):8-14.
REN M, WANG Z D, MU J M, et al. Overview of species and distribution of tobacco germplasm resources in China[J]. Chinese Tobacco Science, 2009, 30(S1): 8-14.
[11]LI H, DURBIN R. Fast and accurate short read alignment with burrows-wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754-1760.
[12]LI H, HANDSAKER B, WYSOKER A, et al. The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079.
[13]WANG K, LI M, HAKONARSON H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data[J]. Nucleic Acids Research, 2010, 38(16): e164.
[14]CHEN K, WALLIS J W, MCLELLAN M D, et al. BreakDancer: an algorithm for high-resolution mapping of genomic structural variation[J]. Nature Methods, 2009, 6(9): 677-681.
[15]ABYZOV A, URBAN A E, SNYDER M, et al. CNVnator: an approach to discover, genotype, and characterize typical and atypical CNVs from family and population genome sequencing[J]. Genome Research, 2011, 21(6): 974-984.
[16]KRZYWINSKI M, SCHEIN J, BIROL ?, et al. Circos: an information aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639-1645.
[17]MCKENNA A, HANNA M, BANKS E, et al. The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Res., 2010, 20(9): 1297-1303.
[18]DANECEK P, AUTON A, ABECASIS G, et al. The variant call format and VCF tools[J]. Bioinformatics, 2011, 27(15): 2156-2158.
[19]唐立群,肖层林,王伟平. SNP分子标记的研究及其应用进展[J]. 中国农学通报,2012,28(12):154-158.
TANG L Q, XIAO C L, WANG W P. Research and application progress of SNP markers[J]. Chinese Agricultural Science Bulletin, 2012, 28(12): 154-158.
[20]王平平,孫渭,张强,等. 烤烟新品种秦烟201的选育及特征特性[J]. 中国烟草科学,2022,43(2):7-11.
WANG P P, SUN W, ZHANG Q, et al. Breeding and characterization of a new flue-cured tobacco variety Qinyan201[J]. Chinese Tobacco Science, 2022, 43(2): 7-11.
[21]常爱霞,卞建锋,蔡宪杰,等. 烤烟新品种中烟207的选育及特征特性[J]. 中国烟草科学,2022,43(3):1-6.
CHANG A X, BIAN J F, CAI X J, et al. Breeding and characterization of a new flue-cured tobacco variety Zhongyan207[J]. Chinese Tobacco Science, 2022, 43(3): 1-6.
[22]李尊强,乔婵,王春军,等. 烤烟新品种龙烟101的选育及特征特性[J]. 中国烟草科学,2022,43(3):14-19.
LI Z Q, QIAO C, WANG C J, et al. Breeding and characterization of a new tobacco variety Longjiang101[J]. Chinese Tobacco Science, 2022, 43(3): 14-19.
