80份绿豆象抗感蚕豆种质资源SSR遗传多样性分析
2023-06-10刘昌燕刘良军韩雪松万正煌沙爱华陈宏伟
孙 虎,刘昌燕,李 莉,刘良军,韩雪松,万正煌,沙爱华,陈宏伟
(1.长江大学农学院/湿地生态与农业利用教育部工程研究中心,湖北 荆州 434025;2. 湖北省农业科学院粮食作物研究所/粮食作物种质创新与遗传改良湖北省重点实验室,武汉 430064)
【研究意义】蚕豆(ViciafabaL.,2n=12)又名胡豆、罗汉豆等,豆科蝶形花亚科巢菜属,为越年生或一年生草本植物,基因组大小约13 Gb[1-2]。蚕豆营养价值丰富,为粮食、蔬菜和饲料、绿肥兼用作物。因为蚕豆与其根瘤菌共生固氮使其在调整农业种植结构、发展畜牧业、提高土壤肥力、保持农田生态平衡中占有重要地位[3]。绿豆象属于鞘翅目豆象科瘤背豆象属。作为一种世界性仓储害虫,绿豆象危害蚕豆、绿豆、豌豆等多种食用豆类,在种植和储藏食用豆的地方均可发生危害。目前,人们主要采用化学方法对绿豆象进行防治,但该方法易产生化学残留污染环境且容易使豆象产生抗药性[4]。因此,培育抗豆象蚕豆品种成为避免豆象侵害的首选。传统育种方法成本高,耗时长,限制性较大。现代分子生物技术的发展,尤其是分子标记技术的发展极大地促进了作物新品种选育进程。因SSR标记具有重复性好、多态性高、共显性遗传、遍布整个基因组等优点,故在水稻、小麦、玉米、大豆等主要作物中,SSR标记被用于作物遗传多样性分析、种质资源鉴定、遗传连锁图谱构建及QTL定位和分子标记辅助选择育种等方面的研究[5-8]。【前人研究进展】迄今为止,大多数已知的蚕豆SSR标记是通过挖掘基因SSR、cDNA测序或SSR富集文库方法的公共数据库开发的[9-18]。前人研究为蚕豆的分子标记开发和功能基因组研究提供了丰富的转录组资源。尽管已经开发了大量的SSR标记,由于蚕豆基因组大小非常大,目前尚无参考基因组可用,还需开发新的蚕豆SSR标记丰富蚕豆的遗传与基因组资源。在蚕豆方面,SSR分子标记已被广泛应用于遗传多样性分析、遗传连锁图谱构建以及关联分析等方面的研究[19-21]。Mohamed等[22]使用SSR标记对来自不同地理区域的35份蚕豆种质资源进行了种质遗传多样性和结构分析,聚类分析与结构分析结果一致,35种蚕豆基因型被分为两类。关于利用SSR分子标记技术对于蚕豆抗豆象方面研究的报道基本没有,人们目前主要通过表型性状对蚕豆抗豆象方面进行研究。段灿星等[23]通过室内人工侵染以及自由选择测验对339份蚕豆材料和100份豌豆材料进行绿豆象抗性研究,研究结果表明对绿豆象抗性与品质成分有关,深色种子与浅色种子相比,受到的侵害更小。Estefanía等[24]为了鉴定蚕豆对豆象抗性的稳定来源并揭示外界气候条件与农艺性状对豆象侵害之间的关系,进行了自然侵染下的田间试验,研究结果表明在蚕豆中存在不同的抗豆象机制;温度、降雨量和湿度似乎是影响蚕豆豆象危害发生的最大气候因素,而在不同的实验中,早熟种子和小粒种子与较低的豆象侵染率相关联。张红岩等[4]在室温条件下,对500份蚕豆种质资源连续2年进行绿豆象饱和侵染处理,最终筛选出H0048、H0092、H0094、H001455、H001471、H001787共6份高抗绿豆象的蚕豆资源。研究表明,食用豆类作物的抗性主要是由于自身携带抗性基因所致。【本研究切入点】目前为止,还没有关于蚕豆对豆象抗性遗传基础相关的研究报道。本研究利用三代全长测序技术测定RNA-seq进而开发新的蚕豆SSR分子标记。利用开发的SSR分子标记对80份蚕豆种质资源进行遗传多样性分析,筛选与蚕豆抗豆象性状相关的SSR位点。【拟解决的关键问题】本文旨在为蚕豆的抗豆象种质资源筛选与鉴定提供依据,进一步挖掘与蚕豆抗豆象性状相关联的SSR分子标记位点,为培育蚕豆抗豆象品种提供理论基础。
1 材料与方法
1.1 试验材料
本试验选用80份不同敏感度的蚕豆抗豆象种质资源材料,所用材料均来自于湖北省农业科学院粮食作物研究所。试验材料见表1。

表1 80份蚕豆材料及其相关信息Table 1 80 faba bean materials and their related information
1.2 试验方法
1.2.1 豆象鉴定 本试验参考程须珍等[25]的方法进行豆象鉴定:每份材料随机选取50粒健康种子分别放入直径5 cm、高12 cm的玻璃瓶中,每个玻璃瓶内接种4~6头绿豆象成虫,让其在种子上随机产卵,至每粒种子落卵量在5个以上时,除去所有的成虫,养虫室温度(29±2) ℃,湿度60%~70%。待感虫材料完全被蛀食后(约40~60 d), 调查每份材料的受害粒数,计算种子被害率,根据种子被害率评价每份材料的抗性等级,评价标准见表2。
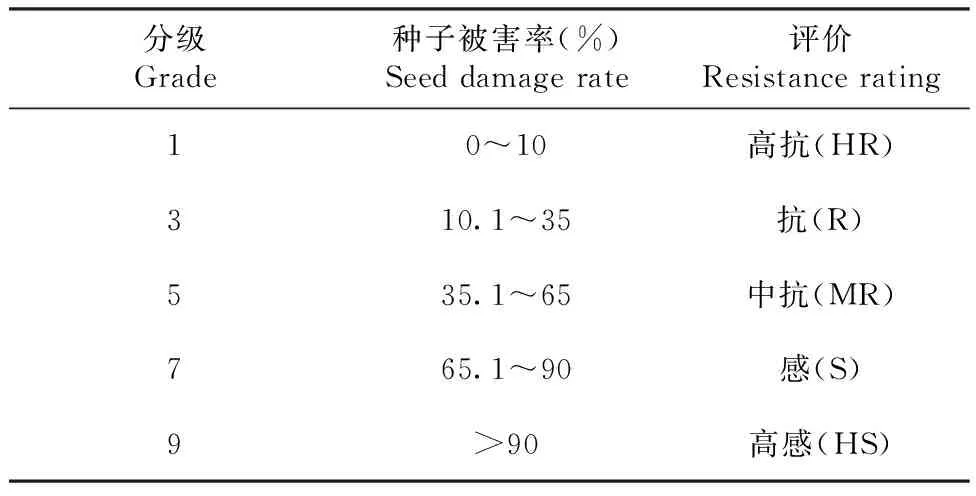
表2 抗性分级标准Table 2 Resistance grading standards
1.2.2 SSR引物选择 对2个蚕豆品种Y134和Y078进行转录组测序,利用MISA软件对转录组测序所得的所有Unigene序列进行SSR位点搜索,寻找含有SSR核心基序的Unigene序列。随后利用Primer 3.0批量设计SSR引物,所得到的引物经过处理后,构建长江大学农学院沙爱华课题组自有蚕豆SSR引物库(未公开)。在批量设计的SSR引物数据库中随机挑选100对SSR引物,由武汉天一辉远生物科技有限公司合成。
1.2.3 蚕豆总DNA的提取 本试验采用经过改进的简化CTAB法[26]提取蚕豆基因组DNA。取蚕豆幼嫩叶片50~100 mg,加入含1%聚乙烯吡咯烷酮的CTAB提取缓冲液700 μL,充分研磨,研磨液转入相对应的1.5 mL离心管;65 ℃水浴30 min,每3~5 min取出轻轻摇动混匀;水浴完成后于通风橱中加入500 μL混合液[v(苯酚)∶v(氯仿)∶v(异戊醇)=25∶24∶1],颠倒混匀;12 000 r/min离心10 min,取上清液转入新的离心管,加入等量混合液[v(氯仿)∶v(异戊醇)=24∶1],颠倒混匀;12 000 r/min离心10 min,取500 μL上清液转入新的离心管,加入2倍体积预冷无水乙醇颠倒混匀后-20 ℃静置30 min;12 000 r/min离心10 min,弃上清液,加入500 μL 70%的乙醇洗涤2次,12 000 r/min离心10 min;弃上清液,室温静置干燥15 min,加入40 μL灭菌水溶解DNA沉淀,室温放置1 d后,使用赛默飞NanoDrop 2000分光光度计和1%琼脂糖凝胶电泳检测DNA浓度及质量。
1.2.4 SSR分析 PCR反应体系(15 μL):DNA模板1.5 μL、ddH2O 9.4 μL、dNTP 0.3 μL、Mg2+0.9 μL、10×Buffer 1.5 μL、上下游引物各0.6 μL(10 (mol/L)、Taq酶0.2 μL(5 U/μL)。PCR扩增程序:95 ℃预变性5 min;95 ℃变性30 s,48~58 ℃(依引物而定)退火30 s,72 ℃延伸45 s,共35个循环;72 ℃延伸10 min,4 ℃恒温保存。PCR扩增产物加入1/2体积(7.5 μL)变性剂,95 ℃变性5 min。6%变性聚丙烯酰胺凝胶电泳,银染显影后观察结果并统计数据。
1.3 数据统计分析
采用人工读带的方法,有带记为“1”,无带记为“0”,缺失记为“9”。利用Power Marker v3.25软件计算引物的多态性信息(Polymorphism information content,PIC);利用Popgen32计算引物的Shannon信息指数(I);利用NTSYS-pc2.10e软件生成遗传相似系数并对80份蚕豆材料进行聚类分析,绘制树状聚类图。
2 结果与分析
2.1 80份蚕豆材料豆象鉴定
采用室内人工接虫的方法对80份蚕豆种质资源进行抗豆象分级鉴定,其中高抗(HR)材料67份,中抗(MR)材料1份,高感(HS)材料9份,感(S)材料3份,详细鉴定结果如表3所示。

表3 80份蚕豆材料抗豆象鉴定结果Table 3 Identification results of 80 faba bean materials for resistance to seed weevil
2.2 蚕豆DNA琼脂糖凝胶电泳检测
由图1-A可知,试验室原CTAB法提取的DNA质量较差,存在DNA降解,对试验结果可能会产生误差;改良CTAB法所提取的蚕豆基因组DNA质量显著提高,降解大幅度降低(图1-B)。
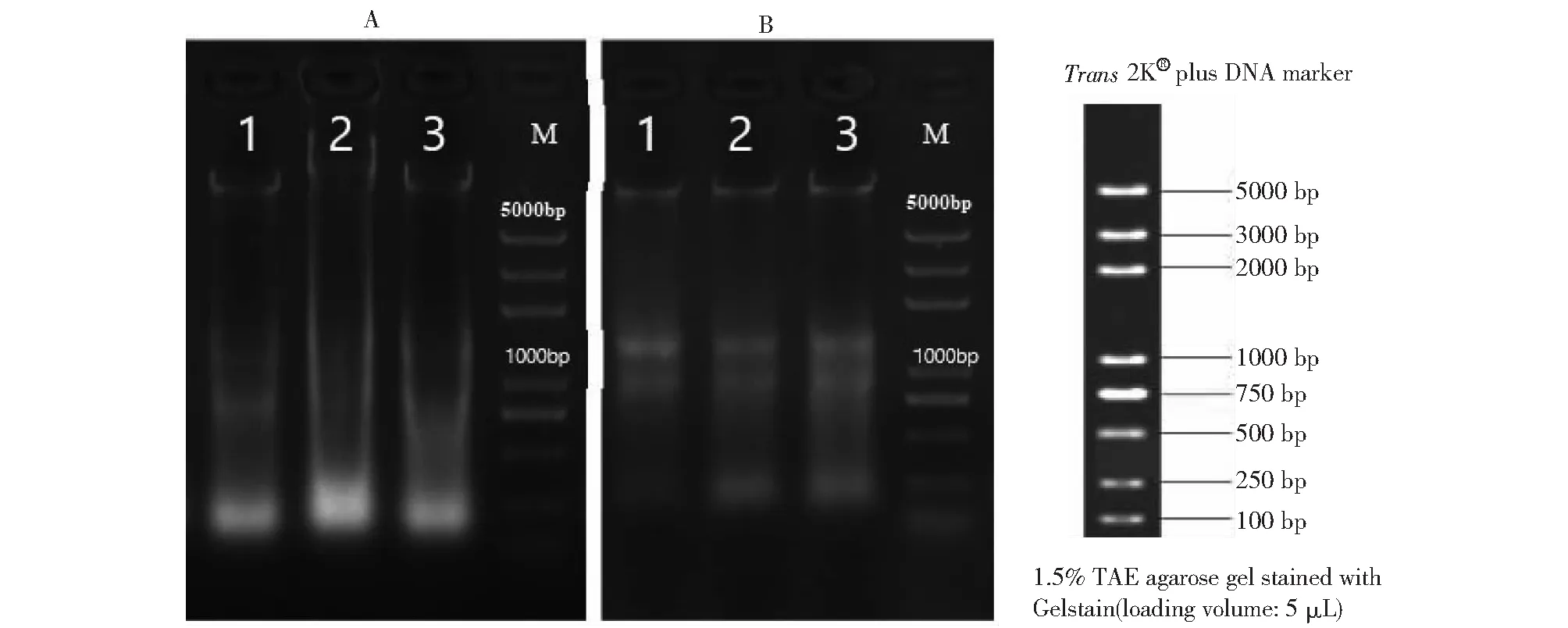
A.原CTAB法提取DNA检测结果;B.改良CTAB法提取DNA检测结果。A. Detection results of DNA extracted by the original CTAB method;B.Detection results of DNA extraction by modified CTAB method.图1 部分蚕豆材料DNA检测结果Fig.1 DNA test results of some broad bean materials
2.3 蚕豆SSR分子标记筛选
为避免品种特异性影响,本试验根据豆象鉴定结果对80份蚕豆材料进行分组,并在不同组别中随机选取32份蚕豆DNA混合后对100对SSR引物进行初筛。初筛结果表明,100对SSR引物中有50对PCR扩增出产物,有效扩增率50%。随后利用80份蚕豆材料对初筛的引物进行PCR扩增,其中32对SSR引物表现出多态性,多态性引物比率占有效扩增引物的64%,占所选取总引物的32%。引物FabaSSR09、27扩增条带清晰,多态性较好,如图2所示。

图2 SSR引物FabaSSR09、FabaSSR27在80份蚕豆材料中的PCR扩增Fig.2 PCR amplification of SSR primers FabaSSR09 and FabaSSR27 in 80 faba bean varieties
2.4 蚕豆SSR分子标记多态性分析
在100对SSR引物中筛选得到32对多态性好的引物,利用这32对引物对80份蚕豆种质资源材料基因组DNA进行PCR扩增,32对SSR多态性引物及多态性信息结果如表4。32对SSR引物在80份材料中共检测到220个等位变异,等位变异变化范围为2~21个,平均每个位点等位基因数为6.875个。多态性位点最丰富的是FabaSSR29标记,有21个等位基因;引物FabaSSR22检测出的等位变异最少,只有2个。多态信息量(PIC)的变幅为0.25(FabaSSR02)~0.82(FabaSSR09),平均值为0.58,PIC值大于平均数的占比为56.25%,表明本研究开发的SSR标记基因多样性较高。其中SSR引物FabaSSR09在高抗和易感材料间扩增出多态性条带(扩增出2条带及以上的材料均为高抗材料),推测该标记与蚕豆抗绿豆象性状相关,可用于蚕豆抗豆象材料的鉴定以及选育抗豆象品种。
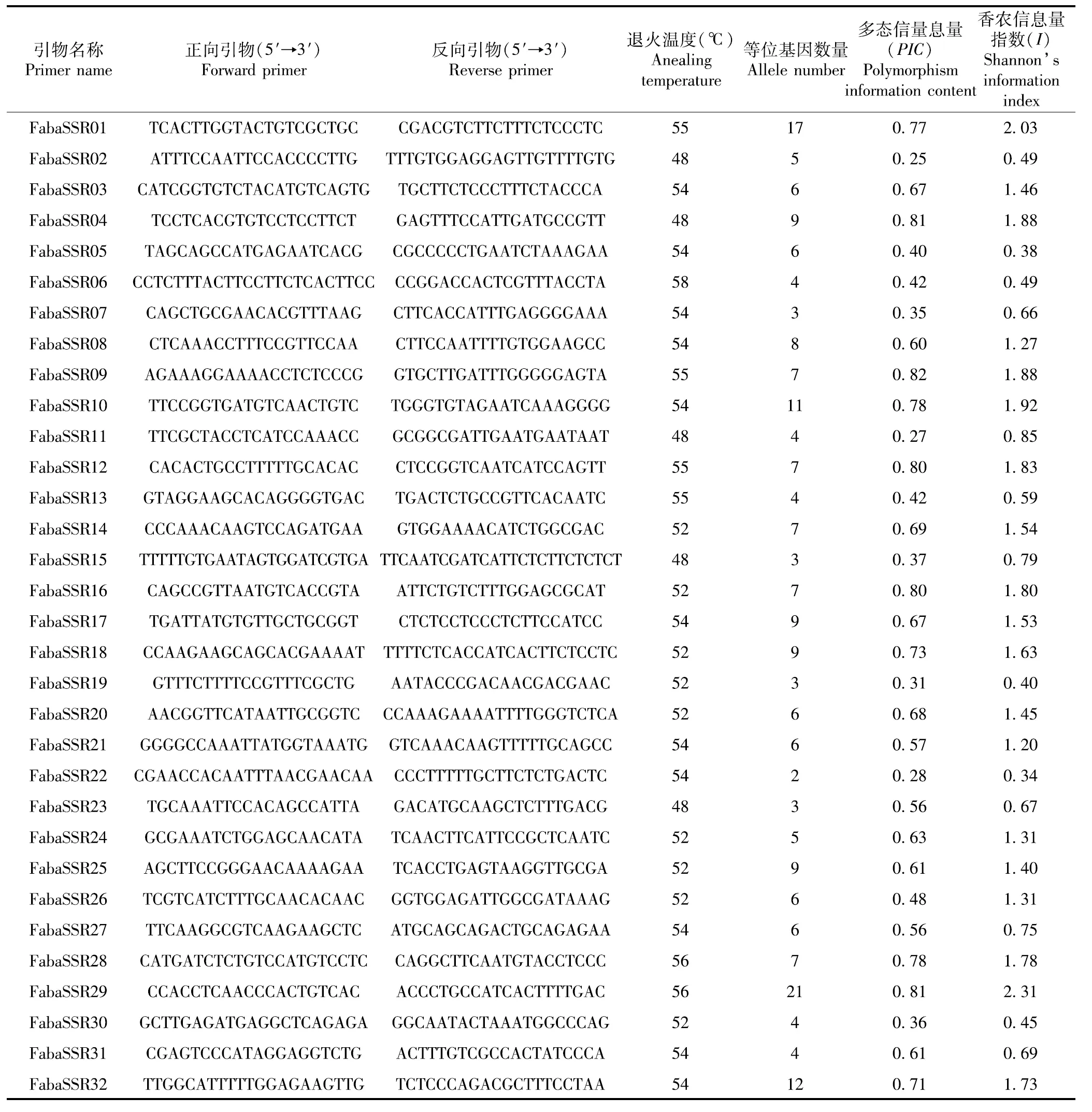
表4 32对多态性SSR引物相关信息Table 4 Correlation information of 32 pairs of polymorphic SSR primers
2.5 蚕豆种质资源材料聚类分析
利用32对SSR引物对蚕豆材料进行鉴定,利用NTSYSpc2.1软件对统计数据进行聚类分析,树状聚类图结果(图3)显示,在相似系数0.70处将80份蚕豆材料可分为两大类。第一大类包含65份材料,其中69(CD0112)、70(CD0283)为HS品种,78(CD0156)为S品种,其余为HR品种;第二大类包含15份材料,其中50(CD0254)、54(CD0279)、65(Y01-102-22)、66(Y04-111-3-1-3)、67(Y05-131-25)为HR品种,68(87-106-1-1)为MR品种,71(Y04-008)、72(Y04-210-1-1)、73(Y04-215-1-1)、74(Y05-131-4)、75(Y05-131-11-1-1)、76(Y10-613)、77(靖04-212-1-4)为HS品种,79(Y01-1021-91)、80(Y01-102-2)为S品种。聚类结果与蚕豆材料的抗性分级关系基本相符。

图3 80份蚕豆材料UPGMA聚类图Fig.3 Dendrogram of 80 faba bean varieties by SSR-UPGMA based on genetic similarities
3 讨 论
种质资源遗传多样性是作物种质资源鉴定和遗传改良的基础。早期蚕豆的遗传多样性研究主要集中在其农艺性状(株高、籽粒大小、有效分支数,百粒重等)等方面。姜翠棉等[27]通过对国内外不同地理来源的637份蚕豆资源的18个形态性状(见花期、株高、单柱荚数、单株粒数、百粒重等)进行调查,研究表明国内春冬性蚕豆资源间,以及国内外蚕豆资源间的遗传变异大;三维主成分分析显示参试资源由三大基因库构成,包括国内春播区资源、中国秋播区资源和国外资源。与其他豆科作物相比,蚕豆的基因组资源相对匮乏。虽然由于DNA测序技术的发展大大降低了测序成本,蚕豆的遗传及基因组信息得到进一步加强。但其庞大的基因组(约13 Gb)阻碍了蚕豆的发展。近年来,转录组测序等技术已被用于组装转录本和量化基因表达,并被广泛用于植物的基因挖掘和引物开发。尽管已经在蚕豆中开发了大量的SSR标记,但相对于蚕豆庞大的基因组而言还远远不够。杨涛等[16]利用罗氏454测序技术从247个春播和秋播蚕豆基因型的混合基因组中构建了一个具有125 559条推定SSR序列的文库,并对其重复类型和长度进行了阐述。设计了28 503对引物,随机选择150对进行验证。其中94对引物在选自不同地理位置的32个蚕豆基因型中表现出多态性。Sima等[28]概述了下一代测序(NGS)技术在挖掘和开发植物新的SSR标记中的应用,表明NGS技术是开发植物种群SSR标记的一种有效方法,特别是对于遗传信息未知的非模式植物。Mokhtar等[29]对前人研究进行了汇总整理,搭建了在线平台VfODB(http://vfodb.easyomics.org/.)。作为一个综合数据库,里面包含了蚕豆的种质信息、各类标记信息、遗传图谱等相关资源,可供研究人员使用。由于目前可用的蚕豆SSR引物不多,所以本研究通过测序开发了一定数量的SSR引物,其中32对SSR引物在供试蚕豆材料中表现出多态性。
随着分子标记技术的发展,人们开始利用分子标记技术进行种质资源遗传多样性分析等方面的研究。杨访问等[30]选取50对SSR引物对41份非洲和湖北蚕豆资源进行SSR遗传多样性分析。研究表明供试41份蚕豆材料有一定遗传多样性,但丰富度不高,蚕豆的遗传多样性与其地理起源相关。目前,蚕豆育种工作者已经针对某些病害成功培育出抗性品种,但进展缓慢,因为蚕豆的科研投入不足,导致对大多数已鉴定抗性的遗传基础以及病虫害的病因和遗传多样性了解不足。虽然已经确定了一系列抗性来源,针对蚕豆锈病、蚕豆枯萎病、列当属植物寄生抗性等已经开发了一些DNA标记。但在大多数情况下,它们的表型表达都没有得到充分的描述,而且它们的遗传基础在很大程度上是未知的。其中没有关于蚕豆豆象抗性遗传信息方面的报道[31]。虽然对于蚕豆抗豆象种质资源的分子遗传基础等方面的研究较少,但在豆科其他作物中有相关的报道。刘长友等[32]以抗豆象栽培绿豆V1128为父本、感豆象栽培品种冀绿7号为母本杂交构建F2分离群体。通过对群体进行抗豆象鉴定以及利用混合群体分离分析法 (BSA法) 筛选抗感池间的多态性标记构建连锁图谱,最终将抗豆象基因Br3定位在绿豆染色体5上,位于标记DMB158和VRBR-SSR033之间。Prakit等[33]对蛾豆种子的绿豆象抗性进行了QTL定位分析,揭示了对绿豆象抗性的一个主效QTL和一个修饰QTL,分别命名为qVacBrc2.1和qVacBrc5.1。qVacBrc2.1 位于SSR标记 CEDG261 和 DMB-SSR160 之间的连锁群 2 上。本试验利用32对SSR标记对80份蚕豆抗豆象相关种质资源进行遗传多样性分析,在一定程度上反映了80份蚕豆材料的亲缘关系,80份蚕豆种质资源的遗传相似系数分布在0.69~0.90,表明不同蚕豆材料之间存在一定差异,但差异不大,各品种之间亲缘关系较近,遗传背景较窄,这与前人的研究结果基本一致[34]。聚类分析表明参试材料的遗传多样性与其抗性分级有关。试验材料的遗传相似系数分布表明参试材料在分子水平上遗传差异不大,遗传背景较狭窄。这个问题在蚕豆育种中普遍存在,可能是因为蚕豆作为常异花授粉植物,天然异交率很高,因此亟需加强对地方种质资源的保护利用以及新种质资源的引进,从而丰富育种基础。从树状聚类图中观察到63和64号蚕豆材料聚类未分离,其原因可能是本研究所用的SSR引物数量有限,缺乏合适的引物区分这2个材料,因此在利用SSR分子标记技术进行遗传多样性分析时,应选取足够多的SSR引物,并且所选SSR引物多态性位点应该丰富,并且要尽可能覆盖植物整个基因组。
4 结 论
以80份对豆象不同抗性分级的蚕豆种质资源为材料,从基于转录组测序技术开发的100对SSR引物中筛选出32对有多态性、条带清晰的引物。利用32对多态性SSR引物分析了80份蚕豆种质资源的遗传多样性,80份蚕豆材料在遗传相似系数0.70处被划分为两大类,分类结果与蚕豆品种抗性分级基本相符。开发的分子标记扩充了蚕豆SSR引物数据库的数量,可用于蚕豆种质资源的鉴定、构建遗传连锁图谱以及分子标记辅助选择育种等方面的研究。通过对蚕豆抗豆象种质资源的遗传多样性分析,可为今后蚕豆种质资源的保护利用和品种鉴定以及培育新品种提供理论参考依据。
