花生根部结瘤性状QTL定位
2023-06-09陈伟刚李威涛喻博伦郭建斌周小静罗怀勇廖伯寿姜慧芳
黄 莉 陈伟刚 李威涛 喻博伦 郭建斌 周小静 罗怀勇 刘 念 雷 永 廖伯寿 姜慧芳
花生根部结瘤性状QTL定位
黄 莉 陈伟刚 李威涛 喻博伦 郭建斌 周小静 罗怀勇 刘 念 雷 永 廖伯寿 姜慧芳*
中国农业科学院油料作物研究所 / 农业农村部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
花生是我国重要的豆科油料作物和经济作物。根瘤是花生共生固氮的重要场所, 研究花生根瘤形成的遗传基础, 有助于更好地研究花生根瘤固氮能力和固氮特性。然而, 关于花生根瘤形成的研究较少, 调控花生根瘤形成的遗传机制不清楚。本研究通过对一个高世代RIL群体的根部结瘤性状进行调查, 鉴定到7份根部不结瘤家系, 根部不结瘤家系的叶绿素含量, 以及株高、单株鲜重、单株干重均显著低于双亲。利用前期构建的SSR标记遗传连锁图, 在A08和B07染色体上各鉴定到1个主效QTL和。通过InDel标记加密, 将QTL的区间由4.7 Mb缩小至1.6 Mb, 遗传变异解释率由9.1%增加至16.4%;的区间由9.9 Mb缩小至1.8 Mb, 遗传变异解释率由7.1%增加至9.9%。根据基因功能注释, 2个QTL区间分别鉴定到4个和2个结瘤素基因存在变异位点。本研究将为解析花生根瘤形成发育以及共生固氮提供理论依据。
花生; 结瘤; QTL定位; 连锁分析
花生(L.)是我国重要的油料与经济作物之一, 是食用植物油和蛋白质的重要来源, 在国民经济和社会发展占有重要地位。我国是世界上花生总产量和消费量最大的国家, 而且花生的总产、单产、单位面积产油量以及种植业产值均居国内油料作物首位[1]。花生作为豆科作物, 根瘤固氮对氮素积累的贡献率高达50%, 是花生氮素供应的主要方式[2]。根瘤固氮能力直接影响着植株地上部的生长和产量的形成, 根瘤固氮能力越强, 植株固氮积累量及产量越高。对花生根瘤固氮相关指标及其与产量的相关性分析表明根瘤数和根瘤鲜重与根瘤固氮积累量、供氮比例和荚果产量呈正相关, 高产品种具有良好的固氮能力[3]。因此, 研究花生根瘤形成的遗传基础, 有助于更好地研究花生根瘤固氮能力和固氮特性, 为花生根瘤高效固氮提供理论依据。
根瘤的形成和发育过程受结瘤因子信号通路(Nod Factor Signaling Pathway)和结瘤自调控信号通路(Autoregulation of Nodulation Signaling Pathway)调控。豆科植物在根际释放代谢产物类黄酮物质, 根瘤菌感知类黄酮信号附着于根毛上, 激活结瘤基因的表达, 释放结瘤因子(Nod Factor, NF)到植物根际。根毛表皮细胞质膜上的NFR1 (Nod Factor Receptor 1)和NFR5 (Nod Factor Receptor 5)受体识别结瘤因子后, 二者形成共受体将信号向下传递, 诱导产生钙震荡, 进而通过依赖于Ca2+、钙调素的激酶CCaMK与其磷酸化底物CYCLOPS的相互作用, 激活下游的转录因子, 如()、()和()等, 最后促进下游早期结瘤相关基因表达, 如()、(), 从而形成根瘤原基, 即根瘤[4-7]。由于根瘤菌的侵染, 根被诱导生成一种CLE类小肽, 通过植物的传导结构向地上运输, 并作为富亮氨酸类受体激酶(LRR-RLK)的配体在茎部与LRR-RLK互作, 并使之激活[8]。类受体蛋白激酶可能介导合成与结瘤有关的信号分子, 通过传导结构被运送至根部, 调控根部结瘤的数量[9-10]。此外, microRNA也参与根瘤的形成与发育,能够靶向切割百脉根的mRNA, 阻碍侵染线的形成并抑制根瘤发育[11]。光信号能够通过诱导大豆地上组织合成TGACG-motif结合因子3/4 (GmSTF3/4)和 FLOWERING LOCUS T (GmFTs)蛋白, 并将这2个蛋白长距离转运至根中从而调控大豆根瘤形成[12]。综上, 豆科植物根瘤的形成发育是一个复杂的过程, 受到多个基因的协同调控。
国内外在百脉根、大豆等根瘤形成上的研究较多, 但花生根瘤已有的研究主要集中在根瘤菌与花生共生互作过程的生理特征, 关于根瘤形成发育的遗传基础研究较少。花生根瘤的形成发育过程可以分为侵染(1 d)、根瘤原基形成(4 d)、根瘤相似结构形成(8 d)、未成熟根瘤形成(12 d)、成熟根瘤(21 d) 5个阶段[13]。模式豆科植物中, 结瘤因子被受体识别后, 诱导根毛变形弯曲, 形成侵染线, 以“侵染线(Infection Thread)”方式进入寄主植物表皮层细胞。根瘤菌在花生中是以“裂隙侵染(Crack-Entry)”方式通过寄主植物表皮伤口, 从细胞间隙侵染进入, 不同于模式豆科植物[13-14]。研究人员认为, 花生根瘤的形成机制与模式豆科植物根瘤形成机制存在不同,花生“裂隙侵染”形成根瘤的方式更为古早[15-16]。早期研究发现通过杂交2个根部结瘤的花生材料, 其重组自交系后代中会出现不结瘤家系材料, 遗传学分析表明花生根瘤数受2个独立基因调控, 当2个隐性等位基因重组在一起时, 出现根部不结瘤的现象[17-18]。对侵染根瘤菌5 d后的根进行转录组分析表明, 结瘤与不结瘤材料之间表达差异显著的基因与一些已知的结瘤因子信号通路基因同源, 如、、、等[19]。
本研究利用一个高世代RIL群体, 通过对其根瘤多年鉴定, 发掘根部不结瘤种质, 对根部不结瘤种质材料的叶绿素含量以及农艺性状进行鉴定, 并通过QTL定位分析, 挖掘花生根部结瘤性状的主效QTL, 为花生根瘤形成发育以及共生固氮提供理论依据。
1 材料与方法
1.1 试验材料
以油料所培育的品种中花10号为母本、资源材料ICG12625为父本进行杂交, 通过单粒传法连续多代自交, 构建了一个高世代的重组自交系群体(recombinant inbred lines, RIL), 共包含140个家系。其中, 中花10号为珍珠豆类型花生, ICG12625为赤道类型花生。
1.2 田间试验及根部结瘤性状考察
亲本及群体材料分别于2017年和2018年种植于湖北武汉中国农业科学院油料作物研究所试验基地, 采用完全随机区组设计, 2次重复, 每个小区单行种植, 每行15株, 行间距30 cm, 每行株距10 cm, 常规田间管理。收获时, 考虑边际效应, 剔除两边的植株, 从每行材料中间选取10株, 用清水洗净根部泥土, 对根部结瘤情况进行观察鉴定。
1.3 根部不结瘤材料叶绿素含量及农艺性状鉴定
将RIL群体中鉴定到的稳定不结瘤的家系分别于2019年和2020年种植于武昌湖北武汉中国农业科学院油料作物研究所试验基地, 采用完全随机区组设计, 2次重复, 每个小区单行种植, 每行15株, 行间距30 cm, 每行株距10 cm, 常规田间管理。亲本和不结瘤家系每份材料各取10株, 利用SPAD- 502型手持式叶绿素仪在饱果期测量主茎上倒数第3完全展开叶。收获时, 调查选取的每株材料的主茎高和单株植株鲜重, 并调查单株荚果干重。
1.4 根部结瘤性状QTL定位分析
根据田间调查的结果, 将结瘤性状分为“结瘤”和“不结瘤”, 视为质量性状, 结合前期构建的该群体SSR分子标记遗传连锁图[20], 利用Windows QTL Cartographer V. 2.5软件复合区间作图法(composite interval mapping, CIM)进行QTL定位分析。LOD值大于3.0的QTL被认为主效QTL。将QTL两侧SSR标记的引物序列在PeanutBase数据库(www. peanutbase.com)中与已发表的栽培种花生基因组序列进行比对, 获得该标记在基因组上的物理位置。利用实验室已有的亲本中花10号和ICG12625的基因组重测序数据, 使用GATK软件包检测QTL区间内两亲本间的SNP和InDel差异位点, 将差异片段>3 bp的InDel位点作为目标InDel, 将其上下游各500 bp的DNA序列从PeanutBase数据库中下载, 并利用Primer 5.0软件设计正向和反向引物。设计完成的引物序列在PeanutBase数据库中与栽培种花生基因组序列比对, 确认该引物是否为目标InDel位点的有效扩增。利用这些开发的InDel标记对RIL群体进行扩增, 对主效QTL进行加密分析。
1.5 QTL区间多态性位点分析
使用ANNOVAR软件对加密后的QTL区间内的变异位点进行注释, 并预测变异对基因功能的影响, 根据变异位点在参考基因组上的位置信息(UTR区、基因间区、基因区或CDS区等), 判断变异对基因的影响(同义突变、非同义突变等)。
1.6 QTL区间基因表达分析
利用R软件包“biomaRt” (https://bioconductor. org/packages/release/bioc/html/biomaRt.html)将已发表的花生多组织转录组测序获得的read counts数据[21]转换成FPKM值, 分析QTL区间内基因在花生根与根瘤中的表达水平。
2 结果与分析
2.1 花生根部结瘤性状的鉴定
连续2年对140份RIL群体及其亲本材料的根部结瘤情况进行调查发现, 亲本中花10号和ICG12625根部均有根瘤, RIL群体中出现不结瘤家系。母本中花10号平均根瘤数为187个, 父本ICG12625平均根瘤数为92个。RIL群体中鉴定到7份家系在2年的环境中稳定地表现为植株根部不结瘤(图1)。
2.2 花生根部不结瘤家系性状鉴定
将这7份不结瘤家系与结瘤家系及亲本进行对比发现, 不结瘤家系叶片偏黄。对这7份不结瘤家系和亲本材料的叶绿素含量进行测定, 不结瘤家系2019年叶片的叶绿素含量变异范围在20.41~29.22 SPAD, 2020年叶片的叶绿素含量变异范围在24.37~ 29.97 SPAD, 而母本材料中花10号2年的叶绿素含量分别为43.91 SAPD和45.80 SPAD, 父本材料ICG12625两年的叶绿素含量分别为40.92 SAPD和46.18 SPAD。不结瘤家系的叶片叶绿素含量均显著低于2个亲本材料的叶绿素含量。株高方面, 除L099家系跟母本中花10号株高无差异, 其他6份不结瘤家系的株高2年均显著低于双亲材料, 2019年株高变异范围为30.33~38.52 cm, 2020年株高变异范围为28.23~39.63 cm。不结瘤家系的单株植株鲜重变异范围为119.0~208.8 g, 单株荚果干重变异范围为16.1~29.6 g, 均显著低于双亲材料(表1)。

图1 花生植株根部结瘤
A: 结瘤的根; B: 不结瘤的根。A: root with nodules; B: root without nodules.
2.3 花生根部结瘤性状QTL定位
将该RIL群体根部结瘤性状分为“结瘤”与“不结瘤”, 通过WinQTLCart2.5进行QTL定位分析, 鉴定到2个主效QTL和, 分别位于连锁群A08和B07。QTL的LOD值为3.7, 遗传变异解释率为9.1%, 区间两侧标记分别为AHGS1880和AGGS1495。QTL的LOD值为3.1, 遗传变异解释率为7.1%, 区间两侧标记分别为Ai07B24086和Ai07B26973。将QTL区间两侧SSR分子标记的引物序列与四倍体栽培种花生Tifrunner基因组序列进行比对, QTL定位到染色体A08上9.5~14.2 Mb区间,定位到染色体B07上118.7~128.6 Mb区间(表2)。
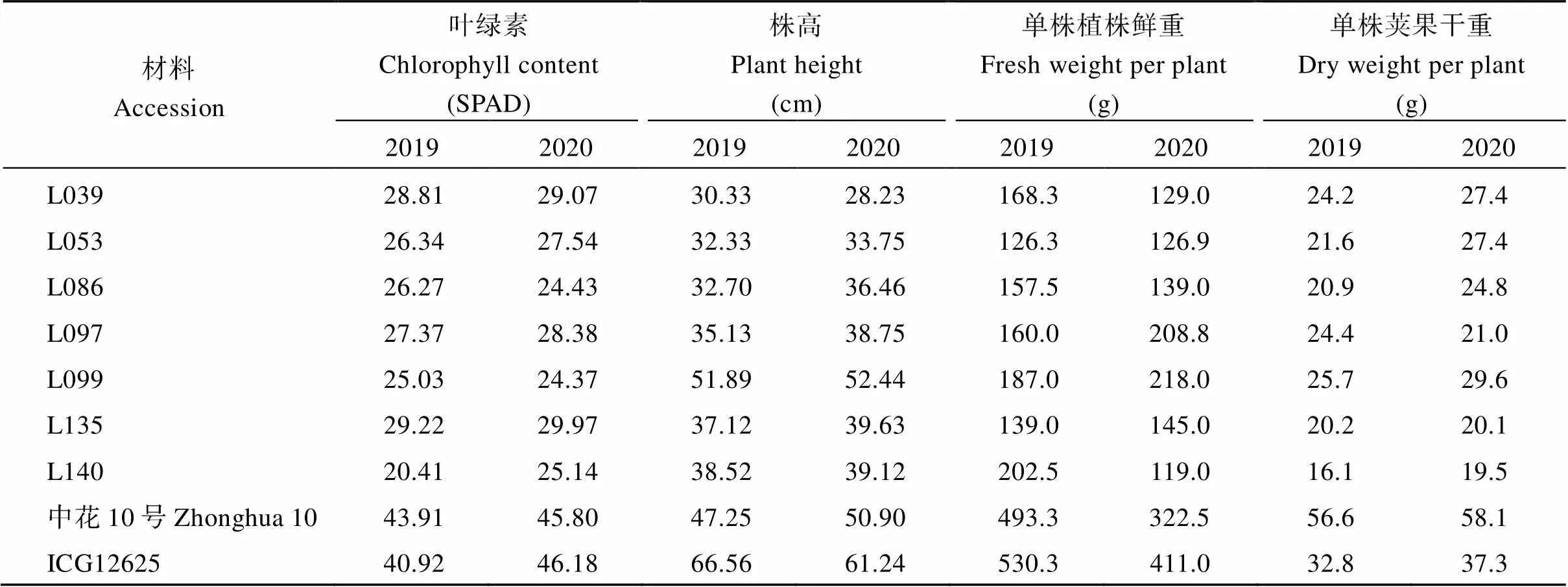
表1 不结瘤家系和亲本的叶片叶绿素含量和农艺性状鉴定

表2 花生根部结瘤性状QTL定位
QTL: quantitative trait locus; PVE: phenotypic variation explained.
2.4 花生根部结瘤性状QTL加密分析
利用亲本中花10号和ICG12625的重测序数据,在染色体A08上9.5~14.2 Mb区间和B07上118.7~128.6 Mb区间鉴定亲本间碱基差异。2个亲本在QTL区间内有647个SNP差异和117个InDel差异, 在区间内有1609个SNP差异和564个InDel差异。基于InDel差异, 在2个QTL区间分别开发了10个和19个InDel标记, 并在RIL群体中进行扩增。利用这些InDel标记在RIL群体中基因型, 在染色体A08上9.5~14.2 Mb区间加密构建了一个包含10个标记、遗传距离17.6 cM的连锁群, 在染色体B07上118.7~128.6 Mb区间加密构建了一张包含19个标记、遗传距离18.8 cM的连锁群。
利用这2个加密的连锁群, 对不结瘤性状进行QTL分析, 在这2个连锁群上仍然鉴定到2个主效QTL (表3和图2)。区间为6.9~13.5 cM, LOD值由3.7增加到5.9, 遗传变异解释率由9.1%增加到16.4%, 比对基因组序列后, 该QTL区间物理位置由原来的4.7 Mb缩小至1.6 Mb。区间为9.9~15.2 cM, LOD值由3.1增加到3.7, 遗传变异解释率由7.1%增加到9.9%, 同时该QTL区间物理位置由原来的9.9 Mb缩小至1.8 Mb。通过标记加密, 2个QTL区间物理位置均缩小至2 Mb以内。

表3 标记加密后的根部结瘤性状主效QTL信息
QTL: quantitative trait locus; PVE: phenotypic variation explained.
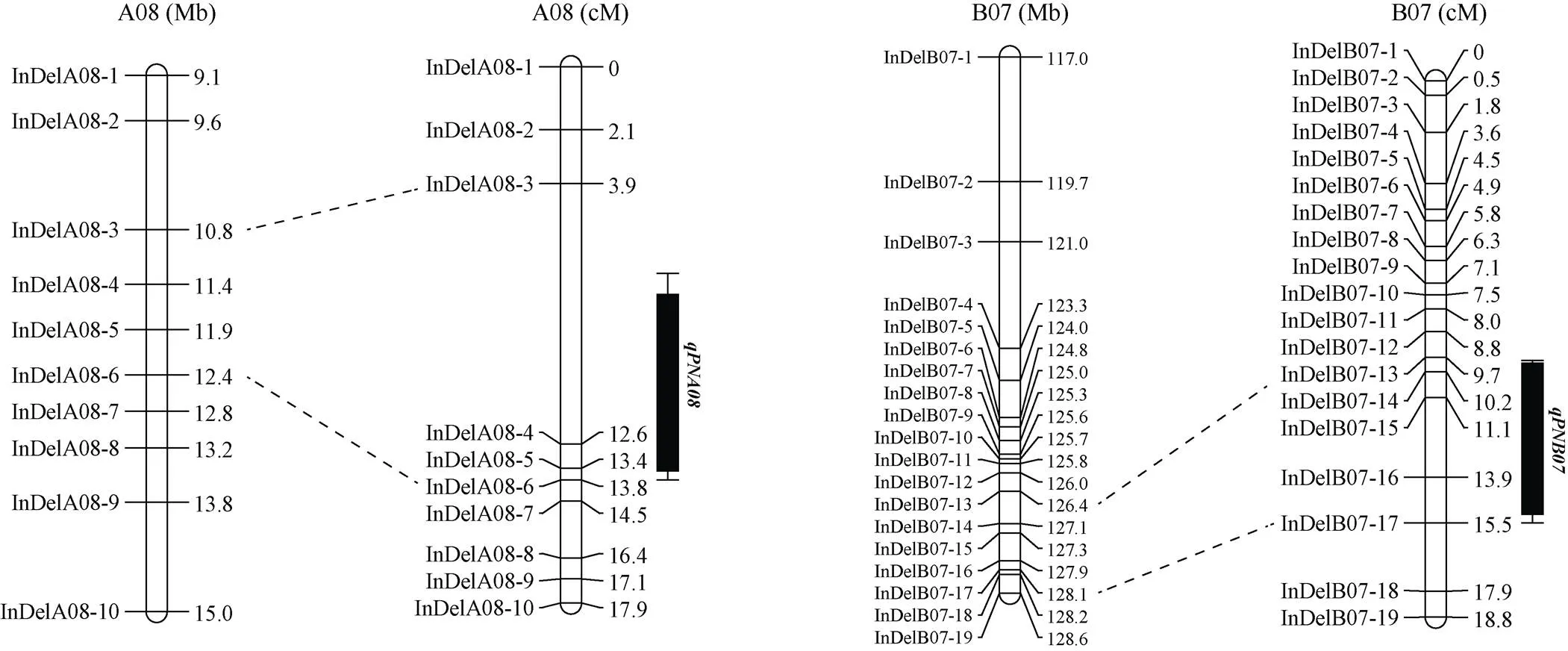
图2 标记加密后的根部结瘤性状主效QTL
2.5 根部结瘤性状QTL加密区间变异位点分析
QTL加密后,区间1.6 Mb内有237个多态性位点, 其中包括198个SNP差异和39个InDel差异。对这些变异位点进行了分析, 接近80%的变异位点位于基因间区, 而在基因内的变异位点中, 存在8个外显子变异位点, 其中有5个变异位点属于非同义突变。区间1.8 Mb内有269个多态性位点, 其中包括199个SNP差异和70个InDel差异。约85%的变异位点位于基因间区, 基因内仅有3个变异位点位于外显子区域, 分别为1个非同义突变、1个翻译提前终止和1个非移码缺失(表4)。

表4 主效QTL候选区间内SNP和InDel注释结果
此外, 加密后的区间和区间各包含88个和80个基因。基于参考基因组基因功能注释, 这2个QTL区间中分别包含6个结瘤素基因, 其中基因、、和在两亲本之间没有差异位点, 其他8个结瘤素基因共有42个变异位点, 包含26个基因间区变异位点和16个基因区变异位点, 重要的变异位点信息见表5。

表5 基因区存在变异位点的结瘤素基因
2.6 根部结瘤性状QTL区间内基因表达分析
利用已发表的花生多组织的转录组数据, 对QTL和区间内的基因进行表达分析。QTL区间内有19个基因在根和根瘤中均不表达, 有8个基因在根和根瘤中基因表达FPKM值均小于2。结瘤素基因和在根中的表达量显著高于根瘤的表达, 而和在根瘤中的表达显著高于根的表达, 并且基因在根中几乎不表达。QTL区间内有22个基因在根和根瘤中均不表达, 有9个基因在根和根瘤中基因表达FPKM值均小于2。结瘤素基因和在根中的表达量显著高于根瘤的表达, 而和在根瘤中的表达显著高于根的表达。同时在根中几乎不表达,在根瘤中几乎不表达(表6)。
3 讨论
花生与根瘤菌互作形成根瘤, 根瘤是花生进行生物固氮的重要场所。本研究连续2年对140份RIL群体及其亲本材料的根部结瘤情况进行调查发现, 亲本材料中花10号和ICG12625根部均有根瘤, RIL群体中出现不结瘤的材料。前人研究分析发现花生不结瘤受2对隐性基因共同调控[17-18]。通过QTL定位分析, 鉴定到2个主效QTL和, 分别位于连锁群A08和B07, 根据加性效应值推测2个主效QTL分别来源于2个亲本。分析不结瘤家系的基因型发现, 父本ICG12625携带的隐性等位基因, 而母本中花10号携带的隐性等位基因。只有2个QTL的隐性等位基因聚合在一起时, 才会抑制根瘤的形成, 导致根部不结瘤。对花生核心种质资源的多年田间观察, 没有观察到不结瘤的材料, 说明双隐性等位基因的组合在自然界中几乎不存在。

表6 主效QTL区间内结瘤素基因在根和根瘤中的表达
基于分子标记和RIL群体的连锁分析是QTL定位研究的重要方法之一。本研究利用前期已构建的SSR分子标记的遗传连锁图[20]和RIL群体的根部结瘤情况, 在A08染色体和B07染色体上分别各定位到一个结瘤性状的主效QTL。但是由于SSR标记的遗传连锁图定位精度较低, 2个QTL在物理图谱上区间还比较大。近年来, 随着高通量测序技术的发展, 基于基因组重测序技术开发SNP标记和InDel标记成为可能。但是由于SNP标记需要通过测序技术检测结果, 而InDel标记的扩增只需通过PAGE胶或者琼脂糖胶检测, 因此InDel标记是成为加密QTL的最快捷有效的标记。本研究利用亲本基因组重测序的数据, 在基于SSR标记检测到的2个主效QTL区间内设计InDel标记进行QTL加密。加密后, 2个主效QTL的LOD值和遗传变异解释率均有所增加, 同时,区间由4.7 Mb缩小至1.6 Mb,区间由9.9 Mb缩小至1.8 Mb。通过InDel标记加密, 将QTL区间均缩小至2 Mb, 这为后续挖掘候选基因提供了理论基础。
根瘤的形成和发育离不开植物基因的调控和参与。结瘤素基因是指一些在根瘤形成发育过程中特异表达并参与共生固氮作用的植物基因, 对根瘤结构的形成及发育、固氮产物的代谢都有重要作用[22]。根据结瘤素基因表达时间的早晚分为早期结瘤素基因()和晚期结瘤素基因()。在根瘤固氮之前表达的植物基因被称为早期结瘤素基因, 一般在根瘤菌侵染几个小时后到根瘤形成、固氮作用之前出现, 主要参与根瘤的形成和发育。而晚期结瘤素基因则在发育完全的、成熟的根瘤中表达, 参与维持根瘤的结构以及共生固氮[23]。的表达参与了皮层细胞分裂, 导致根瘤原基的形成[24-25]; 敲除不影响侵染线的形成, 但会抑制根瘤的形成[26]; 大豆中miR393j-3p可以通过靶向调控而负向调控根瘤的形成[27]。此外, 蒺藜苜蓿中的和[28]、蚕豆中的和[29]、大豆中的[30]均被报道可能涉及豆科植物的根瘤形成和发育。本研究在2个主效QTL区间内均鉴定到8个结瘤素基因在基因区有变异位点, 同时部分结瘤素基因在根和根瘤中的表达有显著差异, 但现有的结果还不能充分确定这些结瘤素基因是否与调控花生根瘤形成的关联性, 还需要后续的试验验证。
4 结论
本研究鉴定到7份根部不结瘤的花生材料, 根部不结瘤家系的叶绿素含量以及株高、单株鲜重、单株干重均显著低于双亲。利用连锁分析, 在A08和B07染色体上各鉴定到1个主效QTL和。
[1] 廖伯寿. 我国花生生产发展现状与潜力分析. 中国油料作物学报, 2020, 42: 161–166. Liao B S. A review on progress and prospects of peanut industry in China., 2020, 42: 161–166 (in Chinese with English abstract).
[2] 吴正锋, 陈殿绪, 郑永美, 王才斌, 孙学武, 李向东, 王兴祥, 石程仁, 冯昊, 于天一. 花生不同氮源供氮特性及氮肥利用率研究. 中国油料作物学报, 2016, 38: 207–213. Wu Z F, Chen D X, Zheng Y M, Wang C B, Sun X W, Li X D, Wang X X, Shi C R, Feng H, Yu T Y. Supply characteristics of different nitrogen sources and nitrogen use efficiency of peanut., 2016, 38: 207–213 (in Chinese with English abstract).
[3] 郑永美, 杜连涛, 王春晓, 吴正锋, 孙学武, 于天一, 沈浦, 王才斌. 不同花生品种根瘤固氮特点及其与产量的关系. 应用生态学报, 2019, 30: 961–968. Zheng Y M, Du L T, Wang C X, Wu Z F, Sun X W, Yu T Y, Shen P, Wang C B. Nitrogen fixation characteristics of root nodules in different peanut varieties and their relationship with yield., 2019, 30: 961–968 (in Chinese with English abstract).
[4] Oldroyd G E, Downie J A. Calcium, kinases and nodulation signalling in legumes., 2004, 5: 566–576.
[5] Smit P, Raedts J, Portyanko V, Debellé F, Gough C, Bisseling T, Geurts R. NSP1 of the GRAS protein family is essential for rhizobia Nod factor-induced transcription., 2005, 308: 1789–1791.
[6] Hirsch S, Kim J, Muñoz A, Heckmann A B, Downie J A, Oldroyd G E. GRAS proteins form a DNA binding complex to induce gene expression during nodulation signaling in., 2009, 21: 545–557.
[7] Ferguson B J, Indrasumunar A, Hayashi S, Lin M H, Lin Y H, Reid D E, Gresshoff P M. Molecular analysis of legume nodule development and autoregulation., 2010, 52: 61–76.
[8] Oldroyd G E, Downie J A. Coordinating nodule morphogenesis with rhizobial infection in legumes., 2008, 59: 519–546.
[9] Krusell L, Madsen L H, Sato S, Aubert G, Genua A, Szczyglowski K, Duc G, Kaneko T, Tabata S, de Bruijn F, Pajuelo E, Sandal N, Stougaard J. Shoot control of root development and nodulation is mediated by a receptor-like kinase., 2002, 420: 422–426.
[10] Searle I R, Men A E, Laniya T S, Buzas D M, Iturbe-Ormaetxe I, Carroll B J, Gresshoff P M. Long-distance signaling in nodulation directed by a CLAVATA1-like receptor kinase., 2003, 299: 109–112.
[11] De Smet I. Lateral root initiation: one step at a time., 2012, 193: 867–873.
[12] Wang T, Guo J, Peng Y, Lyu X, Liu B, Sun S, Wang X. Light- induced mobile factors from shoots regulate rhizobium-triggered soybean root nodulation., 2021, 374: 65–71.
[13] Kanchan K, Anindya K, Zaigam R A, Emeric D, Dany S, Pierre C, Fabienne C, Maitrayee D. Transcriptomic analysis with the progress of symbiosis in ‘crack-entry’ legumehighlights its contrast with ‘infection thread’ adapted legumes., 2019, 32: 271–285.
[14] Sharma V, Bhattacharyya S, Kumar R, Kumar A, Ibanez F, Wang J, Guo B, Sudini H K, Gopalakrishnan S, DasGupta M, Varshney R K, Pandey M K. Molecular basis of root nodule symbiosis between Bradyrhizobium and ‘crack-entry’ legume groundnut (L.)., 2020, 9: 276.
[15] Kinkema M, Scott P T, Gresshoff P M. Legume nodulation: successful symbiosis through short- and long-distance signaling., 2006, 33: 707–721.
[16] Madsen L H, Tirichine L, Jurkiewicz A, Sullivan J T, Heckmann A B, Bek A S, Ronson C W, James E K, Stougaard J. The molecular network governing nodule organogenesis and infection in the model legume., 2010, 1: 10.
[17] Nigam S N, Nambiar P T C, Dwivedi S L, Gibbons R W, Dart P J. Genetics of nonnodulation in groundnut (L.) studies with single and mixed Rhizobium strains., 1982, 31: 691–693.
[18] Essomba N B, Coffelt T A, Branch W D, Van Scoyoc S W. Inheritance of stem color and non-nodulation in peanut., 1991, 18: 126–131.
[19] Peng Z, Liu F, Wang L, Zhou H, Paudel D, Tan L, Maku J, Gallo M, Wang J. Transcriptome profiles reveal gene regulation of peanut (L.) nodulation., 2017, 7: 40066.
[20] Liu N, Chen H, Huai D, Xia F, Huang L, Chen W, Wu B, Ren X, Luo H, Zhou X, Chen Y, Lei Y, Liao B, Jiang H. Four QTL clusters containing major and stable QTLs for saturated fatty acid contents in a dense genetic map of cultivated peanut (L.)., 2019, 39: 23.
[21] Clevenger J, Chu Y, Scheffler B, Ozias-Akins P. A developmental transcriptome map for allotetraploid., 2016, 7: 1446.
[22] van Kammen A. Suggested nomenclature for plant genes involved in nodulation and symbiosis., 1984, 2: 43–45.
[23] Nap J P, Bisseling T. Developmental biology of a plant- prokaryote symbiosis, the legume root nodule., 1990, 250: 948–954.
[24] Gleason C, Chaudhuri S, Yang T, Muñoz A, Poovaiah B W, Oldroyd G E. Nodulation independent of rhizobia induced by a calcium-activated kinase lacking autoinhibition., 2006, 441: 1149–1152.
[25] Tirichine L, Imaizumi-Anraku H, Yoshida S, Murakami Y, Madsen LH, Miwa H, Nakagawa T, Sandal N, Albrektsen A S, Kawaguchi M, Downie A, Sato S, Tabata S, Kouchi H, Parniske M, Kawasaki S, Stougaard J. Deregulation of a Ca2+/calmodulin- dependent kinase leads to spontaneous nodule development., 2006. 441: 1153–1156.
[26] Wan X, Hontelez J, Lillo A, Guarnerio C, van de Peut D, Fedorova E, Bisseling T, Franssen H.andare both involved in nodule initiation and bacteroid development., 2007, 58:2033–2041.
[27] Yan Z, Hossain M S, Arikit S, Valdés-López O, Zhai J, Wang J, Libault M, Ji T, Qiu L, Meyers B C, Stacey G. Identification of microRNAs and their mRNA targets during soybean nodule development: functional analysis of the role of miR393j-3p in soybean nodulation., 2015, 207: 748–759.
[28] Greene E A, Erard M, Dedieu A, Barker D G. MtENOD16 and 20 are members of a family of phytocyanin-related early nodulins., 1998, 36: 775–783.
[29] Vijn I, Yang W C, Pallisgård N, Ostergaard Jensen E, van Kammen A, Bisseling T. VsENOD5, VsENOD12 and VsENOD40 expression during Rhizobium-induced nodule formation onroots., 1995, 28: 1111–1119.
[30] de Blank C, Mylona P, Yang W C, Katinakis P, Bisseling T, Franssen H. Characterization of the soybean early nodulin cDNA clone GmENOD55., 1993, 22: 1167–1171.
Identification of major QTLs for nodule formation in peanut
HUANG Li, CHEN Wei-Gang, LI Wei-Tao, YU Bo-Lun, GUO Jian-Bin, ZHOU Xiao-Jing, LUO Huai-Yong, LIU Nian, LEI Yong, LIAO Bo-Shou, and JIANG Hui-Fang*
Oil Crops Research Institute, Chinese Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture and Rural Affairs, Wuhan 430062, Hubei, China
Peanut is an important oil and economic crop of legumes in China. Nodules are important for symbiotic nitrogen fixation in peanut. Dissecting the genetic basis of nodule formation could promote the understanding of nitrogen fixation ability and characteristics of nodules in peanut. However, there are few studies on nodule formation and the genetic basis of nodule formation remained unclear in peanut. In this study, the nodules were investigated in a high generation RIL population and seven lines were identified without nodule. The chlorophyll content, plant height, fresh weight per plant, and dry weight per plant of the lines without nodule were significantly lower than those of parents. Using the previous genetic linkage map based on SSR markers, major QTLsandwere identified on chromosome A08 and B07, respectively. The InDel markers in the two major QTLs were developed by the genome resequencing data of parents and performed amplification in the RILs. The interval of QTLsandwas reduced to 1.6 Mb and 1.8 Mb, respectively. Meanwhile, the phenotypic variation explained of these two QTLs was increased to 16.4% and 9.9%, respectively. There were four and two nodulin genes variation loci in the two target intervals, respectively. This study provided the understanding for unveiling the genetic basis of formation and development of nodule and symbiotic nitrogen fixation in peanut.
peanut; nodule formation; QTLs mapping; linkage analysis
2023-02-10;
2023-02-22.
10.3724/SP.J.1006.2023.24184
通信作者(Corresponding author):姜慧芳, E-mail: peanutlab@oilcrops.cn
E-mail: huangli5100@126.com
2022-08-09;
本研究由财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-13)和中国农业科学院科技创新工程项目(2022-2060299- 089-031)资助。
This study was supported by the China Agriculture Research System of MOF and MARA (CARS-13) and the Agricultural Science and Technology Innovation Program of Chinese Academy of Agricultural Sciences (2022-2060299-089-031).
URL: https://kns.cnki.net/kcms/detail//11.1809.S.20230221.1719.014.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
