变分自编码的分层解耦卷积脑肿瘤分割网络
2023-05-27苏雅梦
李 锵,苏雅梦,关 欣
变分自编码的分层解耦卷积脑肿瘤分割网络
李 锵,苏雅梦,关 欣
(天津大学微电子学院,天津 300072)
三维卷积神经网络处理图像分割精度高,可以保留更多空间信息,有效解决标签不平衡问题,但存在参数量大的缺点.针对目前三维脑肿瘤分割网络内存资源占用大、硬件设备要求高、计算效率低的问题,将传统3D U-Net网络中的3D卷积替换为分层解耦卷积,能够降低空间环境的计算复杂度和内存占用量,在不提高计算量的前提下显著提高分割精度,提高网络性能.为解决传统自编码器不能自主生成数据的问题,使用结合深度学习和统计学习的变分自编码器,在编码器结果中加入高斯噪声,使得编码器对结果具有鲁棒性,在编码器中加入概率分布防止过拟合,提高算法的泛化性能.采用三线性插值在三维离散采样数据的张量积网格上进行线性插值,有效避免线性方程组不断增大导致计算时间过长的问题.通过对损失函数加权混合,避免梯度弥散时出现学习速率下降现象,解决小区域分割不平衡问题,减少局部性能最优,使网络保持较高运算速度的同时有效提高分割精度,在有限内存空间最大化网络特征提取能力.在脑肿瘤公开数据集BraTS2019上的实验结果表明,该网络在增强型肿瘤、全肿瘤、肿瘤核心上的Dice 值分别可达78.02%、90.05%和83.14%,参数量仅为0.30×106,能够准确、高效地分割出脑肿瘤中各病灶区域,节约硬件设备的算力和内存资源,为临床应用提供可能性.
信号与信息处理;脑肿瘤分割;变分自编码器;三线性插值;分层解耦卷积
脑肿瘤是起源于颅内局部组织细胞增殖形成的新生物,半数以上的脑肿瘤为恶性肿瘤,严重威胁患者的生命和健康[1].最常见的脑肿瘤是胶质瘤,其源自脑组织中的支持性组织胶细胞,根据世界卫生组织(WHO)的分类方法[2]可分为低级别胶质瘤(LGG)和高级别胶质瘤(HGG).放射科医师手工分割脑肿瘤费时且受主观影响,利用医学影像技术可以辅助诊断患者症状并给予治疗.磁共振成像(MRI)是一种用于观察大脑是否有病变的脑部检查,包括4种常见的模态图像:流体衰减反演恢复模态图像(FLAIR)、t1加权图像(T1)、对比度增强t1加权图像(T1ce)和t2加权图像(T2).不同的模态图像具有不同的特点,能够帮助放射科医生提取不同类型的肿瘤信息[3],例如,T1ce模态图像可以观察增强的肿瘤(enhanced tumor,ET),T2模态图像可以识别ET和由坏死和非增强肿瘤组成的肿瘤核心(tumor core,TC),在T2和FLAIR模态图像中,TC的水肿和全肿瘤(whole tumor,WT)表现更为直观.因此,研究半自动或全自动脑肿瘤分割对于脑肿瘤与正常组织的定量分析和评价具有重要意义[4].
近年来,卷积神经网络在医学图像分割领域得到了广泛应用[5-6].其中,U-Net在各种医学图像任务中取得了巨大的成功.Ronneberger等[7]提出的U-Net网络能够在样本数据集相对较小的情况下获得较好的分割结果,成为医学图像分割的主流方法.在此基础上,Dong等[8]采用2D U-Net网络架构,在提高计算效率的同时降低内存使用量,但存在空间上下文缺乏一致性的缺点.Çiçek等[9]使用3D U-Net网络分割脑肿瘤,将整个三维图像输入网络进行训练,该过程不需要进行切片操作,可以有效增强数据,获得较好的分割结果.Chen等[10]利用网络间的信息流引入多路复用模块,减少了时间和空间网络的计算量,但降低了网络的上下文信息.Isensee等[11]降低传统3D U-Net上采样部分的特征维数,使编码器可以训练更多特征,提高了分割精度,但存在内存占用过多、对网络性能要求高等问题.Myronenko[12]使用变分自编码器用于重建原始输入图像,提高分割图像的鲁棒性,但其网络参数量较大,训练时间长,且对硬件设备要求较高.Nazabal等[13]使用变分自编码器利用分布补全缺失数据,通过区分连续、离散以及数字符号,以及处理分类值和序数值等离散值,提高数据信息的完整度.
受上述工作启发,本文提出一种结合变分自编码器(variational auto encoder,VAE)的分层解耦卷积(hierarchical decoupled convolution,HDC)脑肿瘤分割网络HV-Net,旨在平衡网络的分割精度和参数量.首先,在编码阶段,使用变分自编码重采样块将深度学习和统计学习相结合,用概率分布来表示每个潜在特征,有效防止了过拟合问题,提高了算法的泛化性能.其次,为了改善图像内存占用过多和对网络性能要求高的问题,本文提出使用分层解耦卷积块取代3D卷积,可以在空间和信道维度上对标准卷积进行解耦,降低空间环境的计算复杂度和内存占用量.为了提高卷积解码的速度,使用三线性插值(trilinear interpolation)代替编码卷积块中的传统插值,对三维离散采样数据的张量积网格进行三线性插值.最后,为了处理部分较小的脑肿瘤,采用加权混合损失函数避免达到局部极小值和出现稀疏类别不平衡问题.
1 网络模型
1.1 完整网络框架
本文采用减少信道的3D U-Net架构作为网络主要架构,并提出使用一种新型的轻量级分层解耦卷积块取代传统的3D卷积.网络由周期性下移操作(period down-shuffling,PDS)、3D卷积层、分层解耦卷积模块和上采样组成,并添加变分自编码器分支用于重建输入图像,每个HDC模块有分层组卷积(hierarchical group convolution,HGC)和1×3×3的卷积层两部分.上采样由解码卷积块组成,每个解码卷积块有3个卷积单元;周期性下移操作在第1阶段用作下采样,将大小为in×××的高分辨率输入张量in重新排列为大小为out×/2×/2×/2的低分辨率张量out,其中××是in的空间大小,in为通道数.经过PDS操作后,输出空间大小out缩为输入空间的0.5倍,而输出通道数out是in的8倍.此外,在下采样部分,每个编码卷积块都有一次三线性插值[14]运算,同时加入变分自编码器重建输入图像.完整的网络框架如图1所示.在空间域中,通过将3D空间卷积分解为工作在轴向视图、冠状视图和矢状视图3种不同视图上的两个互补2D卷积来引入视图解耦卷积,以便在最小化感知的情况下降低空间复杂性,视图解耦卷积示意如图2所示.在通道域中,对轴向视图的2D卷积应用了一种新的分层组解耦卷积视图,即在具有分层连接特征通道的子组上应用平行轴向视图卷积达到近似3D卷积效果.

图1 结合变分自编码器的解耦卷积脑肿瘤分割网络示意
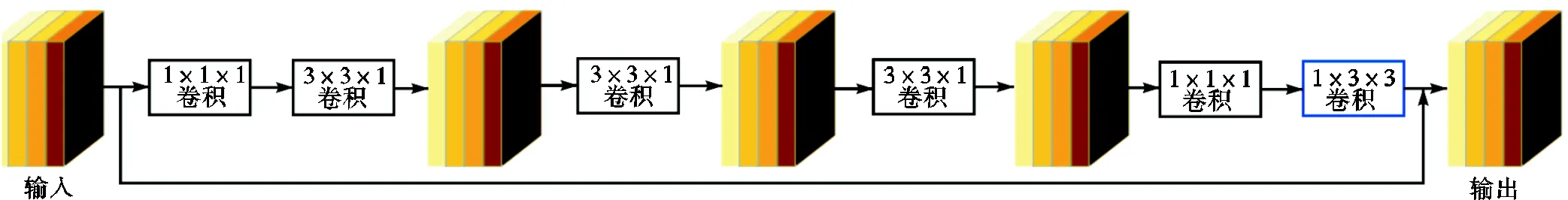
图2 视图解耦卷积示意
1.2 分层解耦卷积模块
本文提出使用的分层解耦卷积模块[15]结构如图3所示,在对多视图和多尺度空间上下文进行编码时,将标准卷积在空间和通道维度上解耦,大大减少计算量.
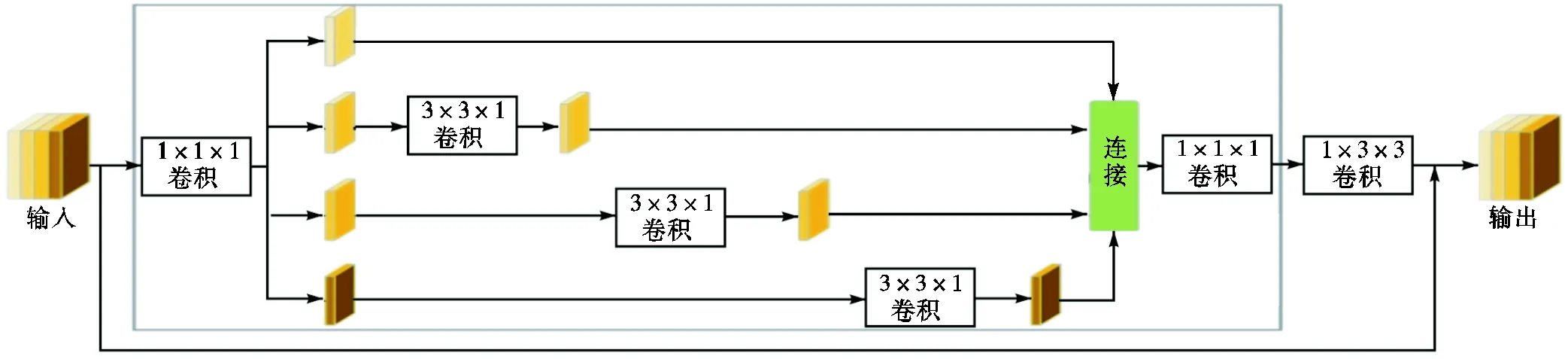
图3 分层解耦卷积模块示意
该模块具有相互连接的多分支结构,同时采用一次和二次卷积,一次卷积应用于并行分支的主卷积,可以更好地在3D体积的焦点视图上分层提取多尺度特征,而二次卷积遵循多分支模块,通过一次卷积来混合多尺度输出,其在互补视图上允许大量输入,能更好地获得远距离空间上下文特征,此外,模块中加入额外下采样,从而能允许大量输入并更好地获得远距离的空间上下文.HDC模块显著减少了用于子序列处理的数据量的大小,同时不考虑下移因素,能利用输入中所有可用的信息.笔者将HDC直接应用到输入数据,其输出将被用作子序列CNN的输入,同时在上采样的解码卷积块中使用三线性插值替代传统线性插值方式,利用被插值位置距离最近的2×2×2个立方体的已知像素点来计算缺失值,提高计算效率,减小计算时间.三线性插值函数示意如图4所示.
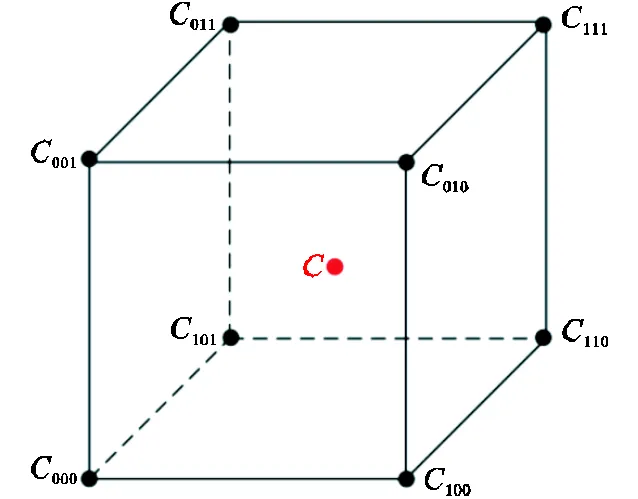
图4 三线性插值函数示意
1.3 变分自编码器
变分自编码器[16]本质是在常规自编码器[17]的基础上增加一个编码器,将对应计算均值的编码器结果加上高斯噪声,使得解码器对噪声具有鲁棒性.为保证计算均值编码器的结果具有零均值,加入KL loss(Kullback-Leibler loss)作为编码器的正则项;另一个对应计算方差的编码器用来动态调节噪声的强度.变分自编码器的结构如图5所示,其中编码器部分用概率分布来表示每一个潜在的特征,能够有效防止过拟合,提高算法的泛化性能;解码器分支在接收编码器输出的同时产生原始输入的重建图像.

图5 变分自编码示意
此外,变分自编码器为每个样本构造专属的正态分布,使得所有的后验分布趋向于标准正态分布,有效防止了噪声为低噪声,KL loss增加,从而重建误差下降,更容易拟合;反之,如果重建误差小于KLloss,即解码器已经得到一定训练,KL loss减少,噪声就会增加,重建误差又开始增加,拟合就更加困难,此时解码器会提升生成能力来平衡重建过程.
使用变分自编码器对输入图像进行重建时,从编码器末端输出开始,本文将输入减少到256的低维空间,从具有给定均值和标准的高斯分布中抽取样本,并按照与解码器相同的架构重建为输入图像维度,不使用来自编码器的跳跃连接,本文使用变分自编码器作为网络分支用于原始图像重建,其结构如表1所示,其中conv代表卷积操作,ReLU为激活函数,GN代表群组归一化(group normalization),FC代表全连接层(fully connected-layer).
表1 变分自编码器分支结构

Tab.1 Structureof variational autoencoding branch
1.4 加权混合损失函数
为了获得分割图像更高置信度的清晰边界,解决脑肿瘤在整个脑部MRI图像中占比小的问题,本文网络提出使用一种加权混合损失函数[18]以减小分割图像与标注图像之间的差异,同时也解决损失函数因为脑肿瘤过小达到局部最小值的问题,避免了稀疏类别不平衡.
焦点损失(focal loss)函数在交叉熵损失(cross entropy loss)函数基础上进行修改,解决了目标检测中正负样本比例严重失衡的问题,其表达式为



(3)
式中:为骰子损失函数;为标签的类别总数;为某个样本的总像素点数;p为第个像素点的概率分布;p为第个元素被预测为类别的概率;g为p对应的标签值.将每个类的dice loss求和取平均,可得到最后的Dice系数.
由于脑肿瘤区域存在高度不平衡的细分,因此本文还使用了广义骰子损失(generalized dice loss,GDL)函数.GDL数学表达式为

式中w为不同的标签集属性提供不变性,定义为

每个标签之间的贡献将通过其体积的倒数进行校正.
基于以上,当目标区域存在高度不平衡的细分时,待分割区域占据整个影像的一小部分,容易造成性能上的局部最优,本文提出加权混合损失函数来平衡这种问题,以提高分割精度.其中平衡因子和用来平衡焦点损失和GDL的权值,实验表明=0.3和=0.7时效果较好.
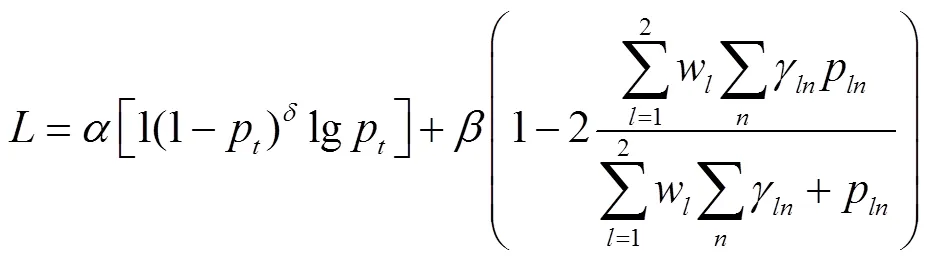
2 实验与结果
2.1 实验环境与数据集
本文的实验环境为CPU Intel® Core i9-9900X 3.5GHz,GPU Nvidia RTX2080Ti (11GB)×4,Ubuntu 16.04 操作系统,使用了开源Pytorch深度学习框架.本文参数设置如表2所示,批处理量为8,最大训练迭代次数为500.
实验数据来自于BraTS2019数据集[20],其中训练集有335个病例,包含259个HGG患者样本,76个LGG患者样本,验证集包含127个无标签患者样本. 每个病例都有4种模态,每种模态的图像大小均为240×240×155×1,真值标签由专业医师共同手动标定,医师将脑肿瘤区域分为背景(background,标记为0)、坏死核心和非增强肿瘤(necrotic and non-enhancing tumor,标记为1)、水肿区域(peritumoral edema,标记为2)和增生性肿瘤(enhancing tumor,标记为4)4类.
表2 实验参数设置

Tab.2 Settings of experimental parameters
2.2 实验评价指标
通常以Dice相似系数、Jaccard相似系数和豪斯多夫(Hausdorff)距离评价分割有效性,以网络的参数量(parameter)和浮点计算量(floating point of operations,FLOPs)评价网络复杂度.
Jaccard相似系数可用来比较预测肿瘤区域和实际肿瘤区域两个集合的差异性,Dice相似系数可用来计算预测肿瘤区域和实际肿瘤区域两个集合的相似性,是分割精度的主要决定因素,表达式为


豪斯多夫距离可用来评价图像边缘点的最大不匹配程度,数学表达式为


式中:为预测结果集;为医师标记的标签集;、分别为两个集合中的元素.
网络的参数量对应算法的空间复杂度,浮点计算量对应算法的时间复杂度,参数量和浮点计算量FP的表达式为
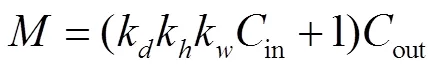

式中:k、k和k分别为卷积核的深度、高度和宽度;in和out分别为输入和输出通道数;、和分别为图像的深度、高度和宽度.
2.3 实验结果与分析
本文的脑肿瘤图像分割实验均在BraTS2019数据集上进行,并在该数据集的在线服务器上进行测试以验证网络的有效性.
为验证本文使用的加权混合损失函数参数对网络性能和分割结果的影响,设置5组不同的取值进行5次实验.表3展示了在参数不同取值下,本文网络在数据集上的分割精度,可以看出当=0.3、=0.7时在测试集中的ET、WT、TC 3个指标上结果为78.02%、90.05%和83.14%,较其他取值时的结果均有提升.在=0.1、=0.9的ET区没有显著差异,在WT和TC两个区域上差距较大.
本文使用3D U-Net作为实验的基准网络,依次加入不同改进模块验证其分割效果.由表4可以看出,使用分层解耦卷积后,网络的整体性能均有所提高,尤其在参数量方面,网络参数量由16.21×106缩至0.29×106,即基准网络3D U-Net参数量是HDC-Net的50倍,说明使用分层解耦卷积能很大程度降低计算成本.加入变分自编码器后,WT的值提升了1.43%,表明VAE能够对编码器产生正则作用,提升整体分割效果.使用三线性插值网络对TC和ET的分割效果提升了1.13%和1.01%,提高网络对较小脑肿瘤的分割效果.融合所有改进模块即为本文所提的HV-Net.
表3 网络在不同参数下的性能对比

Tab.3 Comparison of network performance under dif-ferent parameters
表4 不同模块对网络分割性能影响

Tab.4 Comparison of different modules of HV-Net
为验证本文提出的网络性能和分割结果,将本文所提网络分别与3D U-Net[9]、3D ESPNet[21]、Deep-Medic[22],MFNet和DMFNet[23]等深度学习算法对脑肿瘤图像分割结果进行对比,对比结果如表5所示.由表5结果可知,本文提出的网络在ET、WT、TC上的Dice值别达到了78.02%、90.05%和83.14%,网络的参数量仅有0.30×106.实验结果表明,本文所提网络能够在算法的分割精度保持较高的情况下,较为有效地降低网络参数量.
表5 不同算法在脑肿瘤图像分割中的性能对比

Tab.5 Comparison of different algorithms in brain tumor image segmentation
图6为部分病例使用本文网络的分割结果,分别为胶质瘤在大脑水平面、矢状面和冠状面视图.通过流体衰减反演恢复模态图像和t1加权图像可以看出脑肿瘤各个区域的边界,其中GT(ground truth)代表真实标签,与真实标签对比可知本文网络在该病例TC和ET的预测结果与真实标签相差较小.
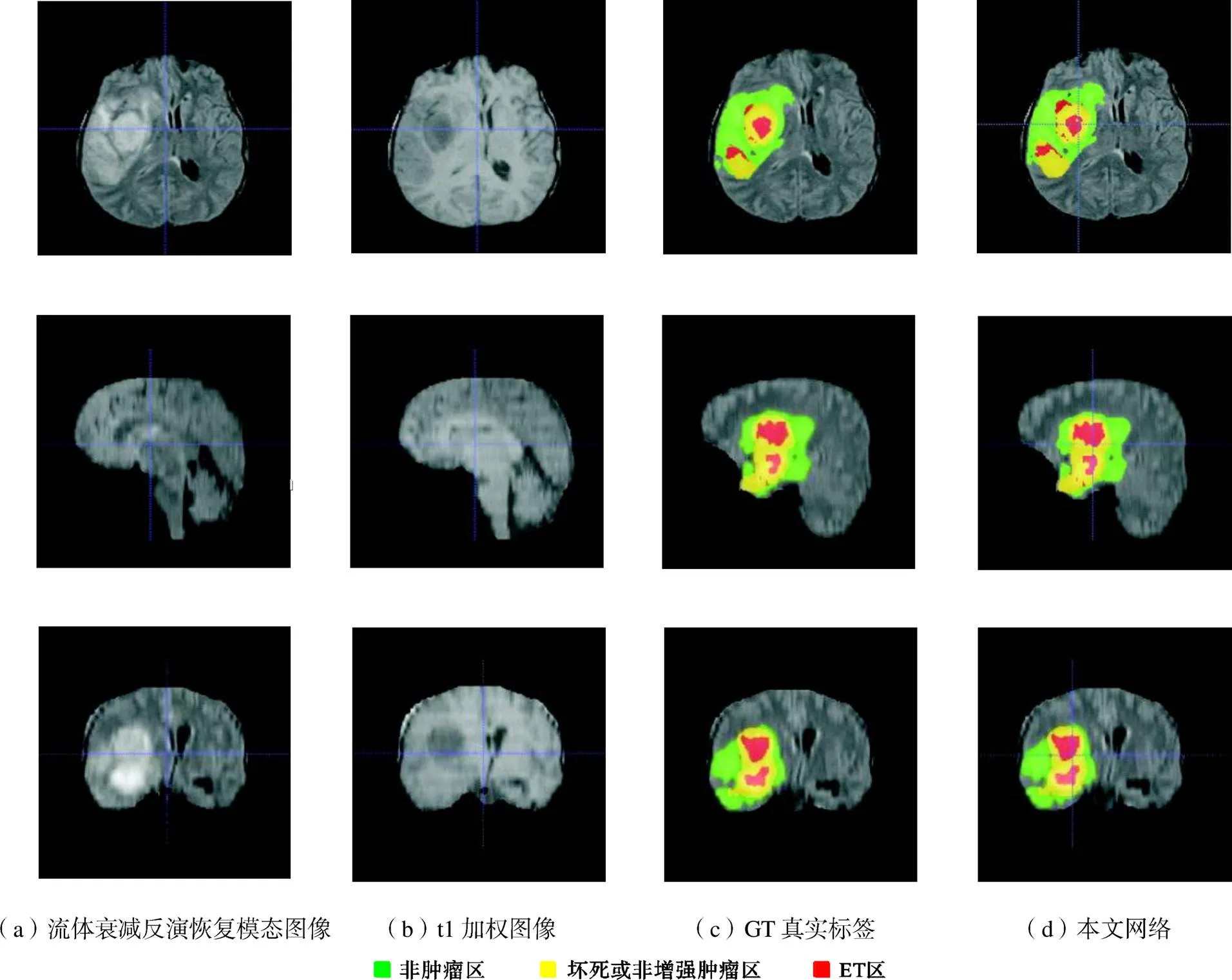
图6 分割结果
3 结 语
本文提出一种结合变分自编码器的分层解耦卷积脑肿瘤分割网络,提高MRI序列中对脑肿瘤各个区域的检测精度.通过使用变分自编码器,对编码器起到正则的效果,利用三线性插值极大地减小了插值计算的总量,解决因为插值计算导致网络计算量增大的问题,同时,本文提出一种加权混合损失函数,提高小目标分割精度,使得在训练过程中能够提高分割效果.
在BraTS2019数据集实验结果表明,本文所提出的网络在增强型肿瘤、全肿瘤、肿瘤核心上平均Dice值得分别达到了78.02%、90.05%和83.14%,参数量仅为0.30×106.从最终的分割精度和算法整体复杂度来看,本文网络优于经典3D U-Net[9]、3D ESPNet[21]、DeepMedic[22]、MFNet和DMFNet[23]等算法,具有计算量小、运行速度快、分割精度高等优点,能够为临床医师分割脑肿瘤提供有效参考.
[1] Menze B H,Andras J,Stefan B,et al. The multimodal brain tumor image segmentation benchmark (BRATS) [J]. IEEE Transactions on Medical Imaging,2015,34(10):1993-2024 .
[2] Louis D N,Perry A,Reifenberger G,et al. The 2016 World Health Organization classification of tumors of the central nervous system:A summary[J]. Acta Neuropathologica,2016,131(6):803-820 .
[3] Naceur M B. Fully automatic brain tumor segmentation using end-to-end incremental deep neural networks in MRI images[J]. Computer Methods and Programs in Biomedicine,2018,166:39-49 .
[4] Wang J,Gao J,Ren J,et al. DFP-ResUNet:Convolutional neural network with a dilated convolutional feature pyramid for multimodal brain tumor segmentation[J]. Computer Methods and Programs in Biomedicine,2021,208:106208.
[5] 李 锵,武昱忻,关 欣,等. 基于 SRD 感受野自调节的肺炎病灶检测[J]. 天津大学学报(自然科学与工程技术版),2021,54(5):508-516.
Li Qiang,Wu Yuxin,Guan Xin,et al. Pneumonia focus detection based on SRD with receptive field self-regulation[J]. Journal of Tianjin University(Science and Technology),2021,54(5):508-516(in Chinese).
[6] ZhangH L,LiQ,GuanX,et al. An improved three-dimensional dual-path brain tumor image segmentation network[J]. Acta Optica Sinica,2021,41(3):60-67.
[7] Ronneberger O,Fischer P,Brox T. U-net:Convolu-tional networks for biomedical image segmentation[C]// International Conference on Medical Image Computing and Computer-Assisted Intervention. Munich,Ger-many,2015:234-241.
[8] Dong H,Yang G,Liu F,et al. Automatic brain tumor detection and segmentation using U-Net based fully convolutional networks[C]//Annual Conference on Medical Image Understanding and Analysis. Edinburgh,UK,2017:506-517.
[9] Çiçek Ö,Abdulkadir A,Lienkamp S S,et al. 3D U-Net:Learning dense volumetric segmentation from sparse annotation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Athens,Greece,2016:424-432.
[10] Chen Y,Kalantidis Y,Li J,et al. Multi-fiber networks for video recognition[C]//Proceedings of the European Conference on Computer Vision (ECCV). Munich,Germany,2018:352-367.
[11] Isensee F,Kickingereder P,Wick W,et al. No new-net[C]//International MICCAI Brainlesion Workshop. Granada,Spain,2018:234-244.
[12] Myronenko A. 3D MRI brain tumor segmentation using autoencoder regularization[C]//International MICCAI Brainlesion Workshop. Granada,Spain,2018:311-320.
[13] Nazabal A,Olmos P M,Ghahramani Z,et al. Handling incomplete heterogeneous data using VAES[J]. Pattern Recognition,2020,107:107501.
[14] Rajon D A,Bolch W E. Marching cube algorithm:Review and trilinear interpolation adaptation for image-based dosimetric models[J]. Computerized Medical Imaging and Graphics,2003,27(5):411-435 .
[15] Luo Z,Jia Z,Yuan Z,et al. HDC-Net:Hierarchical decoupled convolution network for brain tumor segmentation[J]. IEEE Journal of Biomedical and Health Informatics,2020,25(3):737-745 .
[16] Han K,Wen H,Shi J,et al. Variational autoencoder:An unsupervised model for encoding and decoding fMRI activity in visual cortex[J]. NeuroImage,2019,198:125-136 .
[17] Hinton G E,Zemel R S. Autoencoders:Minimum description length and helmholtz free energy[J]. Advances in Neural Information Processing Systems,1994,6:139 .
[18] Ding S,Zhang X,An Y,et al. Weighted linear loss multiple birth support vector machine based on information granulation for multi-class classification[J]. Pattern Recognition,2017,67:32-46 .
[19] Milletari F,Navab N,Ahmadi S A. V-net:Fully convolutional neural networks for volumetric medical image segmentation[C]//2016 Fourth International Conference on 3D Vision (3DV). Palo Alto,USA,2016:565-571.
[20] Henry T,Carré A,Lerousseau M,et al. Brain tumor segmentation with self-ensembled,deeply-supervised 3D U-net neural networks:A BraTS 2020 challenge solution[C]//International MICCAI Brainlesion Workshop. Lima,Peru,2020:327-339.
[21] Nuechterlein N,Mehta S. 3D-ESPNet with pyramidal refinement for volumetric brain tumor image segmentation[C]//International MICCAI Brainlesion Workshop. Granada,Spain,2018:245-253.
[22] Kao P Y,Ngo T,Zhang A,et al. Brain tumor segmentation and tractographic feature extraction from structural MR images for overall survival prediction[C]// International MICCAI Brainlesion Workshop. Granada,Spain,2018:128-141.
[23] Chen C,Liu X,Ding M,et al. 3D dilated multi-fiber network for real-time brain tumor segmentation in MRI[C]// International Conference on Medical Image Computing and Computer-Assisted Intervention. Shenzhen,China,2019:184-192.
A Variational Autoencoding-Hierarchical Decoupled Convolutional Brain Tumor Segmentation Network
Li Qiang,Su Yameng,Guan Xin
(School of Microelectronics,Tianjin University,Tianjin 300072,China)
Three-dimensional convolutional neural networks have the advantages of high segmentation accuracy,can effectively solve the unbalanced label problem,and retain more spatial information. However,they involve complex calculations and several parameters. In view of the 3D brain tumor segmentation network’s high memory resource usage,high-hardware requirements,and the problem of low efficiency of computation,we propose a pseudo 3D U-Net module to replace the traditional 3D convolution with a novel hierarchical decoupled convolution module,which significantly reduces the usage of memory and computation,minimizes the perception while reducing the computational complexity of the space environment,segments the 3D stereo image at once,and significantly improves the segmentation accuracy without increasing the computational cost. To solve the problem that the hidden layer output of the traditional autoencoder is easily to be chaotic and cannot generate data independently,we propose a variational autoencoder that combines deep learning and statistical learning. Gaussian noise is added to the encoder result to make the encoder robust to the result,and the probability distribution is added to the encoder to prevent over-fitting and improve the generalization performance of the algorithm. We use trilinear interpolation to perform linear interpolation on the tensor product grid of 3D discrete sampling data,which effectively avoids the long calculation time caused by the continuous increase in the linear equation system during the calculation. By weighing and mixing the three-loss functions,this study prevent the learning rate decrease caused by the gradient dispersion in a gradient descent calculation,solves the imbalance of small region segmentation,and ensures that the algorithm can effectively improve the segmentation accuracy while maintaining a high computing speed. This study uses the brain tumor open datasetBraTS2019 for testing,and the average Dice_ET,Dice_WT,and Dice_TC reached 78.02%,90.05%,and 83.14%,respectively,with only 0.30×106of the parameters. The results show that this network can accurately and efficiently segment each lesion region of a brain tumor,save the computing power and memory resources of hardware equipment,and provide the potential for clinical application.
signal and information processing;brain tumor segmentation;variational autoencoder;trilinear interpolation;hierarchical decoupled convolution
10.11784/tdxbz202203042
TP391.41
A
0493-2137(2023)07-0767-08
2022-03-21;
2022-05-30.
李 锵(1974— ),男,博士,教授,liqiang@tju.edu.cn.
关 欣,guanxin@tju.edu.cn.
国家自然科学基金资助项目(62071323,61471263,61872267);天津市自然科学基金资助项目(16JCZDJC31100);天津大学自主创新基金资助项目(2021XZC-0024).
Supported by the National Natural Science Foundation of China(No.62071323,No.61471263,No.61872267),the National Natural Science Foundation of Tianjin,China(No.16JCZDJC31100),the Seed Foundation of Tianjin University(No.2021XZC-0024).
(责任编辑:孙立华)
