澳洲坚果种植地土壤固氮微生物群落结构和多样性
2023-05-23王瑞丽施蕊雷娥李雕益包书军熊智
王瑞丽 施蕊 雷娥 李雕益 包书军 熊智






摘要:采用qPCR和高通量测序技术对固氮相关基因(nifH)进行检测,探究农业果园经营形式下土壤固氮微生物的群落结构、多样性及其影响因素。结果表明,种植地pH值在5.0左右,属于典型的酸性土。综合分析发现,澳洲坚果园种植地带养分含量明显升高,并且种植区域养分含量也存在较大差异,说明土壤肥力分布不均。与未种植区域相比,种植区域固氮细菌(相关基因nifH)群落丰富度和多样性指数均呈下降趋势。变形菌门(Proteobacteria)为含nifH基因的固氮细菌群落优势菌门,相对丰度为18.21%~79.69%,并且种植使土壤固氮菌群落结构发生了改变。实时荧光定量PCR结果表明,固氮相关基因(nifH)拷贝数在7.325×106~5.120×107之间。土壤理化因素与功能基因拷贝数相关性结果表明,nifH基因拷贝数与AK、OP、NH+4-N、NO-3-N、TN、OM等的含量均呈极显著负相关。澳洲坚果种植地中固氮细菌以变形菌门(Proteobacteria)为最优势的菌群,这为澳洲坚果种植地土壤肥力恢复和固氮微生物的开发利用提供了理论支持。
关键词:澳洲坚果园;qPCR;高通量测序;固氮细菌;nifH;群落結构;多样性
中图分类号: S154.3 文献标志码: A
文章编号:1002-1302(2023)08-0211-06
基金项目:国家自然科学基金(编号:3166010405);云南省重大科技专项(编号:202002AA1007)。
作者简介:王瑞丽(1995—),女,贵州安顺人,硕士研究生,主要从事澳洲坚果功能微生物研究。E-mail:1393384429@qq.com。
通信作者:熊 智,博士,教授,主要从事微生物学和分子生物学研究。E-mail:zhix-swfu@qq.com。
氮元素是植物生长的必需营养元素之一,对植物光合作用和增产起关键作用[1]。土壤固氮微生物是一类能将大气中游离氮转化为含氮有机物的原核微生物,其数量和群落结构的变化与土壤氮素循环密切相关[2-3]。固氮微生物的固氮作用主要依靠其体内的固氮酶,它们都依赖于nifH编码固氮酶铁蛋白亚基的基因,可广泛用于检测不同环境下固氮作用的基因[4]。大量nifH基因的研究表明,土壤有机质和氮肥会显著影响固氮微生物的物种数量和群落多样性[5]。田永辉等的研究表明,种植年限会影响茶树根际固氮微生物的群落组成和多样性[6]。陈秀波研究发现,植被种类、土壤理化性质、土壤施肥措施等因素会影响固氮微生物群落和多样性的变化[7]。张苗苗等分别利用荧光定量PCR和末端限制性片段长度多样性(T-RFLP)技术研究了长期不同施肥处理对固氮基因的丰度和群落多样性的影响,结果表明,与不施肥处理相比,稻草还田和施氮磷钾(NPK)处理都能显著增加固氮基因的丰度,而秸秆配施NPK处理的nifH基因丰度最高,因而可以增加土壤固氮能力;此外,长期不同施肥处理显著改变了群落结构,但是对群落多样性没有显著改变[8]。Wu等研究了施氮量和水稻品种对根系固氮细菌多样性的影响,结果表明,氮肥和水稻品种都会显著影响根系土壤固氮微生物的群落结构,β-变形菌是最主要的水稻根系固氮菌,施用氮肥会显著降低α-变形菌的相对丰度[9]。Tan等的T-RFLP研究结果表明,施氮肥15 d后水稻根系固氮微生物群落迅速发生改变,即固氮微生物群落受到氮肥的影响,环境条件和水稻品种对根系固氮微生物群落有显著影响[10]。固氮的富集改变了功能性固氮基因的丰度,固氮菌的数量越大,根际氮输入量越大,有利于植物入侵。大量林地生态系统研究表明,氮循环微生物受pH值、土层深度和季节等因素影响。柳建银研究发现,同一领域的红树林土壤样品中,固氮微生物群落组成存在差异[11]。Mergel等通过靶定nifH基因,探究德酸性林地土壤固氮细菌种群,发现固氮菌群数量在0~5 cm土层最高,并随土壤深度增加而降低[12]。Zhou等的研究表明,温度对森林土壤功能菌群变化方面比其他拟议的环境驱动因素起着更主要的作用[13]。
云南省是我国最大的澳洲坚果种植区,主要分布于西双版纳、普洱、临沧、保山、德宏等地市州。目前,澳洲坚果已经成为云南部分地区脱贫攻坚、乡村振兴的重点产业之一[14]。云南省近年大力发展高效林果业,目的是改善澳洲坚果林的生态环境,最终实现澳洲坚果优质高产。并且,澳洲坚果树是云南省主要代表果树种之一,具有滋养水土的作用,对土壤肥力改善和土壤生态恢复具有积极的影响。目前关于人工农业果林生态系统中土壤固氮微生物的研究相对缺乏。
本研究以未种植澳洲坚果树区域与种植区域土壤为研究对象,探讨了种植澳洲坚果树对土壤理化性质、土壤养分和群落特征等方面的影响,并且从基因水平上进一步研究土壤微生物营养元素循环提供参考。另外,可以比较种植地再利用方式的效果,为生态修复、功能微生物开发与利用、土壤肥力恢复等提供科学基础。
1 材料与方法
1.1 土壤样品的采集与处理
土壤样品于2019年6月在云南省江城县嘎勒村澳洲坚果种植基地采集。树种选取2013年种植的澳洲坚果树,土壤采集方法是按“S”形路线取样法,土壤深度为5~30 cm,每棵树分别在东西南北4个方向采集等量的土样,并去除土样中的杂质,混匀后装入高温灭菌的采样袋中,分装2袋,一袋放在-80 ℃冰箱保存,另一袋4 ℃保存用于测定土壤养分。共取4个区域的土样,A0表示非种植区域的土样,A1、A2、A3代表种植地带的土样(表1)。
1.2 试验方法
1.2.1 土壤养分测定 土壤pH值:分别取1袋土样置于pH值为4.01、6.86、9.18的标准缓冲液中,蒸馏水定容至100 mL,摇匀即配制成相应的标准液,然后使用智能型汉显多功能养分速测仪测定土壤pH值。
土壤速效钾(AK)、有效磷(OP)、铵态氮(NH+4-N)和有机质(OM)含量:根据智能型汉显多功能养分速测仪所提供的试剂配制标准溶液,使用特定的显色剂与标准溶液发生显色反应,然后按照相应的操作步骤测定其含量。
硝态氮(NO-3-N)含量:取5 g风干土样于三角瓶内,加入0.5 g CaSO4·2H2O和100 mL蒸馏水,盖塞后在振荡机上振荡10 min。放置5 min后将上清液过滤于干燥洁净的三角瓶内。吸取25 mL上清液于 100 mL 显色管中,分别加入0.05 g CaCO3、酚二磺溶液2 mL、纯水10 mL,1 ∶1氨水先加至溶液呈黄色,再补加3 mL,定容至100 mL后在分光光度计上于410 nm处比色。
1.2.2 土壤固氮微生物群落结构和丰度的测定
1.2.2.1 DNA提取 从-80 ℃冷柜中称取根际土壤0.2 g,按照土壤总DNA提取试剂盒的步骤提取总DNA,DNA纯度和浓度用NanoDrop2000进行检测,DNA提取效果用琼脂糖凝胶电泳技术检测,检测能跑出清晰条带的DNA方可进行下一步试验。
1.2.2.2 高通量分析 根据查得的固氮作用nifH引物序列扩增要求,对提取成功的土壤总DNA进行PCR试验,每个样品3个重复。正向引物F:5′-[JP9]AAAGGYGGWATCGGYAARTCCACCAC-3′;反向引物 R:5′-[JP9]TTGTTSGCSGCRTACATSGCCATCAT-3′。 PCR反應体系:5×FastPfu Buffer缓冲液4 μL,2.5 mmol/L dNTPs 2 μL,正反向引物各0.8 μL,FastPfu聚合酶 0.4 μL,BSA 0.2 μL,DNA模板 10 ng,双蒸水补至20 μL。PCR反应条件:95 ℃ 3 min;95 ℃ 30 s,53 ℃ 30 s,75 ℃ 45 s,37个循环;72 ℃ 10 min,4 ℃保存。由上海美吉生物医药科技有限公司利用美吉生物服务平台完成氮循环功能基因的测序。
1.2.2.3 实时荧光测定PCR 标准曲线制备:每个功能基因实时荧光定量的质粒均按照10倍稀释梯度建立,90 μL稀释液和10 μL质粒,通过对各功能基因预试验,nifH功能基因取10-8~10-2稀释液绘制。反应体系共20 μL:qPCR反应混合物16.5 μL,上下游引物0.8 μL,DNA模板2 μL。PCR循环条件:95 ℃ 3 min;95 ℃ 5 s,58 ℃ 30 s,75 ℃ 1 min,40个循环。完成DNA聚合酶链式反应后,将所有样本放入荧光定量 PCR 仪中反应。定量PCR反应完成后设置溶解曲线用以检验产物的特异性。
1.2.2.4 结果计算 拷贝数(copies/μL)=DNA浓度(ng/μL)×10-9×6.02×1023/(分子量×660),式中分子量指载体的大小加上目的基因的片段大小。
2 结果与分析
2.1 土壤指标分析
由表2可知,未种植区域土壤中AK、OP、NH+4-N、NO-3-N、TN、OM含量明显低于种植区域,差异显著(P<0.05);种植区域各土样除TN含量差异不显著外,其他理化指标都存在显著差异(P<0.05),说明种植区土壤肥力不均。
2.2 参与固氮作用的nifH基因群落结构和多样性
2.2.1 nifH基因群落分类和多样性
高通量测序结果(表3)显示,在种植区域与未种植区域中一共得到74 397条有效序列,其中未种植区域得到 13 661 条有效序列,种植区域每个样本平均获得 20 245 条,测序量能够满足后续的分析需要。未种植区域有效序列条数明显低于种植区域,操作分类单元(OTU)数量明显高于种植区域。所采土样覆盖度均在99.59%以上,说明土样中的固氮细菌均能被检测到,即所测土样中固氮细菌可以完全反映土样情况。
根据相似度对所获得的有效序列进行OTU划分。所有样本序列共分为1 334种OTU,通过与nifH型数据库比对,这1 334种OTU分别归属于3域3界8门13纲21目25科29属38 种。从总体来看,未种植区域固氮作用nifH基因OTU数量最多,说明种植会降低OTU数量,种植区域各土样OTU数量间也存在显著差异,说明土壤肥力不均。
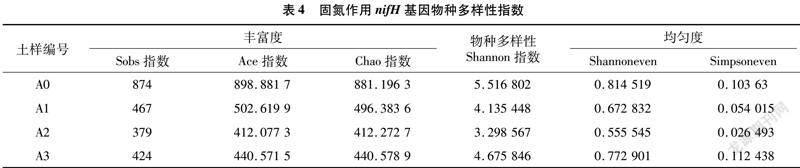
由各样本的多样性指数(表4)可知,未种植区域参与固氮作用的nifH基因的物种丰富度指数明显比种植区域高,说明种植会降低物种丰富度。与未种植区域相比,种植区域物种多样性指数明显降低,并且种植区域间也存在差异,说明种植区域由于长期大量施氮造成土壤肥力相差较大,从而降低了固氮微生物nifH的多样性。从2个均匀度指数可以看出,种植区域土壤的均匀度低于未种植区域,表明种植会降低固氮细菌nifH的均匀度,同时种植区域之间也存在差异,有可能是施肥不均造成。
2.2.2 nifH基因韦恩图 为了便于比较,对种植区域与未种植区域所采土样进行核心OTU分析(即在给定组内所有样品之间共享OTU),它们对应于4组,包括未种植区域(A0)、种植区域(A1、A2、A3)(图1)。未种植区域仅有526个核心OTU局限于单独的组,而种植区域分别有129、78、102个核心OTU分别局限于单独的组,共有89个核心OTU为所有组共享的,种植区域中所采样本与未种植区域共享的OTU分别为205、182、246个,说明种植澳洲坚果树会严重影响土壤固氮微生物群落的相似性。
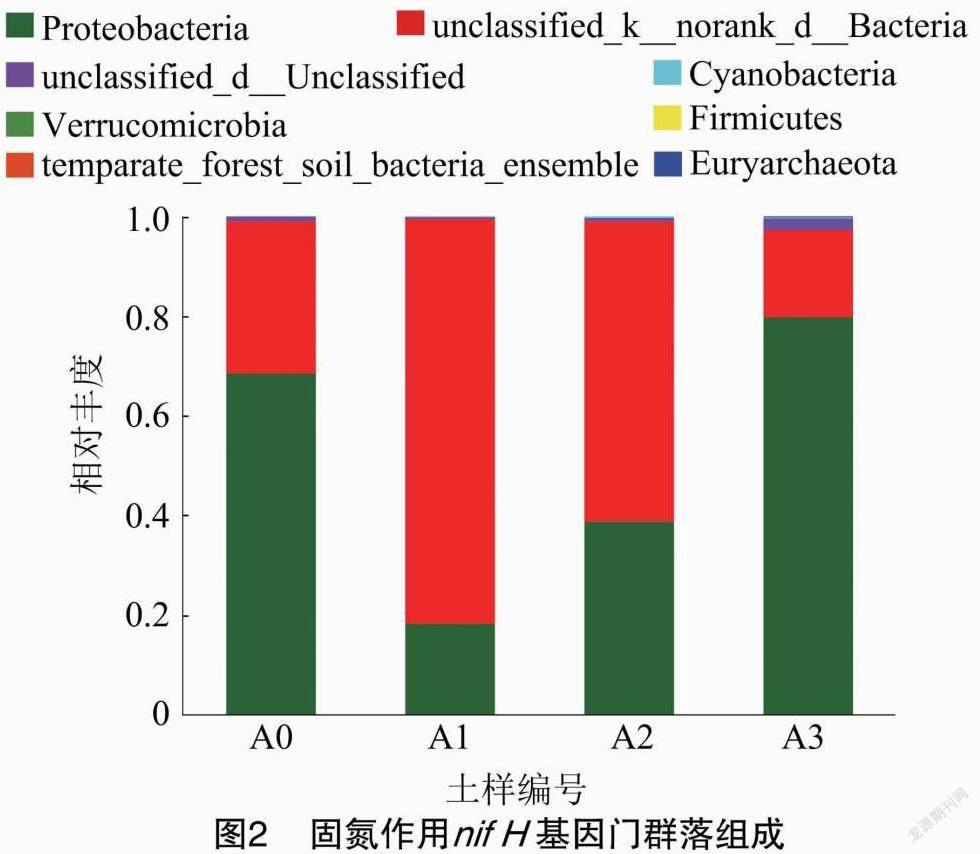
2.2.3 nifH基因的群落柱形图 由图2可知,在门水平上,固氮作用nifH基因的群落共有8类物种组成,其中变形菌门(Proteobacteria)是主要物种,在各个土样中的相对丰度介于18.21%~79.69%之间,尤其在A0、A3土样中分别达到68.44%、79.69%,而在A1、A2土样中分别只有18.21%、38.70%。其次是未分类的物种,相对丰度范围为17.60%~81.29%,所占比例只比变形菌门低15.22%。在A1、A2、A3土样中还分布着蓝藻门(Cyanobacteria),相对丰度分别为0.13%、0.32%、0.18%,同时在A3土样中还发现了厚壁菌门(Firmicutes)、疣微菌门(Verrucomicrobia)和广古菌门(Euryarchaeota),相对丰度分别为0.05%、0.14%、0.02%。
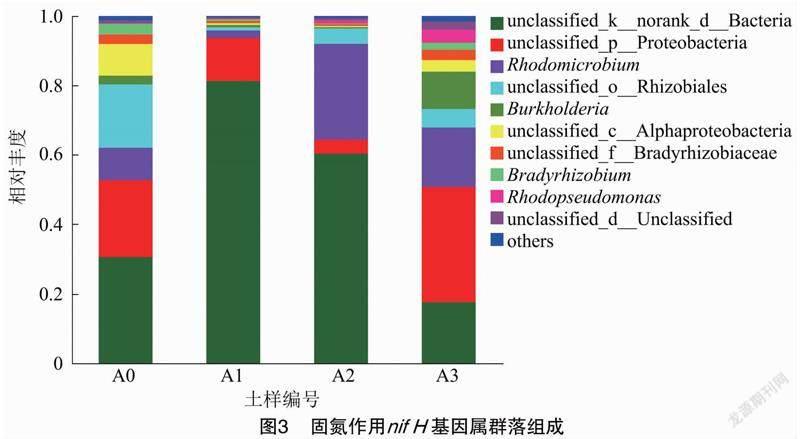
由图3可知,在所有样本中均以未分类的物种相对丰度最高,未种植区域可达30.58%,种植区域最高可达81.29%,最低是17.60%;其次是变形菌门(Proteobacteria),未种植区域丰度可达22.18%,种植区域最高可达33.17%,最低是4.10%。种植区域红微菌属(Rhodomicrobium)的相对丰度明显比未种植区域高,未种植区域的相对丰度为9.3%,而种植区域的分别为2.15%、27.53%、17.13%。未种植区域未分类根瘤菌目(Rhizobiales)的相对丰度明显比种植区域高,其在未种植区域的相对丰度为18.25%,而种植区域分别为0.90%、4.46%、5.34%。总体而言,种植会影响土壤固氮微生物的群落组成和群落间的丰度。
2.2.4 nifH基因的系统发育分析 采用邻接(neighbor-joining,NJ)法选取总丰度排前35位的OTU代表序列与NCBI数据库比对,下载相似性最高的序列,利用MEGA 7.0构建进化树。如图4所示,所得序列与NCBI数据库的基因序列相似性为75.57%~100%,样本OTU根据门水平,可大致分为硝化螺菌门(Nitrospirae)、放线菌门(Actinobacteria)、变形菌门(Proteobacteria)和未知菌类(unclassified)4个门类。在前35位OTU内,A1土样中有35条,A2土样中有23条,A3土样中有28条,A0土样中有23条。与硝化螺菌门相似的序列有17条,A1土样中有3条,A2土样中有3条,A3土样中有9条,A0土样中有3条。与放线菌门相似的序列,A1土样中有16条,A0土样中有2条。与变形菌门相似的序列有4条,A1土样中有3条,A0土样中有2条,序列相似度为76.19%~99.72%。其他序列与未知菌类的nifH基因相似,序列相似度为75.57%~100.00%。
2.3 固氮基因实时荧光定量
2.3.1 nifH基因标准曲线 对所有功能基因进行实时荧光定量,并绘制标准曲线。由图5可知,固氮作用nifH型细菌的决定系数(r2)为0.998 6,说明此次定量的数据精确;扩增效率为99.74%,可以达到绘制标准曲线的要求。根据标准曲线计算,发现澳洲坚果种植地固氮相关基因(nifH)的拷贝数在7.325×106~5.120×107之间。
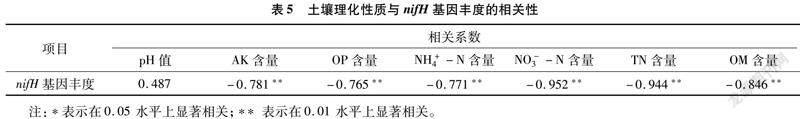
2.3.2 nifH基因与理化因子相关性分析 由表5可以看出,固氮细菌nifH基因的丰度与AK、OP、NH+4-N、NO-3-N、TN、OM[JP+2]等的含量在0.01水平上呈显著负相关。
3 讨论与结论
本研究表明,种植地带固氮基因nifH的多样性和丰富度显著降低,种植地带长期施肥显著影响了含nifH基因的固氮细菌群落结构。取样时避过施肥区域,但种植区域群落结构还是存在差异,说明土壤肥力是区分不同群落结构的最主要因素之一。变形菌门(Proteobacteria)为含nifH基因的固氮细菌群落优势菌门,相对丰度为18.21%~79.69%。此外,在种植区域均发现蓝藻门(Cyanobacteria)的固氮菌群,这与江西红壤中的研究结果[15]相似。董志新等的研究表明,固氮微生物的多样性受土壤因素和种植条件的影响[16]。Hai等的研究表明,高粱秸秆与牛粪配施能增加固氮菌的数量,促进土壤氮素的固定,降低氮素的损失[17]。Wang等发现,表层土壤的土壤有机碳(SOC)含量和铵态氮的濃度与固氮细菌的群落结构有显著的正相关关系,这说明土壤理化性质对表层土壤固氮细菌的群落具有选择性[18]。Sharma等也发现,土壤湿度、有机质含量和矿质氮的浓度是影响固氮细菌群落结构和丰度的主要因素[19]。
本研究通过对固氮细菌实时荧光定量分析,发现澳洲坚果种植地固氮相关基因(nifH)拷贝数在7.325×106~5.120×107之间,孟晗研究发现旱地土壤中细菌的拷贝数在106左右[20],两者结果基本一致。本研究还发现种植澳洲坚果会明显降低固氮菌群nifH的基因拷贝数,种植区域较未种植区域基因拷贝数呈明显的下降趋势,这可能是由于施肥造成的,这与李帆研究青藏高原高寒草甸土壤中自生固氮细菌群落组成的结果[21]一致。
本研究表明,种植区域土壤养分含量明显比未种植区域高。土壤固氮微生物丰度与养分元素的相关性分析结果表明,速效钾、有效磷、铵态氮、硝态氮、全氮、有机质含量对nifH基因拷贝数具有极显著的影响,并且对基因拷贝数呈负相关性,这与李帆研究土壤中自生固氮微生物的群落结构[21]一致。兰鸿珠等研究湿地盐节木泥土中固氮细菌的群落结构和功能基因拷贝数,发现功能基因拷贝数与硝态氮、速效氮含量等存在相关性[22]。
本研究为研究和利用澳洲坚果种植地中的固氮细菌提供了基础依据。另一方面可以比较澳洲坚果种植地再利用方式的效果,为生态修复、固氮微生物开发与利用、土壤肥力恢复等提供科学基础。
参考文献:
[1]马雪峰,高 旻,程治军. 植物氮素吸收与利用的分子机制研究进展[J]. 作物杂志,2013(4):32-38.
[2]Song X Z,Li Q,Gu H H. Effect of nitrogen deposition and management practices on fine root decomposition in Moso bamboo plantations[J]. Plant and Soil,2017,410(1/2):207-215.
[3]Jetten M S M. The microbial nitrogen cycle[J]. Environmental Microbiology,2008,10(11):2903-2909.
[4]Zehr J P,Jenkins B D,Short S M,et al. Nitrogenase gene diversity and microbial community structure:a cross-system comparison[J]. Environmental Microbiology,2003,5(7):539-554.
[5]张 晶,林先贵,尹 睿. 参与土壤氮素循环的微生物功能基因多样性研究进展[J]. 中国生态农业学报,2009,17(5):1029-1034.
[6]田永辉. 不同树龄茶树根际固氮菌组成及多样性研究[J]. 福建茶叶,2000,22(3):19-21,54.
[7]陈秀波. 不同林型红松林土壤微生物群落组成和多样性及与理化性质关系[D]. 哈尔滨:东北林业大学,2020.
[8]张苗苗,刘 毅,盛 荣,等. 稻草还田对水稻土固氮基因(nifH)组成结构和多样性的影响[J]. 应用生态学报,2013,24(8):2339-2344.
[9]Wu L Q,Ma K,Lu Y H. Prevalence of betaproteobacterial sequences in nifH gene pools associated with roots of modern rice cultivars[J]. Microbial Ecology,2009,57(1):58-68.
[10]Tan Z Y,Hurek T,Reinhold-Hurek B. Effect of N-fertilization,plant genotype and environmental conditions on nifH gene pools in roots of rice[J]. Environmental Microbiology,2003,5(10):1009-1015.
[11]柳建银. 红树林根际土壤固氮细菌和功能基因nifH的多样性研究[D]. 武汉:华中农业大学,2009.
[12]Mergel A,Schmitz O,Mallmann T,et al. Relative abundance of denitrifying and dinitrogen-fixing bacteria in layers of a forest soil[J]. FEMS Microbiology Ecology,2001,36(1):33-42.
[13]Zhou J,Deng Y,Shen L,et al. Temperature mediates continental-scale diversity of microbes in forest soils[J]. Nature Communications,2016,7:12083.
[14]杨丽萍,岳 海,何双凌,等. 云南澳洲坚果园土壤和叶片养分评价[J]. 热带作物学报,2021,42(8):2269-2274.
[15]Teng Q H,Sun B,Fu X R,et al. Analysis of nifH gene diversity in red soil amended with manure in Jiangxi,South China[J]. The Journal of Microbiology,2009,47(2):135-141.
[16]董志新,孫 波,殷士学,等. 气候条件和作物对黑土和潮土固氮微生物群落多样性的影响[J]. 土壤学报,2012,49(1):130-138.
[17]Hai B,Diallo N H,Sall S,et al. Quantification of key genes steering the microbial nitrogen cycle in the rhizosphere of sorghum cultivars in tropical agroecosystems[J]. Applied and Environmental Microbiology,2009,75(15):4993-5000.
[18]Wang J C,Zhang D,Zhang L,et al. Temporal variation of diazotrophic community[KG*2/3]abundance and structure in surface and subsoil under four fertilization regimes during a wheat growing season[J]. Agriculture,Ecosystems & Environment,2016,216:116-124.
[19]Sharma S,Singh D K. Temporal variations in diazotrophic communities and nifH transcripts level across the agricultural and fallow land at Jaipur,Rajasthan,India[J]. Indian Journal of Microbiology,2017,57(1):92-99.
[20]孟 晗. 长三角地区土壤不同发育阶段微生物群落结构的变化[D]. 上海:复旦大学,2011.
[21]李 帆. 不同管理模式对青藏高原高寒草甸土壤中自生固氮微生物群落结构影响的研究[D]. 兰州:兰州大学,2014.
[22]兰鸿珠,胡文革,杨 扬,等. 艾比湖湿地盐节木土壤固氮微生物群落结构和丰度的环境异质性特点[J]. 微生物学通报,2019,46(7):1597-1610.
