世界李育种概况
2023-05-12刘硕徐铭刘家成章秋平马小雪刘宁张玉萍张玉君赵海娟刘威生
刘硕,徐铭,刘家成,章秋平,马小雪,刘宁,张玉萍,张玉君,赵海娟,刘威生
世界李育种概况

辽宁省果树科学研究所,辽宁营口 115009
李是全球分布和栽培最广泛的果树之一,主要分为欧洲李()和中国李()两种。中国李起源于中国,具有较高的遗传异质性和果实性状遗传变异的丰富性。据推测,中国的长江流域地区仍有野生中国李的分布。在我国古代,许多书籍中都有李的栽培和品种记载,如秦汉、晋代、北宋和明代。国外也有许多书面记载李的起源和品种,如希腊、法国、芬兰、捷克和俄罗斯。自罗马时代以来,李已逐步传播到欧洲大陆。为了改善李果实品质、提高商品性以满足市场需求,一直在不断地培育新的李品种。世界上最早关于欧洲李品种的记载中选育出成熟期最早的品种,如‘Reine Claude’‘Early Rivers’‘Early Transparent Gage’‘Czar’‘Monarch’和‘Tresident’,目前仍广泛栽培。在中国,已经自主育成并经过品种审定且正式发表的李品种有88个。尽管世界各地的种植者、水果市场和消费者对李品种的改良需求各不相同,但育种性状包括果实大小、果皮颜色、多酚类物质、可溶性固形物、果实成熟期、抗寒性和抗病性能力等,受到共同关注。李育种方式从传统的育种手段,到分子辅助育种、组织培养和转基因育种不断演变。通过育种手段的不断改进,育出许多鲜食品质好、耐贮藏、货架期长和适应性好的优良品种,推动了世界李产业的发展。中国是全球最大的李生产国,李的产量占全球总产量的54.94%,栽培面积占全球总面积的74.75%。我国拥有丰富的李种质资源,这为李的育种创新提供了物质基础。而广泛的地理分布和栽培利用则为李新品种的应用提供了广阔的市场潜力。为了进一步发挥我国李种质资源的优势,提高我国李育种的核心竞争力,本文将梳理世界李的起源、传播、早期栽培利用、育种研究进展以及主要性状的遗传规律,并提出现代李育种策略。
李;育种;品种;策略
0 引言
李是世界上重要的核果类果树之一,属蔷薇科(Rosaceae)李属()。李兼鲜食和加工用途,其果实富含蛋白质、脂肪和碳水化合物,及众多的矿物质、维生素;加工品包括红干、乌干、去皮李干、糖水罐头、李蜜饯、李脯、李酱、话李、果汁和酿酒等[1]。李是世界上分布范围最广泛的果树之一,全球李种类约有19—40种(图1),超过6 000个品种,主要分布于欧亚大陆及美洲温带地区[2]。
李是我国重要的落叶果树之一,野生分布和栽植面积十分广泛。除青藏高原的高海拔地区外,几乎各省份均有分布,从最南部的海南至最北部的黑龙江,从东南沿海至西部的新疆,均有栽培或野生资源,尤其在边远山区、民族地区分布较多,垂直分布最高处可达海拔4 000 m[1]。据FAO(2020)[3]统计资料,世界李栽培面积259.9万hm2,总产量1 210.5万 t;中国是世界上最大的李生产国,种植面积占世界总面积的74.75%(194.3万hm2),产量占世界总产量的54.94%(665.1万t)。李在我国栽培面积略少于苹果(207.16万hm2),高于桃(79.17万hm2)、梨(96.72万hm2)和葡萄(74.48万hm2)[3]。
长久以来,人们为了满足市场对李优质果品的需求,不断地开展李新品种培育,通过提高果实品质、增强商品性促进全球李果品产业发展。为此,本文回顾国内外李育种改良历程,包括世界李的起源传播、早期栽培历程和国内外李育种研究主要目标性状,并提出现代李育种策略,为我国李优异基因资源发掘和育种利用提供借鉴,提高我国李育种效率,实现李育种科技自强自立。
1 李起源与传播
目前,世界李栽培的种类主要为欧洲李()和中国李()。欧洲李被认为起源于黑海和里海之间的高加索南部地区,包括格鲁吉亚、亚美尼亚、阿塞拜疆和伊朗北部高原[2]。Vavilov[4]曾推测该区域六倍体欧洲李与二倍体樱桃李(L.)和四倍体黑刺李(L.)的分布重叠。然而,人为归化后的长期自然生长、天然林分布群落的缺乏以及栽培或野生类型频繁的跨大陆引种和交流等原因使得对欧洲李起源的追溯十分困难,至今它的原始祖先仍存在疑问[5]。19世纪30年代,人们根据形态特征认为高加索地区存在野生欧洲李,随后细胞遗传学分析否定了此假设[6]。20世纪80年代,我国新疆伊犁河谷地区(Ili River)发现野生六倍体李(当地称为‘酸梅’),被认为可能是欧洲李的祖先[7-8]。但是,人们发现新疆‘野生欧洲李’群体的分子遗传多样性水平较低,与野生的遗传多样性较栽培群体高的一般客观规律不相符,推测可能是由于逃逸或归化而形成的野生类型[9]。最新的简化基因组研究表明,欧洲李起源于二倍体樱桃李和四倍体黑刺李的种间杂种,此黑刺李是樱桃李和未知的欧亚李种的种间杂种,可能是早期欧亚种间杂交和人工选择的产物[5]。
中国李起源于中国,曾推测来自我国的长江流域,在湖北、云南等地区至今仍有野生的中国李分布,甚至有上百年生的古树[10]。中国李(Chinese plum)约在200—400年前传入日本开始栽培,称作日本李(Japanese plum)[11]。中国李较高的遗传异质性使育种杂交后代的果实性状遗传变异十分丰富,包括果皮、果肉颜色、果实大小、果实形状等,这使中国李及其衍生材料已成为揭示多年生木本果树遗传规律的经典模式植物。
2 早期李驯化和栽培历史
早期的李驯化主要发生在亚洲、欧洲和北美大陆,史前人类曾用李作为食物[12]。公元前11世纪至公元前6世纪,古代中国人在《诗经》的《王风·秋中有麻》记载有“丘中有李,彼留之子”,可见当时人们对李的生态习性和栽培已有了一定的认识。1949年之后,我国考古发掘出新石器时代或战国时代的李核遗物,证明远在5 000年前,中国人的祖先已采食李果,其栽培历史至少3 000年以上[10]。公元前2世纪,秦汉间的字书《尔雅》中记载了3个李品种:‘无实李’‘接虑李’和‘赤李’;汉代修上林苑搜罗到李的品种达14个;晋代郭义恭《广志》记录了15个李品种;北宋年间《洛阳花木记》记载河南洛阳栽培的李品种达27个;明代《本草纲目》记载栽培的李品种近百个,各品种间的果实大小、果皮色泽等方面均差异很大[10]。我国有些古老的栽培李品种世世代代得到保留,例如浙江嘉兴地区的‘槜李’和中国李变种‘㮈’,前者在古籍《槜李谱》(1857年)提到:“颗以圆整而略带微扁者为上…以指爪破其皮,浆液可一口吸尽…核上有金丝缕缕,粘而不脱”;后者在弘治二年《八闽通志·福州府》(1489年)提及:“奈似林檎差小而长,淡青色,《莆阳志》云即青李也。”[10]。
外国考古人员在瑞士湖畔旁居民住处挖掘到黑刺李和乌荆子李()的果核化石,在高加索山脉和里海地区也发现古代人类曾食用过李。关于李的书面记载,希腊最早始于公元前680—645年,法国于742—814年,芬兰在15—16世纪,捷克在1579年,俄罗斯则在1654年。旧时战争迁移和商品贸易加速了李的传播,古代的罗马人在公元前65年征服了叙利亚的大马士革之后,将李带回国。罗马博物学家和自然哲学家盖乌斯∙普林尼∙塞孔都斯(Gaius Plinius Secundus)在著作《Naturalis Historia》中谈到了李的起源,并对14个品种进行了描述。从古罗马人时代开始,李逐渐分布于欧洲大陆,随后传播到巴尔干半岛西部,特别是前南斯拉夫的一些地区。后来的波斯尼亚(‘波黑’)成为了欧洲李的生产大国[13-14]。
3 国内外李改良历史
3.1 国外李品种改良
世界最早关于欧洲李品种的记载是在1597年的《The Herball or General Historie of Plantes》一书中:“位于英国特威克纳姆(Twickenham)的Vincent Pointer育苗圃收集多种多样的品种”,这表明育种整理工作已经逐步开展。当时,英国托马斯∙安德鲁(Thomas Andrew)选育的成熟期最早的欧洲李品种‘Reine Claude’‘Early Rivers’‘Early Transparent Gage’‘Czar’‘Monarch’和‘Tresident’,这些品种目前仍被广泛栽培[15]。
路德布尔班克(Luther Burbank)是世界著名李育种先驱者之一,是世界级的植物育种大师,培育了近800个品种。他改良了法国的‘Agen’欧洲李品种,选育的‘Miracle’(1903年)品种是×的杂交品种,几乎没有种子,被广泛种植作为珍奇品种。经过几代的杂交后,培育出了‘Conquest’(1916年)品种,其残余种子更小。由此,布尔班克的无核李成为了世界经典[16-19]。此外,他还培育了‘Giant’‘Sugar’和‘Standard’等著名的商业化栽培欧洲李品种[20]。除欧洲李外,他于1884年从日本引进了12个中国李品种(210株幼苗)并种植在加利福尼亚[12],并从中选育出‘Burbank’和‘Satsuma’品种;他将中国李与美国当地种类,包括樱桃李、杏李()、美洲李()、和等进行杂交并得到数十万株幼苗,培育出许多优良品种[21-22]。其中,他选育的‘Santa Rosa’李至今仍是加利福尼亚州第四大生产李品种。美国加利福尼亚州十个优良栽培品种中,有8个品种由他选育[23]。
欧洲李育种者在制定育种计划前常常会预估消费者的需求[24](表1)。从1900年开始,欧洲李新品种选育在13个欧洲国家开展,不同国家育种目标各有不同,摩尔多瓦育种目标为晚花和抗冻性,拉脱维亚育种目标为抗寒能力和自花结实率,瑞典育种目标是培育生长期短的品种,挪威育种目标是耐贮藏性,保加利亚育种目标是延长果实成熟期,罗马尼亚也关注延长果实成熟期和自花结实能力[25-28]。欧洲国家采用不同育种方法,包括杂交育种、诱变育种和实生选种,获得了大约250 000个杂种子代并产生170多个新品种[29]。用于育种改良的亲本材料包括‘Anna Spath’‘Hall’‘Italian Prune’‘Ruth Gerstetter’‘Pozegaca’‘Stanley’‘Mont Royal’‘Pacific’‘Jojo’‘Cacanskalepotica’‘President’‘Cacanskanajbolja’‘d’Ente’‘R. C. Verte’‘Ortenauer’‘Valor’‘French Improved’‘Angeleno’‘Black Star’‘Jubileum’‘Opal’‘Jefferson’‘d’Agen’‘Renclod Althan’‘Tuleu gras’‘Early Rivers’‘Wilhelmina Spath’‘Kirke’和‘Hackman’[26-27,30]。北美早期定居者将欧洲李带入美洲大陆,这些欧洲李只能种植在北部冬季较寒冷地区,导致早期北美地区欧洲李育种进程比较缓慢。
美国农业部(USDA)及私人育种公司(Sun World、Zaiger Genetics和Bradford Farms等)在美国加利福尼亚州地区开展大果、果实硬度高的优良李品种选育工作,包括‘Friar’‘Black-amber’‘Queen Rosa’‘Fortune’‘Red Beauty’和‘Angeleno’等许多品种在世界范围内广泛种植。美国水果市场中的李品种在20世纪80年代之前主要以红色果皮为主,之后则以紫黑色且早熟的‘Friar’‘Black-amber’和晚熟‘Angeleno’为主。佛罗里达州的李育种计划以选育适合亚热带气候的短低温需冷量商业化李品种为主要目标[31]。佛罗里达大学选育的‘Gulf’系列李品种(例如,早熟的‘Gulf beauty’,果实发育期75 d的‘Gulfblaze’和‘Gulfrose’等)对叶缘焦枯病()和细菌性斑点病()具有抗性[32]。
亚洲东部的日本李品种约80%是由私人育种者或种植者选育而成。例如,福岛县的Oishi Roshio选育的‘Oishiwasesumomo’‘Oishinakate’和‘Gekkou’(Masami Yamaguchi pers. comm.)。筑波国立果树科学研究所以延长采收季和提高果实品质为目标,在1970年后选育出‘Honey Rosa’[33]和‘Honey Heart’[34]。韩国国家园艺研究所以提高果品大小、形状、颜色、可溶性固形物和酸度为目标,选育出低酸(<0.5 g∙L-1)性状突出的‘Honey Red’[35]。
在世界其他地区,南非斯泰伦博斯农业研究委员会(ARC Infruitec-Nietvoorbij)满足出口欧洲市场的需要,于20世纪50年代开始选育红色、黄色和黑色果皮,成熟期在11月至次年3月的李品种,包括‘Harry Pickstone’‘Reubennel’‘Laetitia’‘Golden Kiss’和‘Sun Breeze’。南美洲巴西圣卡塔琳娜州农业研究所[36]以低需冷量、抗叶缘焦枯病和细菌性叶斑病为育种目标,选育出‘Camila’和‘Piuna’[37]品种。澳大利亚李育种计划则由昆士兰重要产业部与西澳大利亚农业部Bruce Topp教授负责,选育出抗细菌病、早熟、抗寒且适合出口东南亚的品种[38-40]。新西兰李育种计划旨在选育大果、风味佳、耐贮藏、树体矮化、成熟期不同、抗病的国际化商业品种[30]。
3.2 我国李品种改良
中国李在我国约有800多个品种或类型且分布极广,从云南地区至黑龙江沿岸、台湾岛、东部沿海至青藏高原与天山山脉。我国多种多样的生态、气候、地理使中国李育种具有地域特征,东北李品种群多表现树冠较小、耐寒性强,例如,吉林省农业科学院选育的‘福禄’[41]、‘公主红’[42]等;华北李品种群具势壮、耐寒、果实大特点,代表品种有‘苹果李’‘晚红李’‘帅李’‘伏李’等;华南李品种群长势旺、耐高温多湿,代表品种有‘兴华李’[43]和‘云开1号’[44]等;西南李品种群树体较小、不耐寒且果实小,大多为脆肉类型,代表品种有‘蜂糖李’‘酥李’‘宛青’[45]、‘晚霜脆李’[46]和‘巫溪晚脆李’[47]等。
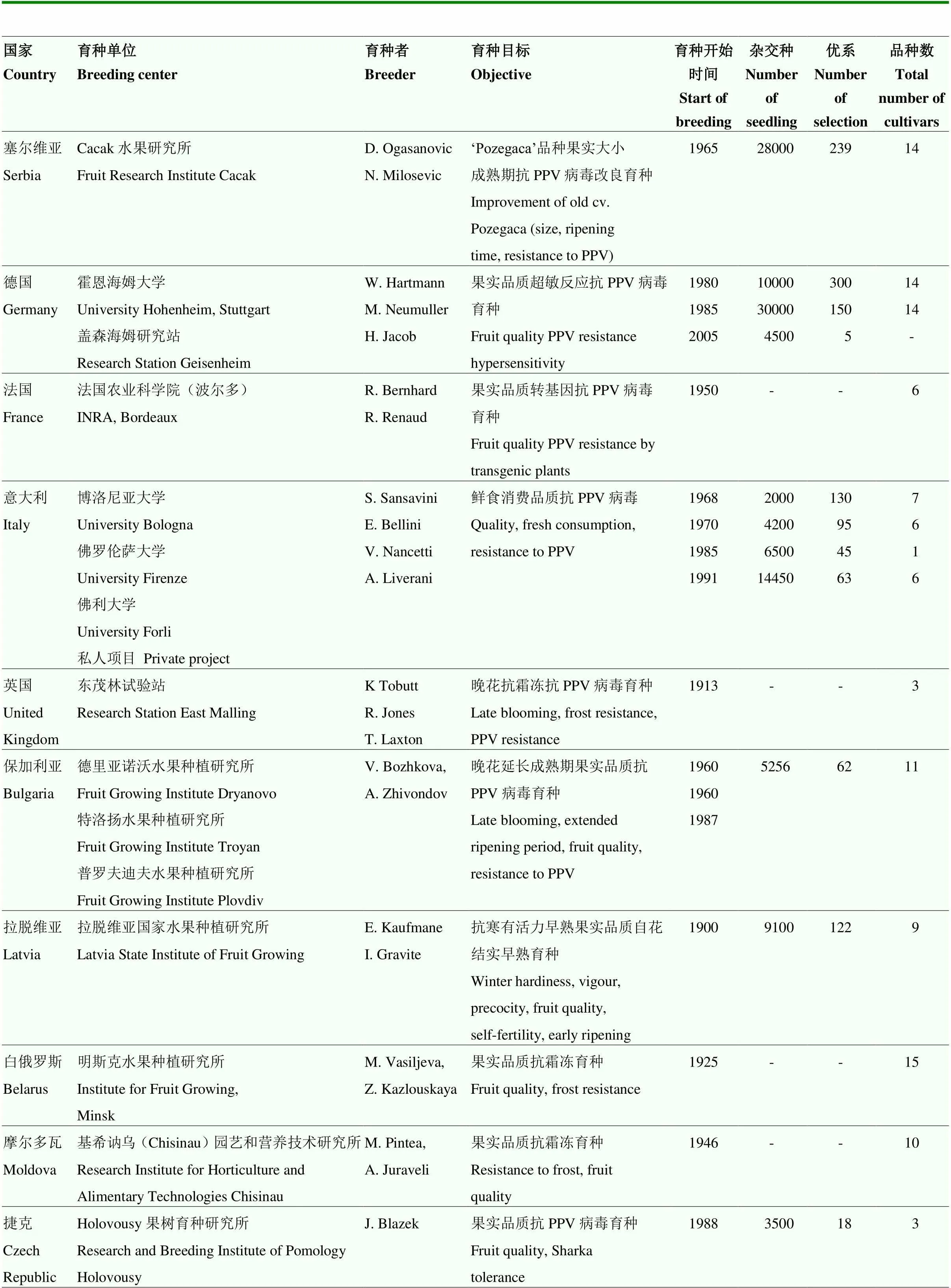
表1 欧洲地区开展李育种主要机构、育种者及育种目标[14,24]
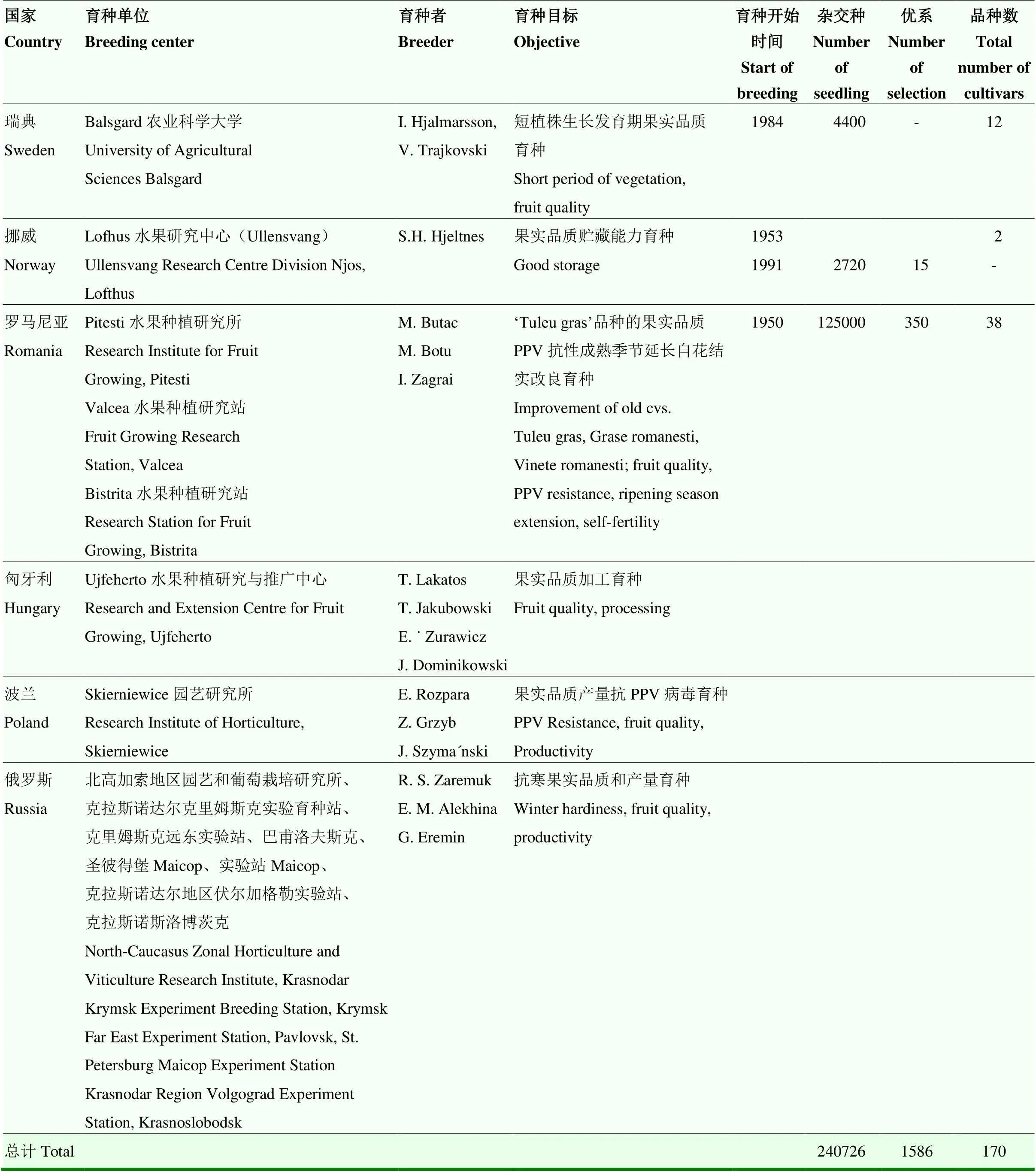
续表1 Continued table 1
我国最早育成的李品种为‘跃进李’(也称六号李)[48],筛选自1956年地方品种‘红干核’实生后代单株。20世纪60—70年代,我国东北地区率先开展了针对寒冷气候的寒地李新品种遗传育种研究,相继培育出‘北方一号’(也称‘绥棱红’)[49]、‘绥李三号’[49]、‘长李7号’[50]、‘17号’[51]等品种,适宜寒地栽培的品种‘长李15号’[52]和‘龙园秋李’[53]等品种在我国李产业发展上发挥了巨大的作用,为我国深入开展李遗传育种研究奠定了坚实的基础。截至2021年不完全统计,我国自主育成通过品种审定并正式发表的李品种有88个,东北三省科研单位育成品种40个,占总数的45.45%,是我国李育种的主要力量,其次是新疆、山西、陕西、福建、重庆等省的科研院所[54]。此外,观赏李新品种也受到园林绿化的重视,包括‘岳寒红叶李’[55]、‘长春彩叶李’[56]、‘北国红’[57]和‘一品丹枫’[58]。
4 现代李育种研究
4.1 李主要性状改良及遗传规律研究
世界范围内种植者、水果市场和消费者对李品种的改良需求各有不同,但共同关注的育种性状包括果实大小、果皮颜色、多酚类物质、可溶性固形物、果实成熟期、抗寒性、抗病性等。
(1)果实大小:李果实大小是影响产量和商品价值的重要农艺性状。在中国李遗传群体中呈现明显的衰退趋势,杂交后代果实总体偏小。以单果质量为34.6 g的‘绥棱红’与单果质量为31.8 g的‘六号李’为亲本,在正、反交的两个群体中,绝大多数后代果实较双亲偏小[59]。中国李自然杂交后代的果实大小平均值小于亲中值,呈小果变异的趋势;而后代果实大小平均值受母本影响较大,具有母性遗传的倾向。选育大果实的杂种,往往依赖于亲本的大果实特征[60]。控制中国李果实大小的QTL位于染色体第七连锁群(LG3)16—20 cM处[61]。
(2)果皮颜色:众多水果中,李的果皮颜色遗传多样性最为丰富,包括绿色、黄色、粉色、红色、紫色、蓝紫色和紫黑色等各种颜色[62]。我国学者研究发现紫红色果皮的‘六号李’后代193株中,紫色与红色果实占74.09%,黄色与绿色占25.9%,有彩色与无彩色比为2.86﹕13;同样,紫红色果皮的‘绥棱红’后代271株中,紫色与红色果实占74.07%,黄色与绿色占25.46%,有彩色与无彩色比2.93﹕1,认为果皮有色/无色的分离比例符合3﹕1遗传[63]。另外,果皮红色×黄色的杂交组合出现黄色果皮的占8.01%,果皮黄色×黄色杂交的组合中81.2%的后代果皮为黄色;亲本果皮均为红色的群体后代中红果皮占96.5%。因此,认为李果皮颜色应主要受1对基因控制,且有色对无色为显性性状[64]。同时,国外学者[61]认为欧洲李和中国李果皮颜色由等位基因系列决定,黄色等位基因对蓝色、红色或紫色等位基因呈隐性遗传。QTL研究表明,控制中国李果皮颜色的基因位于染色体第三连锁群(LG3)52—61 cM处,在第四连锁群(LG4)位点与成熟期性状相邻[61]。
(3)果实多酚类物质:李果实富含抗氧化多酚类化合物——花青素。它是有效的天然自由基清除剂,具有抗氧化、改善肝功能、预防心血管疾病、抗癌、抗炎和保护视力等作用。研究表明,李杂交群体QTL基因座中,花青素-3-葡萄糖苷和花青素-3-芸香苷均位于LG4,总黄酮和原花青素分别位于LG5和LG8,少量花青素化合物定位在LG3和LG4[65]。
(4)可溶性固形物:主要由可溶性糖和有机酸组成,任何与糖和有机酸代谢途径有关的突变都影响其最终结果。对4个李杂交组合的可溶性固形物含量研究发现,在不同组合后代中可溶性固形物含量分离广泛,但不同组合中会出现少量超低亲或超高亲后代[66]。研究表明,控制中国李可溶性固形物的QTL位于染色体第一连锁群54—76 cM处、染色体第六连锁群55—57 cM处[61]。
(5)李果实成熟期:李从开花到果实成熟仅3—4个月。通过遗传育种手段延长或缩短成熟期,培育极早和极晚熟李,可提早或延长李果品货架期。研究发现,中熟与晚熟品种杂交后代群体的成熟期均表现为延后[63];中晚和极晚熟品种杂交F1代的平均果实成熟期比亲中值延后,并且母本对后代成熟期的影响大于父本[59]。中国李成熟期为数量遗传性状,早熟基因位于第4号染色体连锁群6—12 Mb[61]。
(6)抗寒性:李是极少数可在高寒地区生长的核果类果树之一。我国李育种始于20世纪60—70年代,高寒地区的黑龙江、吉林和辽宁率先开展了适宜寒冷气候环境的李新品种选育。多年来,东北地区选育的抗寒李品种约占全国审定并正式发表的57.81%[54]。观察高抗寒‘六号李’与低抗寒‘绥棱红’杂交后代的抗寒表现,发现部分后代在抗寒性方面表现出超亲遗传的现象,因此推测李树抗寒性为多基因控制的数量性状遗传[59]。调查6个杂交组合的抗寒性发现,351个杂交后代抗寒等级与母本相同的株系在各杂交组合中所占比例最大,后代株系的抗寒力大多低于母本,而强于父本,且后代株系的抗寒力平均值高于亲中值[64]。
(7)抗李痘病毒(Sharka):李痘病毒病是被认为果树中最具破坏性的检疫性病毒之一(表2)。病原体PPV(plum pox virus)可感染李属的桃、油桃、扁桃、杏、李、樱桃以及少数非李属物种[67]。李痘病毒最早的报道可追溯到20世纪初,当时Atanassov[68]将李痘状的病症描述为一种新的病毒病,之后,李痘病毒病逐渐蔓延到大多数欧洲地区、地中海盆地周围、近东和中东、南美洲、北美洲以及亚洲地区[69]。在过去的几十年中,李痘病毒病对产业产生了重大影响,并造成了超过100亿欧元的经济损失[70]。李痘病毒病原体PPV分离株根据特定的症状宿主基因组序列和昆虫载体传播性被划分为若干个组[71]。其中,PPV-M(Markus)、PPV-D(Dideron)和PPV-Rec(Recombinant)菌株被认为是最常见的。近期的国际项目(Sharka Containment,首字母缩写词SharCo)框架内,系统收集了来自世界各地的PPV分离株的序列数据(http://www.sharco.eu/)。
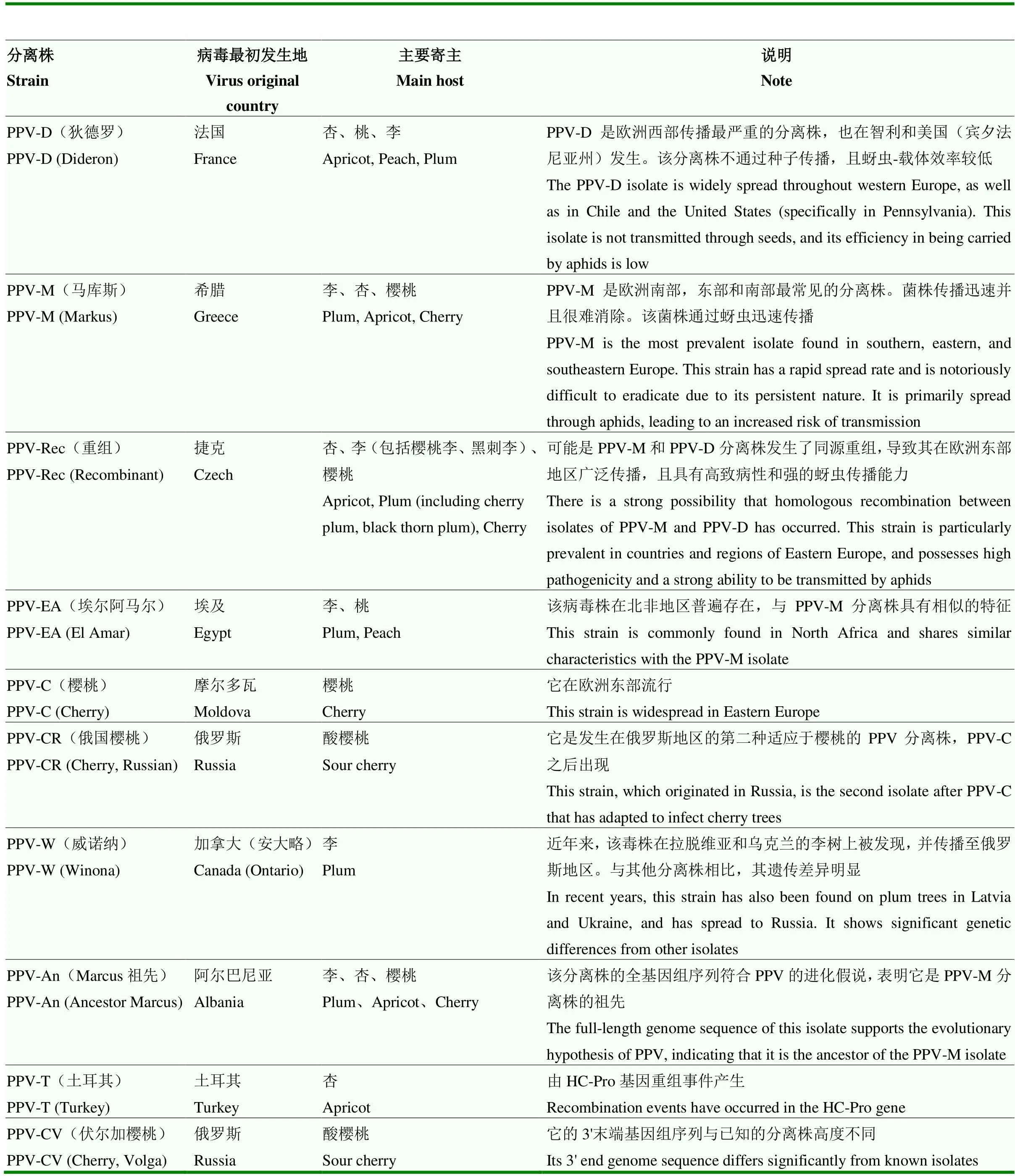
表2 李痘病PPV病毒主要分离株及特征
欧洲李品种‘JoJo’具有针对病毒的超敏反应,可阻止病毒在细胞间传播,在欧洲李常规育种中使用较多[72]。德国李科学家Hartmann和Neumülle[13]详细回顾了李痘病抗性育种的进展,表示至今尚未发现中国李的天然抗源材料。但是,人们对李的近源种——杏()的李痘病毒易感性/抗性的基因研究较多。研究表明,杏对PPV的抗性是一个复杂的过程,涉及基因[73]。Zuriaga等[74]使用定位克隆方法证实了参与了抗性机制,这个过程也可能与其他基因共同参与,这一结果也得到全基因组关联(GWAS)研究证实[75]。Decroocq等[76]基于假定基因ppb0022195中存在的5 bp缺失,开发了名为的单长度多态性(SSLP)标记,该基因编码MATH-TRAF样蛋白,定位在桃基因组(version 1.0)位置8 157 652处。最近,Passaro等[77]利用高分辨率熔解(HRM)实时PCR分析方法检测上述ppb0022195基因的5-bp缺失,该方法的广泛应用可以促进标记辅助选择(marker assisted selection,MAS),将不同抗性品种的抗性等位基因渗入新的商业品种。目前,抗PPV转基因策略已在各种核果类果树中开展和实施,从传统的基于外壳蛋白(CP)的方法到使用替代靶基因的方法[78-79],当下最多采用的是RNA silencing技术[80-81]。
4.2 分子辅助育种研究
标记辅助选择(MAS)是用于提高选定基因型效率的重要分子育种工具[59]。李育种研究中较为成熟的是利用DNA分子标记鉴定李高水平多态性的自交不亲和等位基因(-locus)。Yamane等[82]首先在日本李中克隆了-RNase等位基因。随后,通过PCR在不同品种中鉴定了14个等位基因,并将其命名为a—n[83-85]。Sapir等[86]克隆了另外5个等位基因(1、3—6)。Beppu等[87]发现e等位基因与中国李的自交不亲和相关,将由e开发的分子标记用于杂交后代预选,筛选具有自交性的幼苗[88]。欧洲李具有类似的自交不亲和系统,但由于该物种的六倍体性质,对等位基因的分析更为复杂。对19个栽培欧洲李品种的研究发现,每个基因型有2—6个等位基因[89-90]。
分子遗传图谱是数量性状位点(quantitative trait locus,QTL)定位和MAS育种的基础。目前,只有少数中国李遗传连锁图报道。Vieira等[91]使用扩增片段长度多态性(AFLP)标记的标记构建的亲本‘Chatard’‘Santa Rosa’遗传连锁图仅分别包含56和84个标记,总长度分别为905 cM和1 349.6 cM。Salazar等[61]使用SNP标记通过测序进行基因分型,开发了‘98-99’(479个SNP)和‘Angeleno’(502个SNP)的两个遗传图谱,分别覆盖了688.8 cM和647.03 cM的遗传距离。另一张图谱是由Carrasco等[92]报道的‘Angeleno’בAurora’,使用732个SNP构建,跨度为617 cM,相邻标记之间的平均距离为0.96 cM。近期,Zhang等[93]利用SLAF对来自‘09-16’בFortune’杂交的114株F1代幼苗进行基因分型,构建了含3 341个高质量SLAF和720个基因座的遗传连锁图谱,分为8个遗传连锁群,总长度为869.9 cM,平均距离为1.21 cM。
现代基因组学已成为开展优质高产、高效利用等育种研究的重要手段。近年来,李基因组测序发展十分迅速。Callahan等[94]采用基因编辑抗李痘病(Sharka)的欧洲李‘HoneySweet’[95]为试验材料,使用二代(210x)和三代(55x)测序策略获得了首个包含27 870个scaffolds的基因组,欧洲李基因组草图总长度为1 399.3 Mb。该基因组虽未能满足期望染色体数量要求,但为未来实现多倍体李基因组高质量测序研究提供了重要基础和经验。中国李基因组测序已发布2个基因组版本,均来自我国南方地方品种‘三月李’[96-97]。该品种栽培历史较久,具有早熟、高产、低需冷量等特质。Liu等[97]使用三代测序和Hi-C技术组装的‘三月李’基因组达到8条染色体水平,总长度为284.2 Mb,含有75个scaffolds;Fang等[96]通过三代测序技术组装的‘三月李’基因组总长度为308.06 Mb,并针对早花性状开展转录组学研究。这两版‘三月李’基因组测序,为李基因组学的深入研究提供了重要基础和前提。
与此同时,针对不同研究性状的转录组学和代谢组学也在李物种相继开展。Jo等[98-99]借助‘Akihime’和‘Formosa’品种转录组测序鉴定了植物体内病毒/类病毒种类;Kim等[100]通过对富含多酚和多糖的李品种转录组测序调查李果实成熟过程中的基因表达情况;GONZÁLEZ等[101]建立了‘Angeleno’和‘Lamoon’品种转录组数据库;Fang等[102]利用‘芙蓉李’转录组测序开展了有关果实成熟的研究,并对衍生的简单重复序列(SSR)信息进行描述总结;Farcuh等[103]利用转录组测序手段比较‘Santa Rosa’及其芽变材料‘Sweet Miriam’的果实发育差异变化。Fang等[96]和Liu等[97]在组装基因组过程中也同时开展了‘三月李’的转录组测序辅助基因组注释。此外,Piccolo等[104]对红樱桃李的可溶性固形物、颜色等果实性状开展了代谢组学研究。
4.3 李组织培养与转基因育种
遗传转化是一种强大的植物遗传改良技术,它可以克服果树传统育种中世代周期长、童期长、杂合度高、遗传变异性不足等问题[17]。欧洲李遗传转化和再生技术较为成功[105]。Mante等[106]和Gonzalez- Padilla等[107]使用噻二唑仑(TDZ)和吲哚-3-丁酸(IBA)用于在农杆菌介导的欧洲李下胚轴区段转化后再生芽,在共培养过程中添加了2,4-D可提高转化效率至42%,并能够在6个月后获得转基因植物[105]。对于中国李的遗传转化,Tian等[108]使用IBA结合不同浓度的TDZ苄氨基嘌呤(BAP)使‘Shiro’‘Early Golden’‘Redheart’的下胚轴节段再生并最终获得了再生植株[17]。
李的遗传转化和再生用途多在PPV抗性研究中开展和应用。欧洲李因缺乏天然遗传抗李痘病种质材料,使基因工程成为研发欧洲李抗李痘病的重要方法。作为对经典育种的改进,基因工程被用于生产含有PPV外壳蛋白(CP)基因的转基因克隆,应用病原体衍生抗性的原理。这种新方法创制了转基因欧洲李‘Honey Sweet’。这种转基因李植株对PPV具有高度抗性,并且不表达PPV-CP,几乎检测不到CP mRNA水平。然而,转基因植物也可能会感染病毒,特别是当它被嫁接在对PPV敏感的砧木时,它也会成为PPV的宿主。此外,将Flowering Locus T1()基因通过35S启动子转化到欧洲李中,使转基因植株可在温室中短时间内开花结果,且不会在冷处理或短日照后进入休眠状态;同时表现出一些不同于李的表型特征,如灌木状生长习性和总状花序结构。在李中表达的多态性表型表明该基因对植物生长和发育具有作用。因此,可能是改变温带植物以适应不同气候或新的种植区的基因[94,109-110]。
5 问题与展望
目前,我国地方性中国李品种的特点是鲜食品质好、适应性较好,但大多数在成熟后果肉松软、外观不鲜艳且不耐储运,商品性不佳。2000年前,我国李育种主要以大果、优质为育种目标,而忽视果实硬度(货架期)、外观颜色等商品性好的品种选育;国外育成品种虽外观好、货架期长,但鲜食品质欠佳。21世纪初,辽宁省果树科学研究所在第十次国际李和第十五次国际杏会议上率先提出,为了弥补中国地方品种果肉软、外观差、产量不稳定(即商品性差)和国外引进的改良品种鲜食品质较差等缺点,应利用风味浓郁的中国地方优良品种与商品性好的国外改良品种进行杂交的育种策略,即‘East cross West equal to best’(简称‘West×East’),并依托国家种质资源熊岳李杏圃丰富的种质资源优势,利用我国优良地方品种或育成品种与国外引进品种进行杂交,通过十几年的不懈努力,成功培育出‘国馨’(试验代号‘国峰7’,图2)和‘国色天香’(试验代号‘国峰2’)2个商品性较好的李新品种。‘国馨’是由我国选育的‘龙园秋李’与引进的‘澳14’杂交选育的耐寒、硬肉、浓香的李品种,成熟时硬度达到10.8 kg∙cm-2,在设施栽培条件下可溶性固形物可达21%,露地栽培可达到18%;‘国色天香’是以地方品种‘晚熟香蕉李’与日本品种‘秋姬’杂交育成的硬肉、浓香、色艳,且适宜高温多湿地区栽培的新品种[111]。

图2 ‘国馨’李及果实切面图
现代李育种涉及多方面的理论、方法和技术,面对国内外市场对李新品种的需求,我国李育种还有许多问题需要关注和解决,需要在以下几个方面做进一步的研究和开发:(1)短低温型李育种应对气温升高。气温升高的趋势对我国李栽培生产带来灾难性影响,引起了国内外李育种者的关注,应有针对性地开展李育种改良研究[112]。(2)耐裂果遗传育种研究。近年来,受极端降雨天气影响,我国西南地区的脆肉型李在成熟期裂果现象普遍,严重降低了李果品商品性,经济损失巨大。(3)加强抗晚霜品种选育。李树在春季花期容易受晚霜危害,损害花器官并影响产量,提升李树抗晚霜能力仍是我国李育种的主要目标之一。(4)抗病毒育种。我国李产业存在被李痘病毒侵染的巨大潜在风险,选育抗病毒品种是应对病毒危害的有效手段。(5)开展营养系砧木育种,提高对病虫害和非生物因素,包括温度、水分、营养和耐再植等逆境条件的适应能力。我国疆土幅员辽阔,生态类型多样,野生或栽培李种质资源类型变异丰富、多样性水平高。李种质资源和育种研究者可充分地发挥我国李种质资源优势,充分发掘种质丰富的遗传变异,利用于育种研究,解决中国李产业所处瓶颈问题。
[1] 刘威生, 章秋平, 马小雪, 张玉萍, 刘家成, 张玉君, 刘硕, 刘宁, 徐铭. 新中国果树科学研究70年: 李. 果树学报, 2019, 36(10): 1320-1338.
LIU W S, ZHANG Q P, MA X X, ZHANG Y P, LIU J C, ZHANG Y J, LIU S, LIU N, XU M. Fruit scientific research in New China in the past 70 years: Plum. Journal of Fruit Science, 2019, 36(10): 1320-1338. (in Chinese)
[2] HEDRICK U P, ALDERMAN W H, DORSEY M J, TAYLOR O M, WELLINGTON R. The Plums of New York. Albany J. B. Lyon Company, State Printers, 1911.
[3] 联合国粮农组织数据库[OL]. 2020. 2022-12-23.
Food and Agriculture Organization of the United Nations Database (FAOSTAT) [OL]. 2020. 2022-12-23.
[4] VAVILOV N. Wild progenitors of the fruit trees of Turkistan and the Caucasus and the problem of the origin of fruit trees. Ninth International Horticultural Congress Report, 1930.
[5] ZHEBENTYAYEVA T, SHANKAR V, SCORZA R, CALLAHAN A, RAVELONANDRO M, CASTRO S, DEJONG T, SASKI C A, DARDICK C. Genetic characterization of worldwide(plum) germplasm using sequence-based genotyping. Horticulture Research, 2019, 6: 12.
[6] RYBIN W A. Spontaneous and experimentally produced hybrids between blackthorn and cherry plum and the descent problem of the cultivated plum. Planta, 1936, 25: 22-58.
[7] LIN P, SHI L. The discovery and distribution of Ili wild(Fritsch) in Xinjiang//Proceedings of International Symposium on Horticultural Germplasm, Cultivated and Wild, 1989: 282-286.
[8] LIN S H, PU F S, ZHANG J H, GAO X Y, LI X J. Observation on chromosomes numbers of. China Fruits, 1991, 2: 8-10.
[9] LIU W S, LIU D C, ZHANG A, FENG C, YANG J M, YOON J, LI S H. Genetic diversity and phylogenetic relationships among plum germplasm resources in China assessed with inter-simple sequence repeat markers. Journal of the American Society for Horticultural Science, 2007, 132(5): 619-628.
[10] 张加延, 周恩. 中国果树志-李卷. 北京: 中国林业出版社, 1998: 17-23.
ZHANG J Y, ZHOU E. China Fruit-Plant Monographs, Plum Flora. Beijing: China Forestry Publishing House, 1998: 17-23. (in Chinese)
[11] Yoshida M . The origin of fruits. 2: Plums. 1987, 42(2): 49-53.
[12] Janick J, Moore J N. Fruit Breeding, Volume 1, Tree and Tropical Fruits. Fruit Breeding, 1996: 559-607.
[13] HARTMANN W, NEUMÜLLER M. Plum Breeding//Breeding Plantation Tree Crops: Temperate Species. New York, NY: Springer New York, 2008.
[14] AL-KHAYRI J M, JAIN S, JOHNSON D. Advances in Plant Breeding Strategies: Fruits. Cham: Springer, 2018: 165-215.
[15] RIMPIKA, SHARMA D. Advances in breeding of peach, plum and apricot. Prunus-Recent Advances. Intech Open, 2022.
[16] BURBANK L. How this ‘Miracle’ came to be. Sunset Magazine, 1903-1904(12): 35-36.
[17] BURBANK L, WHITSON J, JOHN R, WILLIAMS H S, SOCIETY L B. Luther Burbank: His Methods and Discoveries and Their Practical Application. New York, N.Y.; London: Luther Burbank Press, 1914.
[18] JANICK J. Luther Burbank: plant breeding artist, horticulturist, and legend. HortScience, 2015, 50(2): 153-156.
[19] CALLAHAN A, DARDICK C, TOSETTI R, LALLI D, SCORZA R. 21st century approach to improving burbank’s ‘stoneless’ plum. HortScience, 2015, 50(2): 195-200.
[20] BRUCE L T, DOUGAL M R, MICHAEL N, MARCO A D, LIU W S. Plum. Fruit breeding, 2012, 15: 571-621.
[21] HOWARD W L. Luther Burbank’s plant contributions//Berkeley, Cal.: Agricultural Experiment Station, 1945.
[22] CROW J F. Plant breeding giants: Burbank, the artist; vavilov, the scientist. Genetics, 2001, 158(4): 1391-1395.
[23] OKIE W R, RAMMING D W. Plum breeding worldwide. HortTechnology, 1999, 9(2): 162-176.
[24] BUTAC M, BOZHKOVA V, ZHIVONDOV A, MILOSEVIC N, BELLINI E, NENCETTI V, BLAZEK J, BALSEMIN E, LAFARQUE B, KAUFMANE E, GRAVITE I, VASILJEVA M, PINTEA M, JURAVELI A, WEBSTER T, HJALMARSSON I, TRAJKOVSKI V, HJELTNES S H. Overview of plum breeding in Europe. Acta Horticulturae, 2013, 981: 91-98.
[25] BELLINI E, NENCETTI V, NATARELLI L. New selections of yellow Japanese plum obtained in Florence at the dofi. Acta Horticulturae, 2010, 874: 321-326.
[26] BLAŽEK J, PIŠTĚKOVÁ I. Initial results from the evaluation of plum cultivars grown in a very dense planting. Acta Horticulturae, 2012, 968: 99-108.
[27] BUTAC M, ZAGRAI I, BOTU M. Breeding of new plum cultivars in Romania. Acta Horticulturae, 201, 874: 51-58.
[28] JACOB H. Plum breeding worldwide//Symposium on Plum of Serbia. 2006: 15.
[29] KNIGHT V H, EVANS K M, SIMPSON D W, TOBUTT K R.Report on a desktop study to investigate the current world resources in Rosaceous fruit breeding programmes. East Malling Research, 2005.
[30] HJELTNES S H, NORNES L. ROGNLI O A. Inheritance of some fruit characters in plum ()//IX International Symposium on Plum and Prune Genetics, Breeding and Pomology, 2008: 45-50.
[31] SHERMAN W B, TOPP B L, LYRENE P M. Breeding low-chill Japanese-type plums for subtropical climates. Acta Horticulturae, 1992, 317: 149-154.
[32] SARKHOSH A, OLMSTEAD M A. Growing plums in florida: HS895/HS250, rev. 1/2016. EDIS, 2020, 2016(2): 12.
[33] YAMAGUCHI M, YOSHIDA M, KYOTANI H, NAKAMURA Y, NISHIMURA K, HAJI T, MIYAKE M. New Japanese plum cultivar ‘Honey Rosa’. Bulletin-National Institute of Fruit Tree Science, 1995.
[34] YAMAGUCHI M, YOSHIDA M, KYOTANI H, NAKAMURA Y, NISHIMURA K, HAJI T, MIYAKE M. New Japanese plum cultivar ‘Honey Heart’. Bulletin-National Institute of Fruit Tree Science, 1999: 15-30.
[35] JUN J H, CHUNG K H, KANG S J, KWACK Y B, PARK K S, YUN H K, JEONG S B. ‘Honey Red’, an early maturing Japanese plum. Journal of the American Pomological Society, 2008, 62: 27-29.
[36] NAKASU B H, BASSOLS M, FELICIANO A J. Temperate fruit breeding in Brazil. Fruit Varieties Journal, 1981, 35: 114-122.
[37] Ducroquet J P, Dalbó M A. SCS 409 Camila e SCS 410 Piuna-Novascultivares de ameixeira com resistência à escaldadura das folhas. Agropecuária Catarinense, 2007, 20: 67-70.
[38] TOPP B L, RUSSELL D M. Breeding early ripening Japanese plums. Acta Horticulturae, 1989, 240: 27-30.
[39] TOPP B L, RUSSELL D M. ‘Queensland bellerosa’ plum. HortScience, 1990, 25(7): 814.
[40] TOPP B L, RUSSELL D M. ‘Queensland earlisweet’ cherry plum. HortScience, 1990, 25(6): 713.
[41] 张艳波, 王雪松, 崔龙, 陈蕾, 李锋. 抗寒李新品种‘福禄’. 园艺学报, 2021, 48(6): 1265-1266.
ZHANG Y B, WANG X S, CUI L, CHEN L, LI F. A new cold resistant plum cultivar ‘fulu’. Acta Horticulturae Sinica, 2021, 48(6): 1265-1266. (in Chinese)
[42] 张艳波, 王雪松, 陈蕾, 崔龙, 李锋, 隋松兵, 张连喜. 抗寒李新品种‘公主红’的选育. 果树学报, 2022, 39(4): 689-691.
ZHANG Y B, WANG X S, CHEN L, CUI L, LI F, SUI S B, ZHANG L X. A new cold-resistant plum variety Gongzhu Hong. Journal of Fruit Science, 2022, 39(4): 689-691. (in Chinese)
[43] 何业华, 杨向晖, 郭翠红, 栾爱萍, 刘朝阳, 夏靖娴, 谢桃, 李楚豪, 曾志. 加工鲜食兼用李新品种‘兴华李’的选育. 果树学报, 2020, 37(10): 1597-1600.
HE Y H, YANG X H, GUO C H, LUAN A P, LIU C Y, XIA J X, XIE T, LI C H, ZENG Z. Breeding report of a new fresh-eating and processing plum cultivar ‘Xinghuali’ (). Journal of Fruit Science, 2020, 37(10): 1597-1600. (in Chinese)
[44] 何业华, 杨向晖, 栾爱萍, 刘成明, 胡桂兵, 林顺权, 秦永华, 夏靖娴, 傅嘉欣, 赵杰堂, 高用顺, 张志珂, 温瑞明, 陈世凯, 罗学优, 池琼云, 卢仕威. 华南李新品种‘云开1号’. 园艺学报, 2020, 47(S2): 2892-2893.
HE Y H, YANG X H, LUAN A P, LIU C M, HU G B, LIN S Q, QIN Y H, XIA J X, FU J X, ZHAO J T, GAO Y S, ZHANG Z K, WEN R M, CHEN S K, LUO X Y, CHI Q Y, LU S W. A new plum cultivar ‘Yunkai 1’. Acta Horticulturae Sinica, 2020, 47(S2): 2892-2893. (in Chinese)
[45] 方波, 赵倩, 黄明, 唐君, 蔡智勇, 张勋, 刘家红, 谭平. 巫山脆李新品种‘宛青’的选育. 果树学报, 2020, 37(7): 1106-1109.
FANG B, ZHAO Q, HUANG M, TANG J, CAI Z Y, ZHANG X, LIU J H, TAN P. ‘Wanqing’, a new late-maturing plum cultivar ofLindl. Wushancuili. Journal of Fruit Science, 2020, 37(7): 1106-1109. (in Chinese)
[46] 周伟, 何才智, 张乃华, 蒲昌权, 汪小伟, 朱根长, 万崇东, 张洪伟, 李丽, 程兰, 何才宏. 李新品种‘晚霜脆李’的选育及栽培技术. 中国果业信息, 2019, 36(3): 55-57.
ZHOU W, HE C Z, ZHANG N H, PU C Q, WANG X W, ZHU G C, WAN C D, ZHANG H W, LI L, CHENG L, HE C H. Breeding and cultivation techniques of a new plum variety ‘Late Frost Crispy Plum’. China Fruit News, 2019, 36(3): 55-57. (in Chinese)
[47] 熊伟, 向芳, 曾明, 王玉柱, 寇琳羚, 李兴婷, 李相进, 何桥, 刘健, 刘世仙, 陈培, 李伟. 李新品种‘巫溪晚脆李’的选育. 中国南方果树, 2020, 49(3): 92-96.
XIONG W, XIANG F, ZENG M, WANG Y Z, KOU L L, LI X T, LI X J, HE Q, LIU J, LIU S X, CHEN P, LI W. Breeding of a new plum variety ‘Wuxi Late Crispy Plum’. South China Fruits, 2020, 49(3): 92-96. (in Chinese)
[48] 宋洪伟, 迟占文, 张艳波, 陶睿, 张冰冰. 寒地李属资源与品种选育. 北方园艺, 2008(6): 105-106.
SONG H W, CHI Z W, ZHANG Y B, TAO R, ZHANG B B. Resources and variety breeding of plum in cold region. Northern Horticulture, 2008(6): 105-106. (in Chinese)
[49] 关连捷, 李久成, 吕映霞. 寒地李树新品种: 绥李三号和绥棱红. 北方果树, 1987(S1): 60-61, 48.
GUAN L J, LI J C, LÜ Y X. New varieties of plum trees in cold region—Suili 3 hao and Suilinghong. Northern Fruits, 1987(S1): 60-61, 48. (in Chinese)
[50] 吴起运. 李新品种: 长李7号. 山西果树, 1991(1): 41.
WU Q Y. Changli No.7, a new plum variety. Journal of Fruit Resources, 1991(1): 41. (in Chinese)
[51] 吴起运. 李新品种: 长李17号. 北方果树, 1991(1): 41.
WU Q Y. Changli 17, a new plum variety. Northern Fruits, 1991(1): 41. (in Chinese)
[52] 李锋. 李抗寒优良新品种(系). 中国果树, 1993(1): 1-2, 5.
LI F. A new cold-resistant plum variety (line). China Fruits, 1993(1): 1-2, 5. (in Chinese)
[53] 曾烨, 牟蕴慧, 金殿义, 刘国华, 刘允中. 李新品种‘龙园秋李’. 中国果树, 1998(2): 26.
ZENG Y, MOU Y H, JIN D Y, LIU G H, LIU Y Z. A new plum variety ‘Longyuan Qiuli’. China Fruits, 1998(2): 26. (in Chinese)
[54] 刘硕, 徐铭, 张玉萍, 张玉君, 马小雪, 章秋平, 刘宁, 刘威生. 我国李育种研究进展、存在问题和展望. 果树学报, 2018, 35(2): 231-245.
LIU S, XU M, ZHANG Y P, ZHANG Y J, MA X X, ZHANG Q P, LIU N, LIU W S. Retrospect, problematical issues and the prospect of plum breeding in China. Journal of Fruit Science, 2018, 35(2): 231-245. (in Chinese)
[55] 唐世勇, 邢英丽, 王永杰, 才丰, 姜永峰. ‘岳寒红叶’李的选育. 北方园艺, 2007(11): 133-134.
TANG S Y, XING Y L, WANG Y J, CAI F, JIANG Y F. Breeding of Yuehan Hongye plum. Northern Horticulture, 2007(11): 133-134. (in Chinese)
[56] 李锋, 张冰冰, 计秀杰, 张艳波, 邢国杰. 抗寒紫叶李新品种‘长春彩叶李’. 园艺学报, 2007, 34(2): 534.
LI F, ZHANG B B, JI X J, ZHANG Y B, XING G J. A new variety of hardy purple-leaf plum ‘Changchun Caiye Li’. Acta Horticulturae Sinica, 2007, 34(2): 534. (in Chinese)
[57] 张艳波, 赵晨辉, 梁英海, 付立中, 李锋, 曹希俊, 计秀杰. 抗寒紫叶李新品种‘北国红’的选育. 北方园艺, 2013(12): 168-169.
ZHANG Y B, ZHAO C H, LIANG Y H, FU L Z, LI F, CAO X J, JI X J. A new variety breeding of hardy purple-leaf plum ‘Beiguohong’. Northern Horticulture, 2013(12): 168-169. (in Chinese)
[58] 陆致成, 李峰, 张静茹, 张艳波, 宋宏伟, 赵晨辉, 孙海龙. 鲜食观赏兼用李新品种‘一品丹枫’. 园艺学报, 2014, 41(12): 2547-2548.
LU Z C, LI F, ZHANG J R, ZHANG Y B, SONG H W, ZHAO C H, SUN H L. A new edible ornamental plum cultivar ‘Yipin Danfeng’. Acta Horticulturae Sinica, 2014, 41(12): 2547-2548. (in Chinese)
[59] 李怀玉, 方玉凤. 李实生后代变异与亲本选择. 北方园艺, 1990(4): 9-13.
LI H Y, FANG Y F. Variation of plum’s offspring and parental selection. Northern Horticulture, 1990(4): 9-13. (in Chinese)
[60] 孙伟, 高庆玉. 中国李自然杂交后代抗寒力、果实大小的遗传与变异. 东北农业大学学报, 2003, 34(3): 250-253.
SUN W, GAO Q Y. Inheritance of cold hardiness and fruit size in Chinese plum (Lindl.). Journal of Northeast Agricultural University, 2003, 34(3): 250-253. (in Chinese)
[61] SALAZAR J A, PACHECO I, SHINYA P, ZAPATA P, SILVA C, ARADHYA M, VELASCO D, RUIZ D, MARTÍNEZ-GÓMEZ P, INFANTE R. Genotyping by sequencing for SNP-based linkage analysis and identification of QTLs linked to fruit quality traits in Japanese plum (lindl.). Frontiers in Plant Science, 2017, 8: 476.
[62] 赵海娟, 刘宁, 张玉萍, 刘威生, 张玉君, 徐铭, 马小雪, 刘家成, 刘硕. 李果皮颜色遗传多样性及其成色因子研究进展. 果树学报, 2022, 39(8): 1479-1489.
ZHAO H J, LIU N, ZHANG Y P, LIU W S, ZHANG Y J, XU M, MA X X, LIU J C, LIU S. Research progress in genetic diversity and related factors of plum peel color. Journal of Fruit Science, 2022, 39(8): 1479-1489. (in Chinese)
[63] 方玉凤, 张风芳, 王官清, 李铎, 李松群, 李怀玉. 六号李、绥稜红李自然杂交后代某些性状的遗传. 沈阳农业大学学报, 1989, 20(1): 15-19.
FANG Y F, ZHANG F F, WANG G Q, LI D, LI S Q, LI H Y. Inheritance of some characters in naturally hybridized progeny from plum 6'X Sui Ling red plum. Journal of Shenyang Agricultural University, 1989, 20(1): 15-19. (in Chinese)
[64] 刘文东. 李树杂交后代亲子性状遗传变异规律. 中国林副特产, 2013(5): 96-97.
LIU W D. Genetic variation of parent-child traits in hybrid progeny of plum tree. Forest by-Product and Speciality in China, 2013(5): 96-97. (in Chinese)
[65] VALDERRAMA-SOTO D, SALAZAR J, SEPÚLVEDA-GONZÁLEZ A, SILVA-ANDRADE C, GARDANA C, MORALES H, BATTISTONI B, JIMÉNEZ-MUÑOZ P, GONZÁLEZ M, PEÑA-NEIRA Á, INFANTE R, PACHECO I. Detection of quantitative trait loci controlling the content of phenolic compounds in an Asian plum (L.) F1population. Frontiers in Plant Science, 2021, 12: 679059.
[66] 焦春雨, 陶可全, 于泽源, 沈铁恒. 中国李果实品质遗传倾向研究. 北方园艺, 1999(2): 23-25.
JIAO C Y, TAO K Q, YU Z Y, SHEN T H. Study on genetic tendency of fruit quality of China plum. Northern Horticulture, 1999(2): 23-25. (in Chinese)
[67] DE MORI G, SAVAZZINI F, GEUNA F. Molecular tools to investigate Sharka disease inspecies. Applied Plant Biotechnology for Improving Resistance to Biotic Stress. Amsterdam: Elsevier, 2020: 203-223.
[68] ATANASSOV D. Plum pox. A new virus disease. Annal of the University of Sofia Faculty Agriculture and Silvicultural, 1932, 11: 49-69.
[69] HADIDI A, BARBA M, CANDRESSE T, JELKMANN W. Virus and Virus-Like Disease of Pome and stone Fruits. APS Press/American Phytopathological Society, 2011.
[70] CAMBRA M CAPOTE N MYRTA A LLÁCER G. Plum pox virus and the estimated costs associated with sharka disease. EPPO Bulletin, 2006, 36(2): 202-204.
[71] GARCÍA J A, GLASA M, CAMBRA M, CANDRESSE T. Plum pox virus and sharka: A model potyvirus and a major disease. Molecular Plant Pathology, 2014, 15(3): 226-241.
[72] NEUMÜLLER M, LANZL S, HARTMANN W, FEUCHT W, TREUTTER D. Towards an understanding of the inheritance of hypersensitivity resistance against the sharka virus in European plum (L.): Generation of interspecific hybrids with lower ploidy levels. Acta Horticulturae, 2009, 814: 721-726.
[73] RUBIO M, RODRÍGUEZ-MORENO L, BALLESTER A R, DE MOURA M C, BONGHI C, CANDRESSE T, MARTÍNEZ-GÓMEZ P. Analysis of gene expression changes in peach leaves in response to Plum pox virus infection using Rna-Seq. Molecular Plant Pathology, 2015, 16(2): 164-176.
[74] ZURIAGA E, SORIANO J M, ZHEBENTYAYEVA T, ROMERO C, DARDICK C, CAÑIZARES J, BADENES M L. Genomic analysis reveals MATH gene (s) as candidate (s) for plum pox virus (PPV) resistance in apricot (L.). Molecular Plant Pathology, 2013, 14(7): 663-677.
[75] MARIETTE S, WONG JUN TAI F, ROCH G, BARRE A, CHAGUE A, DECROOCQ S, GROPPI A, LAIZET Y, LAMBERT P, TRICON D, NIKOLSKI M, AUDERGON J M, ABBOTT A G, DECROOCQ V. Genome-wide association links candidate genes to resistance to plum pox virus in apricot (). The New Phytologist, 2016, 209(2): 773-784.
[76] DECROOCQ S, CHAGUE A, LAMBERT P, ROCH G, AUDERGON J M, GEUNA F, CHIOZZOTTO R, BASSI D, DONDINI L, TARTARINI S, SALAVA J, KRŠKA B, PALMISANO F, KARAYIANNIS I, DECROOCQ V. Selecting with markers linked to the PPVres major QTL is not sufficient to predict resistance to plum pox virus (PPV) in apricot. Tree Genetics & Genomes, 2014, 10(5): 1161-1170.
[77] PASSARO M, GEUNA F, BASSI D, CIRILLI M. Development of a high-resolution melting approach for reliable and cost-effective genotyping of PPVres locus in apricot (.). Molecular Breeding, 2017, 37(6): 74.
[78] ILARDI V, TAVAZZA M. Biotechnological strategies and tools for Plum pox virus resistance: Trans-, intra-, cis-genesis, and beyond. Frontiers in Plant Science, 2015, 6: 379.
[79] LIMERA C, SABBADINI S, SWEET J B, MEZZETTI B. New biotechnological tools for the genetic improvement of major woody fruit species. Frontiers in Plant Science, 2017, 8: 1418.
[80] GARCÍA-ALMODÓVAR R C, CLEMENTE-MORENO M J, DÍAZ- VIVANCOS P, PETRI C, RUBIO M, PADILLA I M G, ILARDI V, BURGOS L. Greenhouse evaluation confirmssharka resistance of genetically engineered h-UTR/P1 plum plants. Plant Cell, Tissue and Organ Culture, 2015, 120(2): 791-796.
[81] SIDOROVA T, PUSHIN A, MIROSHNICHENKO D, DOLGOV S. Generation of transgenic rootstock plum ((L. ×Lindl.) × (Ehrh.)) using hairpin-RNA construct for resistance to the Plum pox virus. Agronomy, 2017, 8(1): 2.
[82] YAMANE H, TAO R, SUGIURA A. Identification and cDNA cloning for S-RNases in self-incompatible Japanese plum (Lindl. cv. Sordum). Plant Biotechnology, 1999, 16(5): 389-396.
[83] BEPPU K, YAMANE H, YAEGAKI H, YAMAGUCHI M, KATAOKA I, TAO R. Diversity of-RNase genes and-haplotypes in Japanese plum (Lindl.). The Journal of Horticultural Science and Biotechnology, 2002, 77(6): 658-664.
[84] BEPPU K, TAKEMOTO Y, YAMANE H, YAEGAKI H, YAMAGUCHI M, KATAOKA I, TAO R. Determination of S-haplotypes of Japanese plum (Lindl.) cultivars by PCR and cross-pollination tests. Journal of Horticultural Science, 2003, 78(3): 315-318.
[85] BEPPU K, YAMANE H, YAEGAKI H, YAMAGUCHI M, TAO R. KATAOKA I. Analysis of S-RNase genes in self-compatible cultivars of Japanese plum, ‘Methley’, ‘Karari’ and ‘Kosyu’. Journal of the Japanese Society for Horticultural Science, 2004, 73(Suppl. 2): 253.
[86] SAPIR G, STERN R, EISIKOWITCH D, GOLDWAY M. Cloning of four new Japanese plum S-alleles and determination of the compatibility between cultivars by PCR analysis. The Journal of Horticultural Science and Biotechnology, 2004, 79(2): 223-227.
[87] BEPPU K, KOMATSU N, YAMANE H, YAEGAKI H, YAMAGUCHI M, TAO R, KATAOKA I.-haplotype confers self-compatibility in Japanese plum (Lindl.). The Journal of Horticultural Science and Biotechnology, 2005, 80(6): 760-764.
[88] BEPPU K, SYOGASE K, YAMANE H, TAO R, KATAOKA I. Inheritance of self-compatibility conferred by the Se-haplotype of Japanese plum and development of Se-RNase gene-specific PCR primers. The Journal of Horticultural Science and Biotechnology, 2010, 85(3): 215-218.
[89] SUTHERLAND B G, ROBBINS T P, TOBUTT K R. Primers amplifying a range ofS-alleles. Plant Breeding, 2004, 123(6): 582-584.
[90] SUTHERLAND B G, TOBUTT K R, ROBBINS T P. Molecular genetics of self-incompatibility in plums. Acta Horticulturae, 2004(663): 557-562.
[91] VIEIRA E A, NODARI R O, DANTAS A C M, DUCROQUET J P H J, DALBÓ M, BORGES C V. Genetic mapping of Japanese plum. Cropp Breeding and Applied Biotechnology, 2005, 5(1): 29-37.
[92] CARRASCO B, GONZÁLEZ M, GEBAUER M, GARCÍA-GONZÁLEZ R, MALDONADO J, SILVA H. Construction of a highly saturated linkage map in Japanese plum (L.) using GBS for SNP marker calling. PLoS One, 2018, 13(12): e0208032.
[93] ZHANG Q P, WEI X, LIU N, ZHANG Y P, XU M, ZHANG Y J, MA X X, LIU W S. Construction of an SNP-based high-density genetic map for Japanese plum in a Chinese population using specific length fragment sequencing. Tree Genetics & Genomes, 2020, 16(1): 18.
[94] CALLAHAN A M, ZHEBENTYAYEVA T N, HUMANN J L, SASKI C A, GALIMBA K D, GEORGI L L, SCORZA R, MAIN D, DARDICK C D. Defining the ‘HoneySweet’ insertion event utilizing NextGen sequencing and agenome assembly of plum (). Horticulture Research, 2021, 8: 8.
[95] SCORZA R, CALLAHAN A, DARDICK C, RAVELONANDRO M, POLAK J, MALINOWSKI T, ZAGRAI I, CAMBRA M, KAMENOVA I. Genetic engineering ofresistance: ‘HoneySweet’ plum-From concept to product. Plant Cell, Tissue and Organ Culture (PCTOC), 2013, 115(1): 1-12.
[96] FANG Z Z, KUI L W, DAI H, ZHOU D R, JIANG C C, ESPLEY R V, DENG C, LIN Y J, PAN S L, YE X F. The genome of low-chill Chinese plum ‘Sanyueli’ (Lindl.) provides insights into the regulation of the chilling requirement of flower buds. Molecular Ecology Resources, 2022, 22(5): 1919-1938.
[97] LIU C Y, FENG C, PENG W Z, HAO J J, WANG J T, PAN J J, HE Y H. Chromosome-level draft genome of a diploid plum (). GigaScience, 2020, 9(12): giaa130.
[98] JO Y, LIAN S, CHO J K, CHOI H, CHU H, CHO W K.transcriptome assembly of two differentcultivars. Genomics Data, 2015, 6: 262-263.
[99] JO Y, CHOI H, LIAN S, CHO J K, CHU H, CHO W K. Identification of viruses infecting six plum cultivars in Korea by RNA-sequencing. PeerJ, 2020, 8: e9588.
[100] KIM H Y, SAHA P, FARCUH M, LI B S, SADKA A, BLUMWALD E. RNA-seq analysis of spatiotemporal gene expression patterns during fruit development revealed reference genes for transcript normalization in plums. Plant Molecular Biology Reporter, 2015, 33(6): 1634-1649.
[101] GONZÁLEZ M, MALDONADO J, SALAZAR E, SILVA H, CARRASCO B.transcriptome assembly of ‘Angeleno’ and ‘Lamoon’ Japanese plum cultivars (). Genomics Data, 2016, 9: 35-36.
[102] FANG Z Z, ZHOU D R, YE X F, JIANG C C, PAN S L. Identification of candidate anthocyanin-related genes by transcriptomic analysis of ‘Furongli’ plum (Lindl.) during fruit ripening using RNA-seq. Frontiers in Plant Science, 2016, 7: 1338.
[103] FARCUH M, LI B S, RIVERO R M, SHLIZERMAN L, SADKA A, BLUMWALD E. Sugar metabolism reprogramming in a non- climacteric bud mutant of a climacteric plum fruit during development on the tree. Journal of Experimental Botany, 2017, 68(21/22): 5813-5828.
[104] PICCOLO E L, ARANITI F, LANDI M, MASSAI R, GUIDI L, ABENAVOLI M R, REMORINI D. Girdling stimulates anthocyanin accumulation and promotes sugar, organic acid, amino acid level and antioxidant activity in red plum: An overview of skin and pulp metabolomics. Scientia Horticulturae, 2021, 280: 109907.
[105] PETRI C, WEBB K, HILY J M, DARDICK C, SCORZA R. High transformation efficiency in plum (L.): A new tool for functional genomics studies inspp. Molecular Breeding, 2008, 22(4): 581-591.
[106] MANTE S, MORGENS P H, SCORZA R, CORDTS J M, CALLAHAN A M.-mediated transformation of plum (L.) hypocotyl slices and regeneration of transgenic plants. Bio/Technology, 1991, 9(9): 853-857.
[107] GONZALEZ-PADILLA I M, WEBB K, SCORZA R. Early antibiotic selection and efficient rooting and acclimatization improve the production of transgenic plum plants (L.). Plant Cell Reports, 2003, 22(1): 38-45.
[108] TIAN L N, WEN Y, JAYASANKAR S, SIBBALD S. Regeneration ofLindl (Japanese plum) from hypocotyls of mature seeds. In Vitro Cellular & Developmental Biology - Plant, 2007, 43(4): 343-347.
[109] RAVELONANDRO M, SCORZA R, POLAK J, CALLAHAN A, KRŠKA B, KUNDU J B, BRIARD P. ‘Honey Sweet’ plum-A valuable genetically engineered fruit-tree cultivar. Food and Nutrition Sciences, 2013, 4(6): 45-49.
[110] SRINIVASAN C, DARDICK C, CALLAHAN A, SCORZA R. Plum () trees transformed with poplar FT1 result in altered architecture, dormancy requirement, and continuous flowering. PLoS One, 2012, 7: E40715.
[111] 刘威生. 最新李、杏新品种(系)简介. 果农之友, 2017(5): 4-6.
LIU W S. Brief introduction of the latest new plum and apricot varieties (lines). Fruit Growers’ Friend, 2017(5): 4-6. (in Chinese)
[112] RUIZ D, COS J, NICOLÁS-ALMANSA M, EGEA J, GARCÍA F, CARRILLO A, RUBIO M, LÓPEZ D, SALAZAR J, GUEVARA A. New promising Japanese plum cultivars for warm areas from CEBAS-CSIC/IMIDA breeding programme. Acta Horticulturae, 2021, 1322: 55-60.
An Overview of the Worldwide Plum Breeding

Liaoning Institute of Pomology, Yingkou 115009, Liaoning
Plum is a significant fruit tree worldwide, with the two main species beingand. Chinese plum, originating from China, exhibits high genetic heterogeneity and abundant genetic variation in fruit traits. It is speculated that wild Chinese plums still exist in the Yangtze River basin. Throughout Chinese history, the plum cultivation and varieties were recorded by many secretaries of the Qin Dynasty, the Han Dynasty, the Jin Dynasty, the Northern Song Dynasty, and the Ming Dynasty. Similarly, in other countries, such as Greece, France, Finland, the Czech Republic, and Russia, there are written records of plum, describing its origin and 14 varieties. Plum was gradually introduced into the European continent during the Roman era, and numerous new varieties have since been cultivated to improve the fruit’s quality, commercial value, and meet market demand. There were currently 88 independently bred plum varieties in China that have passed variety approval and been officially published. Breeding traits of common concern include fruit size, peel color, polyphenols, soluble solids, fruit maturity, cold resistance, disease resistance, etc. Breeding methods range from traditional methods to molecular-assisted breeding, tissue culture, and transgenic breeding. Many excellent varieties with good fresh food quality, storage and transportation resistance, and adaptability have been bred through continuous improvement of breeding methods, promoting the development of the global plum industry. China is the world’s largest plum producer, accounting for 54.94% of total production and 74.75% of total cultivation area worldwide. The rich natural resources of plum in China provide solid material and diversity guarantee for germplasm exploration and breeding application. To further strengthen the international advantages of plum germplasm resources and cultivation areas in China, and to enhance the core competitiveness of national breeding, this study reviewed the global history process of plum breeding and improvement, including the origin, dissemination, early cultivation history of plum in the world, recent research on the main breeding traits, and commercial breeding strategies for modern plum.
plum; breeding; variety; strategy
2022-05-25;
2023-03-01
辽宁省自然科学基金(2021-MS-054)、国家园艺作物种质基础服务平台项目(NHGRC2021-NH10)、中央引导地方科技发展专项(2020JH6/10500070)
刘硕,E-mail:liushuo028@163.com。通信作者刘威生,E-mail:wsliulaas@163.com
10.3864/j.issn.0578-1752.2023.09.011
(责任编辑 赵伶俐)
