表皮蛋白基因参与锈赤扁谷盗磷化氢抗性形成
2023-05-12陈二虎沈丹蓉杜文蔚孟宏杰唐培安
陈二虎,沈丹蓉,杜文蔚,孟宏杰,唐培安
表皮蛋白基因参与锈赤扁谷盗磷化氢抗性形成

南京财经大学食品科学与工程学院/江苏省现代粮食流通与安全协同创新中心/江苏高校粮油质量安全控制及深加工重点实验室,南京 210023
【目的】作为昆虫表皮重要的结构物质,表皮蛋白(cuticle protein,CP)在昆虫对农药表皮穿透抗性形成过程中承担重要作用。锈赤扁谷盗()磷化氢抗性问题日益突出,论文旨在揭示表皮蛋白基因在该害虫磷化氢抗性形成过程中的作用。【方法】基于联合国粮农组织(FAO)推荐的生物测定方法解析5个地理种群(张家港、湘阴、淮安、怀化和太仓种群)锈赤扁谷盗磷化氢敏感性差异。首先通过锈赤扁谷盗转录组数据鉴定获得4个表皮蛋白基因,进而构建系统发育树,并对相应氨基酸序列进行分析。利用基因定量技术(RT-qPCR)解析上述4个锈赤扁谷盗表皮蛋白基因时空(不同发育阶段和成虫不同组织)和不同磷化氢抗性水平下的表达模式,以及表皮蛋白基因对磷化氢胁迫响应的表达模式。使用RNAi(RNA interference)技术对特定的表皮蛋白基因()进行沉默,并研究被有效沉默后锈赤扁谷盗磷化氢敏感性变化情况。【结果】磷化氢敏感性测定结果显示,不同地理种群锈赤扁谷盗磷化氢敏感性水平差异显著,药剂抗性倍数(RR)范围为7.2—1 906.8。系统发育分析显示锈赤扁谷盗4个表皮蛋白均隶属于CPR家族RR2亚家族,且它们氨基酸序列都拥有RR2型几丁质结合域,将其分别命名为、、和。基因表达模式分析显示4个表皮蛋白基因均在锈赤扁谷盗蛹期特异性高表达,且在成虫外周组织中表达水平较高;此外,表皮蛋白基因均在磷化氢极高抗种群中(太仓种群,RR=1 906.8)显著高表达,且锈赤扁谷盗经磷化氢熏蒸胁迫后,表皮蛋白基因可被显著诱导表达。最后,选择进行功能验证,注射dsRNA干扰极高抗种群(TC)的表达后,导致锈赤扁谷盗对磷化氢抗性水平显著降低。【结论】表皮蛋白基因过表达参与锈赤扁谷盗磷化氢抗性形成。
锈赤扁谷盗;磷化氢抗性;表皮蛋白;RNA干扰
0 引言
【研究意义】磷化氢(phosphine,PH3)是国际上使用最为广泛的储粮害虫熏蒸剂,用于防治赤拟谷盗()、锈赤扁谷盗()、谷蠹()等储粮害虫[1-3]。然而,由于粮食仓储行业对这种熏蒸剂的长期过度使用,导致不同种类储粮害虫均对磷化氢产生一定程度的抗性,其中尤以锈赤扁谷盗的抗性问题最为突出,已经成为制约粮食行业健康发展的关键因素[4-5]。尽管磷化氢抗性发生严重,但目前尚无其他熏蒸剂可以取而代之,因此磷化氢抗性已经对国家粮食安全构成巨大威胁,是粮食行业亟需解决的问题。避免和减缓磷化氢抗性发展已成为研究者们关注的焦点,开展磷化氢抗性形成分子机制的研究,对缓解粮食行业面临的困局具有重要意义[6]。【前人研究进展】有关磷化氢作用机制及昆虫抗药性形成机理的研究结果表明,磷化氢主要利用害虫的呼吸作用进入虫体内,其作用靶标为线粒体组织,因而昆虫可通过调节线粒体和呼吸代谢相关基因的表达水平,降低呼吸速率,减少熏蒸剂的吸收,进而使昆虫获得磷化氢抗性[7-8]。例如,赤拟谷盗和谷蠹磷化氢抗性品系的呼吸速率显著低于敏感品系[9-10],而且昆虫磷化氢抗性形成还与线粒体二聚体酶二氢硫辛酰胺脱氢酶基因突变密切相关[11-12]。此外,解毒代谢途径是形成磷化氢抗性的另一个重要机制,包括细胞色素P450、羧酸酯酶(carboxylesterase)、谷胱甘肽硫转移酶(glutathione S-transferase)等解毒代谢酶均被证实参与赤拟谷盗等储粮害虫的磷化氢抗性形成[13-14]。同时,已有研究发现昆虫可通过提高表皮蛋白、几丁质合成酶等基因的转录水平来改变表皮结构和增厚表皮,从而降低表皮穿透性来阻止或减少杀虫剂进入虫体,进而形成抗药性,证明昆虫表皮穿透性亦可介导昆虫抗药性形成[15-17]。例如,德国小蠊()表皮相关基因的过表达引起上表皮碳氢化合物含量增加,表皮通透性显著降低,造成昆虫对高效氯氰菊酯产生抗性[18-19]。昆虫表皮的主要结构物质包括几丁质和表皮蛋白,其中表皮蛋白种类繁多,并且其在昆虫中的表达模式具有明显的时空特异性。表皮蛋白家族基因数量众多,约占基因总数的1%,且结构复杂多样,主要包括CPR(拥有Rebers and Riddiford(R&R)保守基序)、CPAP1(含1个ChtBD2几丁质结合域)、CPAP3(含3个ChtBD2几丁质结合域)、Tweedle(含4个氨基酸保守区域)、CPF(含44个氨基酸的保守区域)等10余个家族类群[20-21]。表皮蛋白在昆虫表皮各层中均有分布和表达[22-23],且前人研究已经证实表皮蛋白基因参与介导昆虫对不同药剂的抗性形成过程[17]。【本研究切入点】表皮蛋白基因介导昆虫对杀虫剂穿透抗性的研究已逐渐成为热点问题,之前的研究发现表皮蛋白基因和参与赤拟谷盗磷化氢抗性形成,进一步证明二者之间存在密切联系[24]。鉴于此,本研究以磷化氢抗性问题突出的锈赤扁谷盗为研究对象,在课题组前期研究的基础上,分别采集不同地理种群试虫,综合运用生物测定、RT-qPCR、RNA干扰等方法探究表皮蛋白基因与磷化氢抗性间的关系。【拟解决的关键问题】阐释表皮蛋白基因介导锈赤扁谷盗磷化氢抗性形成的基本科学问题,为储粮害虫磷化氢抗性治理提供理论依据。
1 材料与方法
试验于2020年3月至2021年6月在南京财经大学食品科学与工程学院储粮害虫防治实验室完成。
1.1 供试昆虫
试验所用5个地理种群锈赤扁谷盗于2016年采自张家港(Zhangjiagang,ZJG)、湘阴(Xiangyin,XY)、淮安(Huaian,HA)、怀化(Huaihua,HH)和太仓(Taicang,TC)储粮生态区,并在实验室内连续饲养20代以上。试验过程中所使用的人工饲料配方为全麦粉﹕酵母粉=19﹕1,无光照,饲养条件为温度(30±1)℃,相对湿度(75±5)%。
1.2 磷化氢敏感性测定
基于联合国粮农组织(1975)推荐的磷化氢生物测定方法:将试虫置于广口熏蒸瓶并密封瓶口,平衡4 h后注入不同浓度磷化氢气体,正常试虫饲养条件下密闭熏蒸20 h;处理结束后于通风橱中散去磷化氢气体(约10 min),待气体散去后统计试虫死亡数量(毛笔轻触虫体尾部,附肢无反应即为死亡),连续统计14 d。生物测定试验设置3次重复,所选试虫均为7日龄成虫,每个重复包含50头试虫。以联合国粮农组织推荐的磷化氢敏感基线(LC50=0.011 mg·L-1)进行锈赤扁谷盗抗性倍数(resistance ratio,RR)的计算。抗性倍数=不同地理种群锈赤扁谷盗LC50/0.011 mg·L-1。其中,抗性倍数介于2.5—10为低抗性,10—40倍为中等抗性,160倍以上为极高抗性。
1.3 生物信息学分析
依据锈赤扁谷盗转录组数据(accession number:SRA245468),鉴定获得4个表皮蛋白基因,分别使用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf. html)、SignalP 4.1(http://www.cbs.dtu.dk/services/ SignalP)和在线软件InterPro(http://www.ebi.ac. uk/interpro/search/sequence-search)预测表皮蛋白基因的完整开放阅读框(open reading frame,ORF)、信号肽和保守特征氨基酸序列。针对系统发育分析研究,首先使用ClustalW软件对昆虫表皮蛋白进行氨基酸序列的多重比对,接着利用邻接法(neighbor- joining,NJ)在Mega 6.0软件中构建锈赤扁谷盗和赤拟谷盗表皮蛋白的进化树,各树分支1 000次重复抽样检验[25]。
1.4 不同发育阶段表皮蛋白基因表达模式分析
锈赤扁谷盗(张家港种群)不同发育阶段样品收集:将虫卵在实验室条件下培养至卵孵化,分别收集1—4龄幼虫、预蛹、蛹和7日龄成虫各30头。用Trizol法提取各样品总RNA(RNA浓度为500—600 ng·μL-1),RQ1 Rnase-Free Dnase清除基因组DNA。通过核酸浓度测定仪进行样本RNA浓度和纯度测定,同时使用琼脂糖凝胶电泳对样品RNA完整性进行检测。通过反转录试剂盒HiScript® II 1st Strand cDNA Synthesis Kit(+gDNA wiper)(南京诺唯赞生物科技有限公司,中国)合成第一链cDNA,所获得的目标产物于-20℃冰箱中进行保存,以备后续使用。
锈赤扁谷盗4个表皮蛋白基因的特异性RT-qPCR引物如表1所示,以和为内参基因[26]。定量PCR试验使用20 μL反应体系,并利用ChamQTMSYBR® qPCR Master Mix(Low Rox Premixed)试剂盒开展试验,反应液包括2 μL Template cDNA、10 μL ChamQ SYBR qPCR Master Mix(Low Rox Premixed)、上下游引物各1 μL、Rnaes-free water 6 μL。反应程序:95℃预变性5 min,95℃变性10 s,60℃延伸30 s(40个循环),最后95℃延伸30 s,整个PCR过程均在ABI 7500 PCR系统(Applied Biosystems,USA)中进行。利用2-∆∆Ct方法进行定量数据分析[27]。
1.5 不同组织表皮蛋白基因表达模式分析
解剖羽化后7日龄成虫50头(张家港种群试虫),分别收集不同组织部位表皮(头、胸、腹)、翅、足、脂肪体的样本。总RNA提取、cDNA合成和RT-qPCR试验均参照1.4节,设置3个生物学重复。
1.6 不同地理种群表皮蛋白基因的表达模式分析
供试锈赤扁谷盗试虫分别采自张家港、湘阴、淮安、怀化和太仓,每个地理种群分别选取30头7日龄成虫为一个样本,总RNA提取、cDNA合成和RT-qPCR试验参照1.4节,共设置3次生物学重复。
1.7 磷化氢胁迫下表皮蛋白基因表达模式分析
选取锈赤扁谷盗张家港种群7日龄成虫开展磷化氢胁迫试验。根据1.2节磷化氢敏感性测定结果,采用LC30作为磷化氢熏蒸浓度,熏蒸处理1、2、6和12 h后快速挑取存活的试虫转入液氮冷冻备用。参照1.4节的方法进行总RNA提取、cDNA合成和RT-qPCR试验,共设置3个生物学重复,每个重复均包含50头试虫。

表1 本研究所用引物序列
1.8 RNA干扰后CfRR2-1表达量检测及磷化氢敏感性测定
选择进行功能验证,根据基因序列信息设计含有T7启动子的引物(表1)进行PCR扩增,根据T7 RiboMAXTMExpress RNA(Promega)试剂盒体外合成ds和ds。参照Huang等[13]的方法,将100 nL(1 μg·μL-1)dsRNA注射到锈赤扁谷盗(太仓种群试虫)蛹内(注射位置为蛹的腹侧第一节或第二节),以注射等量ds和正常饲养的试虫作为对照。待蛹羽化后正常饲养,收集第7天成虫。参照1.4节的方法进行总RNA提取,合成cDNA,并采用基因定量技术检测相应表皮蛋白基因()的沉默效率。最后,被有效沉默后进行磷化氢敏感性检测,选取7日龄成虫,并以磷化氢的亚致死浓度(LC30)对试虫进行处理,并参照1.2节方法进行熏蒸以及统计死亡数量,以此分析磷化氢敏感性变化情况。共设置3次生物学重复,每次重复均包含30头试虫。
1.9 数据统计与分析
数据均表示为平均值±标准差,其中对于基因表达模式、基因沉默效率和试虫死亡率数据均采用单因素方差分析(one-way ANOVA)进行均值多重比较,并利用图基检验法(Tukey’s test)进行显著性分析(<0.05);基于独立样本检验(student-test)对磷化氢胁迫下基因表达模式进行两组数据比较的显著性差异分析(*<0.05)。
2 结果
2.1 不同地理种群锈赤扁谷盗磷化氢敏感性
5个地理种群锈赤扁谷盗对磷化氢抗性倍数介于7.2—1 906.8(表2)。其中张家港(ZJG,抗性倍数=7.2)属于低抗性种群,湘阴(XY,抗性倍数=29.6)属于中等抗性种群,淮安(HA,抗性倍数=325.4)、怀化(HH,抗性倍数=362.7)和太仓(TC,抗性倍数=1 906.8)均属于极高抗性种群。
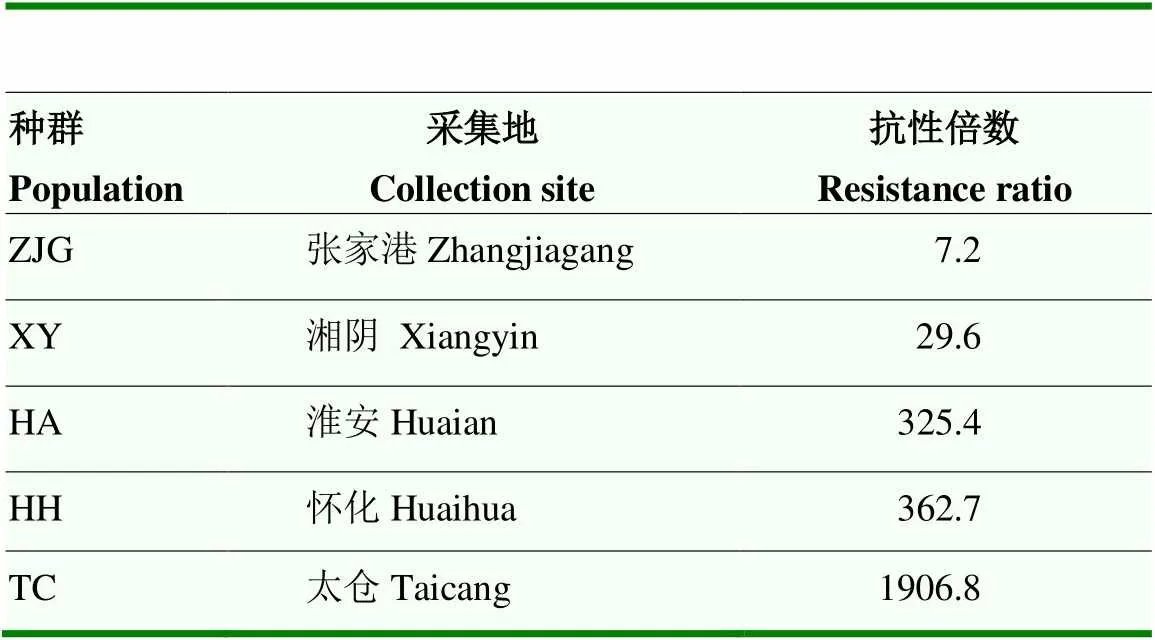
表2 不同地理种群锈赤扁谷盗磷化氢敏感性测定
2.2 锈赤扁谷盗4个表皮蛋白序列分析
表皮蛋白系统发育分析表明锈赤扁谷盗4个表皮蛋白基因均隶属昆虫CPR家族的RR2亚家族,分别命名为、、和(图1-A)。氨基酸序列分析显示上述4个表皮蛋白基因分别编码172、176、145和173个氨基酸(图1-B),其中CfRR2-1蛋白信号肽序列位于第1—20氨基酸位点,CfRR2-2、CfRR2-3和CfRR2-4蛋白信号肽序列均位于第1—18氨基酸位点。保守结构域分析表明CfRR2-1(第63—131个氨基酸)、CfRR2-2(第60—128个氨基酸)、CfRR2-3(第50—118个氨基酸)和CfRR2-4(第59—127个氨基酸)蛋白序列均包含R&R(Rebers and Riddiford)型特征序列(pfam00379),且上述表皮蛋白的保守区域均属于典型的RR2几丁质结合域(图1-B)。
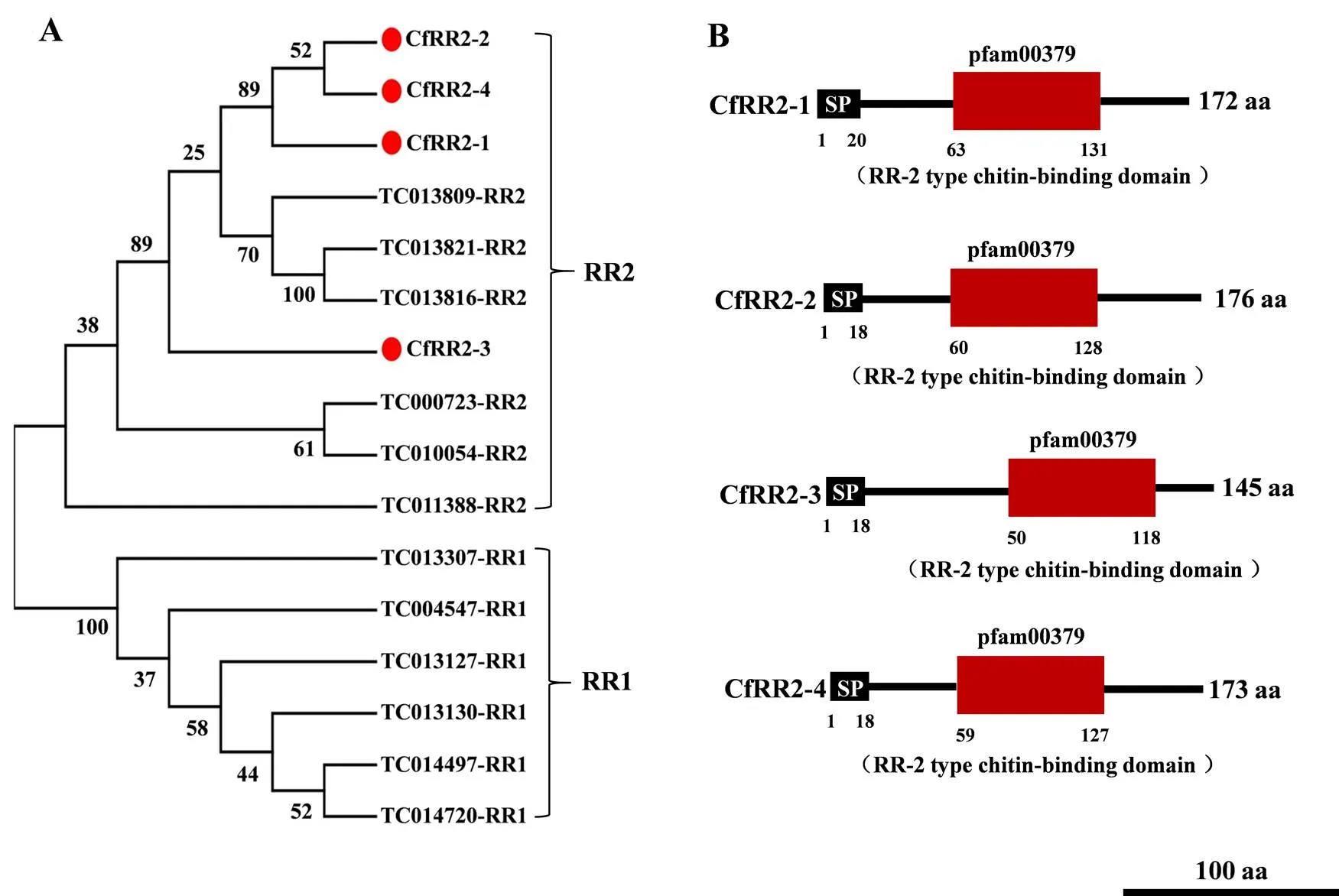
红色圆圈代表锈赤扁谷盗表皮蛋白,黑框代表信号肽序列,红色框代表保守结构域
2.3 表皮蛋白基因在锈赤扁谷盗不同发育阶段的表达模式
表皮蛋白基因在锈赤扁谷盗不同发育阶段的表达模式显示、、和在幼虫期和成虫期表达水平相对较低,均在蛹期表达量最高,呈现特异性高表达,且表达水平均显著高于其他发育阶段(<0.05)(图2)。
2.4 表皮蛋白基因在锈赤扁谷盗不同组织表达模式
锈赤扁谷盗4个表皮蛋白基因均在昆虫外周组织高表达(图3)。其中在锈赤扁谷盗成虫胸部表皮和翅中表达水平显著高于其他组织(<0.05),在头部和腹部表皮的表达量次之,且它们的表达水平均高于内部组织脂肪体(图3-A);在试虫头部表皮组织显著高表达(<0.05),在其他组织间表达量无显著差异(图3-B);除在外周组织翅和足中显著高表达,还在昆虫内部组织脂肪体高表达,且基因表达量显著高于头、胸和腹部表皮组织(<0.05)(图3-C);只在锈赤扁谷盗翅组织特异性高表达(<0.05),在头、胸和腹部表皮组织、足和脂肪体表达量均相对较低(图3-D)。
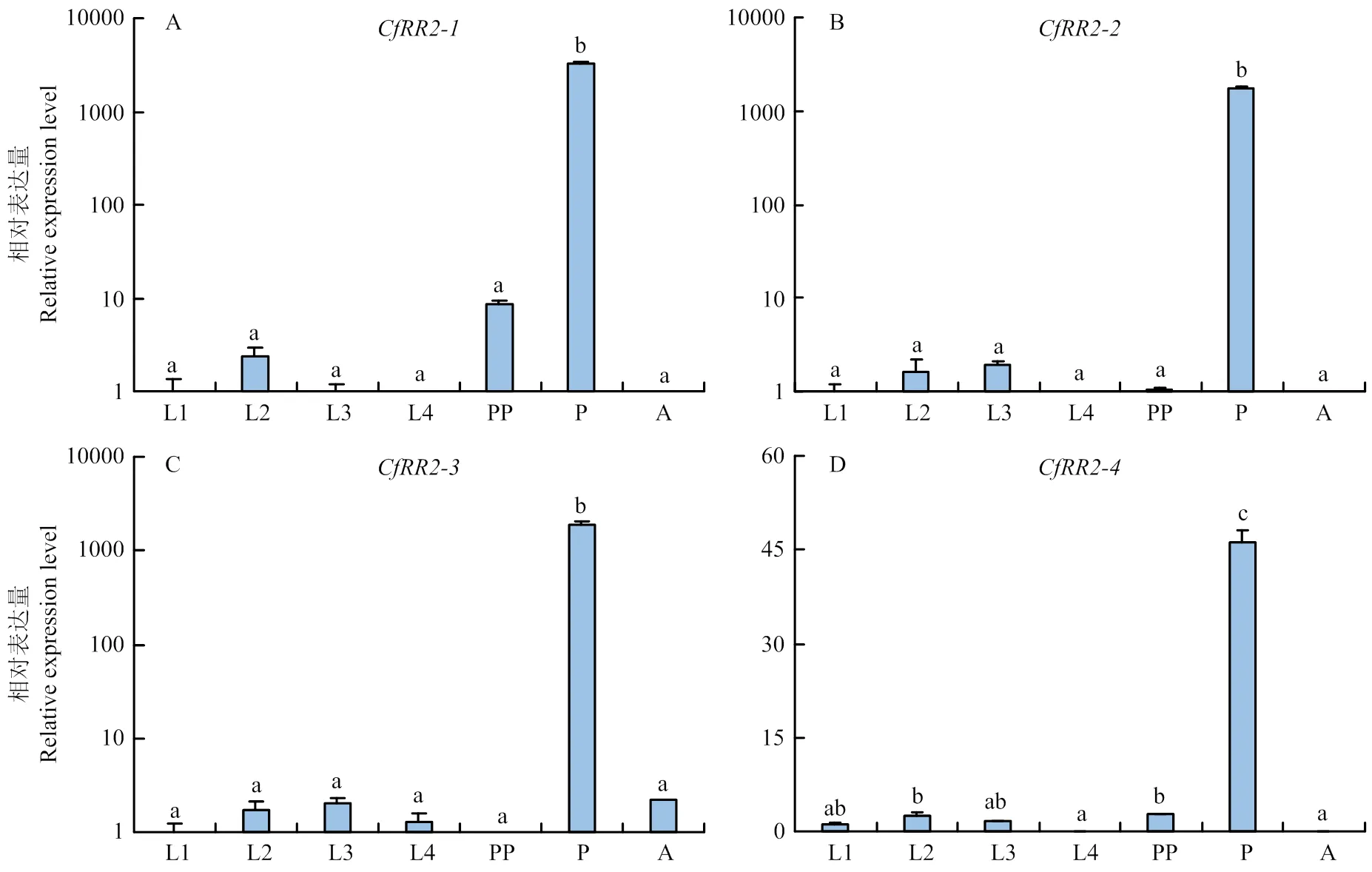
L1—L4:1—4龄幼虫1st-4th instar larva;PP:预蛹期pre-pupal stage;P:蛹期pupal stage;A:成虫adult
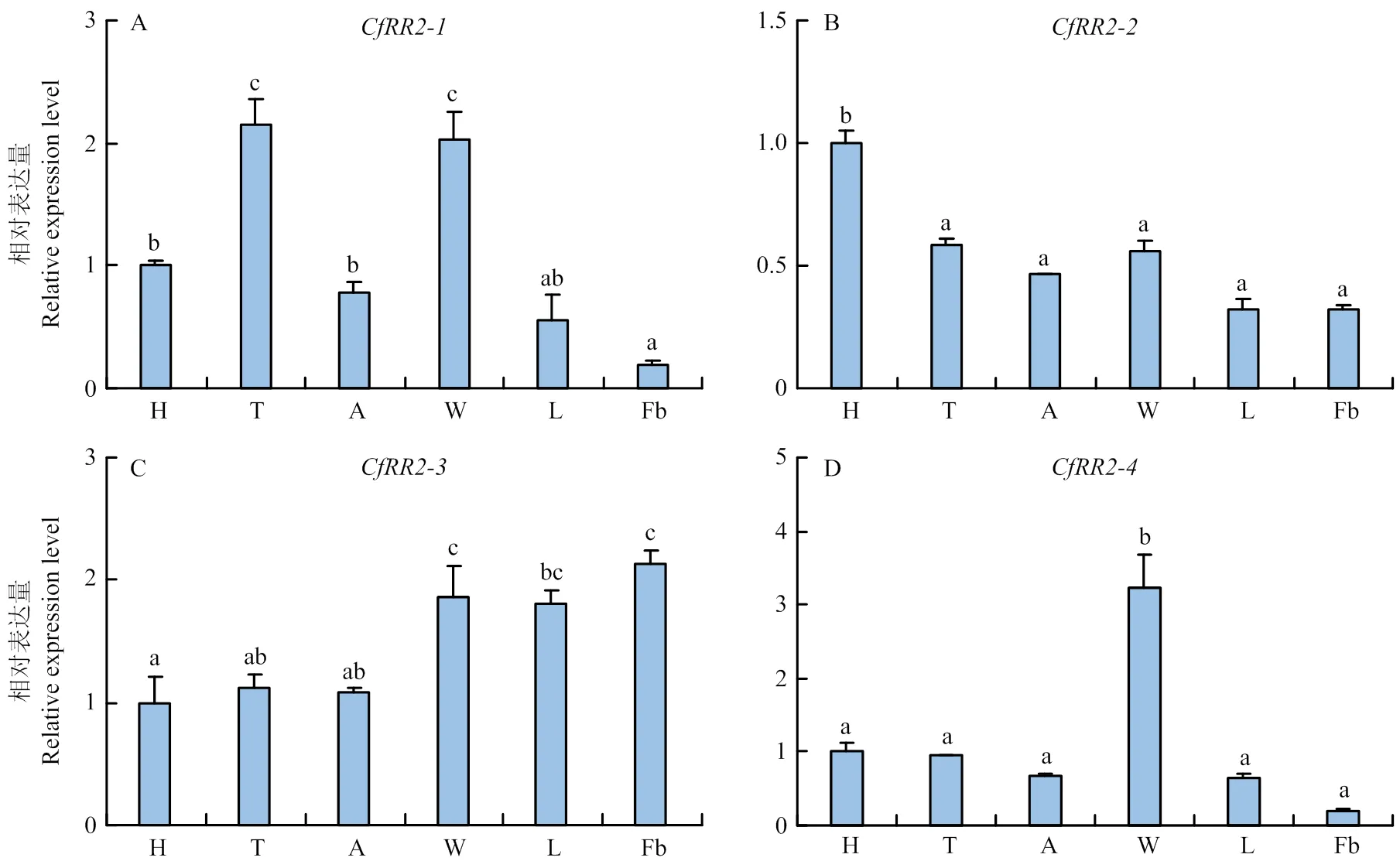
H:头部表皮Head cuticle;T:胸部表皮Thorax cuticle;A:腹部表皮Abdomen cuticle;W:翅Wing;L:足Leg;Fb:脂肪体Fat body
2.5 表皮蛋白基因在不同地理种群锈赤扁谷盗中的表达模式
、、和均在极高抗种群(太仓种群)中特异性高表达,且其表达水平与其他种群间差异显著(<0.05)。在张家港、湘阴、淮安和怀化种群间,上述4个表皮蛋白基因的表达水平均无显著差异(图4)。
2.6 磷化氢胁迫下表皮蛋白基因的表达模式
锈赤扁谷盗4个表皮蛋白基因均可被磷化氢显著诱导表达,且在12 h内基因表达量均呈现先升高后降低的趋势(图5)。其中、和表达量在磷化氢熏蒸后2 h达到峰值,分别是对照组的2.0、3.5和3.5倍;表达量在熏蒸处理6 h达到峰值,是对照组的3.8倍。
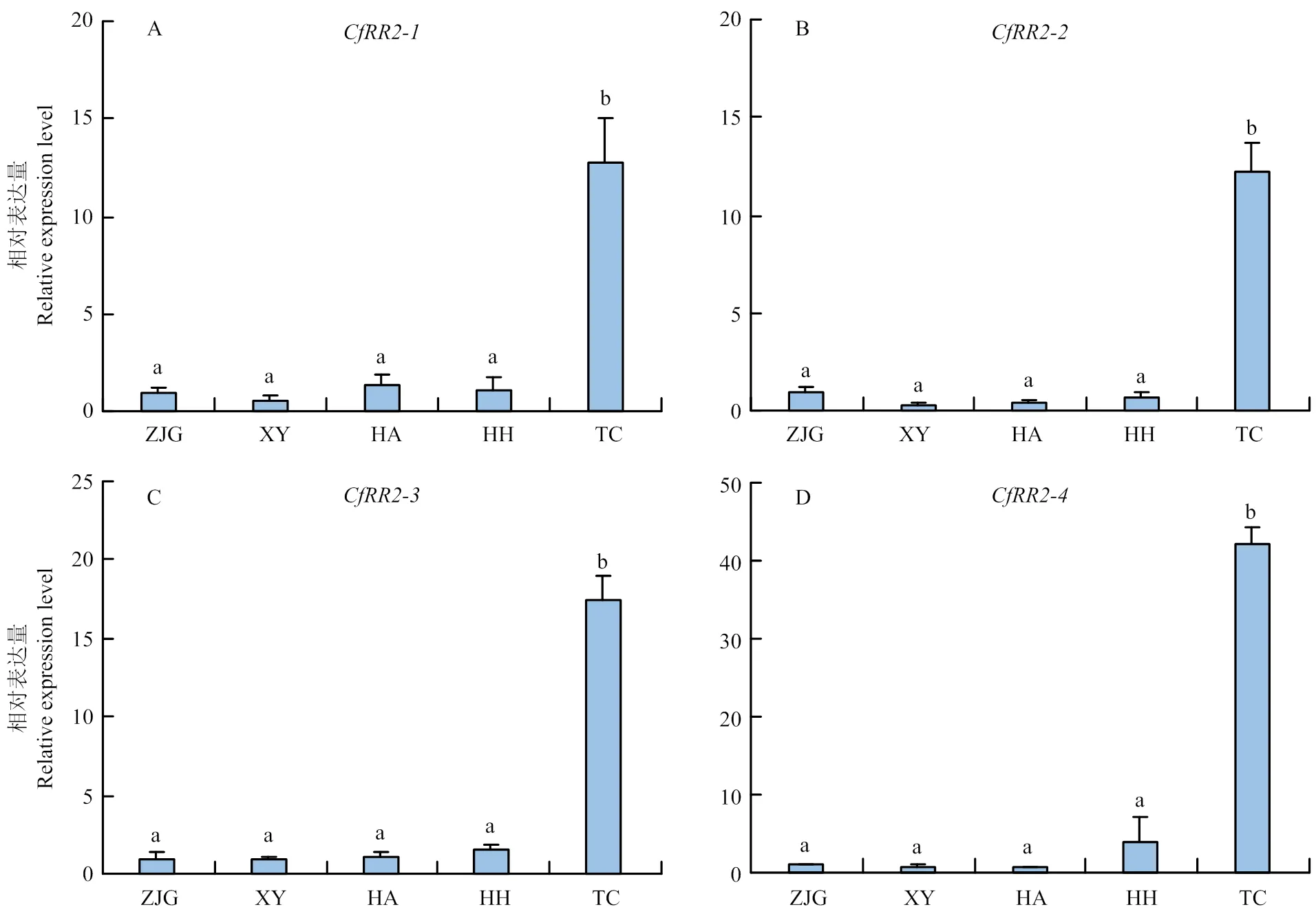
图4 锈赤扁谷盗表皮蛋白基因在不同磷化氢抗性种群中的表达模式
2.7 表皮蛋白基因沉默对锈赤扁谷盗磷化氢敏感性的影响
利用RNAi技术有效沉默极高抗种群(太仓种群)的表达,分析表皮蛋白基因与磷化氢抗性形成的关系。基因定量PCR结果显示RNA干扰能有效沉默的表达,沉默效率达60%,且ds注射组与空白对照组间表皮蛋白基因表达量无显著差异(图6-A)。有效沉默的表达后,锈赤扁谷盗对磷化氢的抗性水平显著降低,磷化氢(LC30)处理后试虫死亡率较对照组显著升高(<0.05),且空白对照组和ds处理组之间试虫死亡率无显著差异(图6-B)。
3 讨论
3.1 锈赤扁谷盗磷化氢抗性发生严重
由于磷化氢熏蒸剂在世界范围内的大量使用,导致众多储粮害虫对磷化氢产生不同程度的抗药性,其中尤以锈赤扁谷盗磷化氢抗性问题最为突出,已逐渐成为限制世界各国粮食储藏行业健康发展的制约因素之一,目前已经在美国、印度、澳大利亚等国家发现锈赤扁谷盗的磷化氢抗性种群,且其抗性水平远高于其他储粮害虫[28-29]。已有研究报道锈赤扁谷盗是磷化氢抗性发生最为严重的粮食仓储害虫之一,且其在世界范围内分布广泛,一旦发生危害便能给国家粮食行业带来巨大经济损失[1,6]。本研究中,分别采集获得5个地理种群的锈赤扁谷盗,进一步的磷化氢生物测定结果显示锈赤扁谷盗磷化氢抗性倍数最高可达1 900余倍(表2)。该研究结果也直接证实了锈赤扁谷盗抗性问题在我国日益严重,抗性水平甚至已经达到难以控制的地步。然而,目前锈赤扁谷盗对磷化氢产生抗性的机理研究却严重滞后,相关磷化氢抗性分子机制尚属未知。
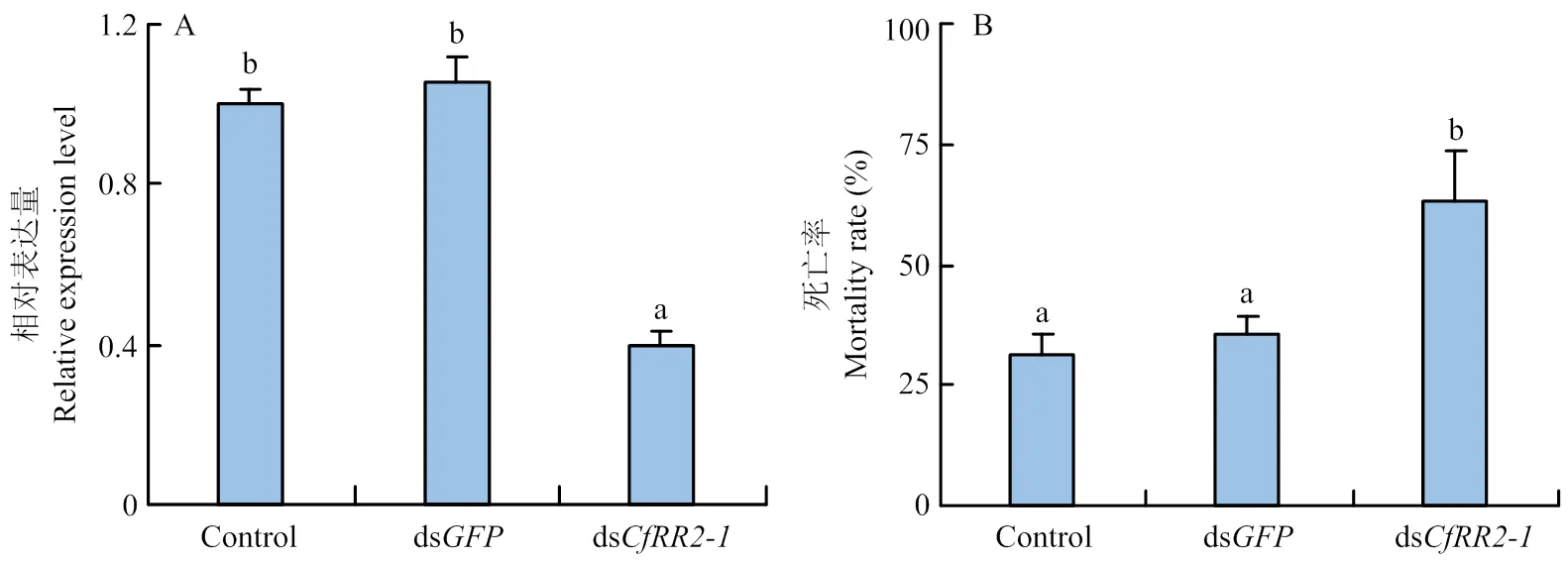
图6 CfRR2-1沉默效率及基因沉默后磷化氢敏感性变化
3.2 锈赤扁谷盗表皮蛋白具有CPR家族典型结构特点
近年来,表皮蛋白基因介导杀虫剂穿透抗性已逐渐成为热点研究问题[17],但表皮蛋白介导磷化氢抗性的报道较为鲜见。值得注意的是,已有研究表明磷化氢不但可以利用呼吸作用伴随空气主动进入昆虫体内,亦可通过扩散作用穿透表皮进入虫体:当昆虫呼吸急促时,呼吸作用表现为主要吸收途径;而当昆虫呼吸作用逐渐减弱时,表皮穿透作用则上升为主要方式,表皮蛋白影响表皮穿透性,因此表皮蛋白基因与磷化氢抗性形成可能存在密切联系[30-31]。本研究在锈赤扁谷盗转录组数据中鉴定获得4个表皮蛋白基因,系统发育分析显示它们均隶属于表皮蛋白CPR家族的RR2亚家族(图1-A)。对家蚕()和双叉犀金龟()的研究表明,CPR家族蛋白与不同类型几丁质间存在相互作用和结合特性[32-33]。氨基酸序列分析表明锈赤扁谷盗4个表皮蛋白均包含保守的RR2型几丁质结合域,具备潜在的几丁质结合能力(图1-B),该特征与昆虫CPR家族蛋白所具备的保守几丁质结合域相符。
3.3 锈赤扁谷盗表皮蛋白基因具有发育阶段和组织表达特异性
表皮蛋白CPR家族主要包含RR1和RR2两个亚家族,研究表明上述两个亚家族基因的表达模式具有明显的组织特异性,即RR1亚家族蛋白主要存在于柔软表皮组织中(如昆虫幼虫表皮),RR2亚家族蛋白通常参与昆虫坚硬表皮组织的形成[34]。与之相似,本研究发现、、和在幼虫阶段的表达均维持在较低水平,而在拥有坚硬外壳的蛹期阶段显著高表达(图2)。成虫不同组织表达水平分析显示,4个表皮蛋白基因均在锈赤扁谷盗外周表皮组织高表达(图3),暗示上述表皮蛋白可能是组成昆虫表皮的重要物质之一,参与表皮形成过程,承担表皮相关的重要功能。大量研究已经证实表皮蛋白基因在昆虫外周组织中具有较强的表达特异性[35],如褐色橘蚜()CPR家族基因在胸腹部表皮的表达量显著高于头部表皮组织[36]。本研究发现锈赤扁谷盗表皮蛋白基因表达模式同样具有复杂性和特异性,如在头胸腹表皮和翅中表达水平显著高于其他组织(图3-A),(图3-B)和(图3-D)分别在头部表皮和翅中特异性高表达。同时,本研究还发现除在外周组织中高表达(图3-C),还在内部器官脂肪体中显著高表达。脂肪体承担昆虫众多生理功能,其中免疫反应尤为重要,例如家蚕脂肪体中发现的高表达表皮蛋白基因,已被证实参与昆虫对大肠杆菌的识别反应[37]。锈赤扁谷盗在成虫脂肪体高表达,据此推断该表皮蛋白可能在昆虫免疫过程中发挥重要作用。
3.4 沉默表皮蛋白基因CfRR2-1显著增强锈赤扁谷盗磷化氢敏感性
表皮蛋白基因在不同地理种群锈赤扁谷盗中的表达量分析显示,、、和均在磷化氢极高抗种群(太仓)中特异性高表达(图4)。在赤拟谷盗[31]、桃蚜()[38]、中华按蚊()[39]、淡色库蚊()[40-41]、冈比亚按蚊()[42]、温带臭虫()[43]等昆虫中同样发现表皮蛋白CPR家族基因在药剂抗性品系中显著高表达。同时,本研究结果显示在经磷化氢熏蒸胁迫处理6 h内,4个表皮蛋白基因均可被显著诱导表达,可能是锈赤扁谷盗响应磷化氢胁迫的应激反应(图5)。类似地,经甲氧虫酰肼药剂胁迫处理后,棉铃虫()2个表皮蛋白CPR家族基因表达量显著升高[44],橘小实蝇()经马拉硫磷处理后,同样发现众多表皮蛋白基因的表达量显著升高[45]。
4个表皮蛋白基因中只有在锈赤扁谷盗外周组织翅和头胸腹表皮中均具有较高表达水平,且表达量都显著高于内部组织脂肪体,暗示其可能在表皮中承担着重要生理功能。基于此,本研究选择开展进一步的功能研究。使用RNAi技术对进行有效沉默,发现经磷化氢(LC30)处理后锈赤扁谷盗的死亡率显著升高(图6),证明表达水平降低后,锈赤扁谷盗对于磷化氢的敏感性显著增强。同样地,淡色库蚊表皮蛋白基因表达水平在拟除虫菊酯抗性品系中显著上调,且该表皮蛋白主要在试虫外周组织足和翅中高表达,利用siRNA沉默该基因导致害虫表皮厚度显著降低,通透性增加,进而杀虫剂在昆虫体表渗透率升高,最终引起试虫对药剂敏感性显著增强[41]。在淡色库蚊溴氰菊酯抗性品系和温带臭虫抗氟氯氰菊酯品系均发现表皮蛋白CPR家族基因显著上调,且利用RNAi技术对表达量上调的表皮蛋白基因进行有效沉默,害虫对农药的抗性水平均显著下降[40,43]。基于上述研究结果可得出特定的表皮蛋白基因参与锈赤扁谷盗对磷化氢抗性形成,进一步推测该害虫可能通过上调特定表皮蛋白基因的表达量来增加表皮厚度或改变表皮结构和组分,从而减小表皮穿透性,降低磷化氢体表渗入,最终形成抗药性[17]。
4 结论
锈赤扁谷盗磷化氢敏感性检测结果表明我国储粮害虫磷化氢抗性发生严重;表皮蛋白CfRR2-1、CfRR2-2、CfRR2-3和CfRR2-4均包含RR2型几丁质结合域,暗示它们具有潜在几丁质结合能力;上述4个表皮蛋白基因具有明显发育阶段和组织表达特异性,均在磷化氢极高抗种群中显著高表达,且磷化氢胁迫可诱导表皮蛋白基因表达;通过RNAi干扰技术沉默特定表皮蛋白基因,锈赤扁谷盗磷化氢敏感性显著升高。综上,表皮蛋白基因的过表达与锈赤扁谷盗磷化氢抗性形成密切相关。
[1] Phillips T W, Throne J E. Biorational approaches to managing stored-product insects. Annual Review of Entomology, 2010, 55: 375-397.
[2] Losey S M, Daglish G J, Phillips T W. Orientation of rusty grain beetles,(Coleoptera: Laemophloeidae), to semiochemicals in field and laboratory experiments. Journal of Stored Products Research, 2019, 84: 101513.
[3] Aulicky R, Stejskal V, Frydova B. Field validation of phosphine efficacy on the first recorded resistant strains ofandfrom the Czech Republic. Journal of Stored Products Research, 2019, 81: 107-113.
[4] Nayak M K, Holloway J C, Emery R N, Pavic H, Bartlet J, Collins P J. Strong resistance to phosphine in the rusty grain beetle,(Stephens) (Coleoptera: Laemophloeidae):its characterisation, a rapid assay for diagnosis and its distribution in Australia. Pest Management Science, 2013, 69(1): 48-53.
[5] Agrafioti P, Athanassiou C G, Nayak M K. Detection of phosphine resistance in major stored-product insects in Greece and evaluation of a field resistance test kit. Journal of Stored Products Research, 2019, 82: 40-47.
[6] Nayak M K, Daglish G J, Phillips T W, Ebert P R. Resistance to the fumigant phosphine and its management in insect pests of stored products: A global perspective. Annual Review of Entomology, 2020, 65: 333-350.
[7] Alzahrani S, Ebert P R. Oxygen and arsenite synergize phosphine toxicity by distinct mechanisms. Toxicological Sciences, 2019, 167(2): 419-425.
[8] Zuryn S, Kuang J, Ebert P. Mitochondrial modulation of phosphine toxicity and resistance in. Toxicological Sciences, 2008, 102(1): 179-186.
[9] Pimentel M A G, Faroni L R D, Tótola M R, Guedes R N C. Phosphine resistance, respiration rate and fitness consequences in stored-product insects. Pest Management Science, 2007, 63(9): 876-881.
[10] Opit G P, Phillips T W, Aikins M J, Hasan M M. Phosphine resistance inandfrom stored wheat in Oklahoma. Journal of Economic Entomology, 2012, 105(4): 1107-1114.
[11] Schlipalius D I, Valmas N, Tuck A G, Jagadeesan R, Ma L, Kaur R, Goldinger A, Anderson C, Kuang J, Zuryn S,. A core metabolic enzyme mediates resistance to phosphine gas. Science, 2012, 338(6108): 807-810.
[12] Schlipalius D I, Tuck A G, Pavic H, Daglish G, Nayak M K, Ebert P R. A high-throughput system used to determine frequency and distribution of phosphine resistance across large geographical regions. Pest Management Science, 2019, 75(4): 1091-1098.
[13] Huang Y, Li F F, Liu M W, Wang Y Z, Shen F, Tang P A. Susceptibility ofto phosphine in China and functions of cytochrome P450s in phosphine resistance. Journal of Pest Science, 2019, 92: 1239-1248.
[14] Yang J O, Park J S, Lee H S, Kwon M, Kim G H, Kim J. Identification of a phosphine resistance mechanism inbased on transcriptome analysis. Journal of Asia-Pacific Entomology, 2018, 21(4): 1450-1456.
[15] Girotti J R, Mijailovsky S J, Patricia Juárez M. Epicuticular hydrocarbons of the sugarcane borer(Lepidoptera: Crambidae). Physiological Entomology, 2012, 37(3): 266-277.
[16] Moussian B. Recent advances in understanding mechanisms of insect cuticle differentiation. Insect Biochemistry and Molecular Biology, 2010, 40(5): 363-375.
[17] Balabanidou V, Grigoraki L, Vontas J. Insect cuticle: a critical determinant of insecticide resistance. Current Opinion in Insect Science, 2018, 27: 68-74.
[18] Chen N, Pei X J, Li S, Fan Y L, Liu T X. Involvement of integument-rich CYP4G19 in hydrocarbon biosynthesis and cuticular penetration resistance in(L.). Pest Management Science, 2020, 76(1): 215-226.
[19] Dang K, Doggett S L, Singham G V, Lee C Y. Insecticide resistance and resistance mechanisms in bed bugs,spp. (Hemiptera: Cimicidae). Parasites & Vectors, 2017, 10(1): 318.
[20] Yang C H, Yang P C, Zhang S F, Shi Z Y, Kang L, Zhang A B. Identification, expression pattern, and feature analysis of cuticular protein genes in the pine moth(Lepidoptera: Lasiocampidae). Insect Biochemistry and Molecular Biology, 2017, 83: 94-106.
[21] 刘晓健, 刘卫敏, 赵小明, 张建珍, 马恩波. 昆虫表皮发育研究进展及展望. 应用昆虫学报, 2019, 56(4): 625-638.
LIU X J, LIU W M, ZHAO X M, ZHANG J Z, MA E B. Progress in the study of insect cuticle development and prospects for future research. Chinese Journal of Applied Entomology, 2019, 56(4): 625-638. (in Chinese)
[22] Qiao L, Xiong G, Wang R X, He S Z, Chen J, Tong X L, Hu H, Li C L, Gai T T, Xin Y Q, Liu X F, Chen B, Xiang Z H, Lu C, Dai F Y. Mutation of a cuticular protein, BmorCPR2, alters larval body shape and adaptability in silkworm,. Genetics, 2014, 196(4): 1103-1115.
[23] Asano T, Taoka M, Shinkawa T, YAMAUCHI Y, ISOBE T, SATO D. Identification of a cuticle protein with unique repeated motifs in the silkworm,. Insect Biochemistry and Molecular Biology, 2013, 43(4): 344-351.
[24] 陈二虎, 孟宏杰, 陈艳, 唐培安. 表皮蛋白基因和参与介导赤拟谷盗对磷化氢的抗性形成. 中国农业科学, 2022, 55(11): 2150-2160. doi: 10.3864/j.issn.0578-1752.2022.11. 006.
CHEN E H, MENG H J, CHEN Y, TANG P A. Cuticle protein genesandare involved in phosphine resistance of. Scientia Agricultura Sinica, 2022, 55(11): 2150-2160. doi: 10.3864/j.issn.0578-1752.2022.11.006. (in Chinese)
[25] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729.
[26] Tang P A, Duan J Y, Wu H J, Ju X R, Yuan M L. Reference gene selection to determine differences in mitochondrial gene expressions in phosphine-susceptible and phosphine-resistant strains of, using qRT-PCR. Scientific Reports, 2017, 7: 7047.
[27] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCTmethod. Methods, 2001, 25(4): 402-408.
[28] Kaur R, Subbarayalu M, Jagadeesan R, Daglish G J, Nayak M K, Naik H R, Ramasamy S, Subramanian C, Ebert P R, Schlipalius D I. Phosphine resistance in India is characterised by a dihydrolipoamide dehydrogenase variant that is otherwise unobserved in eukaryotes. Heredity, 2015, 115(3): 188-194.
[29] KONEMANN C E, HUBHACHEN Z, Opit G P, GAUTAM S, BAJRACHARYA N S. Phosphine resistance in(Coleoptera: Laemophloeidae) collected from grain storage facilities in Oklahoma, USA. Journal of Economic Entomology, 2017, 110(3): 1377-1383.
[30] Price N R. Active exclusion of phosphine as a mechanism of resistance in(F.) (Coleoptera: Bostrychidae). Journal of Stored Products Research, 1984, 20(3): 163-168.
[31] Oppert B, Guedes R N C, Aikins M J, Perkin L, Chen Z, Phillips T W, Zhu K Y, Opit G P, Hoon K, Sun Y,. Genes related to mitochondrial functions are differentially expressed in phosphine-resistant and -susceptible. BMC Genomics, 2015, 16: 968.
[32] Tang L, Liang J, Zhan Z, Xiang Z, He N. Identification of the chitin-binding proteins from the larval proteins of silkworm,. Insect Biochemistry and Molecular Biology, 2010, 40(3): 228-234.
[33] 叶长青, 包涵, 刘田, 杨青. 双叉犀金龟表皮蛋白TdCPR12611与TdCPR7854的表达纯化及特性分析. 昆虫学报, 2021, 64(1): 19-29.
YE C Q, BAO H, LIU T, YANG Q. Expression, purification and characterization of the cuticular proteins TdCPR12611 and TdCPR7854 from(Coleoptera: Scarabaeidae). Acta Entomologica Sinica, 2021, 64(1): 19-29. (in Chinese)
[34] Vannini L, Willis J H. Localization of RR-1 and RR-2 cuticular proteins within the cuticle of. Arthropod Structure and Development, 2017, 46(1): 13-29.
[35] Shahin R, Iwanaga M, Kawasaki H. Cuticular protein and transcription factor genes expressed during prepupal-pupal transition and by ecdysone pulse treatment in wing discs of. Insect Molecular Biology, 2016, 25(2): 138-152.
[36] 丛林, 刘浩强, 李鸿筠, 巴音克西克, 冉春. 褐色橘蚜RR-2型表皮蛋白基因鉴定及功能分析. 植物保护学报, 2020, 47(5): 1078-1087.
CONG L, LIU H Q, LI H J, Bayinkexike, ran c. Identification and function analysis of RR-2 CPR genes in brown citrus aphid(Kirkaldy). Journal of Plant Protection, 2020, 47(5): 1078-1087. (in Chinese)
[37] Liang J, Wang T, Xiang Z, He N. Tweedle cuticular protein BmCPT1 is involved in innate immunity by participating in recognition of. Insect Biochemistry and Molecular Biology, 2015, 58: 76-88.
[38] Silva A X, Bacigalupe L D, Luna-Rudloff M, Figueroa C C. Insecticide resistance mechanisms in the green peach aphid(Hemiptera: Aphididae) II: Costs and benefits. Plos One, 2012, 7(6): e36810.
[39] Zhou D, Duan B, Sun Y, Ma L, Zhu C, Shen B. Preliminary characterization of putative structural cuticular proteins in the malaria vector. Pest Management Science, 2017, 73(12): 2519-2528.
[40] Sun X, Guo J, Ye W, Guo Q, Huang Y, Ma L, Zhou D, Shen B, Sun Y, Zhu C. Cuticle genesandmay confer resistance to deltamethrin in. Parasitology Research, 2017, 116(8): 2175-2179.
[41] Huang Y, Guo Q, Sun X H, Zhang C, Xu N, Xu Y, Zhou D, Sun Y, Ma L, Zhu C L, Shen B.cuticular protein CPLCG5 participates in pyrethroid resistance by forming a rigid matrix. Parasites & Vectors, 2018, 11: 6.
[42] Yahouédo G a, Chandre F, Rossignol M, Ginibre c, Balabanidou V, Mendez N G A, Pigeon O, Vontas J, Cornelie S. Contributions of cuticle permeability and enzyme detoxification to pyrethroid resistance in the major malaria vector. Scientific Reports, 2017, 7: 11091.
[43] Koganemaru R, Miller D M, Adelman Z N. Robust cuticular penetration resistance in the common bed bug (L.) correlates with increased steady-state transcript levels of CPR-type cuticle protein genes. Pesticide Biochemistry and Physiology, 2013, 106(3): 190-197.
[44] 张万娜, 刘香亚, 赖乾, 肖海军. 棉铃虫表皮蛋白基因和的表达特征及其对甲氧虫酰肼的响应. 植物保护学报, 2021, 48(5): 1043-1053.
Zhang W n, Liu X y, Lai Q, Xiao H j. Expression analysis of cuticular protein genesandand their response to the sublethal dose of methoxyfenozide. Journal of Plant Protection, 2021, 48(5): 1043-1053. (in Chinese)
[45] Chen E H, Hou Q L, Dou W, Wei D D, Yue Y, Yang R L, Yang P J, Yu S F, De Schutter K, Smagghe G, Wang J J. Genome-wide annotation of cuticular proteins in the oriental fruit fly (), changes during pupariation and expression analysis of CPAP3 protein genes in response to environmental stresses. Insect Biochemistry Molecular Biology, 2018, 97: 53-70.
Cuticle Protein Genes are Involved in Phosphine Resistance of

College of Food Science and Engineering/Collaborative Innovation Center for Modern Grain Circulation and Safety of Jiangsu Province/Key Laboratory of Grains and Oils Quality Control and Processing of Jiangsu Province, Nanjing University of Finance and Economics, Nanjing 210023
【Objective】As an important structural component of insect cuticle, the cuticle protein (CP) plays an important role in the formation of cuticle penetration resistance to pesticides. The phosphine resistance ofis increasingly prominent, and the current study was conducted to reveal the roles of CP genes in the formation of phosphine resistance in.【Method】According to the phosphine bioassay method that recommended by the Food and Agriculture Organization of the United Nations (FAO), the difference in phosphine sensitivity from five geographical populations (Zhangjiagang, Xiangyin, Huaian, Huaihua and Taicang populations) ofwas analyzed. The four CP genes were identified from the previous transcriptome data of,and then the phylogenetic tree of CPs was constructed and the corresponding amino acid sequence ofCPs was further analyzed. Afterwards, the RT-qPCR was used to analyze the spatio-temporal (different developmental stages and different tissues of adults) expression patterns of four CP genes, and their expression levels under different phosphine resistance levels, as well as the expression patterns of four CP genes in response to phosphine stress were explored. Subsequently, a specific CP gene () was selected to be knocked down by using RNAi (RNA interference) technology, and the change of phosphine sensitivity ofwas determined.【Result】The results of phosphine sensitivity bioassay analysis showed that there were significant differences in phosphine resistance levels of different geographical populations, and the range of insecticide resistance ratio (RR) was 7.2-1 906.8. The further sequence analysis suggested that the four CPs all contained chitin binding domain, which belonged to the RR2 subfamily of CPR family, and they were named as,,and,respectively. The gene expression patterns demonstrated that four CP genes were specifically highly expressed in the pupal stage of, and the high expression levels of four CP genes were detected in the peripheral tissues ofas well. Besides, the CP genes were highly expressed in the phosphine resistant population (Taicang population, RR=1 906.8), and their expression levels could be significantly induced by phosphine in. Lastly, a CP genewas selected for the further functional study. After the gene expression level ofwas significantly knocked down in phosphine resistance (TC) population ofvia the injection of dsRNA, the sensitivity ofto phosphine was significantly increased.【Conclusion】The over-expression of CP gene is involved in the formation of phosphine resistance.
; phosphine resistance; cuticle protein; RNA interference (RNAi)
2023-01-20;
2023-02-24
国家重点研发计划(2021YFD2100604)、江苏省重点研发计划(BE2022377)、国家自然科学基金(32001915,32272388)、江苏高校优势学科建设工程资助项目(YXK2103)、江苏省研究生科研与实践创新计划(KYCX22_1714)
陈二虎,E-mail:erhuchen1104@163.com。通信作者唐培安,E-mail:tangpeian@163.com
10.3864/j.issn.0578-1752.2023.09.007
(责任编辑 岳梅)
