过量表达小麦TaCYP78A5增加花器官的大小
2023-05-12彭海霞卡得艳张天星周梦蝶吴林楠辛转霞赵惠贤马猛
彭海霞,卡得艳,张天星,周梦蝶,吴林楠,辛转霞,赵惠贤,马猛
过量表达小麦增加花器官的大小

1西北农林科技大学风景园林艺术学院,陕西杨凌 712100;2西北农林科技大学生命科学学院,陕西杨凌 712100;3西北农林科技大学农学院,陕西杨凌 712100
【目的】利用表达模式分析、转基因过表达和细胞学观察等策略,解析调控花器官大小的功能和机制,为作物遗传改良提供基因资源和理论基础。【方法】根据EnsemblPlants基因组数据库中不同物种CYP78A家族成员的序列信息,对小麦和其他物种中的同源基因进行序列比对和进化分析;利用生物信息学分析小麦的基因和蛋白结构,以及不同器官的表达模式;通过在拟南芥中组成型过表达和生殖器官局部特异性过表达的策略,明确具有调控花器官大小的功能;利用显微镜观察不同转基因拟南芥花器官的细胞学特征,解析调控花器官大小的细胞学机制;利用小麦转基因过表达策略,明确调控小麦穗部大小等其他穗部性状的功能;利用323份小麦品种的单倍型数据与穗部表型数据进行关联分析,探析不同小麦品种表达量的高低对穗部大小等其他穗部性状的影响。【结果】小麦与拟南芥的基因和蛋白序列相似性较低,但基因和蛋白结构相似性较高。小麦和拟南芥均在多个器官广泛表达,但在花器官中表达量最高。相较于野生型,在拟南芥中组成型过表达能导致花器官增大,花瓣面积显著增加13.5%—35.4%;而且仅在胚珠局部特异性过表达也足以导致拟南芥花器官增大,花瓣面积显著增加9%—22.1%;相反,拟南芥突变体的花器官则显著小于野生型,花瓣面积显著减少27%。在拟南芥中组成型过表达导致花瓣表皮细胞大小较野生型显著增加49%—54%,细胞数目较野生型显著减少11%—19%,局部特异性过表达也导致花瓣表皮细胞大小较野生型显著增加20%—49%,细胞数目较野生型显著减少8%—24%。在小麦中组成型过表达导致小麦穗长增加7.9%—8.9%,颖壳面积增大9.6%—14.7%,穗粒数增加12.4%—23.8%,小穗数和小穗粒数呈现不同程度的变化。单倍型分析结果显示,在323份小麦品种中,具有较高表达量的小麦品种比具有较低表达量的小麦品种的穗长更长,小穗粒数更多,小穗数更少,而穗粒数无显著差异。【结论】以一种非细胞自制的模式促进花器官生长,小麦和拟南芥过量表达该基因均能导致花器官的增大。
小麦;;过量表达;花器官大小;细胞扩张
0 引言
【研究意义】花器官的大小是植物的一个关键形态特征,影响植物交配系统进化和繁殖[1]。在作物中,花器官直接产生了人类所需的大部分食物,其大小深刻影响着作物的经济效应[2]。因此,研究花器官大小相关基因和解析其调控机制具有重要理论意义和实践价值。【前人研究进展】植物器官的发育基于2个不同的过程:细胞的增殖和扩张。它们分别通过影响细胞数量和大小来调控植物器官的形态建成和大小[3]。其中,植物对花器官大小的调控主要通过对细胞增殖或扩张的持续时间和比率来实现[4]。目前,研究发现影响花器官大小的基因主要涉及泛素途径、植物激素信号和转录调控等途径[5]。其中,涉及泛素途径的基因大多负调控花器官的大小。如E3泛素连接酶BIG BROTHER(BB)、泛素受体DA1、E3泛素连接酶DA2等,均通过限制细胞增殖来调节花器官的大小[6-8]。植物激素信号广泛参与多种器官(包括花器官在内)大小的调控。如拟南芥花瓣()的表达受茉莉酸信号的调节,通过限制有丝分裂后细胞的扩张,调节花瓣的生长,而且该基因还与生长素响应基因()互作,共同调节花瓣的发育和大小[9-11]。此外,在拟南芥花发育过程中,ABCE四类基因的不同组合能够协同调控萼片(A+E)、花瓣(A+B+E)、雄蕊(B+C+E)和心皮(C+E)的发育。其中,A类基因()编码AP2/ERF转录因子,E类基因()编码MADS结构域转录因子,其在整个花器官发育中均有表达,协同调控花器官发育的不同过程[12-13]。细胞色素P450家族是植物中最大的基因家族之一,广泛参与植物各项生理活动[14]。其中,CYP78A亚家族成员具有调节多种器官发育的功能。如,拟南芥CYP78A亚家族包含6个成员,其中,功能的丧失导致叶面积、角果长度和种子大小降低[15-18]。和同源性较高,双缺变体表现出胚发育和叶片起始异常[19-20]。、和同源性较高,和双缺突变体均表现为外珠被发育停滞,进而导致种子大小和育性降低[21-22]。其中,和均通过控制母性表皮的细胞增殖影响种子的大小[22-24]。水稻CYP78A亚家族包含8个成员,其中,()主要通过控制分生组织的活性影响叶片的起始和穗分枝的形成[25-26]。()则通过控制胚乳细胞的凋亡调节种子的大小[27-29]。小麦CYP78A亚家族包含4个成员,通过调控种皮细胞增殖影响籽粒的大小[30]。过表达能促进种皮细胞增殖,进而导致籽粒增大。而且,仅在母性表皮中特异过表达,就足以导致籽粒增大、单株产量增加[31]。另一方面,CYP78A家族基因调控器官发育可能涉及激素代谢。如,水稻可能参与脂肪酸和生长素的合成代谢途径[25, 32];拟南芥和小麦在调节器官生长及衰老过程中均检测到生长素和细胞分裂素含量的变化[17, 31, 33]。因此,推测CYP78A亚家族成员依赖下游一个可移动的激素类促生因子,促进其表达部位附近器官的生长[17, 34-35]。【本研究切入点】小麦是重要的粮食作物之一。麦穗是一种特殊的花或果实器官,其大小直接影响小麦的产量。赵惠贤课题组前期研究发现,尽管小麦和拟南芥具有相似的调控植物株型和种子大小的功能,但小麦仅能部分回补拟南芥的功能缺失表型。而且小麦和拟南芥在调控叶片大小、叶起始速率等方面存在显著的差异,表明具有一定的保守性,但在不同物种间的功能存在分化[17, 36]。此外,小麦是否具有调控花器官的功能仍然未知,在调控花器官发育方面是否仍然具有物种间的保守和分化也有待揭示。【拟解决的关键问题】本研究通过比较分析小麦和拟南芥的结构和表达模式,解析在物种间功能保守和分化的潜在基础;进一步系统调查转基因植株花器官的表型,明确小麦和拟南芥具有正调控花器官大小的功能,及其调控机制的差异,为进一步揭示调控植物器官大小的功能和机制奠定基础,为作物遗传改良提供基因资源。
1 材料与方法
1.1 试验材料
拟南芥材料均以哥伦比亚野生型(Columbia)为遗传背景。拟南芥功能缺失突变体购自Arabidopsis Biological Resources(突变体编号:Salk_024697C)。T2组成型启动子和胚珠特异型启动子(promoter of)驱动的过表达拟南芥(::和::)由赵惠贤实验室前期构建,其中,::3个株系(::、::-3和::-6)和::5个株系(::、::、::、::和::)均为纯合株系。转基因材料的构建和鉴定详见前期研究[17, 36]。上述材料均置于温室(昼16 h/夜8 h,70%相对湿度,130—170 μmol·m-2·s-2光照强度)培养。
小麦材料均以春麦JW1为遗传背景。JW1为济麦22和fielder杂交选育的新品系。T3组成型启动子驱动的过表达转基因小麦(::)由赵惠贤实验室前期构建,其中,::2个株系(::和::)均为纯合株系。转基因材料的构建和鉴定详见前期研究[31]。上述材料均置于温室(昼16 h/夜8 h,30%—60%相对湿度,300—500 μmol·m-2·s-2光照强度)培养。
1.2 CYP78A5的生物信息学分析
从EnsemblPlants(https://plants.ensembl.org/)查询下载不同物种CYP78A家族成员的序列。利用DNAMAN(https://www.lynnon.com)软件分析序列相似性。利用InterProScan(https://www.ebi.ac.uk/interpro/ search/sequence/)在线软件分析蛋白结构,利用TMpred(https://sbcb.bioch.ox.ac.uk/TM_noj/TM_noj. html)和PROSITE(https://web.expasy.org/docs/swiss- prot_guideline.html)软件分析蛋白保守结构域,使用SwissModel(https://swissmodel.expasy.org/)软件预测蛋白三维结构。利用CLUSTAL W(https://www.ebi. ac.uk/Tools/msa/clustalw2/)软件进行系统进化树分析。各器官的表达模式数据来源于EnsemblPlants(https:// plants.ensembl.org/)。
1.3 转基因阳性植株的鉴定
转基因阳性拟南芥植株的鉴定:用200 mg·L-1Basta溶液喷施转基因拟南芥(长出2片真叶),隔一天喷施一次(共3次),叶片仍为绿色的植株为转基因阳性拟南芥,可用于后续表型观察。
转基因小麦纯合株系的鉴定:在小麦3叶期,用200 mg·L-1Basta溶液涂抹小麦第一片叶,7 d后,叶片仍为绿色的植株为阳性转基因小麦,可用于后续表型观察。
1.4 花器官的形态学观察
将野生型拟南芥和筛选获得的阳性转基因过表达拟南芥种植于温室,待拟南芥开花后,对花器官形态进行动态观察,记录花器官的整体或花瓣局部形态。采集主茎上完全开放的花朵,拍照,利用Image J(National Institutes of Health)软件测量花瓣面积,每个株系至少检测6个单株,每个单株不少于3朵花。
将野生型小麦以及筛选获得的阳性::小麦种植于温室中,待植株抽穗并扬花12 d后,对其穗部进行形态学观察,记录穗部的整体或外颖壳局部形态。利用Image J软件测量颖壳投影面积,每个株系至少采样检测6个单株,每个单株不少于4个颖壳。
1.5 拟南芥花瓣的细胞学观察
选取温室下长势健康的野生型、突变体以及::和::转基因拟南芥植株,待植株开花后,收集其主茎上相同位置且完全开放花朵的花瓣样品,4℃卡诺固定液(Carnoy Fix Soution,Coolaber,北京)浸泡20 min,然后使用梯度酒精(30%、50%、75%、95%和100%)浸泡30 min,至完全脱色。光学显微镜观察并采集花瓣中部图像,利用Image J软件测定细胞面积,并计算表皮的细胞总数目(表皮的细胞总数目=花瓣面积/细胞面积)。每个株系至少测量6个单株,每个单株不少于3朵花(12个花瓣),每个花瓣中部选取不少于10个细胞用于上述细胞学观察。
1.6 数据统计
用Excel 2016进行数据处理。采用-Test算法进行数据间差异显著性分析。
2 结果
2.1 CYP78A5在花器官高表达且在物种间基因结构保守
小麦基因组包含3个拟南芥的同源基因,分别为(TraesCS2A01G175700.1)、(TraesCS2B01G201900.1)和(TraesCS2D01G183000.1),其核苷酸序列相似性极高,大于98%(图1-A和图2-A)。进化分析显示,CYP78A亚家族成员广泛存在于陆生植物,其中,小麦与水稻和拟南芥序列相似性较高(图1-A—B)。
为了解析小麦与拟南芥在调控多种器官发育方面表现出相似功能的原因,进一步分析小麦与拟南芥的基因和蛋白质结构。结果显示,小麦与拟南芥的编码区均由2个外显子和1个内含子组成。和同编码区的差异主要体现在第二个外显子和内含子存在不同程度的缺失(图1-B)。小麦与拟南芥的编码蛋白序列相似性较低(约55%),但都包含N端的疏水区域和C端的氧结合结构域及血红素结合结构域(图1-B和图2-A)。蛋白质三维结构预测结果显示,小麦与拟南芥的蛋白质空间结构相似性较高(图2-B)。
进一步对小麦与拟南芥在器官间的表达情况进行分析。结果显示,小麦与拟南芥在各器官中均广泛表达,其中,小麦与拟南芥在各器官中的表达模式极为相近,尤其是在早期的花和幼穗中表现出极为相似的高表达水平(图1-C)。以上结果表明,小麦与拟南芥的基因和蛋白序列相似性较低,但蛋白质结构和表达模式相似性较高,可能是小麦与拟南芥在调控多种器官发育方面表现出功能保守和分化的潜在基础。
2.2 过量表达TaCYP78A5导致拟南芥花器官增大
鉴于小麦与拟南芥均在发育初期的花器官中高表达,可能均参与调控花器官的大小。为了进一步验证这个推测,通过调查前期构建的::转基因拟南芥株系花器官的特征,发现与野生型相比,组成型过表达(::)能够导致花器官的显著增大。与之相对应的是,拟南芥突变体植株却表现出明显的花器官减小(图3-A—H)。
为进一步验证对花器官的调控是否以非细胞自制方式促进其表达部位周围器官的生长,通过对前期构建的过表达转基因株系::花器官的表型观察。发现仅在胚珠中特异性过量表达就足以导致花器官显著增大(图3-B和图3-F)。

A:TaCYP78A5(由TaCYP78A5-A推导的蛋白序列)和其他CYP78A家族成员的系统进化树分析;B:TaCYP78A5-A、TaCYP78A5-B和TaCYP78A5-D的外显子和内含子构造,黑色表示蛋白编码区,裂开的地方表示缺失区域,黄色、蓝色和红色分别表示编码疏水区、氧结合位点和亚铁血红素结合位点的序列;C:拟南芥AtCYP78A5和小麦TaCYP78A5的器官表达模式
进一步对不同基因型植株的花瓣面积进行测定,结果显示,与野生型相比,::和::植株的花瓣面积分别显著增大13.5%—35.4%和9%—22.1%,而突变体的花瓣面积则显著缩小27%(图3-I—J)。
综上,的表达能够以一种非细胞自制的模式促进花器官的增大。
2.3 过量表达TaCYP78A5促进拟南芥花瓣细胞的扩张
为了进一步解析调控花器官大小的细胞学机制,分别调查了上述不同基因型拟南芥花瓣的表皮细胞特征。结果显示,与野生型相比,随着花瓣面积的增加,组成型过表达(::)导致表皮细胞大小显著增加49%—54%,而其细胞数目较野生型却显著减少11%—19%(图4-A—H)。相似的,局部特异性过表达(::)也导致花瓣表皮细胞大小和花瓣面积同步增加(增加20%—49%),花瓣表皮细胞数目呈现相反的趋势(减少8%—24%)(图4-A—I)。与之相反,与野生型相比,突变体植株的花瓣细胞面积显著增加19.9%,细胞数目却减少37.8%(图4-H—I)。以上结果表明,的过量表达导致的花器官增大主要归因于细胞的扩张。
2.4 过量表达TaCYP78A5导致小麦穗部增大
为了明确在小麦和拟南芥中对花器官的调控作用是否保守,即在小麦中是否也能够正向调控花器官大小,对前期构建的过表达转基因小麦(::)的穗部特征进行了研究。结果显示,与野生型相比,小麦组成型过表达导致小麦穗长显著增加7.9%—8.9%(图5-A和图5-E)。通过详细调查小麦扬花后12 d的穗部表型,发现在小麦中的过量表达导致小穗和颖壳同步增大,其中,颖壳面积(由外颖壳最大投影面积表示)显著增大9.6%—14.7%(图5-B和图5-I)。增大的穗部可为种子的生长提供更大的物理空间[2],这与过表达导致种子增大的表型相符(图5-B和图5-D)。此外,组成型过表达导致小穗数呈现不同程度的下降(图5-G),而小穗粒数呈现不同程度的上升,但差异不显著(图5-H)。最终伴随着穗部的增大,过量表达转基因植株的穗粒数显著高于野生型,增加12.4%—23.8%。可见,与在拟南芥中对花器官大小的调控作用相似,在小麦中也能够调控穗部的大小和其他穗部性状。
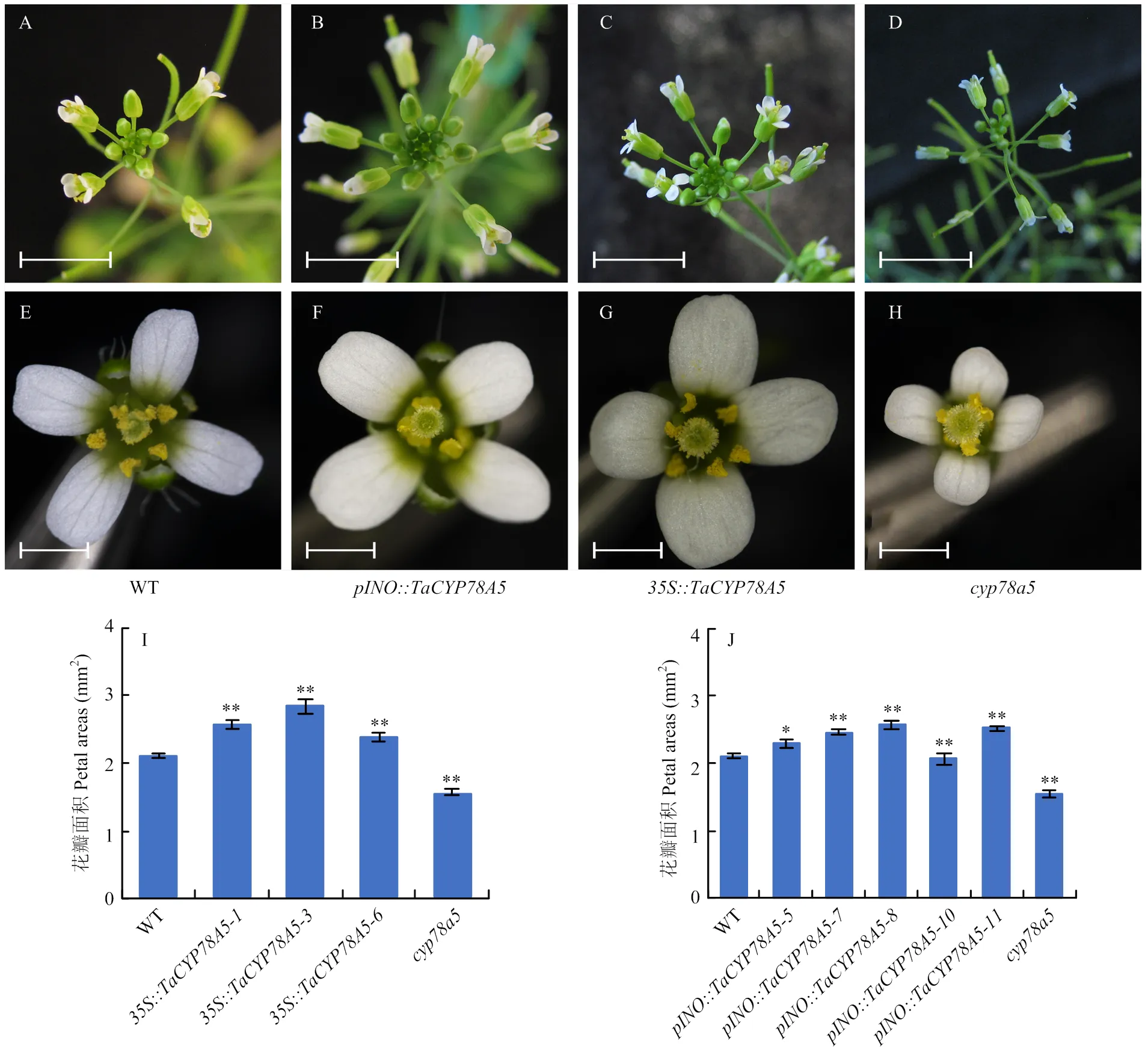
A—H:植株花器官的形态;I—J:植株花瓣面积的统计(n>36),以花瓣最大投影面积作为花瓣面积;*和**分别表示在P<0.05和P<0.01水平时的差异显著。下同。A—D的标尺为0.5 cm;E—H的标尺为500 μm
2.5 不同小麦品种TaCYP78A5的表达对小麦穗部增大的影响
为了进一步明确不同小麦品种中的表达量是否会影响穗部性状,通过对具有较高表达量的小麦品种(简称小麦)和较低表达量的小麦品种(简称小麦)穗部性状进行调查。结果显示,小麦(共计265个小麦品种)的穗长显著高于小麦(共计58个小麦品种),穗长增加约5%(图6-A—B)。此外,与转基因植株的表型一致(图5),拥有较高表达量的小麦小穗数显著低于小麦,而小穗粒数显著高于小麦(图6-C—D)。最终导致在籽粒增大的同时,穗粒数不变(图6-E)。进一步证明表达量的高低影响穗部大小和穗部其他性状。

A:植株花瓣的形态;B—E:植株花瓣中部表皮细胞的形态;F—G:植株花瓣中部表皮细胞面积的统计(n>50);H—I:植株花瓣表皮细胞数目的统计(n>50)。A的标尺为500 μm;B—E的标尺为50 μm
3 讨论
3.1 不同物种间CYP78A5母性调控花器官大小功能的保守和分化
花是种子植物所特有的繁殖器官,是植物从营养生长转向生殖生长的重要阶段。在植物特别是异花授粉的植物中,花器官的大小影响了植物繁殖的成功率[37]。大花通常含有更多的花蜜,而且比小花更显眼,通常能增加传粉者的访问频率。因此,无论是在植物物种内部还是在物种之间,传粉者被大花吸引的行为可以施加强烈的定向选择,使异交植物倾向于产生更大的花[38]。因此,在群体遗传学中如果把能否成功繁殖作为适合度的组成部分,人们发现植物对较大花朵的定向选择在不同物种间普遍存在[39]。尽管小麦、水稻等主要农作物是自花授粉,花器官的大小不再是繁殖成功率的决定因素。但是花器官的大小也影响着最终果实或种子的大小。如子房是花器官的主要组成部分,在多个麦类作物中观测到子房大小与最终籽粒大小之间存在强烈的正相关性[40-42]。因此,前期赵惠贤课题组仅在小麦子房中特异性过表达就足以导致子房和籽粒的同步增大[31]。而且,无论是自花授粉还是异花授粉植物,扬花前母性生殖器官的发育状况都会对扬花后子代果实或者种子的发育产生巨大的影响,而这种母性控制被认为在被子植物中广泛存在[2, 43]。

A—C:扬花后12 d的穗部、颖壳和小穗表型;D:扬花后30 d的籽粒表型;E—I:扬花后12 d的穗长、颖壳面积、小穗数、小穗粒数和穗粒数的统计(n>8),所有植株均在温室中采用相同条件种植。A—D的标尺为1 cm
被认为是一种潜在的母性促生因子,主要通过促进器官母性表皮细胞增殖或扩张来调控器官的大小[17-18, 31, 23]。小麦和水稻是拟南芥的直系同源基因,它们具有保守的结构域(图2)[44],且都可以部分恢复拟南芥的突变表型[28]。在拟南芥和小麦中,均在籽粒发育阶段的母性表皮中高表达;的过量表达能够促进母性表皮细胞的增殖,引起珠被和种皮的协同增大,并最终导致种子增大[16, 31]。番茄的功能缺失会导致果皮的细胞数目降低,果实变小[45]。过量表达小麦能够通过延长母性表皮细胞增殖的时间来促进叶片的增大[17]。本研究发现在小麦或拟南芥中过量表达均能导致花器官和穗部的增大(图3和图5)。其中,过量表达导致拟南芥花瓣表皮细胞大小也较野生型显著增加49%—54%,细胞数目较野生型显著减少11%—19%。利用胚珠特异性启动子局部特异性过表达[46],也导致拟南芥花瓣表皮细胞大小增加20%—49%,花瓣表皮细胞数目减少8%—24%,表明小麦的过量表达导致花器官增大主要归因于母性表皮细胞的扩张。与之相反,拟南芥突变体植株的花瓣细胞面积显著增加19.9%,细胞数目却减少37.8%(图4),表明拟南芥功能缺失导致的花器官变小主要归因于母性表皮细胞增殖的降低。同样,拟南芥和小麦均能正调控叶片大小,而在调控叶片起始率和叶片数目上功能存在差异[17]。上述研究结果表明,不同物种间在调控器官大小方面功能保守,但在调控母性表皮细胞增殖或扩张的机制可能存在差异。

A:小麦Ap-HapⅠ和Ap-HapⅡ的穗部表型;B—E:小麦Ap-HapⅠ(n=265)和Ap-HapⅡ(n=58)穗长、小穗数、小穗粒数和穗粒数的统计。表型数据来源于323个小麦品种在5个环境点的平均值
穗部是母体植株为后代种子发育提供的载体,穗部的发育状况会直接影响着种子大小和数目[2]。与之相对应的,无论是在小麦或拟南芥中过量表达,都会导致花器官和种子的同步增大(图3和图5)[16, 18, 31]。花器官或穗部的增大能够给后期种子的发育提供更大的物理空间上限[2],而这很可能是过量表达导致花器官和种子同步增大的原因之一。另一方面,有限的资源使得母体植株会协调种子的大小和数目[47]。转基因表型和单倍型分析结果均表明,表达量的变化还会影响小穗数、穗粒数和小穗粒数等与种子数目直接相关的穗部性状(图5和图6)。总之,花器官大小的调控可能是母体植株协调种子大小和数目的手段之一,而不同物种间可能通过调控花器官的大小来影响种子大小、数目乃至产量。
3.2 TaCYP78A5调控器官大小可能涉及植物激素代谢途径
除环境因素以外,花器官的大小主要受内在遗传因素控制。其中生长素作为一种最基本的生长调节因子,广泛参与了包括花器官在内的多种器官发育的调控[4]。在拟南芥中组成型过表达生长素诱导基因()能够延长细胞分裂的时间,进而导致花器官增大;而功能缺失则会导致花器官变小[48]。生长素响应基因()的突变会产生更大的花瓣,而花瓣大小的增加是由细胞大小和细胞数量同时增加所致[49]。前人推测家族成员依赖于下游一个类似植物激素的促生因子,以非细胞自制方式促进其表达部位周围器官的生长[17, 34-35]。然而,关于是否涉及生长素代谢途径一直存在争议,因为外源生长素处理不能恢复突变体叶起始速率加快的表型[16]。并且突变体中不仅检测到生长素含量变化,也检测到细胞分裂素等其他植物激素的含量存在波动[33]。
然而,实验室前期利用代谢组学、激素含量测定和生长素响应标记基因::检测转基因小麦和拟南芥突变体,均发现生长素含量发生了明显的变化[31, 36]。相似的生长素累积现象在玉米过表达植株中也被检测到[25, 50]。本研究发现仅在胚珠中特异性过表达就足以导致整个花器官的增大和开花时间的延长,表明是以一种非细胞自制的模式调控花器官的发育和大小。而这种非细胞自制的作用模式是植物激素的典型特征[51]。此外,前人研究发现具有类似生长素维持植株顶端优势及延缓器官衰老的功能[20, 25, 36, 52]。小麦过量表达会加强植株的顶端优势[31];拟南芥功能缺失会导致顶端优势降低[16, 19]。小麦过表达会推迟开花时间[31];而本研究发现过量表达不仅能够增加拟南芥花器官的大小(图3),还能够延长开花时间和延缓衰老(未展示)。上述结果均表明,调控器官大小可能涉及植物激素代谢途径。然而,CYP78A基因家族成员作为一种细胞色素单加氧酶,尽管有证据表明其在脂肪酸等次生代谢物的合成中发挥着重要作用[16, 18],但是否以及如何参与植物激素代谢仍需要更强有力的证据来证明。
4 结论
小麦在幼穗中高表达,在拟南芥和小麦中过量表达均能导致花器官和穗部的显著增大,不同小麦品种中表达量的升高有利于穗长的增加,表明正调控花器官大小。
[1] Goodwillie C, Sargent R D, Eckert C G, Elle E, Geber M A, Johnston M O, Kalisz S, Moeller D A, Ree R H, Vallejo-Marin M, Winn A A. Correlated evolution of mating system and floral display traits in flowering plants and its implications for the distribution of mating system variation. The New Phytologist, 2010, 185(1): 311-321.
[2] MILLET E. Relationships between grain weight and the size of floret cavity in the wheat spike. Annals of Botany, 1986, 58(3): 417-423.
[3] Hepworth J, Lenhard M. Regulation of plant lateral-organ growth by modulating cell number and size. Current Opinion in Plant Biology, 2014, 17: 36-42.
[4] Powell A E, Lenhard M. Control of organ size in plants. Current Biology, 2012, 22(9): R360-R367.
[5] Krizek B A, Anderson J T. Control of flower size. Journal of Experimental Botany, 2013, 64(6): 1427-1437.
[6] Disch S, Anastasiou E, Sharma V K, Laux T, Fletcher J C, Lenhard M. The E3 ubiquitin ligase BIG BROTHER controls Arabidopsis organ size in a dosage-dependent manner. Current Biology, 2006, 16(3): 272-279.
[7] Li Y H, Zheng L Y, Corke F, Smith C, Bevan M W. Control of final seed and organ size by the DA1 gene family in. Genes & Development, 2008, 22(10): 1331-1336.
[8] Xia T, Li N, Dumenil J, Li J, Kamenski A, Bevan M W, Gao F, Li Y H. The ubiquitin receptor DA1 interacts with the E3 ubiquitin ligase DA2 to regulate seed and organ size in. The Plant Cell, 2013, 25(9): 3347-3359.
[9] SzÉcsi J, Joly C, Bordji K, Varaud E, Cock J M, Dumas C, Bendahmane M. BIGPETALp, a bHLH transcription factor is involved in the control ofpetal size. The Embo Journal, 2006, 25(16): 3912-3920.
[10] Brioudes F, Joly C, SzÉcsi J, Varaud E, Leroux J, Bellvert F, Bertrand C, Bendahmane M. Jasmonate controls late development stages of petal growth in. The Plant Journal, 2009, 60(6): 1070-1080.
[11] Li S B, Xie Z Z, Hu C G, Zhang J Z. A review of auxin response factors (ARFs) in plants. Frontiers in Plant Science, 2016, 7: 47.
[12] Ito T, Ng K H, Lim T S, Yu H, Meyerowitz E M. The homeotic protein AGAMOUS controls late stamen development by regulating a jasmonate biosynthetic gene in. The Plant Cell, 2007, 19(11): 3516-3529.
[13] Wuest S E, O'Maoileidigh D S, Rae L, Kwasniewska K, Raganelli A, Hanczaryk K, Lohan A J, Loftus B, Graciet E, Wellmer F. Molecular basis for the specification of floral organs by APETALA3 and PISTILLATA. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(33): 13452-13457.
[14] Doddapaneni H, Chakraborty R, Yadav J S. Genome- wide structural and evolutionary analysis of the P450 monooxygenase genes (P450ome) in the white rot fungus: Evidence for gene duplications and extensive gene clustering. Bmc Genomics, 2005, 6: 92.
[15] Zondlo S C, Irish V F. CYP78A5 encodes a cytochrome P450 that marks the shoot apical meristem boundary in. The Plant Journal, 1999, 19(3): 259-268.
[16] Anastasiou E, Kenz S, Gerstung M, MacLean D, Timmer J, Fleck C, Lenhard M. Control of plant organ size by KLUH/CYP78A5-dependent intercellular signaling. Developmental Cell, 2007, 13(6): 843-856.
[17] Zhou M D, Peng H X, Wu L N, Li M Y, Guo L J, Chen H C, Wu B W, Liu X L, Zhao H X, Li W Q, Ma M. TaKLU plays as a time regulator of leaf growth via auxin signaling. International Journal of Molecular Sciences, 2022, 23(8): 4219.
[18] Adamski N M, Anastasiou E, Eriksson S, O'Neill C M, Lenhard M. Local maternal control of seed size by KLUH/ CYP78A5-dependent growth signaling. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(47): 20115-20120.
[19] Wang J W, Schwab R, Czech B, Mica E, Weigel D. Dual effects of miR156-targeted SPL genes and CYP78A5/KLUH on plastochron length and organ size in. The Plant Cell, 2008, 20(5): 1231-1243.
[20] Poretska O, Yang S Q, Pitorre D, Poppenberger B, Sieberer T. AMP1 and CYP78A5/7 act through a common pathway to govern cell fate maintenance in. Plos Genetics, 2020, 16(9): e1009043.
[21] Sotelo-Silveira M, Cucinotta M, Chauvin A L, CHÁVEZ Montes R A C, Colombo L, Marsch-MartÍnez N, de Folter S. Cytochrome P450 CYP78A9 is involved inreproductive development. Plant Physiology, 2013, 162(2): 779-799.
[22] Ito T, Meyerowitz E M. Overexpression of a gene encoding a cytochrome p450, CYP78A9, induces large and seedless fruit in. The Plant Cell, 2000, 12(9): 1541-1550.
[23] Fang W J, Wang Z B, Cui R F, Li J, Li Y H. Maternal control of seed size by EOD3/CYP78A6 in. The Plant Journal, 2012, 70(6): 929-939.
[24] Sotelo-Silveira M, Cucinotta M, Colombo L, Marsch-Martínez N, de Folter S. Toward understanding the role of CYP78A9 duringreproduction. Plant Signaling & Behavior, 2013, 8(8): e25160.
[25] Miyoshi K, Ahn B O, Kawakatsu T, Ito Y, Itoh J I, Nagato Y, Kurata N. PLASTOCHRON1, a timekeeper of leaf initiation in rice, encodes cytochrome P450. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(3): 875-880.
[26] Feng G N, Zhang C Q, Zhao D S, Zhu K Z, Tu H Z, Xu C W, Liu Q Q. Fine MAPPING AND CLONING OF LEAFY HEAD MUTANT GENE pla1-5 in rice. Rice Science, 2013, 20(5): 329-335.
[27] Nagasawa N, Hibara K I, Heppard E P, Vander Velden K A, Luck S, Beatty M, Nagato Y, Sakai H. GIANT EMBRYO encodes CYP78A13, required for proper size balance between embryo and endosperm in rice. The Plant Journal, 2013, 75(4): 592-605.
[28] Xu F, Fang J, Ou S J, Gao S P, Zhang F X, Du L, Xiao Y H, Wang H R, Sun X H, Chu J F, Wang G D, Chu C C. Variations in CYP78A13 coding region influence grain size and yield in rice. Plant, Cell & Environment, 2015, 38(4): 800-811.
[29] Yang W B, Gao M J, Yin X, Liu J Y, Xu Y H, Zeng L J, Li Q, Zhang S B, Wang J M, Zhang X M, He Z H. Control of rice embryo development, shoot apical meristem maintenance, and grain yield by a novel cytochrome P450. Molecular Plant, 2013, 6(6): 1945-1960.
[30] Ma M, Wang Q, Li Z J, Cheng H H, Li Z J, Liu X L, Song W N, Appels R, Zhao H X. Expression of TaCYP78A3, a gene encoding cytochrome P450 CYP78A3 protein in wheat (L.), affects seed size. The Plant Journal, 2015, 83(2): 312-325.
[31] Guo L J, Ma M, Wu L N, Zhou M D, Li M Y, Wu B W, Li L, Liu X L, Jing R L, Chen W, Zhao H X. Modified expression of TaCYP78A5 enhances grain weight with yield potential by accumulating auxin in wheat (L.). Plant Biotechnology Journal, 2022, 20(1): 168-182.
[32] Sun X H, Cahill J, Van Hautegem T, Feys K, Whipple C, NovÁk O, Delbare S, Versteele C, Demuynck K, De Block J, Storme V, Claeys H, Van Lijsebettens M, Coussens G, Ljung K, De Vliegher A, Muszynski M, InzÉ D, Nelissen H. Altered expression of maize PLASTOCHRON1 enhances biomass and seed yield by extending cell division duration. Nature Communications, 2017, 8: 14752.
[33] Jiang L, Yoshida T, Stiegert S, Jing Y, Alseekh S, Lenhard M, Pérez-Alfocea F, Fernie A R. Multi-omics approach reveals the contribution of KLU to leaf longevity and drought tolerance. Plant Physiology, 2021, 185(2): 352-368.
[34] Eriksson S, Stransfeld L, Adamski N M, Breuninger H, Lenhard M. KLUH/CYP78A5-dependent growth signaling coordinates floral organ growth in. Current Biology, 2010, 20(6): 527-532.
[35] Nobusawa T, Kamei M, Ueda H, Matsushima N, Yamatani H, Kusaba M. Highly pleiotropic functions of CYP78As and AMP1 are regulated in non-cell-autonomous/organ- specific manners. Plant Physiology, 2021, 186(1): 767-781.
[36] 周梦蝶, 李梦瑶, 吴林楠, 郭利建, 王倩, 马猛. 小麦TaKLU基因调控植物株型的研究. 麦类作物学报, 2022, 42(4): 399-406.
Zhou M D, Li M Y, Wu L N, Guo L J, Wang Q, Ma M. Studies on regulation of plant architecture by TaKLU gene in wheat. Journal of Triticeae Crops, 2022, 42(4): 399-406. (in Chinese)
[37] Blarer A, Keasar T, Shmida A. Possible mechanisms for the formation of flower size preferences by foraging bumblebees. Ethology, 2002, 108(4): 341-351.
[38] Parachnowitsch A L, Kessler A. Pollinators exert natural selection on flower size and floral display in. New Phytologist, 2010, 188(2): 393-402.
[39] Kingsolver J G, Hoekstra H E, Hoekstra J M, Berrigan D, Vignieri S N, Hill C E, Hoang A, Gibert P, Beerli P. The strength of phenotypic selection in natural populations. The American Naturalist, 2001, 157(3): 245-261.
[40] Reale L, Rosati A, Tedeschini E, Ferri V, Cerri M, Ghitarrini S, Timorato V, Ayano B E, Porfiri O, Frenguelli G, Ferranti F, Benincasa P. Ovary size in wheat (L.) is related to cell number. Crop Science, 2017, 57(2): 914-925.
[41] Xie Q, Mayes S, Sparkes D L. Carpel size, grain filling, and morphology determine individual grain weight in wheat. Journal of Experimental Botany, 2015, 66(21): 6715-6730.
[42] Yang Z J, van Oosterom E J, Jordan D R, Hammer G L. Pre-anthesis ovary development determines genotypic differences in potential kernel weight in sorghum. Journal of Experimental Botany, 2009, 60(4): 1399-1408.
[43] Li N, Li Y H. Maternal control of seed size in plants. Journal of Experimental Botany, 2015, 66(4): 1087-1097.
[44] Chapple C. Molecular-genetic analysis of plant cytochrome P450- dependent monooxygenases. Annual Review of Plant Physiology and Plant Molecular Biology, 1998, 49: 311-343.
[45] Chakrabarti M, Zhang N, Sauvage C, MUÑOS S, BLANCA J, CAÑIZARES J, Diez M J, Schneider R, Mazourek M, McClead J, Causse, M, van der Knaap E. A cytochrome P450 regulates a domestication trait in cultivated tomato. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(42): 17125-17130.
[46] VillanUeva J M, Broadhvest J, Hauser B A, Meister R J, Schneitz K, Gasser C S. INNER NO OUTER regulates abaxial-adaxial patterning inovules. Genes & Development, 1999, 13(23): 3160-3169.
[47] LAWRENCE V D. Size-number trade-offs and the variation of seed size with plant resource status. The American Naturalist, 1992, 140(2): 287-304.
[48] Mizukami Y, Fischer R L. Plant organ size control: AINTEGUMENTA regulates growth and cell numbers during organogenesis. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(2): 942-947.
[49] Varaud E, Brioudes F, SzÉcsi J, Leroux J, Brown S, Perrot-Rechenmann C, Bendahmane M. AUXIN RESPONSE FACTOR8 Regulatespetal growth by interacting with the bHLH transcription factor BIGPETALp. The Plant Cell, 2011, 23(3): 973-983.
[50] Mimura M, Nagato Y, Itoh J I. Rice PLASTOCHRON genes regulate leaf maturation downstream of the gibberellin signal transduction pathway. Planta, 2012, 235(5): 1081-1089.
[51] Santner A, Calderon-Villalobos L I A, Estelle M. Plant hormones are versatile chemical regulators of plant growth. Nature Chemical Biology, 2009, 5(5): 301-307.
[52] Zhao L H, Cai H Y, Su Z X, Wang L L, Huang X Y, Zhang M, Chen P J, Dai X Z, Zhao H M, Palanivelu R, Chen X M, Qin Y. KLU suppresses megasporocyte cell fate through SWR1- mediated activation of WRKY28 expression in. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(3): E526-E535.
Overexpression of wheatincreases flower organ size

1College of Landscape Architecture and Art, Northwest A&F University, Yangling 712100, Shaanxi;2College of Life Sciences, Northwest A & F University, Yangling 712100, Shaanxi;3College of Agronomy, Northwest A&F University, Yangling 712100, Shaanxi
【Objective】The function and mechanism ofregulating flower organ size was preliminarily analyzed by means of expression pattern analysis, transgenic overexpression and cytological observation. The results provide genetic resources and theoretical basis for crop genetic improvement. 【Method】According to the sequence information of CYP78A family members of different species in EnsemblePlants genome database, sequence alignment and evolutionary analysis were carried out for the homologous genes ofwere analyzed by bioinformatics. Through the strategy of constitutive overexpression and local specific overexpression in reproductive organs of, it is clear thathas the function of regulating flower organ size. The cytological characteristics of flower organs of different transgenicwere observed under microscope, and the cytological mechanism ofregulating flower organ size was analyzed. The function ofexpression on spike size and other spike traits of different wheat accessions. 【Result】The gene and protein sequence similarity of wheatandis low, but the gene and protein structure similarity is high. Wheatandare widely expressed in many organs, but highly expressed in flower organs. Compared with wild type, the constitutive overexpression ofcould lead to the enlargement of flower organs and a significant increase in petal area of 13.5%-35.4%. Moreover, the specific overexpression ofonly in the ovule was enough to cause the enlargement of the flower organ of, and the petal area increased significantly by 9%-22.1%. On the contrary, the flower organ ofmutant was significantly smaller than that of wild type, and the petal area was significantly reduced by 27%. The constitutive overexpression ofinresulted in a significant increase in the size of petal epidermal cells by 49%-54% compared with wild type, and a significant decrease in the number of cells by 11%-19% compared with wild type. Locally specific overexpression ofinalso resulted in a significant increase in the size of petal epidermal cells by 20%-49% compared with wild type, and a significant decrease in the number of cells by 8%-24% compared with wild type. The constitutive overexpression ofexpression level had longer spike length, more grains per spikelet and fewer spikelets per spike than wheat accessions with lowerexpression level, but there was no significant difference in grain number per spike. 【Conclusion】promoted the growth of flower organs in a non cellular self-made mode. The overexpression ofcould lead to the enlargement of flower organs.
wheat;; overexpression; flower organ size; cell expansion
2022-11-28;
2023-02-22
国家自然科学基金(32072003,32072059)
彭海霞,E-mail:penghaixia@nwafu.edu.cn。通信作者马猛,E-mail:mengma5@nwafu.edu.cn
10.3864/j.issn.0578-1752.2023.09.002
(责任编辑 李莉)
