菌糠对棉花黄萎病及棉花根际微生物群落组成的影响
2023-04-29曲远航刘天聪鹿秀云李社增郭庆港马平
曲远航 刘天聪 鹿秀云 李社增 郭庆港 马平
摘要:【目的】評价菌糠对棉花黄萎病的防治效果(防效),明确菌糠对棉花根际微生物群落的影响。【方法】通过温室盆栽试验及田间小区试验评价香菇菌糠对棉花黄萎病的防效;利用实时荧光定量聚合酶链式反应检测棉花根际大丽轮枝菌数量;利用宏基因组测序及生物信息学分析棉花根际微生物群落和功能基因组成。【结果】土壤中添加2%(质量分数)的菌糠对棉花黄萎病的温室防效达到76.8%。添加菌糠后棉花根际大丽轮枝菌数量较空白对照减少81.02%。添加菌糠后棉花根际微生物中微杆菌属(Microbacterium spp.)、中慢生根瘤菌属(Mesorhizobium spp.)、梨形孢属(Serendipita spp.)、毛壳菌属(Chaetomium spp.)等有益微生物丰度显著上升;同时,添加菌糠还改变了棉花根际细菌和真菌的共现模式,在提高细菌物种间关联性的同时降低真菌物种间关联性。利用京都基因和基因组数据库功能注释发现,菌糠处理后棉花根际微生物ABC转运蛋白(ko02010)、双组分调节系统(ko02020)、群体感应(ko02024)等涉及细菌信号识别和定植的基因相对丰度显著提高。在河北和新疆,菌糠对棉花黄萎病的田间防效最高分别为36.84%和43.98%。【结论】菌糠能够有效防治棉花黄萎病并降低棉花根际大丽轮枝菌数量,同时可改变棉花根际微生物群落和功能基因组成。
关键词:菌糠;大丽轮枝菌;棉花黄萎病;根际微生物;宏基因组
Effects of spent mushroom substrate on cotton Verticillium wilt and the cotton rhizosphere microbiome
Qu Yuanhang1, 2, Liu Tiancong1, 2, Lu Xiuyun1, Li Shezeng1, Guo Qinggang1*, Ma Ping1
(1. Institute of Plant Protection, Hebei Academy of Agriculture and Forestry Sciences/Integrated Pest Management Innovation Center of Hebei Province/Key Laboratory of Integrated Pest Management on Crops in Northern Region of North China, Ministry of Agriculture and Rural Affairs, Baoding, Hebei 071000, China; 2. College of Plant Protection, Agricultural University of Hebei, Baoding, Hebei 071000, China)
Abstract: [Objective] The objective of this study is to evaluate the control effect of spent Lentinus edodes substrate (SMS) on cotton Verticillium wilt and to clarify the effect of SMS on microbial community of cotton rhizosphere. [Method] The control effect of SMS on cotton Verticillium wilt was evaluated by pot and plot experiments under greenhouse and field condition. The population of Verticillium dahliae WX-1 in cotton rhizosphere was quantified by real time fluorescent quantitative polymerase chain reaction. The microbiome and functional genes composition in cotton rhizosphere were analyzed by metagenomic sequencing and bioinformatics method. [Result] Soil amended with 2% (mass fraction) SMS could successfully suppress cotton Verticillium wilt, and the greenhouse control effect of SMS on cotton Verticillium wilt was 76.8%. The population of V. dahliae WX-1 in cotton rhizosphere amended with SMS decreased by 81.02% compared with the blank control. The abundances of beneficial microorganisms such as Microbacterium spp., Mesorhizobium spp., Serendipita spp., Chaetomium spp. were significantly increased. Meanwhile, the co-occurrence pattern of bacteria and fungi was also changed in the SMS treatment, and the interspecific association of bacteria was increased while the interspecific association of fungi was decreased. Kyoto Encyclopedia of Genes and Genomes (KEGG) functional annotation showed that the relative abundance of ABC transporter (ko02010), two-component regulatory system (ko02020), and quorum sensing (ko02024) genes involved in bacterial signal recognition and colonization of cotton rhizosphere microorganisms were significantly increased after SMS treatment. In Hebei and Xinjiang, the highest field control effect of SMS on cotton Verticillium wilt was 36.84% and 43.98%, respectively. [Conclusion] SMS can effectively control cotton Verticillium wilt and decrease the population of V. dahliae in the cotton rhizosphere, and change the microbiome and functional gene composition.
Keywords: spent mushroom substrate; Verticillium dahliae; cotton Verticillium wilt; rhizosphere microbiome; metagenomic
由大丽轮枝菌(Verticillium dahliae)引起的棉花黄萎病是棉花生产上危害最为严重的土传病害之一。目前生产上抗黄萎病棉花品种缺乏,且抗性不稳定,加之在没有寄主的情况下大丽轮枝菌能够以微菌核的形式在土壤中存活15年之久[1];因此,通过使用抗病品种和轮作等措施仍然难以有效防治黄萎病,导致黄萎病在我国主产棉区发病严重,亟需研发有效的防治措施。
研究发现,土壤中添加生物有机质能防治作物黄萎病。生物炭是一种作为土壤改良剂的木炭,能促进植物的生长,并提高其抗逆能力。Ogundeji等[2]研究发现,生物炭对茄子(Solanum melongena)黄萎病防效超过70%,且根际细菌多样性和丰度均显著提高,有益微生物如芽孢杆菌(Bacillus spp.)、毛壳菌(Chaetomium spp.)等在茄子根际得到富集。西蓝花残体粉碎后还田能有效防治生菜、马铃薯和棉花黄萎病,异硫氰酸盐及其水解产物氢氰酸是西蓝花残体分解过程中产生的主要抑菌物质[3];此外,西蓝花残体还田能增加作物根际有益菌链霉菌(Streptomyces spp.)和芽孢杆菌的丰度[4]。利用作物秸秆或动物粪便堆肥后的生物有机肥同样能增强土壤生物酶活性,改善土壤微生物区系,防治作物黄萎病[5-6]。
菌糠是种植食用菌后剩余的废料,我国每年生产食用菌后约有菌糠2亿t。菌糠作为农业废弃物如果不加利用随意堆放将造成资源浪费和环境污染。因此,菌糠的回收和科学利用是目前食用菌生产中迫切需要解决的问题,科学有效利用菌糠具有重要的经济效益和生态效益。菌糠富含有机物及矿质元素、有毒元素少、结构疏松、保水能力强,是一种良好的土壤处理剂[7]。另外,菌糠富含食用菌菌丝体及其代谢产物,使其具有多种与微生物互作相关的生物学特性[8]。国内外研究证明,土壤中添加菌糠能有效防治尖孢镰刀菌(Fusarium oxysporum)引起的作物枯萎病。菌糠能增加作物根际有益微生物数量,降低病原菌数量,从而达到控制病害、促进作物生长的效果[9-10]。然而,目前有关菌糠防治作物黄萎病的报道甚少。本研究通过温室盆栽试验和田间小区试验,评价土壤添加菌糠对棉花黄萎病的防治效果,并对棉花根际土壤进行宏基因组分析,从生态学角度解析菌糠防治棉花黄萎病的机理,为菌糠的科学利用以及棉花黄萎病的绿色防控提供依据。
1 材料与方法
1.1 试验材料
试验所用棉花品种为感黄萎病品种“鄂荆1号”,大丽轮枝菌强致病力菌株WX-1由河北省农林科学院植物保护研究所植物病害生物防治实验室提供。所用土壤收集自棉花连作地块。香菇(Lentinus edodes)菌糠(spent mushroom substrate, SMS)收集自河北阜平食用菌厂,菌糠自然风干后粉碎至粒径约150 μm备用。
1.2 病原菌接种液的制备
WX-1在马铃薯葡萄糖液体培养基(potato dextrose broth,PDB;含马铃薯200 g·L-1,葡萄糖20 g·L-1)中,25 ℃、180 r·min-1下培养6 d。培养液经4层纱布过滤,滤液在4 ℃、10 000 r·min-1条件下离心20 min,去除上清液后用无菌水重悬孢子并调整孢子含量至1×107 mL-1。
1.3 菌糠对大丽轮枝菌WX-1的影响
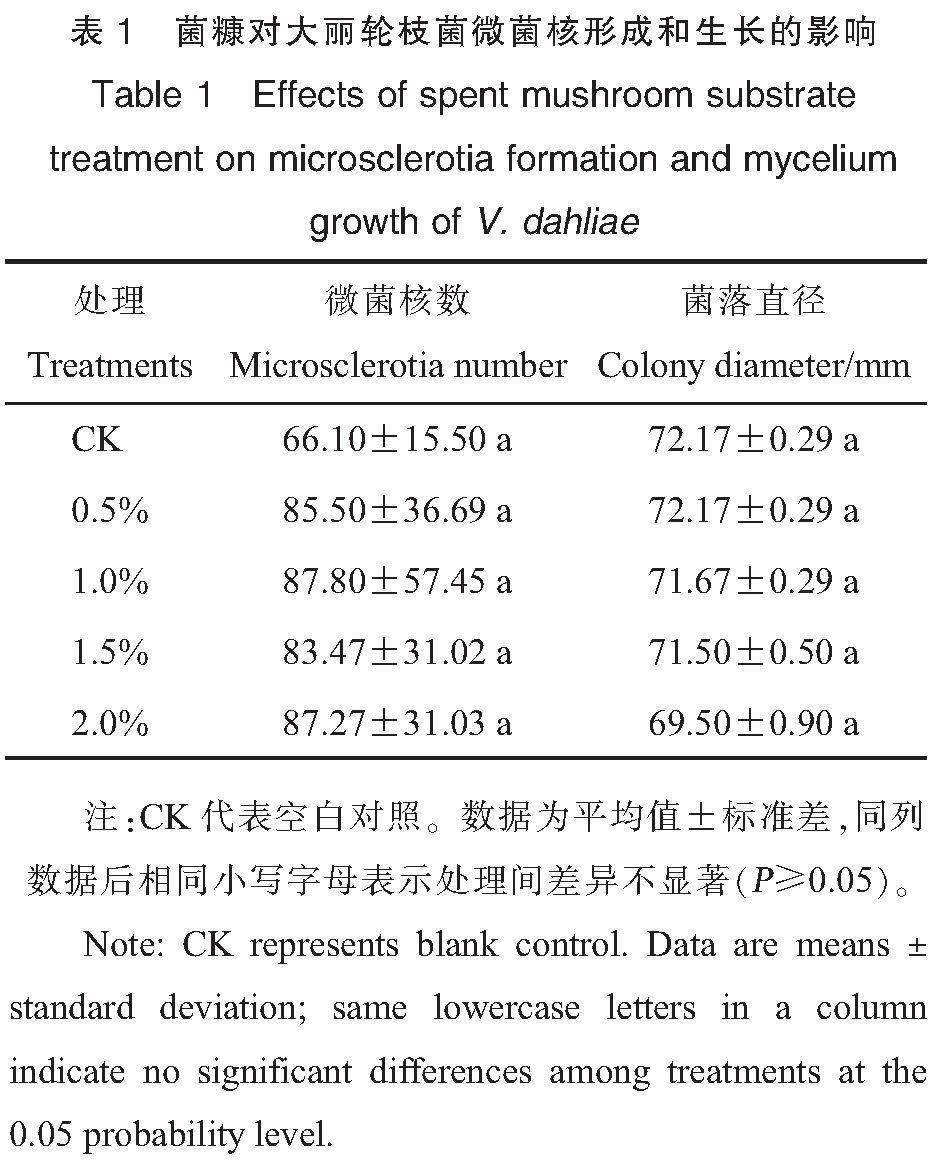
为了分析菌糠对WX-1的抑制作用,在马铃薯葡萄糖琼脂培养基(potato dextrose agar,PDA;主要成分:马铃薯200 g·L-1,葡萄糖20 g·L-1,琼脂15 g·L-1)和PDB培养基中添加不同质量分数(0.5%、1.0%、1.5%和2.0%)的菌糠粉(121 ℃灭菌15 min)。取5 μL WX-1孢子悬浮液(孢子含量为1×107 mL-1,下同)接種于添加不同质量分数菌糠粉的PDA培养皿(直径9 cm)中间,25 ℃黑暗培养20 d,测量菌落直径。按照1%(质量分数)的接种量将WX-1孢子悬浮液接种于添加不同质量分数菌糠粉的PDB培养液中,25 ℃、180 r·min-1黑暗培养6 d,利用血球计数板测量孢子含量。评价菌糠粉对WX-1微菌核形成的影响,在Buffered Minimal Methanol (BMM)培养基(主要成分:5 g·L-1葡萄糖、0.2 g·L-1 NaNO3、0.52 g·L-1 KCl、1.52 g·L-1 KH2PO4、0.52 g·L-1 MgSO4·7H2O、3 μmol·L-1维生素B1、0.1 μmol·L-1维生素B7;pH=8.5)中添加不同质量分数(0.5%、1.0%、1.5%和2.0%)的菌糠粉(121 ℃灭菌15 min),在含有菌糠粉的BMM培养皿上铺1层微孔滤膜(孔径0.2 μm),然后将WX-1孢子悬浮液(100 μL)均匀涂布在滤膜上,25 ℃黑暗培养14 d,利用显微镜观察菌糠粉对WX-1微菌核形成的影响,记录每个视野下微菌核数量。以上每个处理设3个重复,以不添加菌糠粉的培养基作为空白对照。
1.4 菌糠对棉花黄萎病的影响
通过温室盆栽试验评估菌糠对棉花黄萎病的防治效果(防效,control effect, CE)。将2%(质量分数)菌糠粉与土壤充分混匀,装入书式育苗盒中。棉种表面消毒后于25 ℃催芽,每孔播种2粒露白的棉花种子。育苗盒于温室(白天30 ℃,夜间20 ℃)中培养,待出苗后仅保留1株长势一致的幼苗。以土壤中未添加菌糠的处理为空白对照。棉苗长至2叶1心时,采用切根蘸孢子悬浮液的方法接种大丽轮枝菌[11]。接菌后棉苗继续在温室中培养,接菌后21 d调查植株发病情况,并计算病情指数(disease index, DI)。计算公式:DI=∑(n×d)/(N×4)×100。式中,n代表各级病株数,d代表相应病级值,N代表总株数。分级标准[12]如下:0级为植株无症状表现;1级为1~2片真叶发病;2级为3~4片真叶发病;3级为4片以上真叶发病或脱落;4级为全株枯死。根据病情指数计算菌糠处理对棉花黄萎病的防效。计算公式:CE(%)=(DI0-DI1)/ DI0×100。式中,DI0代表空白对照的病情指数,DI1代表菌糠处理的病情指数。
1.5 宏基因组分析
接种病原菌后28 d,打开书式育苗盒并将棉花从中轻轻取出,抖掉根围土后,用毛刷仔细将紧密吸附在根表的根际土刷落并收集。收集到的根际土过孔径280 μm(50目)筛,去除根及其他植物组织,立即冻存于-80 ℃,以备提取土壤DNA。
使用FastDNATM SPIN Kit for Soil(MP Biomedicals, Solon, OH, USA)试剂盒提取根际土壤总DNA。使用NanoDrop 2000分光光度计(Thermo Fisher Scientific Inc., Waltham, MA, USA)测定DNA的质量浓度和质量。宏基因组测序和生物信息分析由上海美吉生物医药科技有限公司利用高通量测序仪HiSeq○R 2000 System(Illumina,美国)完成。每个样品设6个生物学重复。
空白对照和菌糠处理的棉花根际群落结构使用QIIME(quantitative insights into microbial ecology,version 1.9.1)计算距离矩阵(基于Bray-Curtis算法),再使用R软件(version 3.2.1)中vegan包进行主坐标分析(principal coordinate analysis, PCoA)。使用DIAMOND(https://github.com/bbuchfink/diamond)将非冗余基因集与非冗余蛋白质序列数据库(non-redundant protein sequence database, NR)在京都基因和基因組数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)中进行比对并注释,获得物种或功能基因注释信息,然后使用对应的基因丰度总和计算该物种或功能基因丰度,在门、属、直系同源物和代谢通路水平上统计非冗余基因集在各个样品中的丰度,构建相应分类水平上的丰度表,并利用R软件作图。
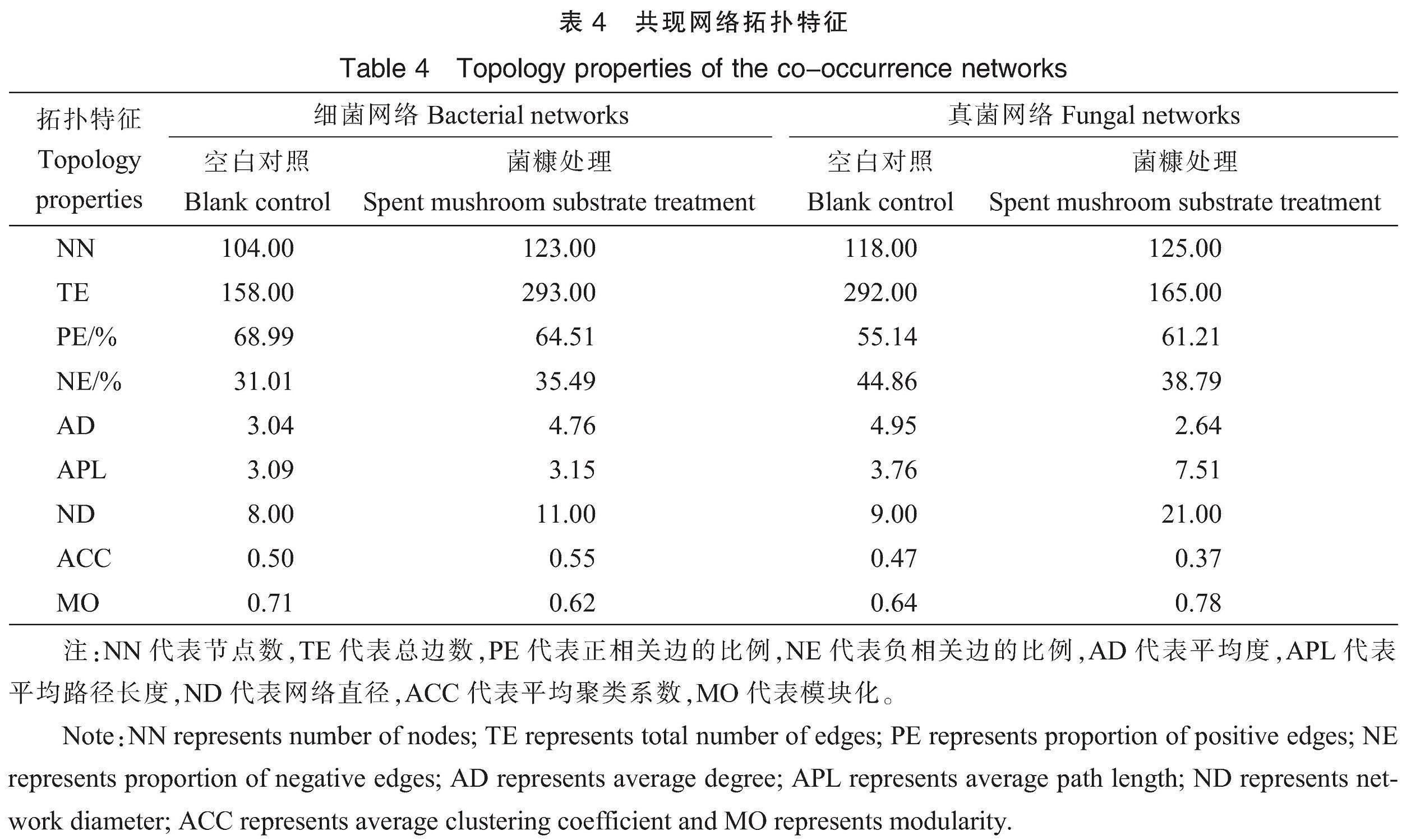
通过Cytoscape平台的CoNet计算根际微生物的相关性和相似性,构建细菌和真菌群落的共现模式。相关分析采用皮尔逊和斯皮尔曼数值为衡量标准,微生物不同属之间利用Bray-Curtis和Kullback-Leibler数值衡量不相似性。当相关系数高于0.8且P<0.01时,认为分类群之间存在统计学上具有强相关关系的共现模式。使用Brownian法将6次测量的P值合并,然后使用Benjamini-Hochberg程序进行调整,以减少获得假阳性结果的概率。使用Gephi软件对共现模式进行可视化,以节点代表微生物组网络中的单个微生物分类群(属),网络边线表示节点之间的成对相关性,表明在生物学上有意义的相互作用。使用Cytoscape平台的MCODE对微生物网络的拓扑特征进行评估,包括节点数(number of nodes, NN)、总边数(total number of edges, TE)、正相关边的比例(proportion of positive edges, PE)、负相关边的比例(proportion of negative edges, NE)、平均度(average degree, AD)、平均路径长度(average path length, APL)、网络直径(network diameter, ND)、平均聚类系数(average clustering coefficient, ACC)和模块化(modularity, MO)。
1.6 根际土中大丽轮枝菌定量检测
使用FastDNATM SPIN Kit for Soil试剂盒提取土壤DNA,通过实时荧光定量聚合酶链式反应(real time fluorescent quantitative polymerase chain reaction, RT-PCR)技术定量检测棉花根际土中WX-1含量[13]。利用大丽轮枝菌特异性引物对WX-1基因组DNA进行PCR扩增,扩增片段插入到pMD19-T载体,经PstⅠ限制性内切酶线性化后进行系列10倍梯度稀释,获得拷贝数为101~107 μL-1的DNA稀释液。以系列稀释液为模板进行 RT-PCR扩增。以质粒拷贝数的对数值为横坐标,以RT-PCR的循环阈值(cycle threshold, Ct)为纵坐标,建立标准曲线。根据标准曲线计算不同处理棉花根际土中WX-1的丰度(以每克土壤的质粒拷贝数计,单位:copies·g-1)。
1.7 菌糠防治棉花黄萎病田间小区试验
为了进一步验证菌糠对棉花黄萎病的防效,分别在河北省高阳县北于八村(简称为“高阳地块”)和新疆生产建设兵团第一师十六团8连3116号地(简称为“第一师地块”)开展田间小区试验。按照30 t·hm-2施用菌糠粉,于播种前翻地时撒施于试验地块。高阳地块所用棉花品种为农大棉36号,新疆试验地块所用棉花品种为塔河2号。在高阳地块和第一师地块分别于2022年8月17日、29日和7月21日、8月25日调查黄萎病发生情况,病情指数分级及防效计算同1.4。第一师地块在8月25日测产,每个小区随机选取6 m2地块,调查棉花铃数,以反映对产量的影响。2个试验地块空白对照和菌糠处理均设置3次重复。
1.8 统计分析
采用单因素方差分析计算菌糠各处理与空白对照之间在防治棉花黄萎病的温室试验中的差异。当单因素方差分析显示有显著差异(P<0.05)时,使用Fisher最小显著差数(least significant difference, LSD)法检验处理间的差异性。所有统计分析均使用SPSS Statistics R26.0.0.0软件(SPSS, Chicago, IL, USA)进行。利用GraphPad Prism 9.0.0作图。
2 结果与分析
2.1 香菇菌糠对大丽轮枝菌的影响
抑菌试验结果(表1)表明,与空白对照相比,不同质量分数(0.5%、1.0%、1.5%和2.0%)菌糠处理对WX-1微菌核形成和菌落生长没有显著影响。
2.2 菌糠对棉花黄萎病的防治效果
温室盆栽试验结果(图1)显示:接菌后21 d,空白对照棉花黄萎病病情指数为42.19,而2%菌糠(SMS)处理的棉花黄萎病病情指数仅为9.78。SMS处理对棉花黄萎病的防效达到76.8%。表明土壤添加2%菌糠对棉花黄萎病具有较好防效。
2.3 菌糠处理对棉花根际WX-1数量的影响
利用RT-PCR技术对不同处理棉花根际土壤中大丽轮枝菌数量的测定结果(图2)表明,空白对照中根际WX-1丰度为3.62×106 copies·g-1;2%SMS处理后棉花根际WX-1数量为6.87×105 copies·g-1,与空白对照相比极显著下降81.02%。
2.4 菌糠对棉花根际微生物群落的影响
对棉花根际土壤中微生物进行宏基因组分析,通过Hiseq高通量测序,每个土壤样品产生约6 Gb的数据量,对原始序列进行质控处理,去除低质量序列、模糊序列和嵌合体序列后,空白对照和SMS处理分别得到5.28×107和5.25×107条质控序列(表2)。所有处理覆盖率均超过97%,表明分析数据基本覆盖环境样品中全部微生物,能够比较全面真实地反映棉花根际土壤微生物群落组成。
与空白对照相比,SMS处理对棉花根际细菌和真菌群落影响差异明显。主坐标分析表明,2种处理方式下细菌(图3A)和真菌(图3B)群落组成在分類学基础上分别沿第一和第二坐标轴分开聚集,第一主成分(PC1)分别解释所有变量的95.82%和70.93%;第二主成分(PC2)分别解释所有变量的0.79%和5.07%,累积贡献率分别为96.61%和76.00%,说明能够表征细菌和真菌群落组成的特征。SMS处理与空白对照处理在细菌和真菌群落上沿PC1显著分开,说明菌糠是引起棉花根际细菌和真菌群落差异的主要因素。
在门水平,共注释到152个细菌门和8个真菌门。细菌群落中,变形菌门(Proteobacteria,48.62%)占比最高,其次分别为放线菌门(Actinobacteria,31.84%)、酸杆菌门(Acidobacteria,5.72%)、拟杆菌门(Bacteroidetes,5.64%)、芽单胞菌门(Gemmatimonadetes,1.78%)、疣微菌门(Verrucomicrobia,1.77%)、浮霉菌门(Plancto-mycetes,1.15%)和绿弯菌门(Chloroflexi,1.00%),相对丰度小于1%的类群归入其他(图4A)。真菌群落中,子囊菌门(Ascomycota,61.75%)占主导地位,其次分别为担子菌门(Basidiomycota,27.67%)、毛霉门(Mucoromycota,6.42%)、壶菌门(Chytridiomycota,2.30%)和捕虫霉门(Zoopagomycota,1.67%),相对丰度小于1%的类群归入其他(图4B)。
在属水平,共注释到3 380个细菌属和347个真菌属。细菌群落中,14个属相对丰度大于1%(表3)。其中,相对丰度较高的属有类诺卡氏菌属(Nocardioides spp.,11.02%)、罗河杆菌属(Rhodanobacter spp.,4.04%)、溶杆菌属(Lysobacter spp.,2.90%)、德沃斯氏菌属(Devosia spp.,2.90%)、慢生根瘤菌属(Bradyrhizobium spp.,2.32%)和鞘氨醇单胞菌属(Sphingomonas spp.,2.05%)。真菌群落中,13个属相对丰度大于1%(表3)。其中,相对丰度较高的属有梨形孢属(Serendipita spp.,13.76%)、轮枝孢属(Verticillium spp.,12.59%)、油脂酵母属(Lipomyces spp.,4.64%)、曲霉属(Aspergillus spp.,4.19%)和小菇属(Mycena spp.,4.01%)。
属水平差异分析显示,空白对照和SMS处理中,3 380个细菌属中有1 922个属相对丰度差异显著,347个真菌属中有87个属相对丰度差异显著。相对丰度前10的差异物种中,类诺卡氏菌属、德沃斯氏菌属、诺尔氏菌属、微杆菌属、中慢生根瘤菌属细菌(图5A)和梨形孢属、毛壳菌属、Thermothielavioides spp.、柄孢壳属真菌(图5B)相对丰度在菌糠处理(SMS)中显著提高,而罗河杆菌属、溶杆菌属、慢生根瘤菌属、鞘氨醇单胞菌属、链霉菌属细菌和轮枝菌属、油脂酵母属、曲霉属、小菇属、毛霉属、根孢囊霉属真菌相对丰度在SMS处理中则显著降低。
2.5 棉花根际微生物共现网络分析
网络分析表明,与空白对照相比(图6A、C),菌糠处理后根际土壤细菌网络更加复杂,物种间联系紧密(图6B);而真菌网络则更简单,物种间联系松散(图6D)。从拓扑特征来看(表4),不同处理节点数相近,而总边数差异较大。与空白对照相比,菌糠处理后细菌网络总边数提高了85.44%,真菌网络总边数降低了43.49%;从正负相关边的比例(PE和NE)来看细菌群落以协同作用为主,而真菌群落协同和竞争作用共存;细菌网络模块化(MO)水平降低了12.68%,真菌网络模块化(MO)水平提高了21.88%,说明与对照相比,菌糠处理的细菌网络更稳定而真菌网络波动性更强。
2.6 菌糠对棉花根际微生物功能基因的影响
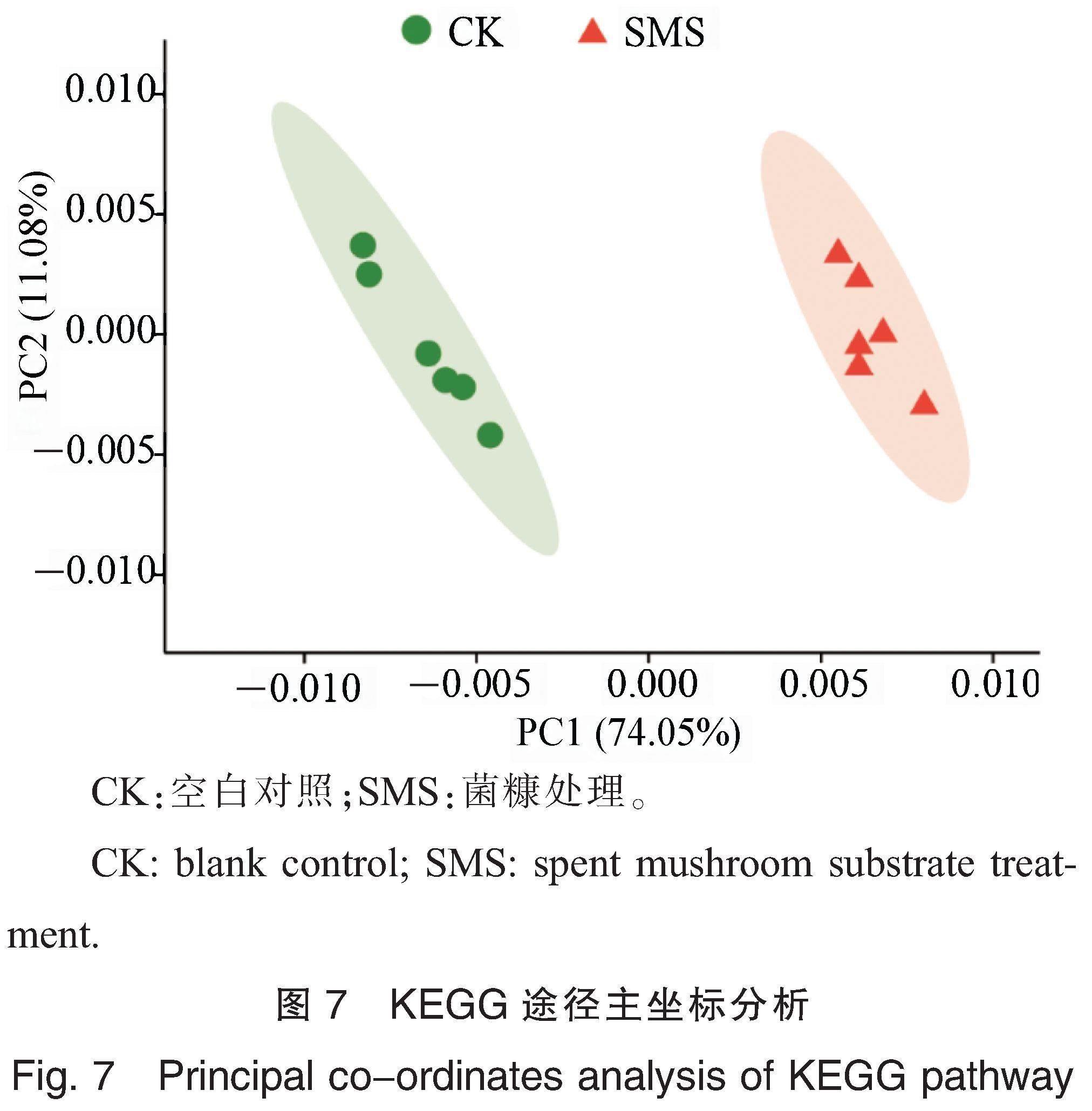
通过比对KEGG数据库获得基因对应的功能注释并进行统计,共获得333条KEGG途径。PCoA结果(图7)显示,菌糠处理和空白对照的根际微生物群落功能基因沿第一坐标轴分开聚集,第一主成分(PC1)解释所有变量的74.05%,第二主成分(PC2)解释所有变量的11.08%;累积贡献率为85.13%,说明能够表征根际微生物功能基因组成的特征。菌糠处理在功能基因上与空白对照处理沿PC1显著分开,说明菌糠是引起功能基因差异的主要因素。
在去除非微生物KEGG途径后,61条KEGG途径的相对丰度(38.54%)在空白对照和菌糠处理间差异显著。其中,相对丰度较高的15类KEGG途径中(图8),与ABC转运蛋白(ATP-binding cassette transporter, ABC transporters, ko02010)、双组分调节系统(two-component regulatory system, ko02022)和群体感应系统(quorum sensing, ko02024)相关基因的丰度在菌糠处理后显著提高,而与氨基酸的生物合成(biosynthesis of amino acids,ko01230)、碳代谢(carbon metabolism, ko01200)、丙酮酸代谢(pyruvate metabolism, ko00620)等12条途径相关基因的丰度在菌糠处理中则显著降低。
2.7 菌糠对棉花黄萎病的田间防效及结铃影响
在河北高阳地块和第一师地块的田间小区试验中,2个试验地块的2次病害调查中菌糠对棉花黄萎病均表现出较好的防效(表5)。在高阳地块的2次调查中防效分别为34.48%和36.84%;在第一师地块的2次调查中防效分别为43.98%和41.81%,整体好于高阳地块。第一师地块的调查结果显示,菌糠处理地块的单位面积结铃数增加5.44%。
3 讨论
菌糠作为一种来源广泛且廉价的农业废弃物,作为土壤添加剂防治作物土传病害的潜力受到越来越多关注。以往研究证明菌糠对由尖孢镰刀菌引起的经济作物枯萎病具有良好防效,而菌糠防治其他土传病害的报道较少。本研究发现,土壤添加菌糠能有效防治棉花黄萎病,显著降低棉花根际大丽轮枝菌的数量。菌糠作为培养食用菌的废弃物,其中含有食用菌菌丝体及其代谢产物,因此,菌糠具有与微生物互作相关的生物学特性。本研究中,温室盆栽试验表明0.5%、1.0%、1.5%和2.0%的香菇菌糠对大丽轮枝菌的生长和微菌核形成没有显著影响,据此推测,菌糠防治棉花黄萎病与其抑菌活性无关。
植物根际富集有益微生物可能是防治作物土传病害的机理之一[14]。本研究分析了菌糠处理对棉花根际微生物群落结构的影响,结果表明,与空白对照相比,菌糠处理增加了棉花根际德沃斯氏菌属、微杆菌属、中慢生根瘤菌属、梨形孢属、毛壳菌属等潜在有益微生物的丰度,在黄瓜上的应用中得到类似结果[9]。有益微生物在维持作物健康生长,抵御病原菌侵染中扮演重要角色[15]。Pereira等[16]利用食石油微杆菌(M. oleovorans)处理玉米种子能够减少玉米出苗后根内拟轮枝镰孢菌(Fusarium verticillioides)数量,从而缓解病害嚴重程度。植物内生真菌印度梨形孢(S. indica)定植于多种植物的根部,可以增强这些植物的生长和抗逆能力,Cheng等[17]研究发现印度梨形孢能诱导香蕉对香蕉枯萎菌(F. oxysporum f. sp. cubense)产生抗性。毛壳菌是一类重要的生防菌,能有效防治多种植物真菌病害[18]。Ogundeji等[2]研究发现,毛壳菌在茄子根际的富集可能是生物炭防治茄子黄萎病的主要机理之一。以上结果表明,棉花根际富集有益微生物可能是菌糠防治棉花黄萎病的机理之一。
研究证明,根际微生物间较高的关联性有助于减轻病原微生物的侵染[19]。模块化可在一定程度上反映网络的稳定性,较高的模块化代表种群间竞争的加剧导致网络稳定性降低[20]。共现网络分析发现,菌糠是导致微生物群落联系改变的主要因素,而变化模式在细菌和真菌群落中则截然相反。在细菌共现网络中,菌糠提高了网络规模,节点数增加,物种间关联性提高,协同效应增强;而真菌群落共现网络规模减小,物种间关联性降低。菌糠促进了细菌群落间联系,降低了模块化,使得网络更加稳固。
根际微生物群落基因功能注释表明多条涉及微生物信号识别和定植的通路在菌糠处理中富集。双组分调节系统是芽孢杆菌和假单孢菌等生防细菌中一类重要的信号传递系统,调控抑菌物质的合成、生物膜形成与根际定植等重要生防相关性状[21]。群体感应指微生物群体在其生长过程中,由于群体密度的增加,导致其生理和生化特性的变化,是细菌中一类重要的调控系统,参与细菌抗生素产生、生物膜形成以及根际定植等生防性状[22-23]。本研究发现,土壤添加菌糠提高了群体感应相关基因的丰度,推测菌糠处理可富集棉花根际有益微生物,有益微生物数量增加能激活群体感应系统,提高生防菌在根际的定植能力和抗生素产生能力,从而发挥生防效果。此外,植物根际存在丰富多样的微生物,相互之间主要通过产生抑菌物质而相互竞争营养和空间位点。ABC转运蛋白参与调控细菌对抗生素的抗性,激活ABC转运蛋白能提高细菌的抗药性[24]。Armalyte等[25]分析了土壤微生物群落组成和抗生素耐药性的关系,发现鞘氨醇单胞菌通过ABC转运蛋白发挥耐药性并成为优势种群。Muller等[26]通过转座子插入突变证明ABC转运蛋白相关基因在芽孢杆菌与黄色黏球菌(Myxococcus xanthus)的竞争中起到关键作用。由此推测菌糠处理可激活根际有益菌ABC转运蛋白表达,提高有益菌的根际竞争能力及环境适应力,发挥生防效果。
4 结论
土壤中添加菌糠可有效防治由大丽轮枝菌引起的棉花黄萎病,降低根际大丽轮枝菌的数量。增加根际有益微生物丰度、改变微生物共现模式是菌糠防治棉花黄萎病的重要作用机理。该研究从生态调控角度揭示了菌糠防治棉花黄萎病的机理,为创制高效防控作物黄萎病的生防制剂提供了理论依据。
参考文献:
[1] 张文琦. 大丽轮枝菌Fungaltrans转录因子VdFTF1调控致病性功能研究[D]. 北京: 中国农业科学院, 2017.
Zhang Wenqi. Study on pathogenic mechanisms of fungal_trans transcription factor VdFTF1 in Verticillium dahliae[D]. Beijing: Chinese Academy of Agricultural Sciences, 2017.
[2] Ogundeji A O, Li Y, Liu X J, et al. Eggplant by grafting enhanced with biochar recruits specific microbes for disease suppression of Verticillium wilt[J/OL]. Applied Soil Ecology, 2021, 163: 103912 (2021-02-28)[2022-09-05]. https://doi.org/10.1016/j.apsoil.2021.103912.
[3] Inderbitzin P, Ward J, Barbella A, et al. Soil microbiomes associated with Verticillium wilt suppressive broccoli and chitin amendments are enriched with potential biocontrol agents[J/OL]. Phytopathology, 2018, 108(1): 31-43 (2018-01-31)[2023-09-05]. https://doi.org/10.1094/PHYTO-07-17-0242-R.
[4] 趙卫松, 郭庆港, 苏振贺, 等. 马铃薯健株与黄萎病株根际土壤真菌群落结构及其对碳源利用特征[J/OL]. 中国农业科学, 2021, 54(2): 296-309 (2021-02-25)[2023-06-25]. https://doi.org/10.3864/j.issn.0578-1752.2021.02.006.
Zhao Weisong, Guo Qinggang, Su Zhenhe, et al. Characterization of fungal community structure in the rhizosphere soil of healthy and diseased-Verticillium wilt potato plants and carbon source utilization[J/OL]. Scientia Agricultura Sinica, 2021, 54(2): 296-309 (2021-02-25)[2023-06-25]. https://doi.org/10.3864/j.issn.0578-1752.2021.02.006.
[5] Antoniou A, Tsolakidou M D, Stringlis I A, et al. Rhizosphere microbiome recruited from a suppressive compost improves plant fitness and increases protection against vascular wilt pathogens of tomato[J/OL]. Frontiers in Plant Science, 2017, 8: 2022 (2017-11-29)[2023-06-25]. https://doi.org/10.3389/fpls.2017.02022.
[6] Tubeileh A M, Stephenson G T J C P B. Soil amendment by composted plant wastes reduces the Verticillium dahliae abundance and changes soil chemical properties in a bell pepper cropping system[J/OL]. Current Plant Biology, 2020, 22: 100148 (2020-06-12)[2023-06-25]. https://doi.org/10.1016/j.cpb.2020.100148.
[7] Leong Y K, Ma T W, Chang J S, et al. Recent advances and future directions on the valorization of spent mushroom substrate (SMS): a review[J/OL]. Bioresource Technology, 2022, 344: 126157 (2021-10-31)[2023-06-25]. https://doi.org/10.1016/j.biortech.2021.126157.
[8] Hanafi F H M, Rezania S, Taib S M, et al. Environmentally sustainable applications of agro-based spent mushroom substrate (SMS): an overview[J/OL]. Journal of Material Cycles and Waste Management, 2018, 20(3): 1383-1396 (2018-12-28)[2023-06-25]. https://doi.org/10.1007/s10163-018-0739-0.
[9] Wang H W, Xu M, Cai X Y, et al. Application of spent mushroom substrate suppresses Fusarium wilt in cucumber and alters the composition of the microbial community of the cucumber rhizosphere[J/OL]. European Journal of Soil Biology, 2020, 101: 103245 (2020-11-30)[2023-06-25]. https://doi.org/10.1016/j.ejsobi.2020.103245.
[10] Ocimati W, Were E, Tazuba A F, et al. Spent Pleurotus ostreatus substrate has potential for managing Fusarium wilt of banana[J/OL]. Journal of Fungi, 2021, 7(11): 946 (2021-11-30)[2023-06-25]. https://doi.org/10.3390/jof7110946.
[11] 趙卫松, 李社增, 鹿秀云, 等. 西兰花植株残体还田对棉花黄萎病的防治效果及其安全性评价[J/OL]. 中国生物防治学报, 2019, 35(3): 449-455 (2019-07-23)[2023-06-25]. https://doi.org/10.16409/j.cnki.2095-039x.2019.03.012.
Zhao Weisong, Li Shezeng, Lu Xiuyun, et al. Safety evaluation of broccoli residues returning on cotton growth and its control effect on Verticillium wilt[J/OL]. Chinese Journal of Biological Control, 2019, 35(3): 449-455 (2019-07-23)[2023-06-25]. https://doi.org/10.16409/j.cnki.2095-039x.2019.03.012.
[12] 赵丽红, 冯自力, 李志芳, 等. 棉花抗黄萎病鉴定与评价标准的商榷[J/OL]. 棉花学报, 2017, 29(1): 50-58 (2017-03-14)[2023-06-25]. https://doi.org/10.11963/issn.1002-7807.201701006.
Zhao Lihong, Feng Zili, Li Zhifang, et al. Development of an improved standard for identifying and evaluating Verticillium wilt resistance in cotton[J/OL]. Cotton Science, 2017, 29(1): 50-58 (2017-03-14)[2023-06-25]. https://doi.org/10.11963/issn.1002-7807.201701006.
[13] 赵卫松, 郭庆港, 李社增, 等. 西兰花残体还田对棉花黄萎病防治效果及其对不同生育时期土壤细菌群落的影响[J/OL]. 中国农业科学, 2019, 52(24): 4505-4517 (2020-06-11)[2023-06-25]. https://doi.org/10.3864/j.issn.0578-1752.2019.24.006.
Zhao Weisong, Guo Qinggang, Li Shezeng, et al. Control efficacy of broccoli residues on cotton Verticillium wilt and its effect on soil bacterial community at different growth stages[J/OL]. Scientia Agricultura Sinica, 2019, 52(24): 4505-4517 (2020-06-11)[2023-06-25]. https://doi.org/10.3864/j.issn.0578-1752.2019.24.006.
[14] Jiao X R, Takishita Y, Zhou G S, et al. Plant associated rhizobacteria for biocontrol and plant growth enhancement[J/OL]. Frontiers in Plant Science, 2021, 12: 634796 (2021-03-17)[2023-06-25]. https://doi.org/10.3389/fpls.2021.634796.
[15] Ab Rahman S F S, Singh E, Pieterse C M J, et al. Emerging microbial biocontrol strategies for plant pathogens[J/OL]. Plant Science, 2018, 267: 102-111 (2018-02-28)[2023-06-25]. https://doi.org/10.1016/j.plantsci.2017.11.012.
[16] Pereira P, Nesci A, Etcheverry M G. Efficacy of bacterial seed treatments for the control of Fusarium verticillioides in maize[J]. Biocontrol, 2009, 54(1): 103-111.
[17] Cheng C Z, Li D, Qi Q, et al. The root endophytic fungus Serendipita indica improves resistance of banana to Fusarium oxysporum f. sp. cubense tropical race 4[J/OL]. European Journal of Plant Pathology, 2020, 156(1): 87-100 (2019-10-31)[2023-06-25]. https://doi.org/10.1007/s10658-019-01863-3.
[18] Soytong K, Kanokmedhakul S, Kukongviriyapa V, et al. Application of Chaetomium species (Ketomium) as a new broad spectrum biological fungicide for plant disease control: a review article[J]. Fungal Diversity, 2001, 7: 1-15.
[19] Mendes L W, Mendes R, Raaijmakers J M, et al. Breeding for soil-borne pathogen resistance impacts active rhizosphere microbiome of common bean[J/OL]. Isme Journal, 2018, 12(12): 3038-3042 (2018-12-31)[2023-06-25]. https://doi.org/10.1038/s41396-018-0234-6.
[20] Faust K, Raes J J N R M. Microbial interactions: from networks to models[J]. Nature Reviews Microbiology, 2012, 10(8): 538-550.
[21] Haas D, Keel C, Reimmann C. Signal transduction in plantbeneficial rhizobacteria with biocontrol properties[J]. Antonie Van Leeuwenhoek, 2002, 81(1): 385-395.
[22] 王培培, 郭慶港, 李社增, 等. 群体感应调控因子ComA对枯草芽胞杆菌NCD-2抑菌物质产生和生物膜形成的影响[J/OL]. 植物保护学报, 2016, 43(2): 193-200 (2016-05-23)[2023-06-25]. https://doi.org/10.13802/j.cnki.zwbhxb.2016.02.003.
Wang Peipei, Guo Qinggang, Li Shezeng, et al. Functional analysis of quorum sensing regulator ComA on the production of antifungal active compounds and biofilm formation in Bacillus subtilis NCD-2[J/OL]. Acta Phytophylacica Sinica, 2016, 43(2): 193-200(2016-05-23)[2023-06-25]. https://doi.org/10.13802/j.cnki.zwbhxb.2016.02.003.
[23] Wei H L, Zhang L Q. Quorum-sensing system influences root colonization and biological control ability in Pseudomonas fluorescens 2P24[J]. Antonie Van Leeuwenhoek, 2006, 89(2): 267-280.
[24] 孙铭优, 吴照晨, 王斌, 等. ABC转运蛋白及其相关的多药抗性研究现状[J/OL]. 植物保护学报, 2022, 49(1): 374-382 (2022-06-22)[2023-06-25]. https://doi.org/10.13802/j.cnki.zwbhxb.2022.2021100.
Sun Mingyou, Wu Zhaochen, Wang Bin, et al. Research status of ATP-binding cassette transporters and related multidrug resistance[J/OL]. Acta Phytophylacica Sinica, 2022, 49(1): 374-382 (2022-06-22)[2023-06-25]. https://doi.org/10.13802/j.cnki.zwbhxb.2022.2021100.
[25] Armalyte J, Skerniskyte J, Bakiene E, et al. Microbial diversity and antimicrobial resistance profile in microbiota from soils of conventional and organic farming systems[J/OL]. Frontiers in Microbiology, 2019, 10: 892 (2019-04-26)[2023-06-25]. https://doi.org/10.3389/fmicb.2019.00892.
[26] Muller S, Strack S N, Ryan S E, et al. Identification of functions affecting predator-prey interactions between Myxococcus xanthus and Bacillus subtilis[J/OL]. Journal of Bacteriology, 2016, 198(24): 3335-3344 (2016-12-31)[2023-06-25]. https://doi.org/10.1128/JB.00575-16.
