小样本医学关系三元组抽取
2023-04-27蒋帅何良华
蒋帅 何良华
关键词:深度学习;小样本;关系三元组抽取;对抗训练
1 概述
深度神经网络,尤其是预训练语言模型(如BERT) 的使用,使得关系抽取任务获得了极大的性能提升。然而,现有的很多方法往往依赖大量的标注数据,且很难解决数据的长尾分布问题,如关系分类问题,训练数据集中不同类别的样本数量不同,某些关系类别样本非常少,对于这些关系难以准确分类。又如,由于医学中的罕见疾病病人数量非常少,难以获得大量数据样本。
为了解决这些问题,一些基于Few-Shot的实体识别和关系分类数据集和算法被提出。小样本学习结合原型网络,能够很好地学习类别不断变化情况下模型的泛化能力。对于目标领域样本较少的情况,通常可以借助另一较大的数据集(源域)学习通用知识,然后应用到目标域上。此外,在源域上训练时结合领域判别对抗训练,可以学习源域和目标域共性特征,能有效提升模型的泛化能力。结合这些方法和思想,本文的主要工作如下:
1) 结合域判别对抗训练在Wiki域的数据集上预训练模型,为小样本医学关系三元组抽取学习共性的知识;
2) 提出一个基于残差连接的原型网络模块应用于小样本医学关系三元组抽取问题,取得了很好的抽取效果。
2 相关工作
2.1 小样本关系三元组抽取
小样本学习(Few-shot Learning) 模型大致分为三类:基于模型的方式、基于度量的方式和基于优化的方式。基于模型的方式致力于改进模型的结构,使得在少量样本上快速更新模型的参数,基于度量的方式(如原型网络[1-3]) 通过度量测试样本和支持集样本的距离完成分类,基于优化的方式则致力于改进参数的优化方法。当前,在小样本关系三元组抽取领域,大多数算法都采用基于度量的方式解决分类问题。
Haiyang Yu 等人[4]在2020 年提出了MPE(Multi-Prototype Embedding) 模型,应用于小样本关系三元组抽取,先采用序列标记的方式抽取实体,然后根据support样本学习实体原型和句子原型用于表征关系原型,关系分类准确度非常高。然而,这种方式使得整体关系三元组的抽取效果强依赖于实体抽取结果,尽管能够取得较高的关系分类性能,但是由于实体抽取结果较差,导致最终关系三元组抽取结果不理想。
Xin Cong等人[5]在2022年提出了RelATE模型,基于原型学习和注意力网络先对关系进行分类,然后在每个关系下学习实体的START标记原型和END标记原型,即识别出实体的第一个token和最后一个token 位置,从而抽取出实体,然后和之前的关系组合成关系三元组。此算法相对于MPE,采用关系指导的方式,一方面避免了实体对后续模块的影响,另一方面将实体和关系进行了一定的语义结合。同时,实践证明,基于原型网络抽取实体比直接采用序列标记方式在小样本情况下效果更好。但是相对地,关系分类的准确度有一定的下降。
2.2 跨域小样本学习
对于当前域(称为目标域)样本较少的情况,通常可以借助于另一个较大的域(称为源域)学习一些共性的知识,然后将其应用于目标域以提升性能。此外,域判别对抗训练能够学习不同域的共性特征,有助于提升模型的泛化能力,如文献[6]中的Proto-ADV(BERT)网络,基于原型学习和对抗训练的方式提升小样本关系抽取医学域适应模型的性能。
3 小样本医学关系三元组抽取模型
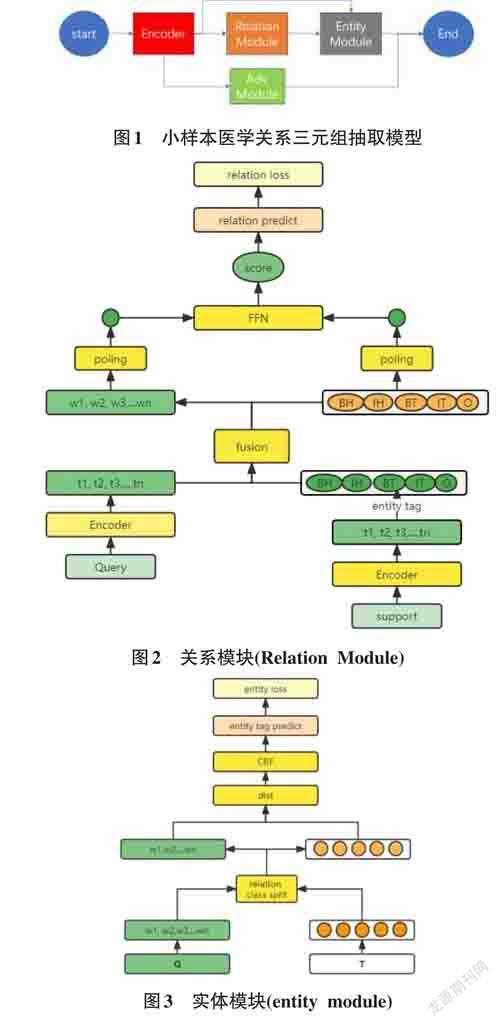
小样本医学关系三元组(Few-Shot Bio Triple Ex⁃traction) 的总体模型如图1所示,包含4个主要模块:编码器模块、关系分类模块、实体识别模块以及域判别对抗训练模块。本文主要介绍当前所做的医学域适应工作,用于解决小样本医学关系三元组抽取问题,包括一个残差连接的实体标记原型网络以及域判别对抗训练模块,其余模块包括关系分类模块和实体识别模块,见图2和图3。
3.1 问题描述
依据传统的Few-shot任务设定,将小样本关系三元组抽取问题定义为NwayKshot问题,其中N对应每次分类时关系的类别数,K表示每个关系类别Support 样本的数量。对于每个Query语句,关系分类问题即对N个类别进行分类;而实体识别问题,使用传统的实体标记(BH、IH、BT、IT、O分别对应头实体第一个to⁃ken、头实体其他token、尾实体第一个token、尾实体其他token、非实体token) 对句子进行序列标记,实体识别即建模为标记预测问题。
3.2 残差连接的实体标记原型网络
对于每个query语句经过encoder层,获得每个to⁃ken的特征表示Qori(t1,t2,…,tn),其中n 为句子的长度,类似的每个support语句经过encoder层得到Sori(t1,t2,…,tn)。对于support样本,由于每个token的实体标记已知,本文对同一关系类别的K 个样本的同类实体标记对应的token特征向量做平均池化,得到實体标记原型Tori(N,5,D)的特征矩阵,其中“5”是实体标记的类别共5 类,D 是特征向量长度。Qori(t1,t2,…,tn)和Tori(N, 5, D)基于注意力机制得到Attention之后的特征表示Qatt(t1,t2,…tn) 和Tatt(N, 5, D),则实体识别模块的输入:query的token特征矩阵Q(t1,t2,..tn)= Qori(t1,t2,…,tn) || Qatt(t1,t2,…tn),实体标记原型T(N,5,D)= Tori(N, 5, D) ||Tatt(N, 5, D),其中||表示拼接操作。
3.3 域判别对抗训练
域判别对抗训练模块的总体流程如图4所示。从源域(Wiki) 和目标域(Bio) 中分别选取M个样本构造无标记样本集合W和B,每个batch分别从两个域选取m个样本,经过encoder层对句子进行编码,选取CLS作为句子表征,2m个特征构成特征矩阵E(2m,D),经过FFN 层得到预测结果Y(2)=W2*ReLU(W1E(2m,D)+B1)+B2,其中D 表示特征向量长度,“2”表示有2个域。最终,域判别对抗训练模块的损失:
4 实验与结果分析
4.1 实验数据集
本文实验均基于Fewrel 2.0 da(domain adaption) 数据集(详见文献[6]) 。该数据集包含来自Wikipedia 语料库和Wikidata知识库采集的80种关系每种关系包含700个样本,以及从PubMed数据库采集的10种医学关系,每种关系包含100个医学样本。
4.2 实验设置
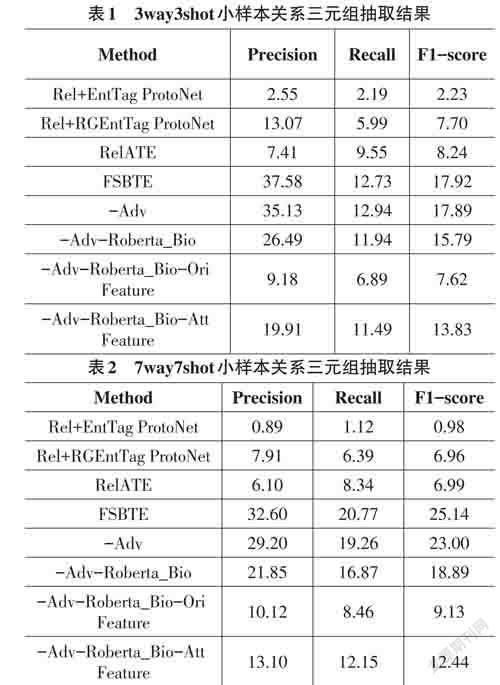
所有实验基于Python3.8 和Pytorch1.7 框架,在NVIDIA GEFORCE 3090 GPU上進行训练和测试。实验随机选取Wiki域的50种关系训练模型(35 000个样本),随机选取PubMed域的3种关系作为验证数据集(300个样本),余下7种关系作为测试数据集(700个样本)。所有实验均在Wiki域迭代训练10 000次,batch⁃size固定为1,每次迭代以5way5shot方式采样support 样本和query样本,在验证集上测试3way3shot关系三元组抽取的F1-score,保存取得最优结果时的模型,然后在测试集上分别以3way-3shot 和7way-7shot 方式随机采样1 000次,计算关系三元组预测结果的pre⁃cision、recall、F1-score的均值。
对比实验设置如下:
1) Rel+EntTag ProtoNet:关系采用ProtoNet,实体采用BIO标记原型(即不区分关系);
2) Rel+RGEntTag ProtoNet:关系采用ProtoNet,实体采用关系指导的实体BIO标记原型;
3) RelATE:文献[3]中的方法;
4) FSBTE,本文方法;
5) FSBTE-Adv,减去域判别对抗训练模块;
6) FSBTE-Adv-Roberta_Bio,在5)的基础上进一步将Roberta-Bio语言模型[7]替换为Bert语言模型,作文Encoder模块;
7) FSBTE-Adv-Roberta_Bio-Ori Feature,在6)的基础上进一步减去残差连接模块中Bert得到的token 特征表示,仅根据关系模块中attention得到token特征表示计算实体标记原型;
8) FSBTE-Adv-Roberta_Bio-Att Feature,在6)的基础上进一步减去残差连接模块中关系模块的atten⁃tion得到token特征表示,仅根据Bert得到的token表示计算实体标记原型。
4.3 实验结果
3way3shot和7way7shot医学关系三元组抽取实验结果如表1 和表2 所示。根据Rel+EntTag ProtNet、Rel+RGEntTag ProtoNet和RelATE三组实验结果可以看出,采用关系指导的方式将实体根据关系区分学习实体的原型表示,可以显著地提升关系三元组抽取的性能;根据FSBTE和FSBTE-Adv两组对比实验可以看出,域判别对抗训练方式对于提升模型的泛化能力依然是十分有效的手段;根据最后三组对比实验可以看出,本文提出的残差连接模块极大地提升了Wiki域适应到医学域泛化性能,表明其对于小样本医学关系三元组抽取问题的有效性。
5 总结
本文提出了一个基于残差连接的原型网络模块,应用于小样本医学关系三元组抽取,同时结合域判别对抗训练,提升了网络域适应能力。多组对比实验证明了本文方法的有效性。
