亚麻FAD基因家族的生物信息学鉴定分析
2023-04-20侯静静赵利王斌
侯静静 赵利 王斌




摘要:不饱和脂肪酸为人体提供基本代谢所必需的能量,须从膳食中补充。脂肪酸去饱和酶FAD(fatty acid desaturase)是植物不饱和脂肪酸合成途径中的关键酶,植物体内脂肪酸的各组分比例和不饱和度与FAD的去饱和作用息息相关。为探究亚麻FAD基因家族的表达与进化,为其在亚麻高品质育种中的应用提供理论依据。运用生物信息学方法对亚麻全基因组FAD基因家族的43个LuFADs基因进行分析。结果显示,该家族成员编码的蛋白质大小为152~453个氨基酸,大部分为碱性不稳定亲水蛋白。与拟南芥FADs蛋白序列构建系统发育树,可分为4个主要亚家族:Δ12/ω-3去饱和酶、“前端”去饱和酶、Δ7/Δ9去饱和酶和SAD去饱和酶。保守结构域和外显子-内含子结构分析得出,同一亚组中的家族成员具有较为相似的基因结构。染色体定位分析呈随机性分布。亚细胞定位预测得出,叶绿体上的家族成员最多。启动子顺式作用元件分析发现,该家族成員中抗氧化反应元件(ARE)数量最多。
关键词:亚麻;FAD基因家族;生物信息学分析
中图分类号:S565.9 文献标志码:A 文章编号:2097-2172(2023)03-0246-08
doi:10.3969/j.issn.2097-2172.2023.03.011
Abstract: Unsaturated fatty acids provide the human body with the energy necessary for basic metabolism and must be supplemented from the diet. FAD (fatty acid desaturase) is a key enzyme in the biosynthesis of unsaturated fatty acids in plants. The proportion and unsaturation of fatty acids in plants are closely related to the desaturation of FAD. In order to explore the expression and evolution of flax (Linum usitatissimum L.) FAD gene family and to provide theoretical basis for its application in high-quality flax breeding, in this study, bioinformatics method was used to analyze 43 LuFADs genes of flax. Results showed that the size of proteins encoded by the members of this family were between 152 to 453 amino acids, most of which were alkaline unstable hydrophilic proteins. The phylogenetic tree was constructed with the FADs protein sequence of Arabidopsis thaliana, which could be divided into four main subfamilies: Δ12/ω-3 desaturase, 'front-end' desaturase, Δ7/Δ9 desaturase and SAD desaturase. The analysis of conserved domain and exon-intron structure showed that the family members in the same subgroup had relatively similar gene structure. The chromosome localization analysis showed random distribution, and the subcellular localization predicted that the most family members were on the chloroplast. Analysis of promoter cis-acting elements showed that the number of antioxidant response elements (ARE) were the largest among this family.
Key words: Linum usitatissimum L.; FAD gene family; Bioinformatics analysis
亚麻(Linum usitatissimum L.) 是一年生自交草本植物,在我国甘肃、内蒙古、山西、宁夏和河北等地广泛种植[1 ]。亚麻可分为油用、纤维用和油纤兼用3种类型,其中油用亚麻俗称胡麻,胡麻籽含油率为40%左右,具有较高的食用营养价值[2 ]。胡麻籽油的脂肪酸组成包括2种脂肪酸(棕榈酸及硬脂酸)和3种不饱和脂肪酸 (油酸、亚油酸及α-亚麻酸),这5种脂肪酸占到胡麻油粗脂肪含量的99%,其中α-亚麻酸达到54%左右[3 ]。α-亚麻酸是人体必需脂肪酸,属于ω-3系不饱和脂肪酸,具有降血脂、抗血栓肿瘤、健脑明目和改善肌肤等多种保健功能[4 ]。
不饱和脂肪酸的合成以饱和脂肪酸硬脂酸为底物,在脂肪酸延长酶(fatty acid elongase,FAE)和脂肪酸去饱和酶FAD(fatty acid desaturase)的作用下,通过一系列的脱氢、延长、去饱和作用形 成[5 ]。其中FAD是植物不饱和脂肪酸合成的关键酶,植物体内脂肪酸各组分的比例及其不饱和度与FAD的去饱和作用息息相关[6 ]。FAD家族主要在质体和内质网中催化油酸生成亚油酸、亚油酸生成亚麻酸[7 ]。依据辅因子和亚细胞定位的不同,FAD可划分为膜结合和可溶性2种类型。膜结合FAD包括ω-6 FAD(催化油酸形成亚油酸)和ω-3 FAD(催化亚油酸形成a-亚麻酸),可溶性FAD包括Δ4 FAD(催化二十碳五烯酸EPA生成十二碳六烯酸DHA)、Δ6 FAD(催化亚油酸生成γ-亚麻酸)及Δ9 SAD(催化硬脂酸生成油酸)[8 ]。学者们对芝麻、油菜、向日葵、油茶等油料作物的FAD基因家族进行了生物信息学分析[9 - 12 ],在其他作物中,例如藜麦、荞麦、茄子等植物中也有该基因家族分析相关报道[13 - 15 ]。相关研究发现,FAD与细胞膜的稳定性和流动性也相关,进而对植株抗逆性产生影响[16 ]。亚麻FAD基因家族研究基于转录组数据的分析[17 ],但随着高通量测序技术的发展以及亚麻全基因组数据的更新,对于脂肪酸合成途径相关基因家族的分析有待进一步完善。我们运用生物信息学方法对亚麻全基因组的FAD基因家族进行分析,包括蛋白理化性质分析、系统进化分析、保守结构域及基因结构分析、染色体定位分析、亚细胞定位预测和启动子顺式作用元件分析,以期为其在亚麻高品质育种中的应用提供理论依据。
1 材料与方法
1.1 序列来源
从NCBI上获得亚麻(version 2.0)15条染色体的基因序列(登录号为CP027619-CP027633)[18 ],从 phytozome数据库(https://phytozome.jgi.doe.gov/pz/ portal.html#)中下载蛋白质序列。
1.2 研究方法
1.2.1 LuFADs基因家族成员筛选与鉴定 使用以下2种方法鉴定亚麻FAD蛋白基因。第1种是基于拟南芥基因组(Version10.0,https://www.arabidopsis.org/)中的27个参考序列,通过BLASTP来鉴定亚麻基因组中的FAD蛋白,E值设置为1.0E-10。第2种是从Pfam蛋白家族数据库 (http://pfam.xfam.org/)下载与FA_Desaturase (PF00487)、FA_Desaturase 2(PF03405)和TMEM189(PF10520)结构域对应的隐马尔可夫模型(HMM)文件作为query,使用HMMER v3.3(http://eddylab.org/software/hmmer/hmmer-3.3.tar.gz)中的hmmsearch工具來查找亚麻蛋白数据库中所有可能的FAD蛋白序列,E值 < 1e-5。
1.2.2 亚麻LuFADs基因的生物信息学分析 利用ClustalW对亚麻、拟南芥FAD蛋白序列进行多重序列比对,接着在MEGA11软件中使用邻接法进行系统进化树构建。使用Bioperl工具(https://bioperl.org/)计算亚麻FAD家族成员的氨基酸数量、蛋白理论等电点(PI)和分子量大小(Mw)等指标[19 ]。利用本地MEME工具(版本 4.11.2,http://alternate.meme-suite.org/tools/meme)搜索亚麻FAD家族成员序列中的保守基序。通过在线软件(MG2C, http://mg2c.iask.in/mg2c_v2.1/)和(GSDS, http://gsds.cbi.pku.edu.cn/) 分别构建亚麻FAD家族成员的染色体位置图和外显子-内含子结构图。使用CELLO (http://cello.life.nc-tu.edu.tw/)进行LuFAD家族成员亚细胞定位预测。采用Perl脚本提取亚麻FAD家族成员基因编码区上游1 500 bp的序列,并利用在线数据库PlantCARE(http: //bioinformatics.psb.ugent.be/webtools/plantcare/html/) 对该基因家族成员中的顺式作用元件进行鉴定[20 ]。
2 结果与分析
2.1 LuFADs基因家族成员鉴定及蛋白理化性质分析
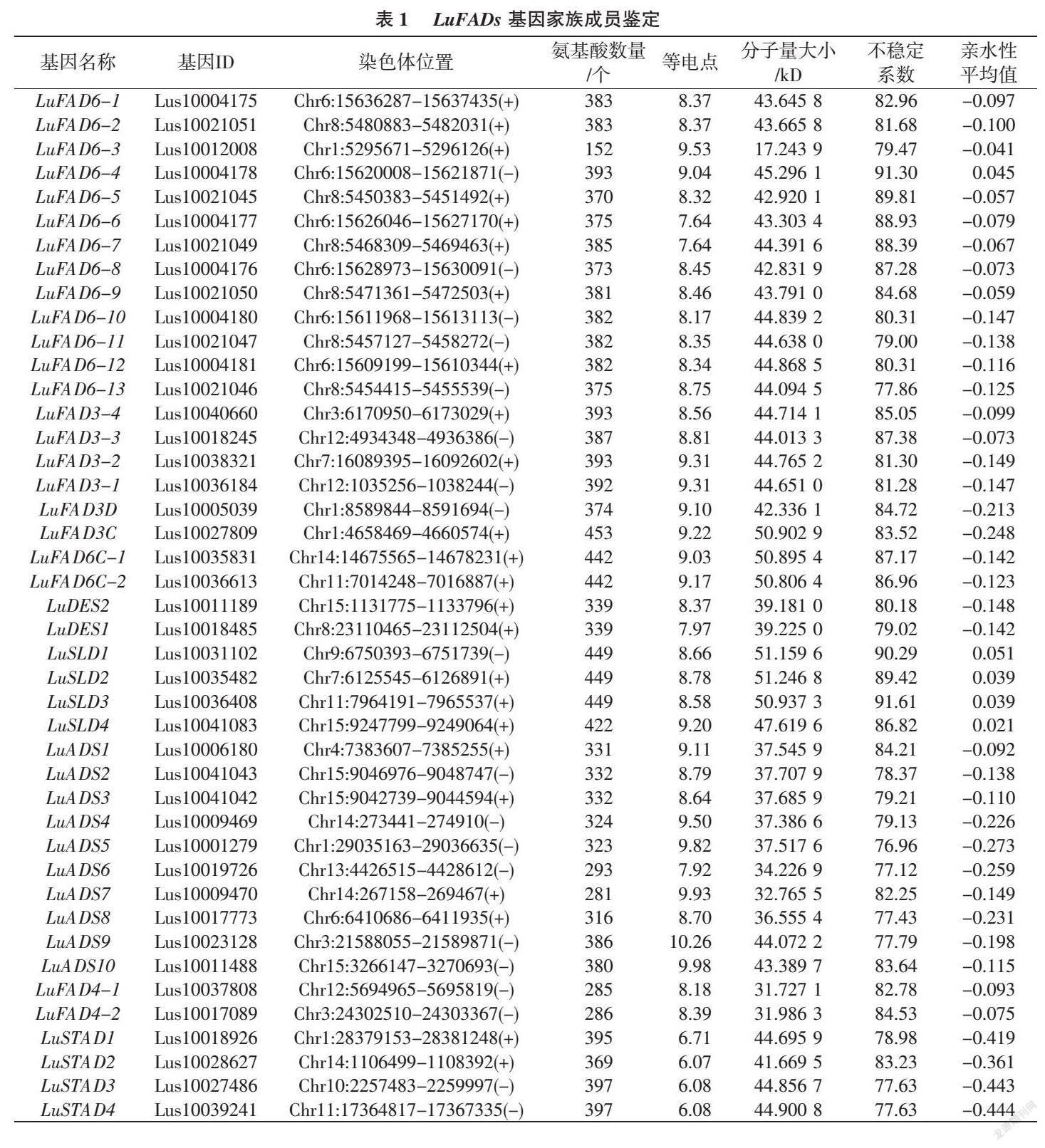
综合2种方法的筛选结果,运用SMART (http://smart.embl.de/)和NCBI-CDD(https://www.ncbi.nlm.nih.gov/cdd/)数据库进一步验证筛选结果中是否具有FAD结构域组成,去除有错误、短小 (<100 aa)和没有FAD结构域的蛋白质序列,最终鉴定了43个LuFADs基因,并依据它们在15条染色体位置信息和系统发育分析结果进行命名(表1、图1)。编码的蛋白质范围为152~453个氨基酸。LuFADs的理论分子量为17.243 9(LuFAD6-3)~51.246 8 kD(LuSLD2),等电点(pI)为6.07~10.26,LuSTAD(1~4)系列的pI < 7.00,呈酸性,其余39个成员pI均大于7,呈碱性。LuFADs基因蛋白的不稳定性指数范围为76.96~91.61,均为不稳定蛋白。亲水性平均值 (GRAVY)为正数的有5个,分别为LuSLD(1~4)系列和LuFAD6-4,表现为不亲水性,其余38个家族成员均为亲水蛋白(表1)。
2.2 LuFADs家族成员系统进化分析
运用邻接法将LuFADs蛋白序列(43)与拟南芥FADs蛋白序列(27)构建系统发育树(图1),可分为4个主要亚族:Δ12/ω-3去饱和酶亚族、“前端”去饱和酶亚族、Δ7/Δ9去饱和酶亚族和SAD去饱和酶亚族。
Δ12/ω-3去饱和酶亚家族包括3个分支:FAD6、FAD3和FAD6C。其中FAD6分支包括拟南芥的AtFAD6和亚麻的LuFAD6(1-13);FAD3分支为膜结合ω-3FAD系列基因,包括拟南芥的AtFAD3、AtFAD3C、AtFAD3D和亚麻的LuFAD3C、LuFAD3D、LuFAD3-1、LuFAD3-2、LuFAD3-3、LuFAD3-4;FAD6C分支包括拟南芥的AtFAD6C和亚麻的LuFAD6C-1、LuFAD6C-2。其余3个亚族中,亚麻基因成员和拟南芥基因成员均表现较高的聚集性,特别是在Δ7/Δ9去饱和酶亚族中,亚麻基因LuADS(1-10)和拟南芥AtADS系列基因分别各自聚为1个分支。
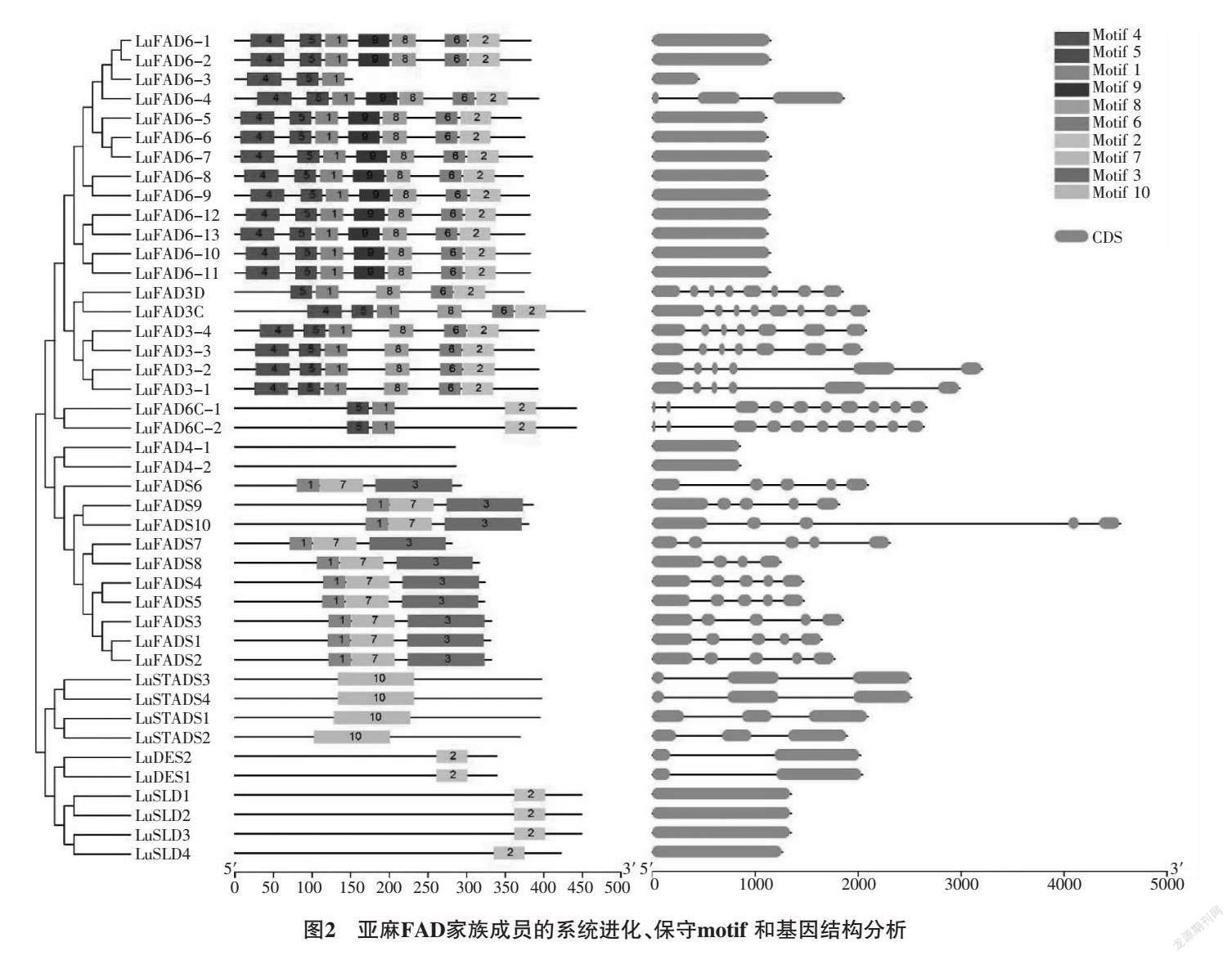
2.3 亚麻FAD家族保守结构域和基因结构分析
对亚麻LuFADs基因家族进行保守结构域分析 (表2,图2)可知,同一亚族中保守结构域的分布较为相似,LuADS亚组中均包含motif-1、motif-3和motif-7;LuSTAD亚组中均只含motif-10;LuDES和LuSLD亚组中含有motif-2;LuFAD6亚组中,除LuFAD6-3外其他基因均含有motif-1、motif-2、motif-4、motif-5、motif-6、motif-8和motif-9;LuFAD4-1和LuFAD4-2不含有motif。
对LuFADs基因家族进行外显子-内含子结构分析(图2)显示,在LuFAD6亚组中,只有LuFAD6-4含有2个内含子,其余12个基因均只有外显子;LuFAD4-1、LuFAD4-2和LuSLD系列没有内含子,剩余基因有不同数量(1~7,9)内含子。LuSLD(1~3)的CDS区最长,大于1 000 bp;LuADS10的基因长度最长,约为4 500 bp,LuFAD6-3基因最短,不足500 bp。
综合家族系统进化可以看出,同一亚组中的基因具有较为相似的结构。LuSTAD亚组中均含有2个内含子。LuADS亚组中,除LuADS8的显子数量为4外,其余均为5。LuFAD6C-1和LuFAD6C-2内含子最多,具有9个。LuADS10的内含子序列最长。
2.4 LuFADs基因家族成员染色体定位和亚细胞预测
由图3可知,LuFADs基因家族随机分布于除第2号和5号染色体(Chr2、Chr5)之外的所有亚麻染色体上,分布广泛。Chr4、Chr9、Chr10和Chr13上各自有1个基因成员,Chr7上有2个基因,Chr3、Chr11和Chr12上各分布着3个基因,Chr14上有4个基因,Chr1和Chr15各为5个基因。Chr6和Chr8的LuFADs家族成员最多,均为7个,其中在Chr6上LuFAD6-1、LuFAD6-4、LuFAD6-6、LuFAD6-8、LuFAD6-10和LuFAD6-12距離较近,在Chr8染色体上LuFAD6-2、LuFAD6-5、LuFAD6-7、LuFAD6-9、LuFAD6-11和LuFAD6-13的距离较为接近。
对LuFADs家族成员进行亚细胞定位预测(表3)可知,定位结果分为4类,包括叶绿体、细胞质、细胞核和线粒体。定位在叶绿体上的家族成员最多,共32个基因。细胞质上7个基因成员,细胞核上3个基因,线粒体上1个基因。其中LuFAD6、LuFAD4和LuSTAD成员的基因均定位在叶绿体上。
2.5 LuFAD启动子顺式作用元件分析
对LuFADs进行启动子顺式作用元件分析 (图4)可知,该基因家族含有的元件可分为4类,光响应元件(G-box、Box 4、GATA-motif、I-box、TCT-motif、Sp1、TCCC-motif、GT1-motif)、激素响应元件(TGACG-motif、CGTCA-motif、ABRE、TGA-element、AuxRR-core、TCA-element、P-box、GARE-motif、TATC-box)、胁迫响应元件(MBS、TC-rich repeats、LTR、ARE、GC-motif)和生长发育相关元件(A-box、O2-site、CCAAT-box)。其中抗氧化反应元件ARE数量最多(89),其次是G-box(77),Box 4(74)排第3,TGACG-motif和CGTCA-motif在基因家族中的含量分布数量相同(72)。基因LuFAD3-3含元件数最多,为35个;LuADS3和LuADS7的元件数最少,均为6个。
3 讨论与结论
近年来,FAD基因家族在多种植物物种中得到鉴定验证,其家族成员数量不尽相同,如甜荞麦中10个[14 ],大豆中29个[21 ],陆地棉中41个[22 ]。本研究分析鉴定了亚麻43个LuFADs基因家族成员,gDNA长度为456~4 547 bp,编码的氨基酸为152~453个。等电点(pI)6.07~10.26。除LuSLD(1~4)系列和LuFAD6-4,其余38个家族成员均为亲水蛋白。有研究者对茄子的FAD家族序列进行理化性质分析,表明分子量与成员所含氨基酸数量成正比,大部分家族成员为亲水性蛋白,这与本研究结果相似[15 ]。系统发育树分析聚为4类,同一亚族中保守结构域的分布较为相似,在Δ7/Δ9去饱和酶亚族中均含motif-1、motif-3和motif-7,在SAD去饱和酶亚族中均只含motif-10;在“前端”去饱和酶亚族中只含motif-2,在Δ12/ω-3去饱和酶亚族中LuFAD6、LuFAD6C、LuFAD3C和LuFAD3D各分支的保守结构域相似。Liu等[23 ]用最大似然法将核桃FADs蛋白序列与拟南芥FADs蛋白序列一起构建系统发育树,分析JrFADs基因家族,也分为4个亚家族,与本研究相一致。
对基因外显子-内含子结构分析表明,同一亚组中的基因具有较为相似的基因结构。Xue 等[10 ]对油菜FAD基因家族进行基因结构分析,各个FAD亚家族的外显子-内含子结构高度保守。Liu等[23 ]分析JrFADs基因家族,结果表明同一亚家族中的基因具有相似的基因结构。Cheng等[24 ]对香蕉同一亚科中的MaFAD家族成员进行分析,显示出相似的内含子/外显子结构和内含子相,并且它们编码的蛋白质由相似的基序组成。
染色体定位和亚细胞预测发现,LuFADs基因家族随机地分布在除Chr2和Chr5之外的所有亚麻染色体上。亚细胞定位在叶绿体上的家族成员最多,共有32个基因。Xue等[10 ] 的研究表明,油菜3个芸薹属物种中FAB2、FAD4、FAD6和FAD8蛋白质的所有成员均位于叶绿体中。启动子顺式作用元件分析得出,抗氧化反应元件ARE的数量最多,为89个,基因LuFAD3-3所含元件数最多。Yasemin等[25 ]对向日葵的FAD基因家族的研究表明,主要定位在叶绿体和内质网膜中,其中FAB2家族成员位于叶绿体中,可能与FAD在细胞不同部位脂肪酸去饱和中的作用有关。综合分析向日葵、芝麻、油菜、可可树和亚麻的FAD基因家族,在氧化还原过程、脂肪酸生物合成过程或脂质代谢过程中发挥作用,这与本研究结果相似。
参考文献:
[1] 伍叶娜,潘 根,姜 慧,等. 亚麻全基因组关联分析研究进展[J]. 中国麻业科学,2022,44(2):109-118.
[2] 张 辉,贾霄云,高凤云,等. 胡麻[M]. 北京:中国农业科学技术出版社,2021.
[3] 廖振林,李倩滢,陈俊杰,等. 亚麻籽油组分的功能活性研究进展[J]. 现代食品科技,2021,37(11):379-389;337.
[4] 唐佳芮,魏 冰,石珊珊. α-亞麻酸分离纯化技术研究进展[J]. 中国油脂,2020,45(8):28-31;38.
[5] LEE K R, CHEN G Q, KIM H U. Current progress towards the metabolic engineering of plant seed oil for hydroxy fatty acids production[J]. Plant Cell Reports, 2015, 34(4):603-15.
[6] POVKHOVA L V, MELNIKOVA N V, ROZHMINA T A, et al. Genes associated with the flax plant type(oil or fiber) identified based on genome and transcriptome sequencing data[J]. Plants, 2021, 10(12): 2616.
[7] CHELLAMUTHU M, KUMARESAN K, SUBRAMANIAN S. Increase in alpha-linolenic acid content by simultaneous expression of fatty acid metabolism genes in Sesame(Sesamum indicum L.)[J]. Physiology and Molecular Biology of Plants, 2022, 28(3): 559-572.
[8] 吴 端,王力军,杨仕梅,等. 植物种子a-亚麻酸形成及调控机理研究进展[J]. 植物遗传资源学报,2020,21(1):49-62.
[9] WANG L H, YU S, TONG C B, et al. Genome sequencing of the high oil crop sesame provides insight into oil biosynthesis[J]. Genome Biology,2014,15(2):39-46.
[10] XUE Y, CHEN B, WANG R, et al. Genome-wide survey and characterization of fatty acid desaturase gene family in brassica napus and its parental species[J]. Applied Biochemistry and Biotechnology, 2018, 184(2): 582-598.
[11] 于海峰,韩平安,李美娜,等. 基于RNA-Seq技术的向日葵油酸形成的转录组学分析[J]. 中国油料作物学报,2018,40(6):769-776.
[12] 江 南,谭晓风,张 琳,等. 基于RNA-Seq的油茶种子α-亚麻酸代谢途径及相关基因分析[J]. 林业科学,2014,50(8):68-75.
[13] 刘 江,王育川,董艳辉,等. 藜麦FAD2基因鉴定及生物信息学分析[J]. 分子植物育种,2022,20(3):715-721.
[14] 赵训超,魏玉磊,丁 冬,等. 甜荞麦脂肪酸脱氢酶基因(FeFAD)家族的鉴定与分析[J]. 东北农业科学,2021,46(1):36-41.
[15] 朱宗文,张爱冬,吴雪霞,等. 生物信息学鉴定分析茄子脂肪酸去饱和酶(FAD)基因家族[J/OL]. 分子植物育种:1-15(2021-05-12)[2022-05-18]. http://kns.cnki.net/kcms/detail/46.1068.S.20210512.0826.002.html
[16] DOMINGUEZ T, HERNANDEZ M L, PENNYCOOKE J C, et al. Increasing ω-3 desaturase expression in tomato results in altered aroma profile and enhanced resistance to cold stress[J]. Plant Physiology, 2010, 153(2):655-665.
[17] YOU F M, LI P C, KUMAR S, et al. Genome-wide identification and characterization of the gene families controlling fatty acid biosynthesis in flax(Linum usitatissimum L.)[J]. Journal of Proteomics & Bioinformatics, 2014, 7(10):310.
[18] YOU F M, XIAO J, Li P, et al. Chromosome-scale pseudomolecules refined by optical, physical and genetic maps in flax[J]. Plant Journal, 2018, 95(2): 371-384.
[19] 劉畅畅,黄 敏. 玉米LEC1基因家族的鉴定与生物信息学分析[J]. 甘肃农业科技,2022,53(1):31-37.
[20] 张国琴,葛玉彬,张正英. 高粱耐冷基因全基因组鉴定及进化分析[J]. 甘肃农业科技,2020(12):37-41.
[21] CHI X Y, YANG Q L, LU Y D, et al. Genome-wide analysis of fatty acid desaturases in soybean(Glycine max)[J]. Plant Molecular Biology Reporter, 2011, 29(4): 769-783.
[22] FENG J, DONG Y, LIU W, et al. Genome-wide identification of membrane-bound fatty acid desaturase genes in Gossypium hirsutum and their expressions during abiotic stress[J]. Sci Rep, 2017, 1(7):1-12.
[23] LIU K, ZHAO S, WANG S, et al. Identification and analysis of the FAD gene family in walnuts(Juglans regia L.) based on transcriptome data[J]. BMC Genomics, 2020, 21(1):299-303.
[24] CHENG C, LIU F, SUN X, et al. Genome-wide identification of FAD gene family and their contributions to the temperature stresses and mutualistic and parasitic fungi colonization responses in banana[J]. International Journal of Biological Macromolecules, 2022, 204: 661-676.
[25] YASEMIN C A, NECDET M U, MEHMET C B, et al. Comparative identification and evolutionary relationship of fatty acid desaturase(FAD)genes in some oil crops: the sunflower model for evaluation of gene expression pattern under drought stress[J]. Biotechnology & Biotechnological Equipment, 2018, 32(4): 846-857.
