基于表型性状和SSR分子标记构建甘薯核心种质
2023-03-23陈伊航唐朝臣张雄坚姚祝芳江炳志王章英
陈伊航 唐朝臣 张雄坚 姚祝芳 江炳志 王章英,*
基于表型性状和SSR分子标记构建甘薯核心种质
陈伊航1,2唐朝臣1张雄坚1姚祝芳1江炳志1王章英1,*
1广东省农业科学院作物研究所/ 广东省农作物遗传改良重点实验室, 广东广州 510640;2西北农林科技大学草业与草原学院, 陕西杨凌 712100
为更好地保存、研究和利用甘薯种质资源, 本研究以国家甘薯种质资源圃(广州)保存的1091份甘薯种质为材料, 分别采用欧氏距离和Nei’s距离进行NJ聚类分组, 组内随机取样, 构建核心种质。利用均值、方差、香农多样性指数、变异系数等指标对核心种质的表型性状数据进行代表性评价, 以及利用有效等位基因、Nei’s遗传多样性指数、Shannon’s多样性指数等指标对核心种质的SSR分子标记数据进行代表性评价; 并利用主成分分析对核心种质进行确认。结果表明, 构建的甘薯核心种质包含289份材料, 占全部种质的26.49%; 在<0.05概率下, 核心种质中表型性状以及SSR分子标记的相关指标与全部种质无显著差异, 且二者的表型频率分布基本一致; 主成分分析表明核心种质具有与全部种质相似的遗传多样性和群体结构。建立的甘薯核心种质很好地代表了全部种质的遗传变异和群体结构, 可为甘薯的品种改良、优良基因挖掘以及种质创新奠定良好基础。
甘薯; 核心种质; 表型性状; SSR分子标记; 种质资源
甘薯((L.) Lam.)是旋花科甘薯属的一年生或多年生的双子叶草本植物, 起源于美洲, 于16世纪末从福建和广东传入中国[1]。随着时间的推移, 自然杂交和选择导致了本地不同种类甘薯的进化, 使得国内保留了大量的基因库[2]。随着种质收集工作的开展, 大量的甘薯种质资源得以保存。由于甘薯为无性繁殖作物, 为了保存种质, 每年都需要对甘薯进行繁苗和更新。甘薯种质数量日益增多, 给种质资源的整理保存、鉴定评价以及研究利用等工作带来了巨大的挑战。
Frankel和Brown[3]于1984年提出了核心种质(core collection)概念, 即采用一定的方法选择整个种质资源的部分种质, 以最小的数量和遗传重复来代表整个种质资源。水稻[4]、小麦[5]、玉米[6]、芝麻[7]等多种作物均已构建核心种质。目前已有对甘薯种质资源遗传多样性的相关研究[8-10], 并已经对甘薯种质资源的SSR分子标记进行鉴定与开发[11], 建立了甘薯相关DNA指纹图谱[12]。甘薯核心种质的构建尚无报道, 需要构建核心种质以改善甘薯种质资源保存与利用的现状。
近年来, 国内外学者构建核心种质主要基于表型性状和SSR分子标记2个方面或其中之一[13]。基于表型性状构建核心种质有郑福顺等[14]对480份番茄的20个表型性状进行遗传多样性分析, 构建核心种质; 汪磊等[15]对422份向日葵的11个表型数据进行分析, 成功构建核心种质; 侯志强等[16]基于250份菊芋的19个表型性状成功构建核心种质。基于SSR分子标记构建核心种质如徐超华等[17]利用15对SSR分子标记成功构建割手密核心种质; 张馨方等[18]用21对SSR分子标记对161份板栗进行分析, 成功构建核心种质。在甜荞[19]、苎麻[20]、云南松[21]、紫苏[22]等物种中有同时基于表型性状和SSR分子标记构建核心种质的报道。目前, 基于表型性状和SSR分子标记其中之一构建核心种质较多, 同时基于二者构建核心种质较少。
本研究从国家甘薯种质资源圃(广州)选取1091份甘薯种质资源为材料, 以20个表型性状数据为基础, 结合10对多态性较好的SSR分子标记对其进行遗传多样性分析, 构建甘薯核心种质, 并对其代表性进行评价, 最终确定核心种质, 旨在为甘薯种质资源的保护、优异种质挖掘及品种的遗传改良提供理论依据。
1 材料与方法
1.1 试验材料
1.2 数据采集
1.2.1 表型数据采集 参照《甘薯种质资源描述规范和数据标准》[23]进行质量性状调查, 其中地上性状调查于栽插后40~50 d进行, 地下性状调查于栽插后130~140 d进行。调查的质量性状及其相关指标见表1。采用田间观测法进行表型数据采集, 用柯尼卡美能达CM-700d/600d分光测色计采集顶叶色, 采用紫光(UNIS) Uniscan M1快速平板扫描仪采集叶色和薯肉主色。

表1 甘薯20个质量性状赋值

(续表1)
1.2.2 DNA提取及毛细管电泳 于栽插后45 d采集1091份资源的叶片样品, 应用CTAB法提取样本的基因组DNA。引物利用课题组开发的SSR引物序列GDAAS0911、GDAAS0819、GDAAS0922、GDAAS0782、GDAAS0338、GDAAS0940、GDAAS0871和GDAAS0694[12]以及Meng等[24]开发的SPGS2和SPGS3。
SSR-PCR反应体系共20 μL, 包含ddH2O 14.8 μL、dNTP 0.4 μL、buffer 2 μL、正反向引物各0.3 μL、DNA模板2 μL、DNA连接酶0.2 μL。SSR-PCR扩增程序: 94℃预变性5 min; 94℃变性30S, 54℃复性35 s, 72℃延伸40S, 共35个循环; 最终72℃延伸3 min。
毛细管电泳方法: 将甲酰胺与分子量内标按100︰1的体积比混匀后, 取15 μL加入上样板中, 再加入1 μL稀释10倍的PCR产物, 然后使用3730XL测序仪进行毛细管电泳。
1.3 取样方法
以样本数量大, 取样比例小, 样本数量小, 取样比例大为原则, 从全部种质中抽取核心种质。其中, 每一组抽取样本应在该组中均匀分布, 且确保每一组中的各个小组均能抽取样本; 特殊样本或者极端样本采取全部保留的策略, 以确保抽取的核心种质的代表性。具体抽样比例见表2。
1.4 数据分析
利用Genemarker中的Fragment (Plant)片段分析软件对测序仪得到的原始数据进行分析, 将各泳道内分子量内标的位置与各样品峰值的位置作比较分析, 采用0/1赋值法, 在相同的迁移位置上有扩增条带记为1, 无扩增条带记为0, 构建0、1二元数据矩阵。
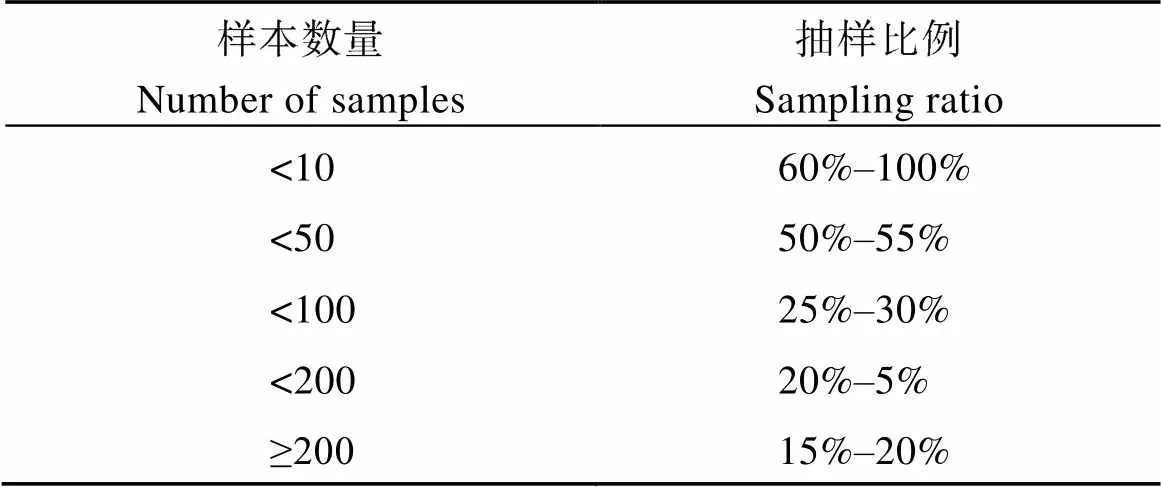
表2 核心种质抽样
采用ArcMap 10.7软件绘制来源地分布图。采用SPSS 26软件统计20个表型性状的平均值、方差和极差, 并通过单因素ANOVA检验对表型性状进行检验和检验; 利用Microsoft Excel 2019计算变异系数(CV)和Shannon’s多样性指数() [式(1)~(2)]。
现代有轨电车为依靠司机瞭望驾驶,采用沿轨道行驶的电力牵引的低地板有轨电车车辆,并按地面公交模式组织运营的公共交通系统[1]。由于现代有轨电车在城市道路上行驶,车辆依靠司机瞭望运行,其运营组织形式更加灵活多样,因此有轨电车车站配线设计不但要满足运营功能的需求,更要结合道路条件进行设置,同时应充分发挥有轨电车网络化运营特征,满足网络的灵活调度管理需求。
利用SPSS 26软件计算表型性状数据各个种质间的欧氏距离, 并将欧氏距离矩阵导入MEGA 11软件得到表型性状NJ (Neighbor-Joining)聚类结果, 并绘制聚类图; 利用NTSYSpc 2.10软件对SSR分子标记数据进行处理, 得到SSR分子标记的Nei’s距离矩阵, 将Nei’s距离矩阵导入MEGA 11软件得到表型性状NJ (Neighbor-Joining)聚类结果, 并绘制聚类图。SSR分子标记的平均等位基因数()、平均有效等位基因数()、Nei’s遗传多样性指数()、Shannon’s多样性指数()等遗传多样性指标均通过软件POPGENE32得到。利用Origin 2021软件绘制20个表型性状的频率分布图以及表型性状和SSR分子标记的PCA图。


式中,P表示某性状第级的分布频率,为总分级数。
2 结果与分析
2.1 种质资源分析
2.1.1 来源地分析 在保存的全部甘薯种质中, 国内种质共有914份, 占比83.79%, 在24个省(自治区)均有分布。其中来自广东省数量最多, 占比50.87%, 其次是福建、海南、江苏、广西4个省(自治区), 台湾、山东、四川等省份数量较少。国外引进品种来自12个国家, 共有177份材料, 占比16.21% (图1)。
2.1.2 基于表型性状的聚类分析 基于20个表型性状对全部种质进行聚类分析, 以欧氏距离为6.51作为分组依据, 将全部种质1091份材料分为10组, 分别标记为Group 1~Group 10 (图2-A)。Group 1有230份材料, 占比21.08%, 主要表现为脉基色和柄基色为绿色; Group 2有24份材料, 占比2.20%, 主要表现为顶叶形状为缺刻, 顶芽色为褐色, 叶侧脉色为浅绿, 脉基色为绿色; Group 3有16份材料, 占比1.47%, 主要表现为顶叶形状和叶片形状为心形, 叶侧脉色为绿色; Group 4有57份材料, 占比5.22%, 主要表现为顶芽色为紫色, 顶叶形状和叶片形状为心形, 薯皮主色为黄色; Group 5有189份材料, 占比17.32%, 主要表现为顶叶形状为心形, 叶片形状为缺刻, 柄基色、茎色和茎主色为紫色, 薯皮主色为红色; Group 6有42份材料, 占比3.85%, 主要表现为顶叶形状和叶片形状为心形, 无茎次色; Group 7有23份材料, 占比2.11%, 主要表现为叶主脉色为紫斑, 叶侧脉色为绿色, 茎端茸毛多; Group 8有96份材料, 占比8.80%, 主要表现为脉基色、柄基色和茎次色为紫色, 无茎端茸毛, 薯肉主色为淡黄; Group 9有1份材料, 占比0.09%; Group 10有413份材料, 占比37.86%, 主要表现为顶叶形状和叶片形状为缺刻, 薯皮主色为黄色。
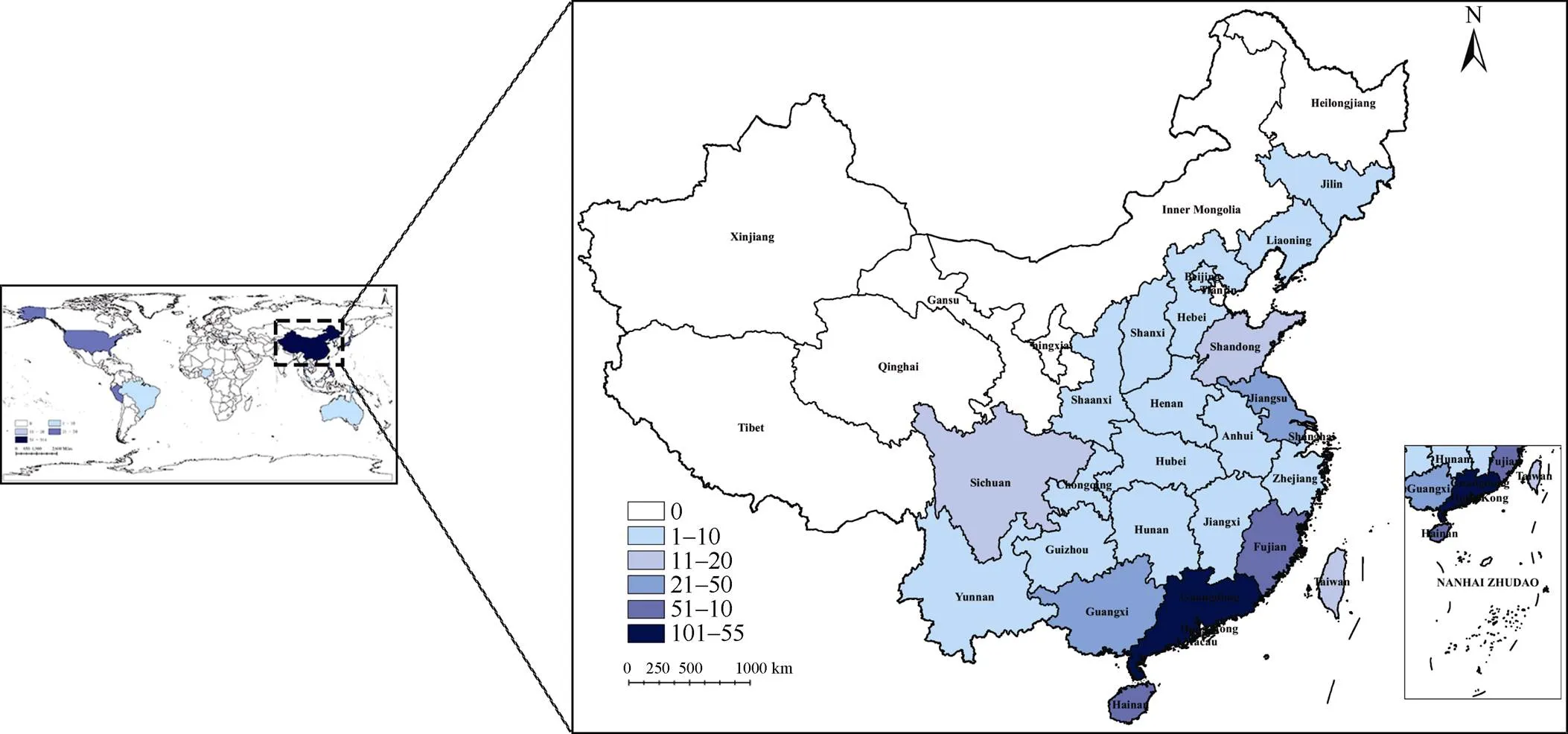
图1 全部种质资源来源地信息
2.1.3 基于SSR分子标记的聚类分析 基于SSR分子标记对全部种质进行聚类分析, 以Nei’s距离为0.54作为分组依据, 将全部种质共1091份材料分为10组, 分别标记为Group 1~Group 10 (图2-B)。Group 1~Group 10的样本数量依次为: 639 (58.57%)、90 (8.25%)、200 (18.23%)、22 (2.02%)、42 (3.85%)、37 (3.39%)、4 (0.37%)、19 (1.74%)、4 (0.37%)、34 (3.12%)。部分SSR分子标记毛细管电泳结果见附图1。
2.2 核心种质构建
基于聚类分析结果, 以表型性状和SSR分子标记将全部种质分为12组, 并根据分组结果对全部种质进行抽样(表3)。结合抽取样本的结果, 将2组样本共有的品种只保留1份, 不是共有样本的品种全部保留, 最终得到289份核心种质, 占全部种质26.49%。289份核心种质的聚类结果见附图2。

图2 基于表型性状和SSR分子标记的全部种质聚类图
A: 基于表型性状; B: 基于SSR分子标记。
A: cluster dendrogram based on phenotype traits; B: cluster dendrogram based on SSR molecular markers.
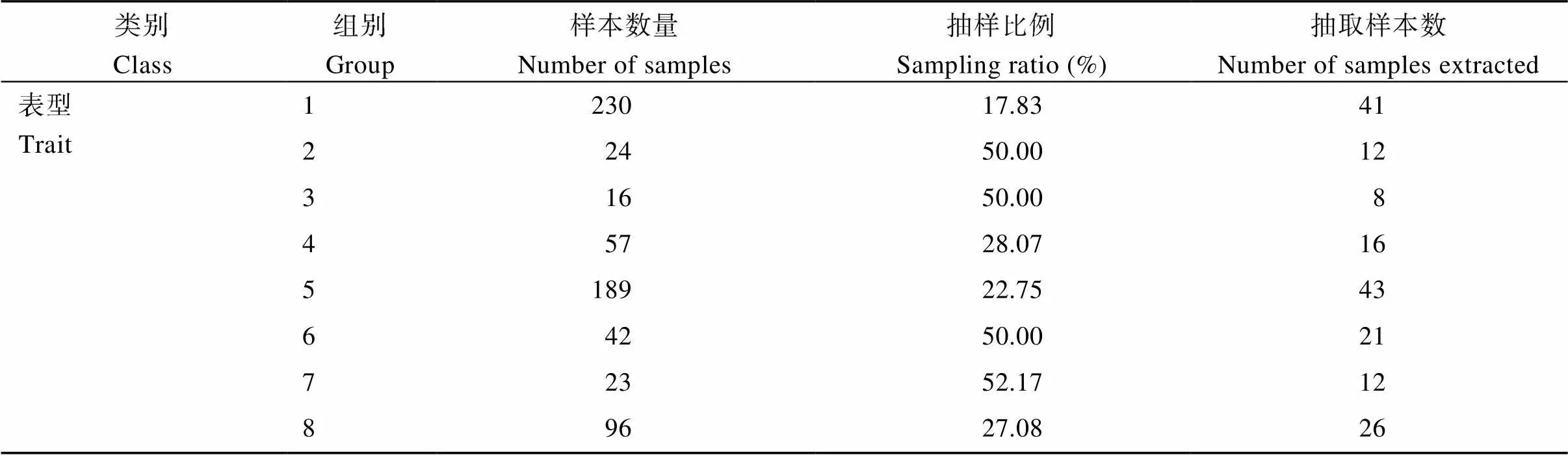
表3 基于表型性状和SSR分子标记抽样结果
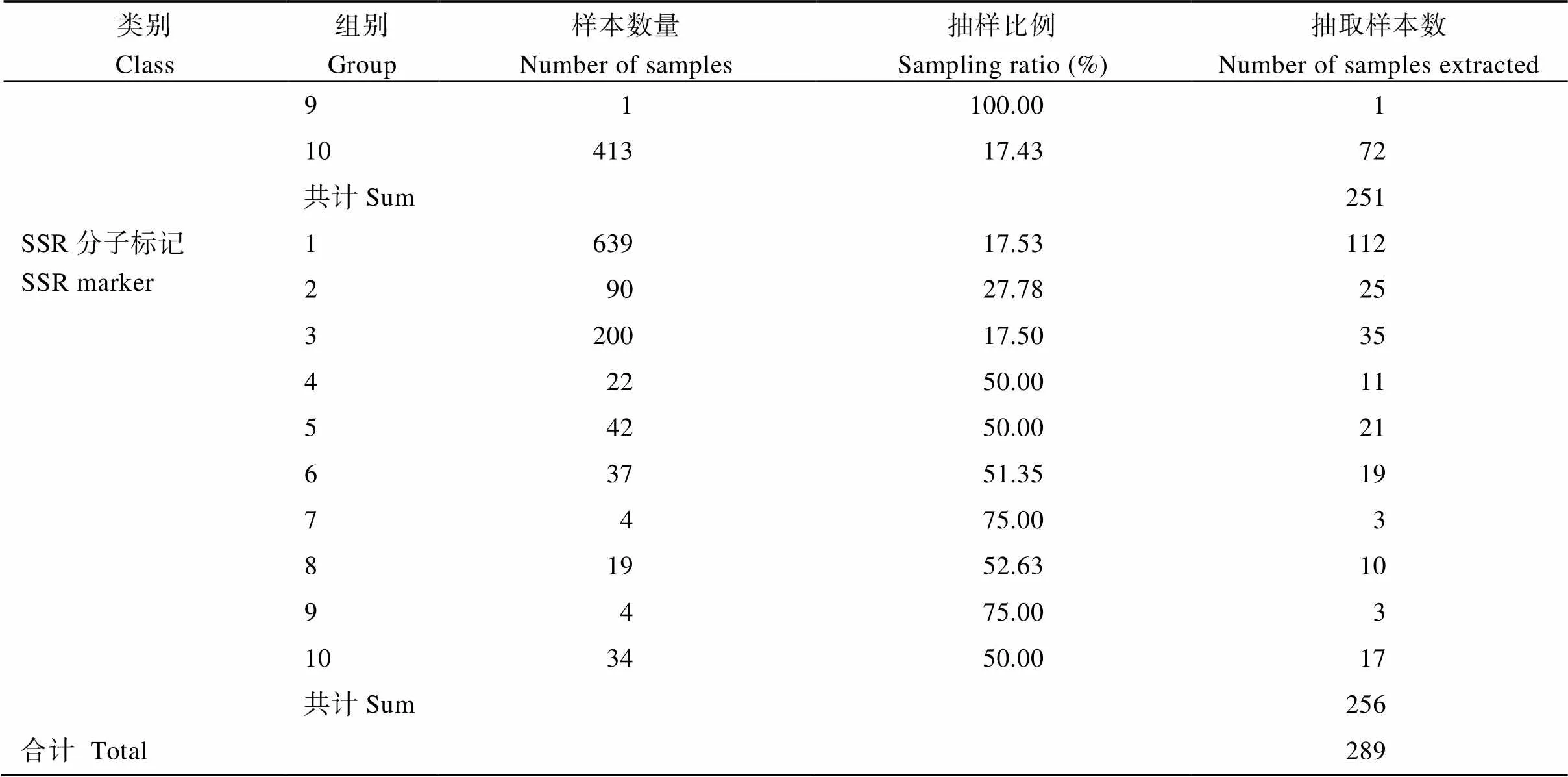
(续表3)
2.3 核心种质代表性评价
2.3.1 基于表型数据的核心种质代表性检验 单因素ANOVA检验表明, 核心种质共有18个表型性状与全部种质无显著性差异。此外, 核心种质中多数性状的方差高于全部种质, 表明核心种质中的遗传冗余度明显减小, 变异率更高。极差分析表明, 全部种质有17个表型性状的变异范围100%保留在核心种质中, 其余3个性状的保留比例分别为: 顶叶色88.89%、顶叶形状80.00%和叶色83.33% (表4)。由此可见, 核心种质对全部种质的性状的变异幅度具有良好的代表性。从20个表型性状的频率分布图(图3)可以看出, 所有性状在全部种质和核心种质中的分布较为吻合, 说明核心种质很好地保留了全部种质的遗传多样性和群体结构。
Shannon’s多样性指数和变异指数常用来比较不同样品的表型特征、等位基因的丰富度和均匀度。全部种质与核心种质20个性状的和变异系数是非常相似的, 说明核心种质的样本有效地去除了全部种质中的遗传冗余, 保留了全部种质的遗传多样性, 变异均匀度显著提高(表4)。
2.3.2 基于SSR分子标记的核心种质代表性评价 通过对比2个种质SSR分子标记的等位基因数、平均有效等位基因数、Nei’s遗传多样性指数、Shannon’s多样性指数以及多态性位点百分率, 比较核心种质与全部种质的遗传多样性。全部种质的平均等位基因数、平均有效等位基因数和多态性位点百分率分别为1.88、1.25和88.15%, 核心种质分别为1.92、1.32和92.08%, 稍高于全部种质。全部种质的Nei’s遗传多样性指数和Shannon’s多样性指数分别为0.15和0.24, 核心种质分别为0.19和0.30, 分别对2个指标进行检验, 均为差异不显著(表5), 说明核心种质与全部种质的遗传多样性水平十分相似。
2.4 核心种质的确定
利用主成分分析对构建的核心种质进行确认, 从全部种质和核心种质的主成分分布图可知, 核心种质与全部种质基于表型性状的样本主要呈上下分布, 且核心种质的样本较为均匀地分布在全部种质的样本中; 基于SSR分子标记的样本分布相对集中, 但不难看出核心种质的样本在全部种质样本中呈均匀分布(图4)。因此, 核心种质很好地保留了全部种质的遗传多样性和群体结构, 确保了核心种质的有效性和代表性。
3 讨论
作物种质资源是国家的宝贵财富, 是作物遗传和育种研究的基础。本研究利用1091份甘薯种质材料的20个表型性状以及10组多态性较好的SSR分子标记数据构建甘薯核心种质, 采用分组取样, 取样比例为26.49%, 最终获选核心种质材料289份。
3.1 取样策略对核心种质构建的影响
构建代表性强的核心种质, 采用的取样方法很关键。若想维持原始种质的遗传多样性形式, 随机取样不失为一种比较合适的取样策略[25], 但随机取样并不能考虑到原始种质中的特殊、极端材料和稀有表型材料。通过系统聚类对原始种质进行分组, 相同或者相似的种质能够聚在一起。针对其中的极值材料、特殊材料, 采用优先取样法[26]将极值材料以及特殊材料优先抽出, 剩下的较为均匀的种质采用随机取样法进行取样, 不仅可以维持原种质的遗传多样性形式, 还能最大限度地保留原种质的遗传变异。原始种质的分组通常会根据种源地信息、表型性状和分子标记等进行聚类分组, Katinas等[27]认为如果仅以种源地理信息为依据进行分组, 可能不能达到预期效果, 同时结合表型或者遗传信息进行聚类分组, 构建核心种质更具有代表性。本研究分别以表型性状和SSR分子标记数据对原始种质分组,最终分组结果表明相似性较高的种质均聚集在同一组中, 特殊材料能够单独成组, 并能明显分出极值材料。

图3 全部种质与核心种质20个表型性状频率分布图
缩写同表1。图中黑色表示核心种质的频率分布, 浅灰色表示全部种质的频率分布。
Abbreviations are the same as those given in Table 1. In this figure, the black and light gray represent the frequency distribution of the core collection and the frequency distribution of the entire collection, respectively.


表5 全部种质和核心种质遗传多样性比较
图4 基于表型性状以及SSR分子标记的全部种质和核心种质主成分分布图
Fig. 4 Principal component plots for the entire collection and core collection based on phenotype traits and SSR molecular markers
A: 基于表型性状; B: 基于SSR分子标记。在该图中, 红色表示核心种质的频率分布, 浅灰色表示全部种质的频率分布。
A: principal component plot based on phenotype traits; B: principal component plot based on SSR markers. In this figure, the red and light gray represent the frequency distribution of the core collection and the frequency distribution of the entire collection, respectively.
构建的核心种质最终的种质库规模与采用的取样比例有关。原始种质数量较大且遗传多样性较高, 需采用较高的取样比例, 反之采用较低的取样比例; 如果表型鉴定性状较少, 不能充分展现遗传变异, 应适当提高取样比例[16]。国内外不同作物构建的核心种质的取样比例基本为5%~30%[28], 一般在15%左右。崔竣杰等[29]采用18.48%的取样比例构建苦瓜核心种质; Kumar等[30]构建水稻核心种质, 原始种质为3004份, 核心种质为520份, 取样比例约为17.31%。本研究从1091份甘薯材料中共选取了289份核心种质, 取样比例约26.49%。本研究基于前人构建核心种质的经验, 将方法初次利用在甘薯上, 构建的核心种质取样比例较大, 但仍处在5%~30%这一比例范围内, 说明核心种质的构建是成功的。如果要达到核心种质的规模, 还需要在核心种质的基础上进一步筛选, 在确保维持原始种质遗传多样性形式和保存原始种质的遗传变异的基础上, 缩减取样比例, 构建规模更小的核心种质。
3.2 核心种质的评价
构建核心种质的数据来源主要有表型性状和分子标记两大类。表型性状具有直观、简易的优点, 但容易受到外界环境的干扰。DNA分子标记技术具有高效、稳定的特点, 相比于表型性状能更真实地反映材料之间的差异和亲缘关系, 却不能够直观地体现材料之间的表型差异。在不同类型的分子标记中, SSR分子标记具有多等位基因、共显性和高度多态性等优点[31], 能快速鉴别物种之间的亲缘关系。结合表型性状与分子标记的数据构建核心种质, 既能直观地反映材料之间的表型差异, 又能够使结果更加真实、可靠以及更具有代表性。对核心种质的评价就是检验其代表性和有效性, 主要包括表型性状数据和分子标记数据2个方面。表型性状数据的评价主要包括遗传多样性指数、变异系数、表型方差、表型分布频率等[28,32]; 分子标记数据的评价主要包括有效等位基因数、Nei’s遗传多样性指数、Shannon’s多样性指数等[7,17-18,33]。最终, 本研究选取遗传多样性指数、变异系数、平均值、方差、极差作为表型性状指标和有效等位基因数、Nei’s遗传多样性指数、Shannon’s多样性指数作为SSR分子标记指标评价甘薯核心种质的代表性。通过检验和检验对核心种质与全部种质比较表明, 除了叶尖形状和茎叶生长势2个性状之外, 多数性状的均值、方差、香农指数、变异系数等均无显著差异。相比其他的表型性状, 叶尖形状以及茎叶生长势均只有3个赋值指标, 其余的表型性状大多有6个赋值指标,最多的能有10个赋值指标, 使得全部种质中叶尖形状和茎叶生长势的比例分布较集中, 而构建核心种质的取样比例较小, 导致核心种质中的叶尖形状和茎叶生长势的分布情况与全部种质出现差异。如果在原来3个指标的基础上, 新增加1~2个指标对这2个性状进行赋值, 这一现象应当会得到改善。
3.3 甘薯核心种质的不足与展望
本研究从表型性状以及SSR分子标记2个方面对构建的甘薯核心种质进行代表性评价, 结果表明构建的核心种质代表性良好。甘薯种质应当从生物学特性、植物学特征、品质性状和抗性等多个维度进行评价[34]。本研究选取的表型性状主要为植物学特征, 并未从生物学特性、品质性状以及抗性等其他维度对甘薯种质进行鉴评。甘薯核心种质包含了全部种质的绝大多数表型性状以及基因型, 利用该种质对甘薯中重要的表型性状进行鉴定评价, 比利用部分随机或特殊种质更具有代表性。基因型鉴定是充分认识和利用种质资源的重要基础, 利用重测序对全基因组进行基因型鉴定是种质资源的主流技术[35]。Oh等[36]通过全基因组重测序对黑稻中的花青素相关基因进行了鉴定; Han等[37]通过全基因组重测序在棉花中鉴定出9个FOV7抗性位点; Yamakawa等[38]利用全基因组重测序技术开发与马铃薯和甘薯育种紧密相关的DNA标记。在前人的基础上, 可将核心种质用于甘薯种质资源的研究中, 挖掘甘薯种质资源中的重要性状和关键基因、深入了解甘薯种质资源的遗传背景、扩大育种中亲本选择的范围以提高先进品种的遗传多样性、提高特殊种质以及野生近缘种的利用率。
4 结论
本研究基于表型性状和SSR分子标记数据, 分别采用欧氏距离和Nei’s距离进行NJ聚类分组, 组内随机取样, 构建了289份甘薯核心种质, 占全部种质的26.49%。核心种质表型性状数据的均值、方差、变异系数、香农指数等与全部种质无显著差异, 表型频率分布与全部种质一致; SSR分子标记数据的Nei’s遗传多样性指数、Shannon’s多样性指数等与全部种质无显著差异。最后通过主成分分析进一步确认了核心种质与全部种质的群体结构相似性。最终得到的甘薯核心种质与全部种质之间的差异不显著, 具有较好的代表性。
[1] 陆漱韵, 刘庆昌, 李惟基. 甘薯育种学. 北京: 中国农业出版社, 1998. pp 19–27.
Lu S Y, Liu Q C, Li W J. Sweetpotato Breeding. Beijing: China Agriculture Press, 1998. pp 19–27 (in Chinese).
[2] 房伯平, 张雄坚, 陈景益, 安康. 我国甘薯种质资源研究的历史与现状. 广东农业科学, 2004, (增刊1): 3–5.
Fang B P, Zhang X J, Chen J Y, An K. The history and status of sweetpotato germplasm research in China., 2004, (S1): 3–5 (in Chinese with English abstract).
[3] Frankel O H, Brown A H D. Plant genetic resources today: a critical appraisal. In: Holden J H W, Williams J T, eds. Crop Genetic Resources: Conservation and Evaluation. London: George Allen and Unwin, 1984. pp 249–257.
[4] 张洪亮, 李自超, 曹永生, 裘宗恩, 余萍, 王象坤. 表型水平上检验水稻核心种质的参数比较. 作物学报, 2003, 29: 252–257.
Zhang H L, Li Z C, Cao Y S, Qiu Z E, Yu P, Wang X K. Comparison of parameters for testing the rice core collection in phenotype., 2003, 29: 252–257 (in Chinese with English abstract).
[5] Hao C Y, Zhang X Y, Wang L F, Dong Y S, Shang X W, Jia J Z. Genetic diversity and core collection evaluations in common wheat germplasm from the northwestern spring wheat region in China., 2006, 17: 69–77.
[6] 姚启伦, 方平, 杨克诚, 潘光堂. 基于SSR标记构建西南玉米地方品种核心种质的方法. 湖南农业大学学报(自然科学版), 2009, 35(3): 225–228.
Yao Q L, Fang P, Yang K C, Pan G T. Methods of constructing a core collection of maize landraces in southwest China based on SSR data.(Nat Sci), 2009, 35(3): 225–228 (in Chinese with English abstract).
[7] 刘艳阳, 梅鸿献, 杜振伟, 武轲, 郑永战, 崔向华, 郑磊. 基于表型和SSR分子标记构建芝麻核心种质. 中国农业科学, 2017, 50: 2433–2441.
Liu Y Y, Mei H X, Du Z W, Wu K, Zheng Y Z, Cui X H, Zheng L. Construction of core collection of sesame based on phenotype and molecular markers., 2017, 50: 2433–2441 (in Chinese with English abstract).
[8] 刘中华, 林志坚, 李华伟, 许泳清, 李国良, 邱永祥, 邱思鑫, 汤浩. 甘薯种质资源遗传多样性的ISSR分析. 南方农业学报, 2019, 50: 2392–2400.
Liu Z H, Lin Z J, Li H W, Xu Y Q, Li G L, Qiu Y X, Qiu S X, Tang H. Genetic diversity analysis of sweetpotato [(L.) Lam.] by ISSR molecular markers., 2019, 50: 2392–2400 (in Chinese with English abstract).
[9] 李慧峰, 陈天渊, 黄咏梅, 吴翠荣, 李彦青, 滑金锋, 范继征. 甘薯种质资源形态标记遗传多样性分析. 西南农业学报, 2015, 28: 2401–2407.
Li H F, Chen T Y, Huang Y M, Wu C R, Li Y Q, Hua J F, Fan J Z. Genetic diversity of sweetpotato germplasm resources revealed by morphological traits., 2015, 28: 2401–2407 (in Chinese with English abstract).
[10] 易燚波, 岑亮, 郭小路, 张启堂, 傅玉凡, 唐云明. 甘薯种质资源的遗传多样性分析. 西南大学学报(自然科学版), 2008, 30(4): 156–162.
Yi Y B, Cen L, Guo X L, Zhang Q T, Fu Y F, Tang Y M. Genetic diversity of sweetpotato germplasm resources.(Nat Sci), 2008, 30(4): 156–162 (in Chinese with English abstract).
[11] Wang Z Y, Li J, Luo Z X, Huang L F, Chen X L, Fang B P, Li Y J, Chen J Y, Zhang X J. Characterization and development of EST-derived SSR markers in cultivated sweetpotato ()., 2011, 11: 139–139.
[12] 罗忠霞, 房伯平, 李茹, 王章英, 黄立飞, 陈景益, 张雄坚, 李育军, 陈新亮, 黄实辉. 基于EST-SSR标记的甘薯种质资源DNA指纹图谱构建. 植物遗传资源学报, 2014, 15: 810–814.
Luo Z X, Fang B P, Li R, Wang Z Y, Huang L F, Chen J Y, Zhang X J, Li Y J, Chen X L, Huang S H. Construction of DNA fingerprint database based on EST-SSR markers for sweetpotato germplasm., 2014, 15: 810–814 (in Chinese with English abstract).
[13] 王建成, 胡晋, 黄歆贤, 徐盛春. 植物遗传资源核心种质新概念与应用进展. 种子, 2008, 27(5): 47–50.
Wang J C, Hu J, Huang X X, Xu S C. New concept and application on core collection of plant germplasm resources., 2008, 27(5): 47–50 (in Chinese with English abstract).
[14] 郑福顺, 王晓敏, 李国花, 李洪磊, 周鹏泽, 王林, 白圣懿, 刘珮君, 张雪艳, 胡新华, 付金军, 高艳明, 李建设. 基于表型性状的宁夏番茄种质资源核心种质构建. 浙江大学学报(农业与生命科学版), 2021, 47: 171–181.
Zheng F S, Wang X M, Li G H, Li H L, Zhou P Z, Wang L, Bai S Y, Liu P J, Zhang X Y, Hu X H, Fu J J, Gao Y M, Li J S. Core collection construction of Ningxia tomato germplasm resources based on phenotypic traits.(Agric Life Sci), 2021, 47: 171–181 (in Chinese with English abstract).
[15] 汪磊, 王姣梅, 汪魏, 王玲, 王力军, 严兴初, 谭美莲. 基于表型多样性构建向日葵核心种质. 中国油料作物学报, 2021, 43: 1052–1060.
Wang L, Wang J M, Wang W, Wang L, Wang L J, Yan X C, Tan M L. Development of a core collection in sunflower (L.) germplasm using phenotypic diversity., 2021, 43: 1052–1060 (in Chinese with English abstract).
[16] 侯志强, 王丽慧, 赵孟良, 杨世鹏, 孙雪梅, 高洁铭, 钟启文. 基于表型数据的菊芋核心种质初步构建. 分子植物育种, 2021, 19: 3463–3472.
Hou Z Q, Wang L H, Zhao M L, Yang S P, Sun X M, Gao J M, Zhong Q W. Preliminary construction of core collection of Jerusalem artichoke based on phenotypic data., 2021, 19: 3463–3472 (in Chinese with English abstract).
[17] 徐超华, 刘新龙, 毛钧, 刘洪博, 林秀琴, 陆鑫, 苏火生. 基于SSR分子标记数据构建割手密核心种质库. 湖南农业大学学报(自然科学版), 2020, 46: 657–663.
Xu C H, Liu X L, Mao J, Liu H B, Lin X Q, Lu X, Su H S. Construction of a core-collection ofbased on SSR molecular markers.(Nat Sci), 2020, 46: 657–663 (in Chinese with English abstract).
[18] 张馨方, 张树航, 李颖, 郭燕, 王广鹏. 基于SSR标记构建燕山板栗核心种质. 华北农学报, 2021, 36(增刊1): 31–38.
Zhang X F, Zhang S H, Li Y, Guo Y, Wang G P. Construction of core collection of Yanshan chestnut germplasm based on SSR markers., 2021, 36(S1): 31–38 (in Chinese with English abstract).
[19] 李金龙, 范昱, 赵梦雨, 康珍, 杨克理, 张凯旋, 周美亮. 基于表型性状和SSR分子标记构建甜荞初级核心种质. 植物遗传资源学报, 2021, 22: 1240–1247.
Li J L, Fan Y, Zhao M Y, Kang Z, Yang K L, Zhang K X, Zhou M L. Construction of primary core collection of buckwheat germplasm resources based on Phenotypic traits and SSR., 2021, 22: 1240–1247 (in Chinese with English abstract).
[20] Luan M B, Chen Y M, Wang X F, Xu Y, Sun Z M, Chen J H, Wang J S. Core collection of ramie comprising 1151 germplasms based on simple sequence repeats and phenotypic markers., 2018, 41: 859–866.
[21] Wang X L, Cao Z L, Gao C J, Li K. Strategy for the construction of a core collection forFranch. to optimize timber based on combined phenotype and molecular marker data., 2021, 68: 3219–3240.
[22] Sa K J, Kim D M, Oh J S, Park H, Ju K L. Construction of a core collection of nativegermplasm collected from South Korea based on SSR markers and morphological characteristics., 2021, 11: 23891.
[23] 张允刚, 房伯平. 甘薯种质资源描述规范和数据标准. 北京: 中国农业出版社, 2006. pp 10–60.
Zhang Y G, Fang B P. Descriptors and Data Standard for Sweetpotato [(L.) Lam.]. Beijing: China Agriculture Press, 2006. pp 10–60 (in Chinese).
[24] Meng Y S, Zhao N, Hui L I, Zhai H, Shao-Zhen H E, Liu Q C. SSR fingerprinting of 203 sweetpotato ((L.) Lam.) varieties., 2018, 17: 86–93.
[25] Hu J, Zhu J, Xu H M. Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops., 2000, 101: 264–268.
[26] 刘遵春, 张春雨, 张艳敏, 张小燕, 吴传金, 王海波, 石俊, 陈学森. 利用数量性状构建新疆野苹果核心种质的方法. 中国农业科学, 2010, 43: 358–370.
Liu Z C, Zhang C Y, Zhang Y M, Zhang X Y, Wu C J, Wang H B, Shi J, Chen X S. Study on method of constructing core collection ofbased on quantitative traits., 2010, 43: 358–370 (in Chinese with English abstract).
[27] Katinas L, Crisci J V. Agriculture biogeography: an emerging discipline in search of a conceptual framework., 2019, 42: 513–529.
[28] 闫彩霞, 王娟, 张浩, 李春娟, 宋秀霞, 孙全喜, 苑翠玲, 赵小波, 单世华. 基于表型性状构建中国花生地方品种骨干种质. 作物学报, 2020, 46: 520–531.
Yan C X, Wang J, Zhang H, Li C J, Song X X, Sun Q X, Yuan C L, Zhao X B, Shan S H. Developing the key germplasm of Chinese peanut landraces based on phenotypic traits., 2020, 46: 520–531 (in Chinese with English abstract).
[29] 崔竣杰, 程蛟文, 曹毅, 胡开林. 基于SSR标记和表型性状构建苦瓜核心种质的研究. 中国蔬菜, 2022, (2): 25–32.
Cui J J, Cheng J W, Cao Y, Hu K L. Construction of core bitter gourd germplasm based on SSR markers and phenotypic traits., 2022, (2): 25–32 (in Chinese).
[30] Kumar A, Kumar S, Singh K B M, Prasad M, Thakur J K. Designing a mini-core collection effectively representing 3004 diverse rice accessions., 2020, 1: 100049.
[31] Rajput S G, Santra D K. Evaluation of genetic diversity of proso millet germplasm available in the United States using simple- sequence repeat markers., 2016, 56: 2401–2409.
[32] 李秀诗, 付瑜华, 周祥, 黎青, 刘凡值, 杨成龙, 周明强. 基于表型性状的薏苡初级核心种质库构建. 热带作物学报, 2020, 41: 669–675.
Li X S, Fu Y H, Zhou X, Li Q, Liu F Z, Yang C L, Zhou M Q. Establishment ofL. core germplasm collection based on phenotypic characters., 2020, 41: 669–675 (in Chinese with English abstract).
[33] 黄雨芹, 尹光天, 杨锦昌, 余纽, 邹文涛, 李荣生. 基于SSR分子标记的闽楠()核心种质的构建. 分子植物育种, 2020, 18: 2641–2648.
Huang Y Q, Yin G T, Yang J C, Yu N, Zou W T, Li R S. Developing a mini core germplasm ofbased on SSR molecular marker., 2020, 18: 2641–2648 (in Chinese with English abstract).
[34] 中华人民共和国农业部. 农作物种质资源鉴定技术规程甘薯, NY/T 1320–2007, 2007.
Ministry of Agriculture, The People’s Republic of China. Technical specification for identification of crop germplasm resources-sweetpotato [(L.) Lam.], NY/T 1320–2007, 2007 (in Chinese)
[35] 黎裕, 王天宇. 美国植物种质资源保护与研究利用. 作物杂志, 2018, (6): 1–9.
Li Y, Wang T Y. Conservation and utilization of American plant germplasm resources., 2018, (6): 1–9 (in Chinese).
[36] Oh J H, Lee Y J, Byeon E J, Kang B C, Kyeoung D S, Kim C K. Whole-genome resequencing and transcriptomic analysis of genes regulating anthocyanin biosynthesis in black rice plants., 2018, 8: 115.
[37] Han W L, Zhao J Y, Deng X J, Gu A X, Li D L, Wang Y X, Lu X S, Zu Q L, Chen Q, Chen Q J, Zhang J F, Qu Y Y. Quantitative trait locus mapping and identification of candidate genes for resistance to Fusarium wilt race 7 using a resequencing- based high density genetic bin map in a recombinant inbred line population of., 2022, 13: 815643.
[38] Yamakawa H, Haque E, Tanaka M, Takagi H, Tamiya S. Polyploid QTL-seq towards rapid development of tightly linked DNA markers for potato and sweetpotato breeding through whole genome resequencing., 2021, 19: 2040–2051.

附图1 部分SSR分子标记毛细管电泳结果
Fig. S1 Results of partial SSR molecular marker capillary electrophoresis
A~D分别为GDAAS0694、GDAAS0338、GDAAS0871和GDAAS0922的检测峰图。横坐标为条带位点, 纵坐标为相对荧光单位。
A–D are the detection peaks of GDAAS0694, GDAAS0338, GDAAS0871, and GDAAS0922 respectively. The abscissas represented the banding site and the ordinates represented the relative intensity fluorescence.
附图2 基于表型性状和SSR分子标记的核心种质聚类图
Fig. S2 Cluster dendrogram of the core collection based on phenotype traits and SSR molecular markers
A: 基于表型性状; B: 基于SSR分子标记。
A: based on phenotype traits; B: based on SSR molecular markers.
Construction of core collection of sweetpotato based on phenotypic traits and SSR markers
CHEN Yi-Hang1,2, TANG Chao-Chen1, ZHANG Xiong-Jian1, YAO Zhu-Fang1, JIANG Bing-Zhi1, and WANG Zhang-Ying1,*
1Crops Research Institute, Guangdong Academy of Agricultural Sciences / Guangdong Provincial Key Laboratory of Crop Genetic Improvement, Guangzhou 510640, Guangdong, China;2College of Grassland Agriculture, Northwest A&F University, Yangling 712100, Shaanxi, China
To better preserve, study, and utilize sweet potato collection resources, 1091 sweetpotato germplasms preserved in the National Sweetpotato Germplasm Nursery (Guangzhou) were used as the materials in this study. Euclidean distance and Nei’s distance were used for NJ cluster grouping, respectively, and random sampling was conducted within the group to construct the core collection. Mean, variance, Shannon’s diversity index, coefficient of variation, and other indicators were used to evaluate the representativeness of the core collection based on phenotypic traits data, and effective alleles, Nei’s genetic diversity index, Shannon’s diversity index, and other indicators were used to evaluate the representativeness of the core collection based on SSR markers data. The results showed that the constructed sweetpotato core collection contained 289 materials, accounting for 26.49% of the entire collection. At< 0.05, there was no significant difference in the related indicators of phenotypic traits and SSR molecular markers between the core collection and the entire collection, and the phenotypic frequency distribution of the two germplasm was basically the same. The principal component analysis revealed that the core collection had similar genetic diversity and population structure to the entire collection. In conclusion, the established core collection of sweetpotato well represented the entire collection’s genetic variation and population structure, which could lay a good foundation for variety improvement, excellent genes mining, and germplasm innovation of sweetpotato.
sweetpotato; core collection; phenotypic traits; SSR markers; germplasm resource
10.3724/SP.J.1006.2023.24122
本研究由国家重点研发计划项目(2019YFD1000700, 2019YFD1000701), 财政部和农业农村部国家现代农业产业技术体系建设专项, 广东省甘薯马铃薯产业技术体系建设项目(2021KJ111)资助。
This study was supported by the National Key Research and Development Program of China (2019YFD1000700, 2019YFD1000701), the China Agriculture Research System of MOF and MARA, and the Guangdong Modern Agricultural Industry Technology System (2021KJ111).
王章英, E-mail: wangzhangying@gdaas.cn
E-mail: chenyihang2022@126.com
2022-05-21;
2022-09-05;
2022-09-22.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220919.1753.006.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
