生物信息学分析幽门螺杆菌感染差异基因表达情况
2023-03-10程明璟樊玉娟赵卫东
程明璟,严 萍,樊玉娟,赵卫东*
(1.大理大学临床医学院,云南大理 671000;2.大理大学第一附属医院消化内科,云南大理 671000)
幽门螺杆菌(Helicobacter pylori,H.pylori)感染与胃炎、消化性溃疡、淋巴瘤和胃癌等疾病相关,全球每年新发胃癌病例中约60%与H.pylori 慢性感染相关〔1-2〕。因此,H.pylori 慢性感染的防治已成为国内外研究的热点问题。深入研究H.pylori 慢性感染机制,根除H.pylori 感染,是预防胃癌的重要方法〔3〕。针对H.pylori 慢性感染的免疫学机制探讨根除H.pylori 感染的有效治疗方法,是国内外感染与免疫学领域面临的重大课题。近年来,借助生物信息学分析方法寻找各类疾病发生、发展过程中重要的靶标分子已经成为生物医学领域新的研究热点〔4〕。基于此,本研究利用公开发表数据库中H.pylori 感染的相关数据资料,借助生物信息学方法分析H.pylori 感染过程中发生显著变化的核心基因及其影响的重要生物学过程,以期为H.pylori 感染的根除以及对胃癌的防治提供理论参考。
1 材料与方法
1.1 数据收集利用关键词“Helicobacter pylori”“Homo sapiens”在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)创建的基因表达综合(Gene Expression Omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo)进行检索,共检索到665 个数据集,经筛选后,3 个数据集(GSE6143、GSE60427、GSE60662)成为目标数据集,随后下载3 个数据集的基因表达文件,其中,GSE6143 数据集来源于GPL193 测序平台,共包括24 个样本(8 个H.pylori 阴性样本,16 个H.pylori阳性样本);GSE60427 数据集来源于GPL17077 测序平台,共包括32 个样本(8 个H.pylori 阴性样本,24 个H.pylori 阳性样本);GSE60662 数据集来源于GPL13497 测序平台,共包括16 个样本(4 个H.pylori 阴性样本,12 个H.pylori 阳性样本)。
1.2 差异表达基因(differentially expressed genes,DEGs)的筛选利用NCBI 网站提供的在线分析软件GEO2R 对GSE6143、GSE60427 和GSE60662 数据集进行DEGs 分析,分析时以H.pylori 阴性样本为对照组,H.pylori 阳性样本为实验组,并计算出每个基因变化倍数的绝对值(|lgFC|)和校正P 值,当基因同时满足|lgFC|≥1.0 和P<0.05 时,该基因被视为DEGs,利用韦恩图表示以上3 个数据集DEGs 中共同的核心基因。
1.3 KEGG 通路富集分析和GO 分析为了探索核心基因影响的细胞通路及基因本体特点,使用在线软件Enrichr(https://maayanlab.cloud/Enrichr/)对核心基因进行KEGG 通路富集和GO 分析,根据综合得分排名将前10 条信息进行展示。
1.4 蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络分析为了进一步弄清H.pylori 感染人体后核心基因表达的蛋白质之间的相互作用,利用在线软件STRING(https://string-db.org/)建构PPI 网络图,当配对分数>0.4 时,认为2个蛋白质之间存在相互作用;利用Cytoscape 软件分析配对蛋白质,并计算每个蛋白质的度值,当度值≥5 分时,将其确定为重要蛋白质。
2 结果
2.1 DEGs 筛选结果以基因表达|lgFC|≥1.0 和P <0.05 为筛选标准,GSE6143、GSE60427 和GSE60662 中分别有178 个、256 个和130 个DEGs。利用韦恩图表示3 个数据集中共有的DEGs,共筛选到29 个核心基因,分别为IFNAR2、TRAF1、ICAM1、CASP1、TLR4、TGFB1、ENG、CCL19、CXCL3、CR2、C3、EMP3、ICAM3、CCL4、MFNG、GATD3A、ATP2A3、IGF1R、PTPN21、PTPN14、DKK3、HDAC5、FGFR4、PAK1、BAG1、IL1RL2、TLR5、PTPN9 和IL9R。见图1。

图1 核心基因筛选韦恩图
2.2 核心基因的KEGG 通路富集分析为进一步探索29 个核心基因的通路富集情况,对其进行了KEGG 通路富集分析,富集结果显示,核心基因主要涉及军团菌病、疟疾、核因子κB(nuclear factor-κB,NF-κB)信号通路、炎症性肠病、类风湿性关节炎、利什曼病和百日咳等方面。见表1。
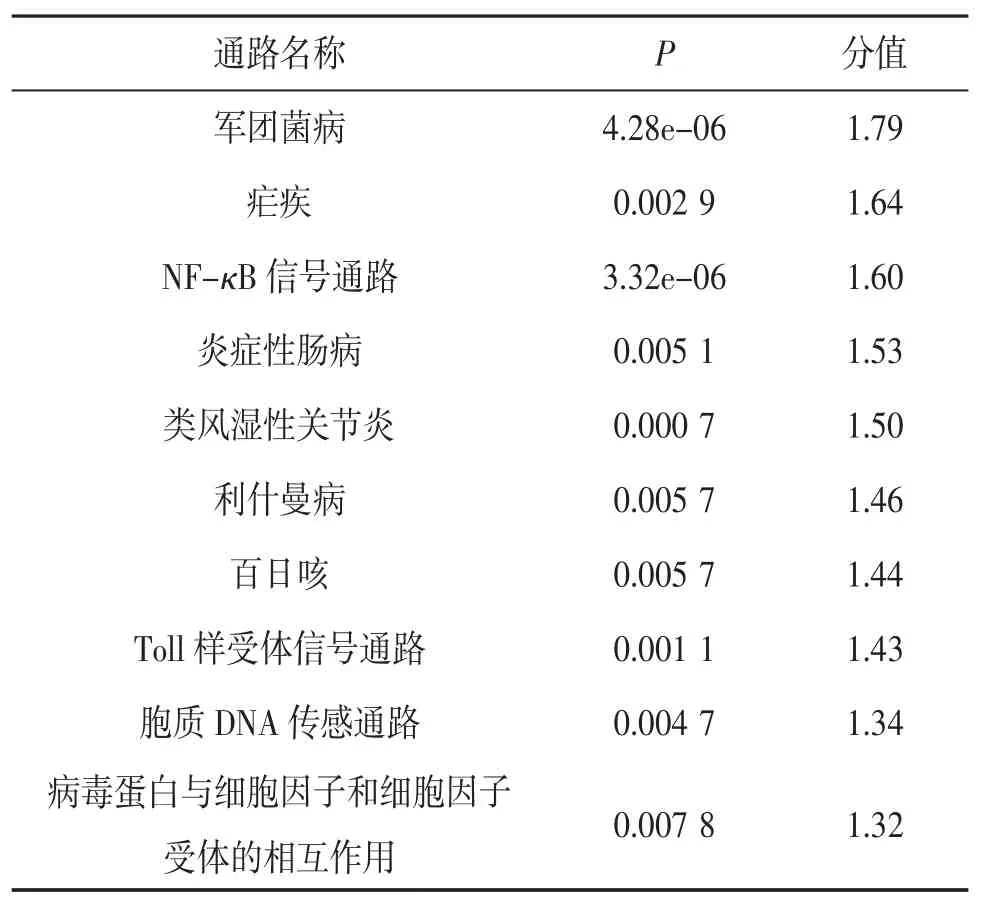
表1 核心基因的KEGG 通路富集情况
2.3 核心基因的GO 分析为了进一步探索29 个核心基因的特性及其生物学功能,对其进行了GO分析,包括细胞组成分析、生物过程分析和分子功能分析。细胞组成分析结果显示,核心基因主要存在于蛋白激酶复合物、转移酶复合物、血小板致密管状网络膜、血小板致密管状网络和肌浆网膜中;生物过程分析结果显示,核心基因主要参与补体激活替代途径、心内膜垫形成的上皮向间充质转化的调节、细胞对转化生长因子β 刺激的反应调节、核质转运的调节和细胞外基质分解的调节等过程;分子功能分析结果显示,核心基因的分子功能包括Ⅱ型转化生长因子β 受体结合、Ⅰ型转化生长因子β受体结合、半胱氨酸型内肽酶激活剂活性参与凋亡过程、成纤维细胞生长因子激活受体活性和干扰素受体活性等。见表2。

表2 核心基因的GO 分析情况
2.4 PPI 网络分析为进一步了解核心基因所表达蛋白质之间的相互关系,使用在线软件STRING 对其进行PPI 网络分析。见图2A。根据度值高低进行排序,度值越高表明与之相关联的蛋白质数量越多,其在PPI 网络中越重要。PPI 网络分析结果显示,排名第1 位的蛋白质为TLR4(度值为13),第2位至第13 位的蛋白质依次为ICAM1(度值为12),CCL4(度值为7),TLR5、CCL19、CR2(度值均为5),TGFB1、CASP1、CXCL3(度值为4),C3、ENG(度值均 为3),ICAM3、TRAF1(度值均为2)。使 用Cytoscape 软件将度值≥5 分的重要蛋白质进行可视化展示,相互作用关系见图2B。
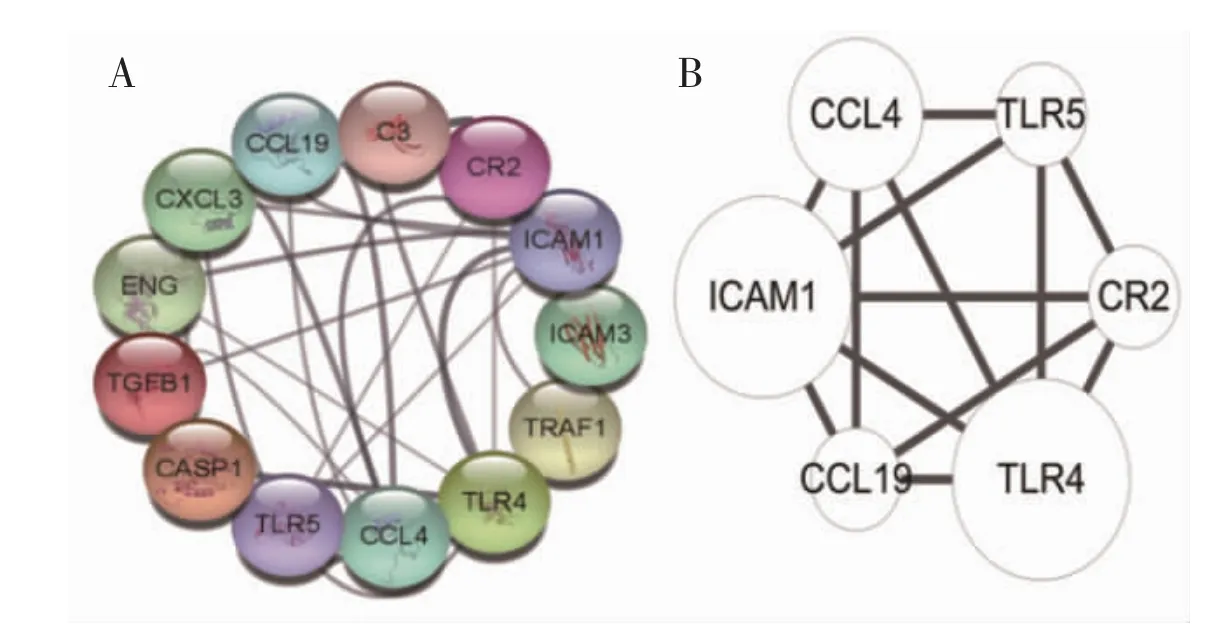
图2 核心基因表达蛋白的PPI 网络分析
3 讨论
本研究通过分析公共数据库中H.pylori 感染相关的数据资料,筛选出H.pylori 感染者与未感染者之间的DEGs,并对这些DEGs 进行了KEGG 通路富集分析和GO 分析。分析结果显示,H.pylori 感染机体后激活了炎症免疫反应。在PPI 网络分析中发现H.pylori 感染机体后趋化因子(如TLR4、ICAM1、CCL4、TLR5、CCL19、CR2 等重要蛋白质)较为活跃。
TLR4,又称Toll 样受体4,是Poltorak 等于1998 年发现的,可作为脂多糖、脂磷壁酸等病原相关分子模式受体,进一步激活天然免疫反应〔5〕。TLR4被认为参与了多种疾病的发生、发展,Hu 等〔6〕通过泛癌研究发现TLR4 的表达与胃癌、肾透明细胞癌、黑色素瘤和子宫内膜癌患者预后相关。此外,TLR4 亦可通过激活JNK 信号通路促进肝脏炎症反应〔7〕。在H.pylori 感染中,TLR4 介导了机体对H.pylori的识别,并引起宿主的抗H.pylori 免疫反应〔8〕。在TLR4 基因多态性研究方面,rs4986790 多态性可显著增加H.pylori 感染风险〔9〕,而rs1057317 多态性亦可明显增加H.pylori 感染所致的胃癌发生风险〔10〕。
ICAM1,又称细胞间黏附分子1 或CD54,主要参与细胞信号转导、细胞生长、炎症和血管生成,对白细胞活化及迁移具有重要作用〔11〕。因此,ICAM1被认为参与了多种癌症的转移和浸润,包括乳腺癌〔12〕、前列腺癌〔13〕和结直肠癌〔14〕等。早在2002年,Innocenti 等〔15〕发现H.pylori 感染后可显著促进黏附相关因子ICAM1 的表达。随后,De Jesus等〔16〕研究也表明H.pylori 的尿素酶会引起ICAM1表达升高,而ICAM1 进一步促进了H.pylori 对胃黏膜的黏附。
CCL4,又称巨噬细胞炎性蛋白1β,主要由活化的单核细胞分泌,可趋化淋巴细胞至炎症部位。在多种癌症中CCL4 亦可通过招募细胞毒T 淋巴细胞而加强抗肿瘤免疫〔17〕。此外,有研究〔18〕发现H.pylori 感染后胃黏膜中的CCL4 表达明显增强,并且CCL4 升高水平与感染部位炎症细胞浸润高度相关。
TLR5,又称Toll 样受体5,其是识别鞭毛蛋白的主要胞外受体。H.pylori 感染后,其菌毛相关蛋白CagY可活化TLR5,从而引起机体的固有免疫反应〔19〕。而H.pylori 的菌毛蛋白FlaA 可逃避TLR5 介导的先天免疫反应〔20〕。在胃活检组织中,Pachathundikandi 等〔21〕发现TLR5 表达与H.pylori 感染和胃黏膜病变相关。
CCL19,又称巨噬细胞炎性蛋白3β,主要由次级淋巴组织中的T 淋巴细胞分泌,可趋化成熟树突状细胞和幼稚T 淋巴细胞。H.pylori 感染后,胃黏膜细胞分泌CCL19,趋化CCR7+树突状细胞至淋巴结,最终诱导特异性免疫反应〔22〕。而CCL19 在H.pylori 感染相关胃癌中的研究较少,仍需进一步探究。
CR2,又称Ⅱ型补体受体,分布于单核细胞和B细胞表面,主要调节B 细胞的增殖和分化,此外也是EB 病毒的受体〔23〕。目前,虽然关于CR2 与H.pylori感染之间的直接研究较少,但是有学者发现H.pylori感染可促进EB 病毒对胃黏膜细胞的黏附〔24〕,具体机制仍有待研究。
综上所述,H.pylori 感染机体后引起机体免疫反应,尤其是机体固有免疫反应。同时TLR4、ICAM1、CCL4、TLR5、CCL19 和CR2 在H.pylori 感染相关疾病中也发挥了重要作用,可为进一步研究H.pylori 感染相关疾病提供参考。
