基于EST-SSR分子标记的广西海伦兜兰遗传多样性研究*
2023-03-01朱舒靖柴胜丰唐健民唐凤鸾陈泰国
朱舒靖,邹 蓉,柴胜丰,唐健民,唐凤鸾,陈泰国,3,韦 霄**
(1.广西师范大学生命科学学院,广西桂林 541006;2.广西壮族自治区中国科学院广西植物研究所,广西桂林 541006;3.桂林医学院药学院,广西桂林 541000)
兰科(Orchidaceae)兜兰属(Paphiopedilum)的所有植物已被列入《濒危野生动植物物种国际贸易公约》(CITES)附录Ⅰ和我国2021年版的《国家重点保护野生植物名录》,同时也是《中华人民共和国野生植物保护条例》的重点保护对象,备受国际的关注与保护[1]。海伦兜兰(Paphiopedilumhelenae)是兰科兜兰属植物,为多年生常绿草本植物,叶色深绿,花色金黄,观赏价值非常高,是杂交育种不可多得的种质资源材料,其经济价值和科研价值极高[2]。海伦兜兰于1996年首次被报道,发现于越南北部高平省;而我国于2005年在广西西部发现有分布,并于2007年正式报道,其种群数量非常少,首次报道时仅发现35株。海伦兜兰为地生兰花,主要生长在石灰岩地区,分布地海拔较高,常常分布在近山顶处常绿阔叶林的悬崖石缝土壤中[3]。
自然种群之间和内部的遗传变异对物种的长期生存尤为重要,尤其是濒危物种。了解濒危物种的遗传多样性与变异,有助于揭示其进化机制和濒危原因,对其保护和管理至关重要[4,5]。因此,对海伦兜兰的遗传多样性与变异进行系统的研究,可为其种质资源、良种选育和保护策略等提供有效参考,避免海伦兜兰植物种质资源濒临灭绝。简单重复序列(SSR)或微卫星DNA是由1-6个单元核苷酸组成的重复的DNA序列,普遍存在于原核生物和真核生物的基因组中[6]。SSR标记技术具有多等位基因、共显性、信息量大、相对丰度高、成本低和试验重复性好等特点,在植物遗传分析方面具有广泛的应用前景[7]。目前,有众多研究者通过SSR分子标记对兰科植物的遗传多样性进行了研究,如春兰品种与其园艺关系[8]、变异品种鉴定[9]、大花蕙兰(Cymbidiumhybrid)遗传多样性[10]、中国兰花的亲缘关系与遗传多样性[11]等,而关于海伦兜兰遗传多样性与遗传结构的研究尚未见报道。因此,本研究采用EST-SSR分子标记技术对海伦兜兰遗传多样性与遗传结构进行评估,并提出相应的保护策略,以期为该种质资源的有效保护与利用提供科学理论依据。
1 材料与方法
1.1 材料
海伦兜兰采自广西邦亮长臂猿国家级自然保护区的靖西市(JX)、靖西市四角山(SJS)居群,广西弄岗国家级自然保护区的三联站(SLZ)、民强站(MQZ)居群以及广西崇左市龙州县下冻镇春秀村(LZ)居群(表1),每个居群采集7-22个植株(每个植株间距离在2 m以上),每个植物选择2片健康无病虫害的叶片。将采集植株的新鲜叶片用变色硅胶干燥,共71份样本。样品由广西壮族自治区中国科学院广西植物研究所韦霄研究员鉴定为海伦兜兰(Paphiopedilumhelenae)。
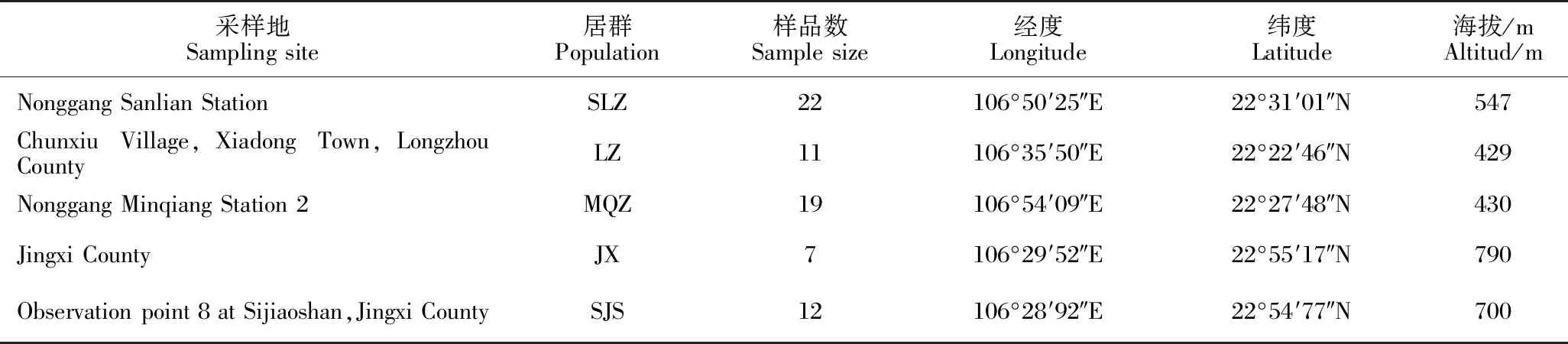
表1 海伦兜兰5个居群的详细信息
1.2 DNA提取和EST-SSR分型
使用改良十六烷基三甲基溴化铵(CTAB)法[12]提取样品的DNA,并利用1.2%琼脂糖凝胶电泳进行质检。本研究选取Xu等[13]利用亨利兜兰(P.henryanum)开发的34对引物进行引物筛选,其中大部分引物在兜兰属的7个物种中均具有良好的扩增效果[13]。选取16个样本进行引物筛选验证,最终筛选出10对扩增成功的、峰形良好的引物开展遗传多样性分析(表2)。PCR聚丙烯酰胺凝胶电泳扩增采用20 μL反应体系:1 μL模板DNA(30 ng/μL),10 μL 2×TaqPCR Marter Mix,正、反引物各0.5 μL (10 μmol/L),8 μL ddH2O补齐至20 μL。PCR扩增程序:94 ℃预变性3 min;94 ℃变性30 s,51-58 ℃退火30 s,72 ℃延伸30 s,35个循环;最后72 ℃延伸10 min,4 ℃保存[14]。使用GeneMapper v4.1软件在ABI 3730 xl DNA分析仪(Applied Biosystems,美国)上读取SSR基因型。

表2 SSR 引物信息 Table 2 SSR primer information
1.3 数据分析
1.3.1 遗传多样性与遗传分化
使用GeneAlEx v6.5.1[15]软件计算所有位点和每个居群的观测等位基因(Na)、有效等位基因(Ne)、香农信息指数(I)、观测杂合度(Ho)、期望杂合度(He)、基因流(Nm)、居群间遗传分化系数(Fst)和多态位点百分比(PPB),利用PowerMarker[16]软件计算所有位点的多态性信息含量(PIC)。
1.3.2 遗传结构分析
采用3种方法分析群体间的遗传结构:(1)使用主坐标分析(PCoA)中的主坐标成分来说明个体间的遗传距离;(2)利用GeneAlEx v6.5.1软件进行分子方差分析(AMOVA),研究居群间和居群内的总遗传变异;(3)使用Structure[17]对全部个体进行贝叶斯聚类分析,设置K=1-20,Burn-in周期为10 000,MCMC (Markov Chain Monte Carlo)设为100 000,每个K值运行10次,并利用在线工具Structure Harvester计算出最佳ΔK值,即为最佳群体分层情况,根据最佳K值作图。
1.3.3 系统发育分析
利用PowerMarker软件计算两两样品之间的Nei′s遗传距离,基于Nei′s遗传距离矩阵,采用MAGA v6[18]的非加权组平均法(UPGMA)构建所有个体的系统发育树。
2 结果与分析
2.1 EST-SSR标记多态性
利用10对引物对海伦兜兰进行多态性分析,结果见表3。由表3可知,71份样品共检测到31.8个观测等位基因,观测等位基因数为1.600 (DL034)-5.600 (DL020),平均每个位点存在3.180个等位基因;有效等位基因数为1.177(DL034)-4.101(DL020),平均值为2.191;香农信息指数为0.208 (DL034)-1.510 (DL020),平均值为0.801;观察杂合度为0.153 (DL034)-0.771(DL032),平均值为0.490;期望杂合度为0.124(DL034)-0.740 (DL020),平均值为0.451;EST-SSR基因多态性信息含量为0.154 (DL034)-0.817 (DL020),平均值为0.444。综上,本研究使用的EST-SSR引物大部分多态性良好,可用于后续试验进行遗传信息分析,其中引物DL034多态性显著低于其他引物。
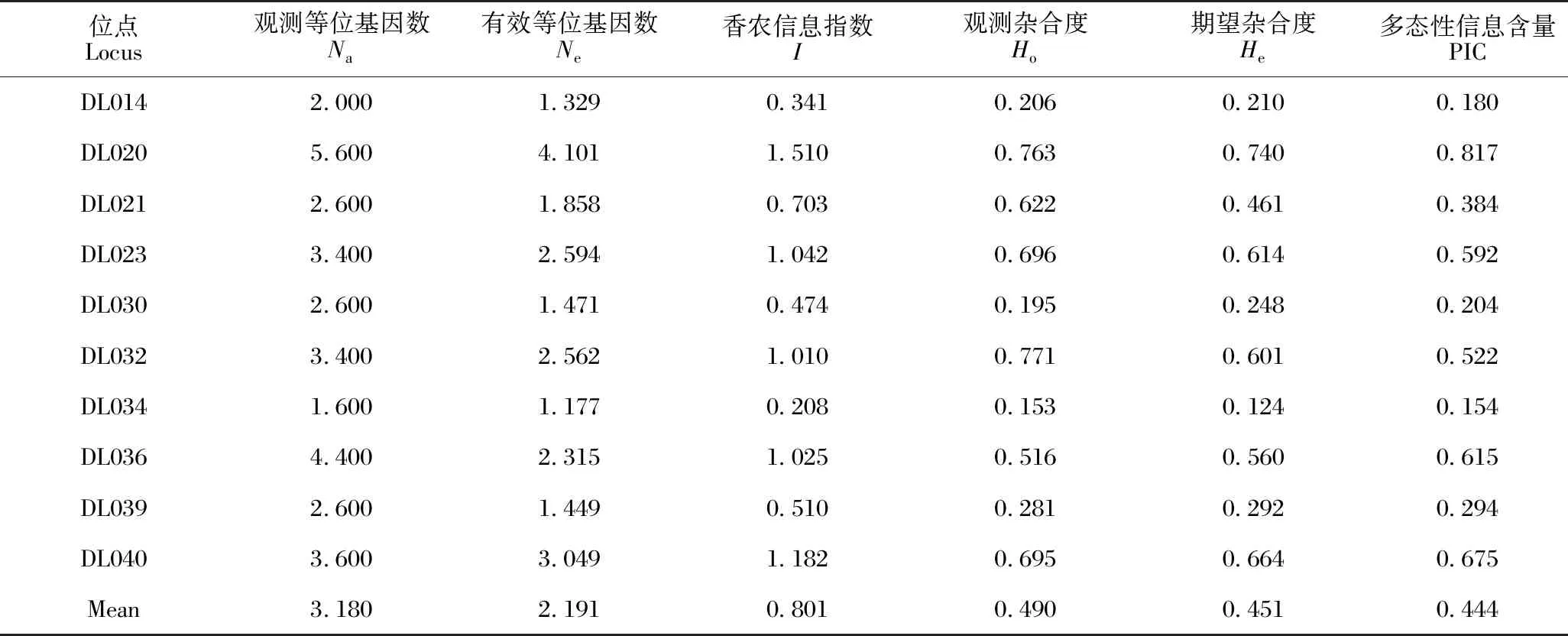
表3 10对引物多态性分析
2.2 遗传多样性分析
海伦兜兰5个居群的遗传多样性信息结果见表4。由表4可知,观测等位基因数为2.900 (JX、MQZ)-3.600 (SLZ),平均值为3.180。有效等位基因数为2.020 (SLZ)-2.316 (JX),平均值为2.191。香农信息指数为0.749(MQZ)-0.861(JX),平均值为0.801。观测杂合度为0.404 (SLZ)-0.603 (LZ),平均值为0.490。期望杂合度为0.403 (SJS)-0.503 (JX),平均值为0.451。多态位点百分比为90%-100%,平均值为92%。综上,海伦兜兰的遗传多样性较丰富,其中靖西市(JX,I=0.861、He=0.503、PPB=90%)和龙州县下冻镇春秀村(LZ,I=0.855、He=0.501、PPB=100%)居群的遗传多样性较高,广西邦亮长臂猿国家级自然保护区的靖西市四角山(SJS,I=0.773、He=0.403、PPB=90%)居群的遗传多样性相对较低。

表4 海伦兜兰5个野生居群的遗传多样性水平
2.3 遗传结构与分化
分子方差分析结果(表5)表明,海伦兜兰居群间的遗传变异仅占7%,而居群内遗传变异占93%,说明海伦兜兰的变异主要发生在居群内,居群内遗传分化程度较高,居群间遗传分化程度较低。由表6可知,各居群间遗传分化系数(Fst)为0.022-0.127,遗传分化程度不高。其中,SJS与LZ、SLZ以及SLZ与LZ居群之间的Fst值均小于0.05,表现出低遗传分化,表明这些居群之间具有较近的亲缘关系;而其他各个地区之间的遗传分化系数均为0.050-0.150,表现出中等遗传分化;SJS与JX的遗传分化系数最高(0.127),表明这两个居群间亲缘关系最远。海伦兜兰居群间Nm值为1.718-11.103,平均值为4.916,基因流水平较高。

表5 海伦兜兰群体的分子方差分析

表6 海伦兜兰5个居群间遗传分化系数(Fst,对角线下方)和基因流(Nm,对角线上方)
利用Structure软件中的贝叶斯聚类方法对海伦兜兰群体的遗传结构进行分析,结果如图1所示。当K=3时,ΔK取得最大值,表明海伦兜兰5个居群的基因型可分为3种(图1),但这3种基因型在基因池中分布不均且不能按照基因型分布(图2)。
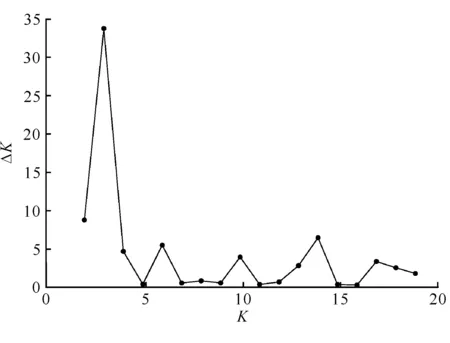
图1 海伦兜兰各居群群体结构分析的ΔK值分布
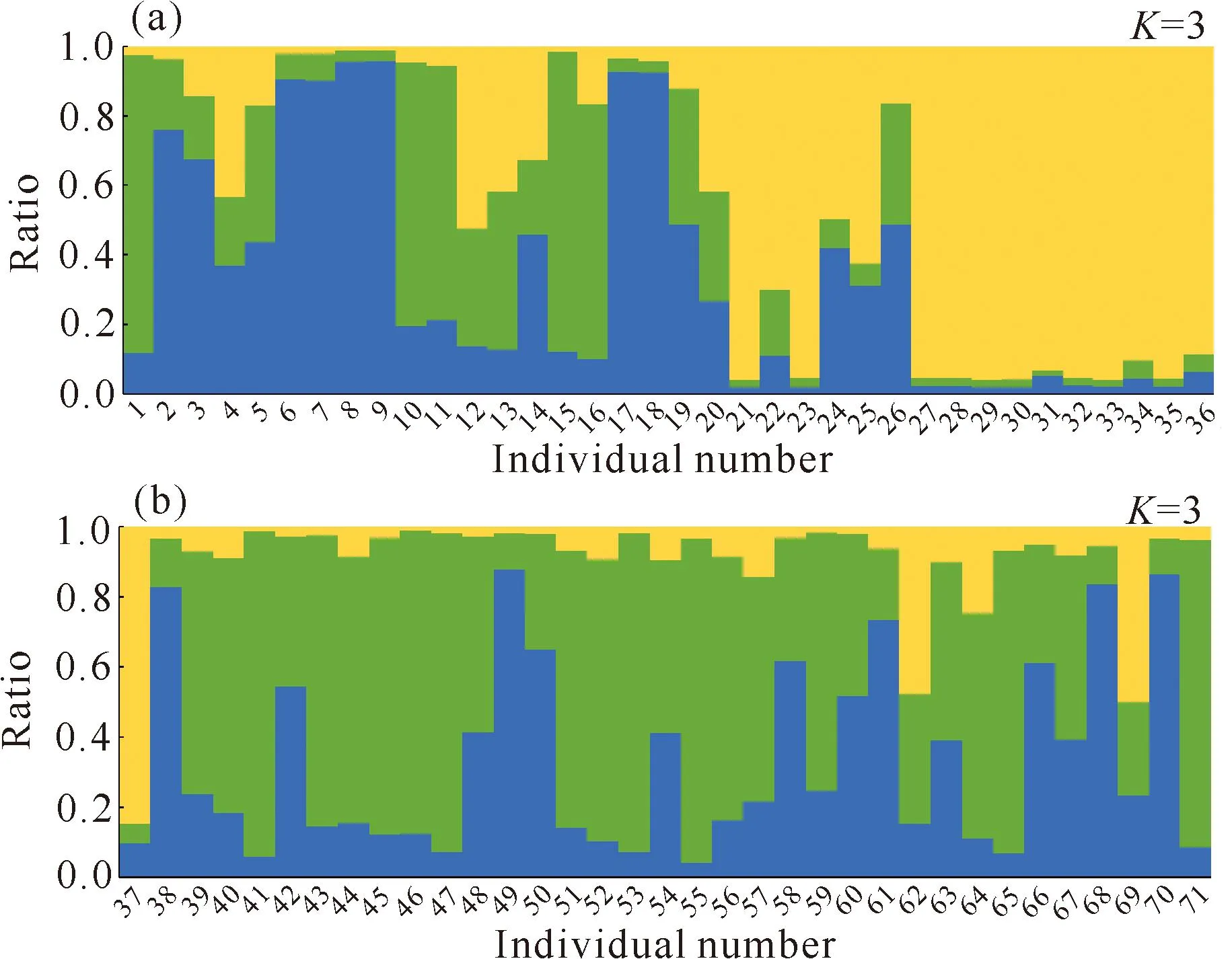
图2 海伦兜兰群体的遗传结构
2.4 系统发育关系分析
基于Nei′s遗传距离矩阵,采用UPGMA对71份海伦兜兰样品进行聚类分析(图3)。结果显示,2份JX和1份MQZ聚为一支(Ⅰ);剩余的5份JX、11份LZ、11份MQZ、22份SLZ和12份SJS聚为一支(Ⅱ),Ⅱ支又划分为Ⅱ-1、Ⅱ-2,其中Ⅱ-1包括4份LZ和1份SLZ,Ⅱ-2包括5份JX、8份LZ、11份MQZ、21份SLZ和12份SJS。LZ居群少数样品与SLZ居群1份样品亲缘关系近,JX居群少数样品与MQZ居群1份样品亲缘关系较近。主坐标分析结果显示,第一主坐标和第二主坐标分别占总遗传变异的15.57%和11.31%(图4)。综上可知,UPGMA聚类分析结果与PCoA主坐标分析结果基本一致,即海伦兜兰明显划分为3支,但未按照地理位置明显划分。

图3 海伦兜兰种质资源遗传关系的进化树
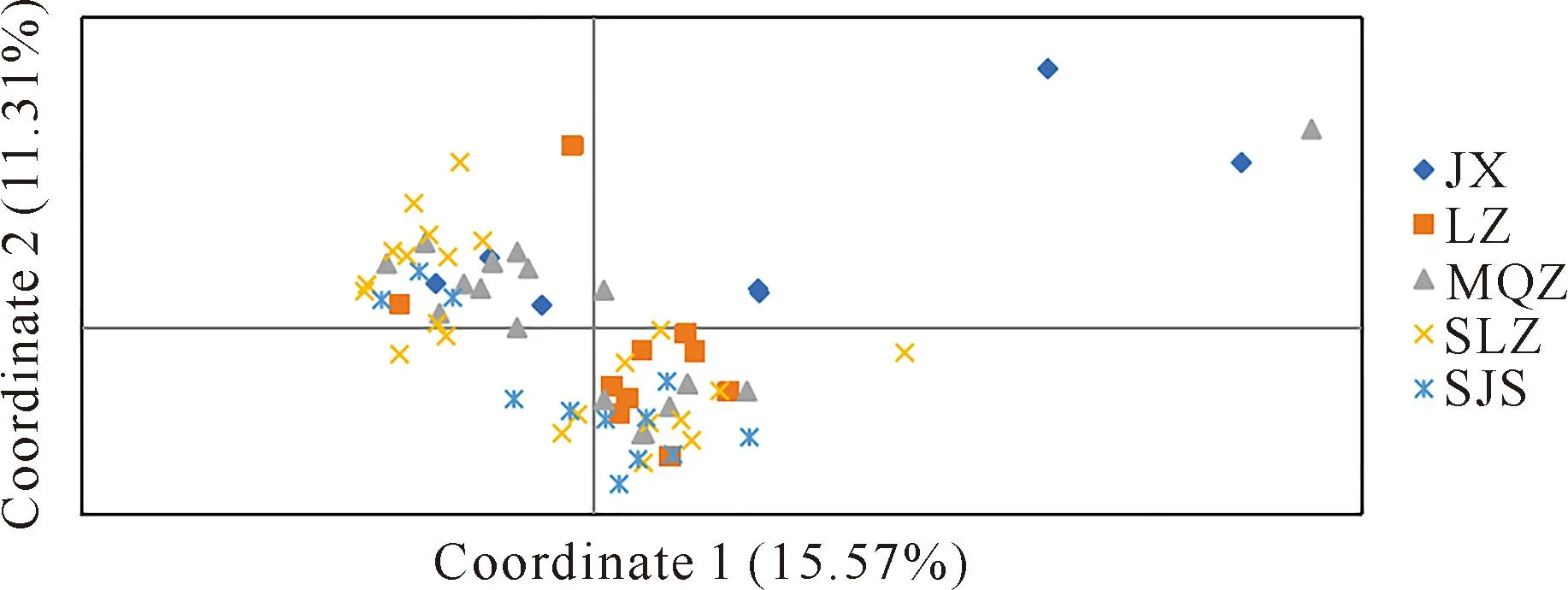
图4 海伦兜兰群体主坐标分析
3 讨论
3.1 遗传多样性分析
EST-SSR分子标记技术具有灵活性高、成本低、多态性丰富、重复性好等优势,利用该技术来分析海伦兜兰的遗传多样性,其结果更能反映DNA水平上真实的遗传变异,为海伦兜兰的品种选育与保护提供参考依据[19,20]。遗传多样性是物种进化潜力和抵抗外界环境变化的一种体现,物种遗传多样性越丰富,其适应外界环境的能力越强,反之则越弱[21]。本研究结果表明,海伦兜兰的微卫星位点均表现出较高的多态性,PIC平均值为0.444,比野生春兰(Cymbidiumgoeringii)的多态性(PIC=0.805 0)低[22]。海伦兜兰的PPB平均值为92%,高于硬叶兜兰(P.micranthum,PPB=81.25%)[23]、贵州南部的8种野生兜兰(PPB=80.26%)[24]和贵州北盘江流域6种兜兰(PPB=84.54%)[25]的多态性平均水平。此外,海伦兜兰的遗传多样性平均水平(He=0.451、I=0.801)低于蕙兰(C.faberi,He=0.600 0、I=1.049 0)[26],高于带叶兜兰(P.hirsutissimum,He=0.420 5、I=0.742 5)[21]、铁皮石斛(Dendrobiumofficinale,He=0.344、I=0.508)[27],表明海伦兜兰具有中等遗传多样性。
植物的遗传多样性通常取决于其繁育系统和生活型,稀有和特有物种的遗传多样性水平低于广布物种,一般而言,分布广、种子由动物传播的多年生草本植物具有较高的遗传多样性[28,29]。海伦兜兰为地生型草本植物,分布范围有限,种群数量极少[2],本研究结果显示海伦兜兰具有中等遗传多样性。SJS和SLZ居群遗传多样性相对较低,可能是由于栖息地遭受破坏、长期保持小群体规模和群体破碎化导致遗传多样性丧失[1,3,30]。杨舒婷等[31]研究发现,海拔越高,物种的遗传多样性也越高。海伦兜兰对生境的要求极高,分布地区海拔较高,尤其是JX居群位于海拔790 m,人为干扰相对困难,因此保留较高的遗传多样性。
3.2 遗传结构和遗传分化
从遗传结构上来看,海伦兜兰遗传变异仅有7%来自居群间,绝大部分遗传变异发生在居群内,这与徐言等[21]研究发现的兜兰属植物遗传变异主要发生在居群内的研究结果一致。海伦兜兰具有中等的遗传多样性,群体间遗传变异较低,可能与海伦兜兰具有艳丽的花色而实施欺骗性授粉策略以及具有沙尘状的种子等特征有关,这些特征增加了群体内的基因交流频率[32]。董丽敏等[33]研究表明,Fst值为0.00-0.05的群体,各亚群间分化可忽略不计;Fst值为0.05-0.15时,则被视为中度分化;Fst值为0.15-0.25时,则被认为是高度分化。本研究发现,海伦兜兰遗传分化处于中等水平(Fst=0.071),低于六盘北野生兜兰属植物(Gst=0.536 9)[25]。此外,基因流是影响种群遗传分化的重要因素之一[34]。海伦兜兰的基因流较高(Nm=4.916),较高的基因流可以阻止由遗传漂变引起的种群遗传分化。种群间的基因流动一般受到种子和花粉传播的限制[35]。兰花的典型特征是种群遗传分化低,这通常归因于兰花多为尘埃状种子,虽然大多数兰花种子通常落在母株附近,但它可以依赖风进行长距离传播[36]。
3.3 系统发育关系
本研究中UPGMA聚类树、Structure和PCoA的分析结果基本一致,海伦兜兰可分为3支,但并未严格按照地理位置划分,这与秦惠珍等[37]对白花兜兰(P.emersonii)的研究结果一致,说明海伦兜兰5个居群之间遗传分化较小,亲缘关系较近。JX居群与MQZ居群在地理位置上较SJS远,但JX居群与MQZ居群的部分个体优先聚集在一起;同理LZ部分个体优先与SLZ居群部分个体聚集在一起,而不是与同一个保护区内距离较近的MZQ居群聚集在一起。出现这一结果的原因可能是由于各居群间基因交流频繁或是受到人为活动的影响,如海伦兜兰在公开后被非法大量采集,导致海伦兜兰植株在不同地区之间迁移。
4 结论
本研究结果表明,海伦兜兰的遗传多样性(He=0.451,I=0.801)处于中等水平;JX和LZ这两个居群的遗传多样性较高,应作为重点保护单元进行保护;绝大多数遗传变异存在于居群内,仅有7%的遗传变异存在于居群间,居群内的遗传变异大于居群间的变异。目前,除龙州县下冻镇春秀村的LZ居群外,其他居群(JX、SJS、SLZ和MQZ)均在保护区内。因此,对LZ居群,林业部门应尽快建立保护小区;而对JX、SJS、SLZ和MQZ居群应加强保护区建设,加大管护和巡护力度,避免生境破坏和人为采挖。此外,海伦兜兰分布区狭窄,种群个体数量极少,抗干扰能力弱,为保护其遗传多样性,有必要开展人工授粉,促进种群的有性繁殖,防止物种退化,建议从居群间选择有代表性植株的子代进行迁地保护,最大限度地保护其种群的遗传多样性。
