大型迪庆藏猪不同生长阶段背脂与腹脂脂质代谢差异基因及调控网络分析
2023-02-27欧阳晓芳严达伟董新星
王 琳,马 黎,张 博,邓 俊,张 浩,欧阳晓芳,严达伟*,董新星*
(1.云南农业大学动物科学技术学院,昆明 650201; 2.云南农业职业技术学院,昆明 650212;3.中国农业大学动物科学技术学院,北京 100193; 4.云南省畜牧总站,昆明 650224)
脂肪组织是机体重要的能量储存场所和内分泌器官,对维持机体代谢平衡及能量稳态起重要作用[1-2]。在畜禽生产中,机体每沉积1 g脂肪所需要的能量是肌肉的4倍,脂肪大量沉积会降低饲料转化效率[3]。另外,肌内脂肪含量直接影响肉的嫩度、风味和口感,因此,解析畜禽脂肪沉积的调控机制,对畜禽生产中脂肪的靶向调控以促进畜禽养殖的高质量发展具有重要意义。
脂肪在机体的不同部位以不同的速度在不同时期沉积,导致全身脂肪分布随年龄增长有明显的变化[4],一系列基因、转录因子及信号通路介导脂肪沉积[5],涉及多基因表达、信号转导、网络精细调控等复杂过程[6]。对沙子岭猪和大约克猪的肌肉组织转录组分析发现,糖脂代谢基因(ENO1、ENO3和ATP5B)在不同品种猪中差异表达[7]。不同部位脂肪的基因表达模式、增殖分化潜力也不尽相同,皮下脂肪高表达的基因主要是参与脂质代谢和碳水化合物代谢的基因[8],内脏脂肪高表达的基因不仅参与脂质代谢过程[9],还参与免疫应答相关过程[10]。随着分子生物学技术的发展,C/EBPs[11]、SREBP-1c[12]、CREB[13]、FOXOs[14]、KLFs[15]、PPAR[16-17]等脂肪沉积的候选基因及转录因子相继被发现。迪庆藏猪是高原型猪种之一,具有沉脂力强、肉质好的特点[18],是解析脂质代谢的理想猪种,目前对迪庆藏猪的报道主要是肌肉生长[19]、胴体性能[20]、低氧适应[21],尚未见迪庆藏猪不同生长阶段腹膜与皮下脂质代谢差异的功能基因及其调控机制的报道。本研究以大型迪庆藏猪为对象,筛选不同阶段背脂和腹脂脂质代谢差异的关键基因并分析其调控网络,为差异化靶向调控猪脂肪沉积奠定基础,为迪庆藏猪的选育利用提供参考。
1 材料与方法
1.1 试验材料
选择胎次相同,出生日期相近、体重10 kg左右、去势后的大型迪庆藏猪36头,随机分为3组(S1组:10~40 kg;S2组:10~80 kg;S3组:10~120 kg),在云南省香格里拉市绿源生态种养专业合作社藏猪养殖基地进行育肥试验,S1组、S2组、S3组分别在体重达40、80、120 kg左右时屠宰,测定胴体性能,每组采集3头猪的背部皮下脂肪和腹膜脂肪,液氮速冻运回实验室,-80 ℃冰箱保存备用。
1.2 方法
1.2.1 表型数据处理 背膘厚等胴体数据用SAS软件(Version 9.2)的GLM过程进行最小二乘分析,结果以“平均值±标准差”表示,所用数学模型为:
Yij=μ+ai+eij
式中,Yij为性状观测值,μ为群体均数,ai为不同阶段(组合)效应,eij为随机残差。
1.2.2 总RNA提取和质量检测 用TRIzol法提取总RNA,10 g·L-1琼脂糖凝胶电泳检测RNA质量,NanoDropND-2000紫外分光光度计检测RNA浓度和纯度。
1.2.3 cDNA文库构建与RNA测序 取3 μg检测合格的RNA样品,按Illumina TruSeq RNA Library Preparation Kit说明书的方法构建cDNA文库,用Agilent 2100 Bioanalyzer对构建好的cDNA文库进行质检,检测合格的文库在Illumina HiseqTM2000上机测序。
1.2.4 测序数据处理 对下机的原始测序数据(Raw data)进行质控,去除污染和低质量片段,获得Clean reads,用HISAT2软件将Clean reads与猪参考基因组(Susscrofa11.1)比对,用StringTie软件对转录本进行组装和定量,利用DEGseq2软件以|log2fold change|>1且P<0.05为标准筛选差异表达基因。
1.2.5 差异基因层级聚类分析 依据差异基因表达量,利用R语言的Pheatmap软件包对差异基因进行聚类分析。
1.2.6 差异基因GO和KEGG富集分析 用在线软件DAVID对筛选的差异基因进行GO和KEGG分析,筛选与脂肪代谢相关的差异基因,以富集度count≥2且矫正P<0.05为差异基因显著富集的阈值。
1.2.7 差异基因互作网络分析 以GO和KEGG分析筛选出的与脂肪代谢相关的差异基因的表达量为基础,用R语言的stats程序包计算差异基因间的相关性,用Cytoscape 3.7.1软件构建基因互作网络图。
1.2.8 qPCR 用Primer Premier 5.0软件设计qPCR引物(引物信息见表1),利用反转录试剂盒将RNA反转录为cDNA,以猪GAPDH基因作为内参,对筛选到的差异基因进行实时荧光定量检测。
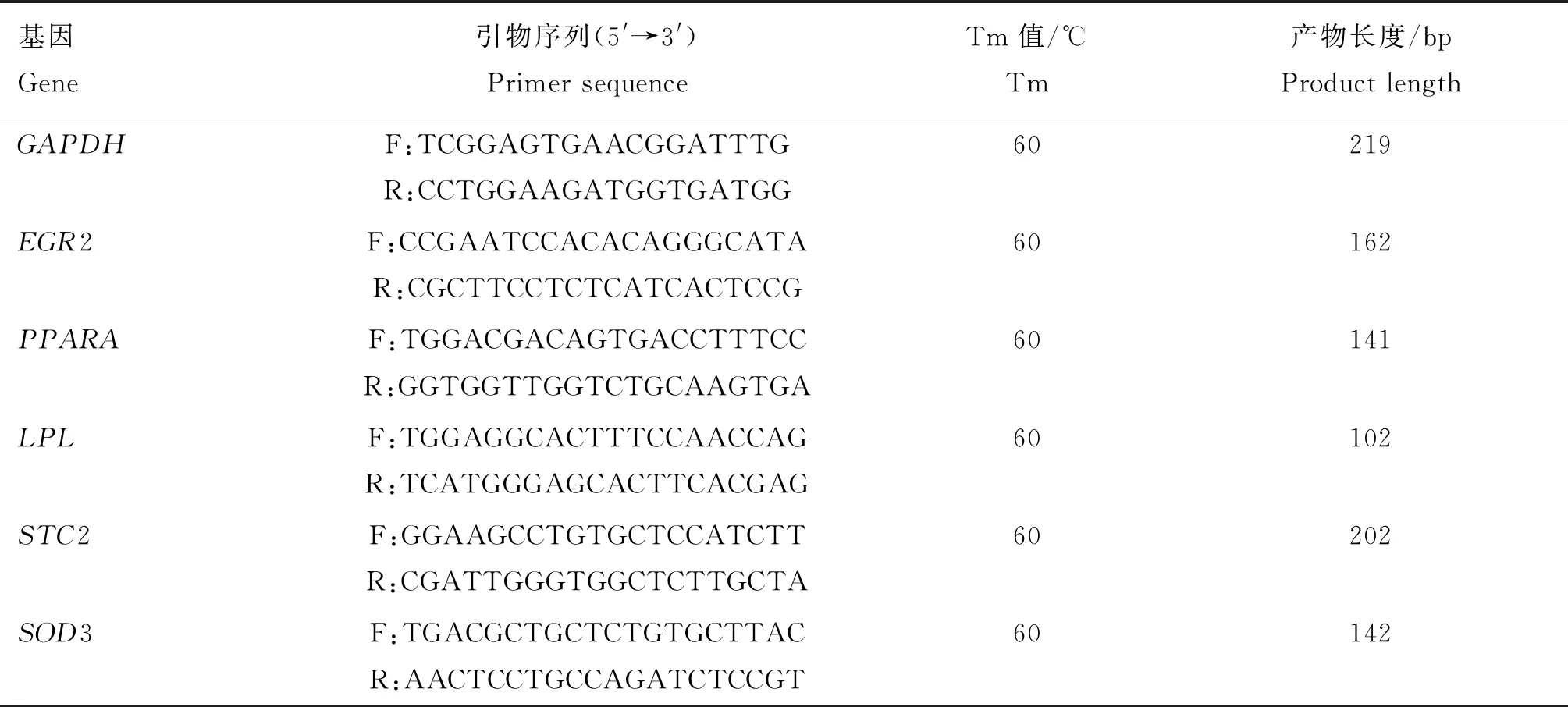
表1 差异表达基因扩增引物
2 结 果
2.1 不同体重大型迪庆藏猪的背膘厚和腹脂率比较
由表2可知,平均背膘厚S3组显著高于S2组(P<0.05)、极显著高于S1组(P<0.01),S2组极显著高于S1组(P<0.01);6-7肋间膘厚S3组显著高于S2组(P<0.05)、极显著高于S1组(P<0.01),S2组极显著高于S1组(P<0.01);板油率S3组极显著高于S2组和S1组(P<0.01),S2组极显著高于S1组(P<0.01)。可以看出,随着大型迪庆藏猪阶段性生长,平均背膘厚、6-7肋间膘厚和板油率均随体重增加而显著增加。

表2 不同体重大型迪庆藏猪背膘厚和腹脂率
2.2 大型迪庆藏猪背脂和腹脂转录组测序数据统计
RNA-Seq测序结果显示(表3),每个样品Raw reads数均在46 M以上,clean reads比率均在90%以上,clean Q30比例均在94%以上,mapping率均在87%以上,符合生物信息学分析要求。
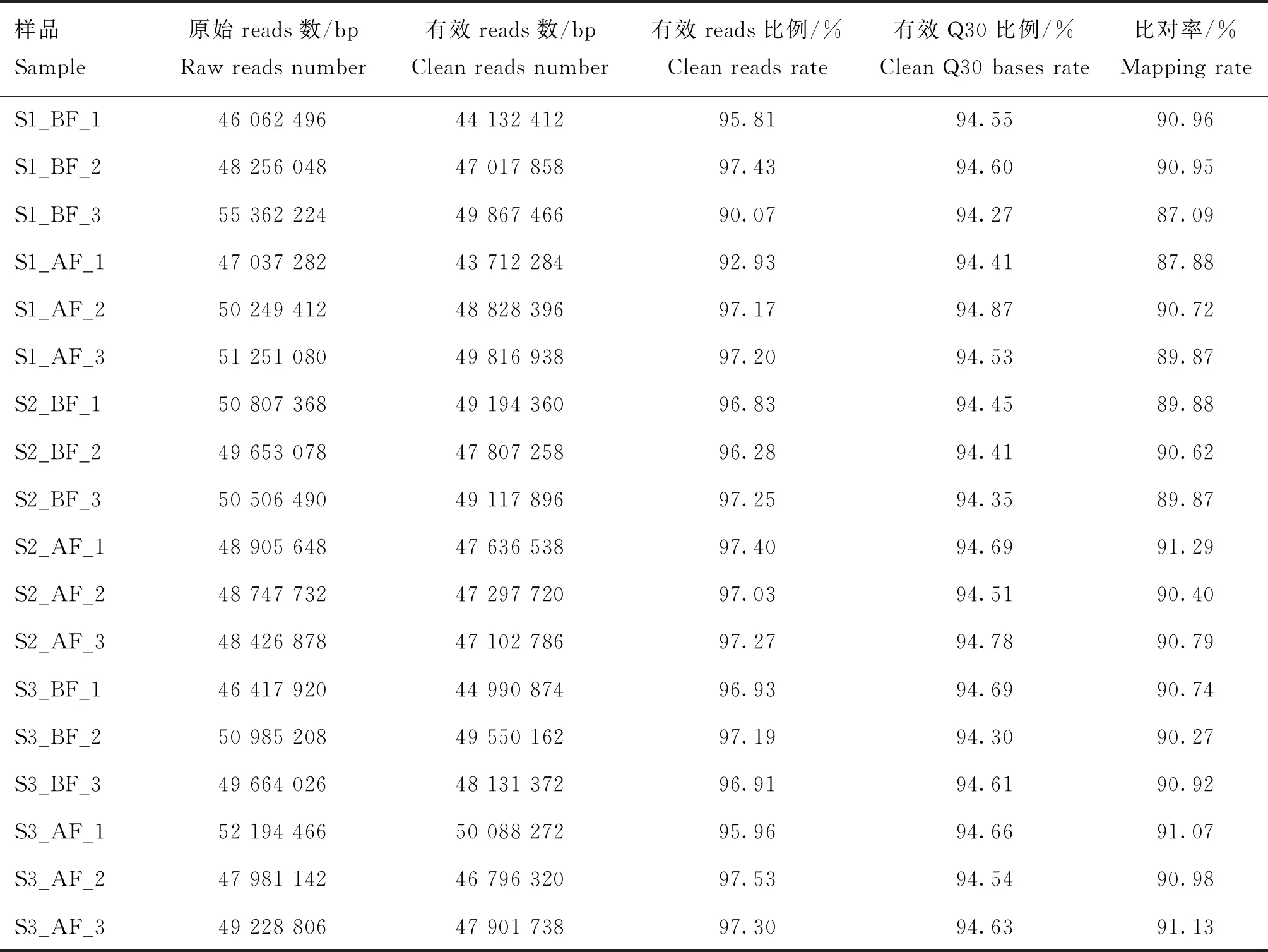
表3 RNA-Seq数据统计
2.3 大型迪庆藏猪背脂和腹脂差异基因聚类结果
差异基因层级聚类显示(图1),3个组背脂和腹脂差异基因聚成两个大类,与生物学分组一致,可进行后续分析。
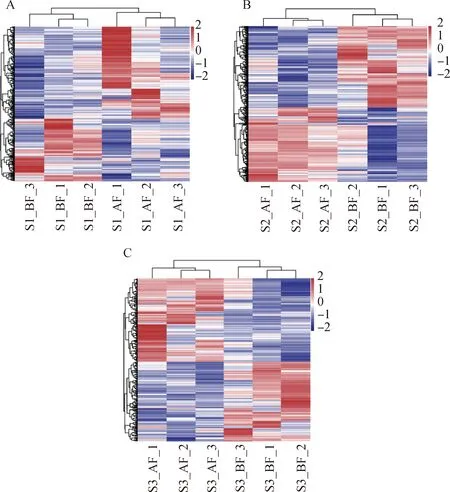
A.S1组背脂和腹脂差异基因聚类热图;B.S2组背脂和腹脂差异基因聚类热图;C.S3组背脂和腹脂差异基因聚类热图
2.4 大型迪庆藏猪背脂和腹脂差异表达显著基因筛选
以|log2fold change|>1且P<0.05为筛选差异表达显著基因的标准,与背脂相比,S1组腹脂差异表达显著基因486个(显著上调基因255个、显著下调基因231个,图2A),S2组腹脂差异表达显著基因765个(显著上调基因367个、显著下调基因398个,图2B),S3组腹脂差异表达显著基因339个(显著上调基因173个、显著下调基因166个,图2C)。

A.S1_BF vs. S1_AF差异表达显著基因火山图;B.S2_BF vs. S2_AF差异表达显著基因火山图;C.S3_BF vs. S3_AF差异表达显著基因火山图
2.5 背脂和腹脂差异表达显著基因定量验证
随机挑选EGR2、PPARA、LPL、STC2、SOD3共5个差异表达显著基因进行qPCR验证,5个基因的定量验证结果与RNA-Seq结果一致(图3)。
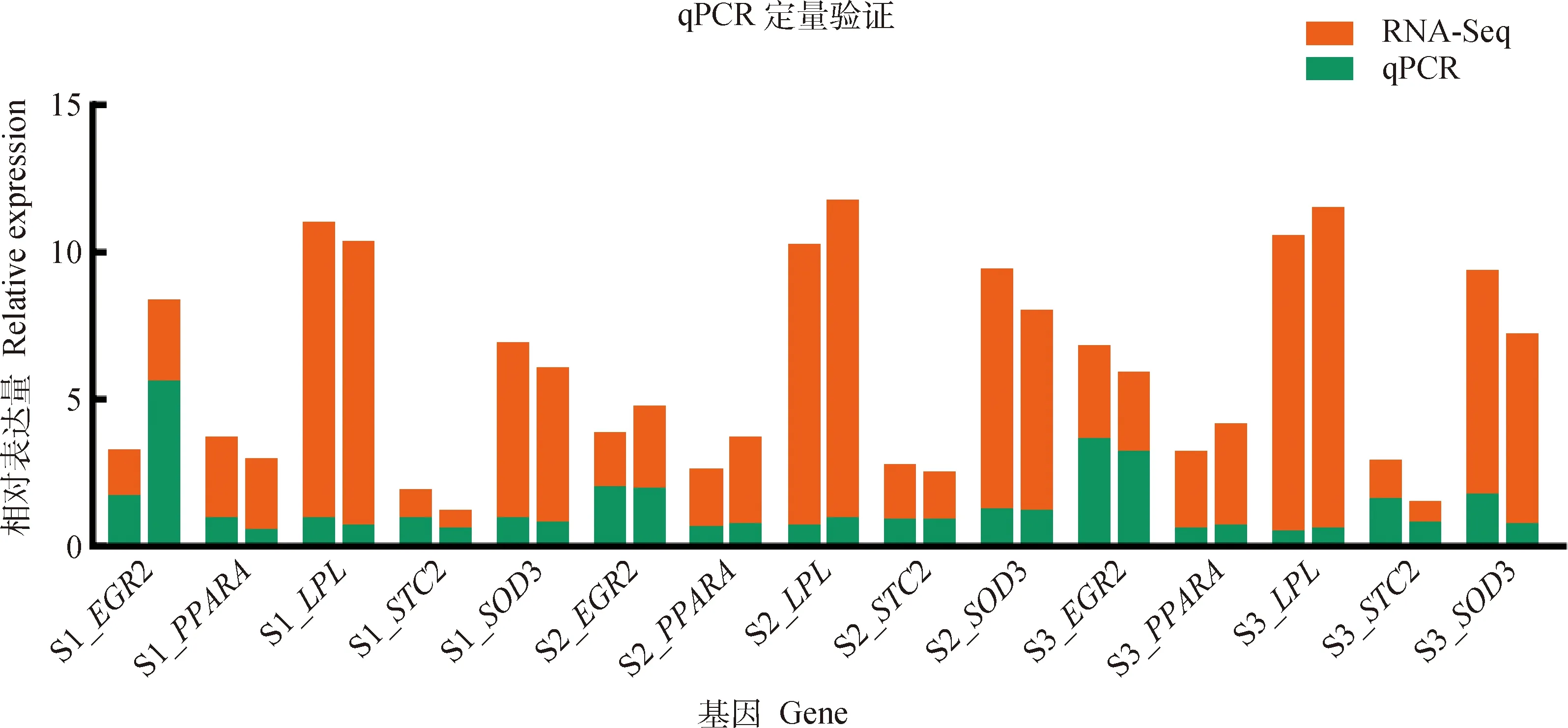
图3 大型迪庆藏猪背脂和腹脂差异表达显著基因qPCR验证
2.6 不同体重大型迪庆藏猪背脂与腹脂差异表达显著基因功能富集分析
40 kg组,差异基因主要富集于肌肉收缩、调节骨骼肌收缩等GO条目(图4A),心肌收缩、心肌细胞肾上腺素能信号传导等KEGG通路(图4B),EGR2、RARRES2、TMOD4、SFRP2基因在以上通路中富集。
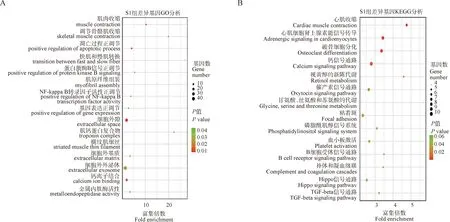
A.S1组差异表达显著基因GO分析气泡图;B.S1组差异表达显著基因KEGG分析气泡图
80 kg组,差异基因主要富集于细胞黏附、葡萄糖反应等GO条目(图5A),PI3K-Akt信号通路、破骨细胞分化等KEGG通路(图5B),THBS1、PPARA、NRIP1、LPL基因在以上通路中富集。
120 kg组,差异基因主要富集于间充质细胞增殖正调节、饥饿的细胞反应等GO条目(图6A),Hippo信号通路、TGF-beta信号通路等KEGG通路(图6B),HTRA1、TSHR、LRRK2、STC2、SHOX2、SOD3基因在以上通路富集。
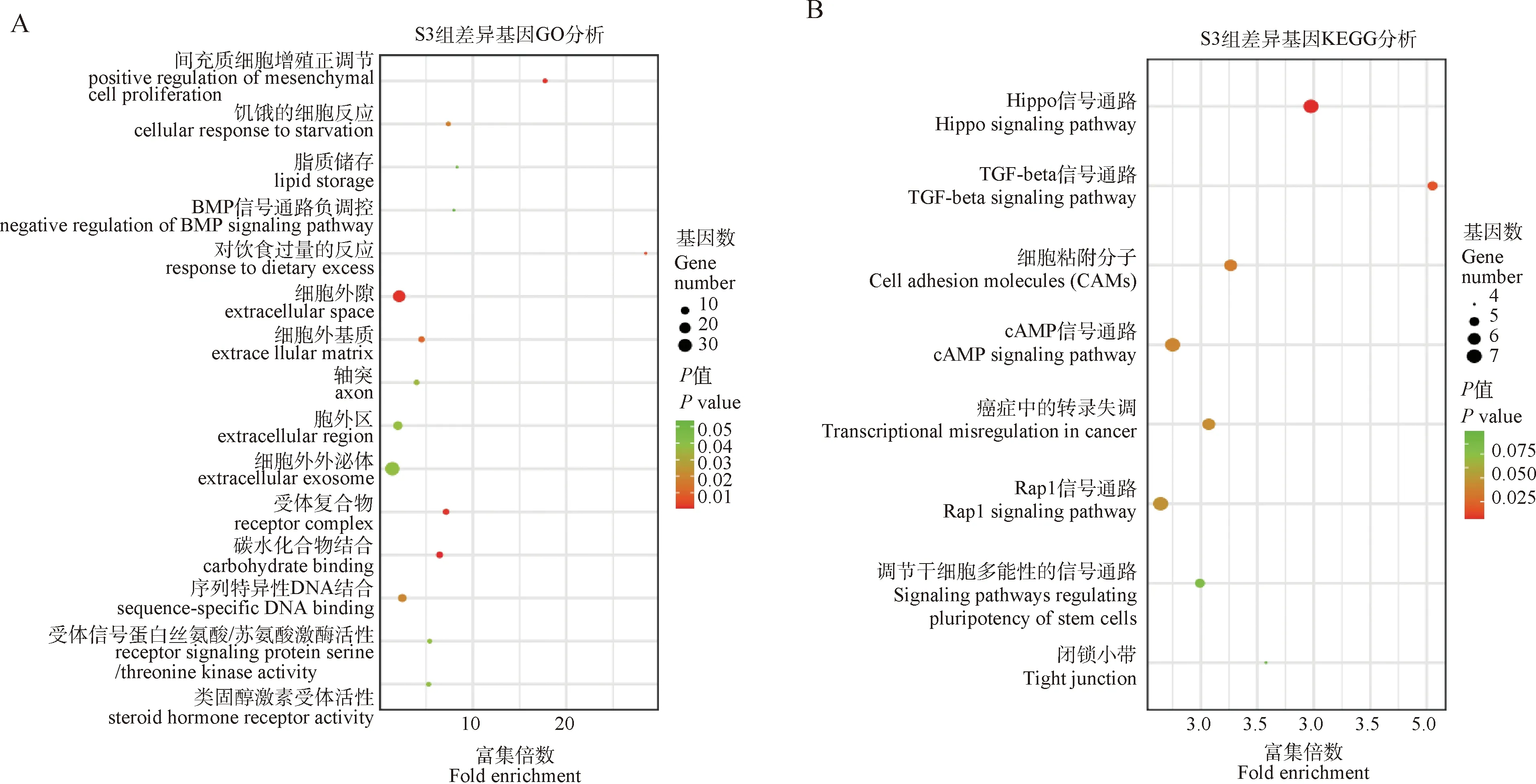
A.S3组差异表达显著基因GO分析气泡图;B.S3组差异表达显著基因KEGG分析气泡图
2.7 不同体重大型迪庆藏猪背脂与腹脂差异表达显著基因互作网络分析
2.7.1 S1组差异表达显著基因互作网络 S1组脂肪代谢相关通路差异表达显著基因互作网络图(图7)显示:EGR2、RARRES2、TMOD4、SFRP2基因位于网络核心,EGR2、RARRES2、SFRP2在腹脂表达上调,TMOD4表达下调。
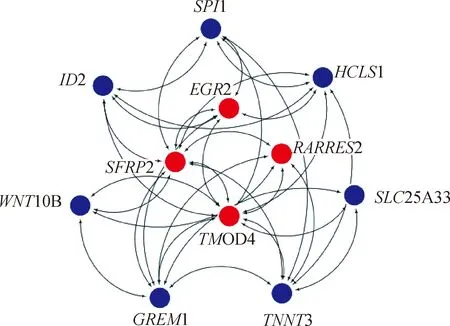
红色节点表示位于网络核心的基因,蓝色节点表示位于网络外周的基因,下同
2.7.2 S2组差异表达显著基因互作网络 S2组脂肪代谢相关通路差异表达显著基因互作网络图(图8)显示,THBS1、PPARA、NRIP1、LPL基因位于网络核心,THBS1、PPARA、NRIP1、LPL在腹脂表达上调。

图8 大型迪庆藏猪S2组背脂和腹脂差异表达显著基因互作网络
2.7.3 S3组差异表达显著基因互作网络 S3组脂肪代谢相关通路差异表达显著基因互作网络图(图9)显示,HTRA1、TSHR、LRRK2、STC2、SHOX2、SOD3基因位于网络核心,LRRK2、TSHR在腹脂表达上调,SHOX2、SOD3、STC2、HTRA1表达下调。
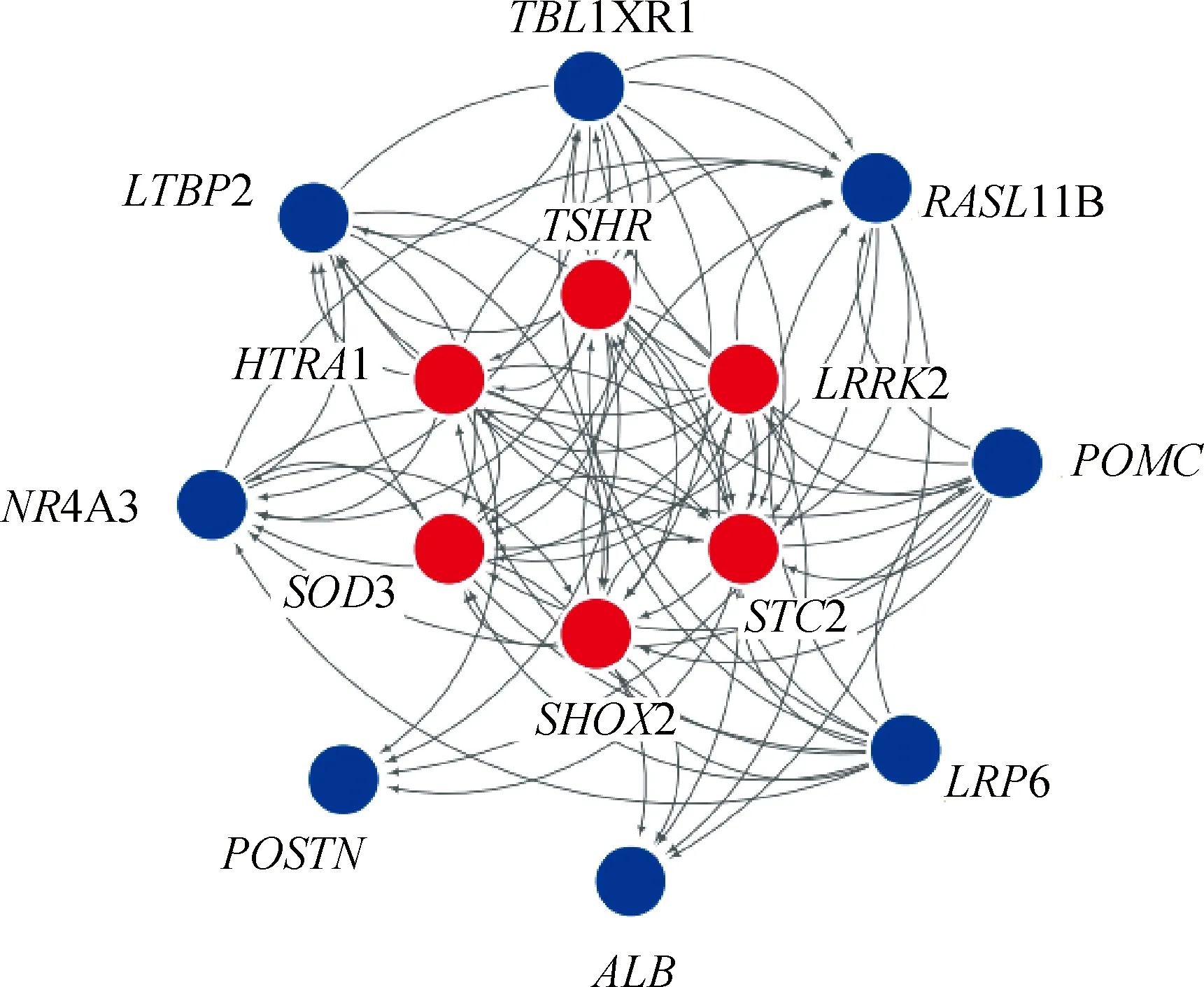
图9 迪庆藏猪S3组背脂和腹脂差异表达显著基因互作网络
3 讨 论
3.1 大型迪庆藏猪S1阶段背脂、腹脂差异的核心基因与脂质代谢
S1阶段,EGR2、RARRES2、SFRP2在腹脂显著上调,TMOD4显著下调。EGR2编码锌指转录因子,是脂肪形成的早期诱导因子,通过诱导成脂转录因子C/EBPβ表达而促进脂肪细胞早期分化[22];在3T3-L1脂肪细胞分化早期,EGR2通过C/EBPβ-PPARγ途径促进脂肪细胞分化[23];过表达EGR2的细胞系,脂肪开始积累时间更早,形成的脂滴更大,脂肪形成标记基因aP2、PPARγ表达量更高[24]。RARRES2编码的趋化素蛋白是一种脂肪因子,与G蛋白偶联受体CMKLR1结合,促进Akt/mTOR和ERK1/2磷酸化,进一步激活Akt/mTOR和ERK1/2信号通路而促进前脂肪细胞分化、增殖[25],参与脂肪代谢和能量稳态[26]。RARRES2缺失小鼠,皮下脂肪显著增加而内脏脂肪显著降低,进一步研究发现,RARRES2对皮下和内脏脂肪的作用不同是由TIMP1表达差异引起,TIMP1是脂肪形成的负调控因子,主要在巨噬细胞中表达,抑制3T3-L1脂肪细胞分化,RARRES2缺失小鼠通过减少皮下脂肪巨噬细胞浸润,降低TIMP1表达,促进皮下脂肪形成,而RARRES2缺失小鼠内脏脂肪中TIMP1表达增加,抑制脂肪生成[27]。SFRP2编码包含一个富含半胱氨酸的结构域循环的可溶性蛋白,与Wnts结合位点同源,Wnts家族分泌蛋白通过降低成脂转录因子PPARγ和C/EBPs表达而抑制脂肪细胞增殖、分化,SFRP2通过结构上有同源的结构域与Wnts的特异性受体竞争性结合而抑制Wnts活性从而减少对成脂转录因子的抑制,促进脂肪细胞增殖、分化[28-29];Crowley等[30]研究表明,内脏脂肪SFRP2的mRNA和蛋白表达量高于皮下脂肪。TMOD4是TMOD家族成员,TMOD4启动子区有脂肪转录因子C/EBPδ和肌细胞分化调控因子MEF2A的结合位点,这2 个位点的靶基因分别与肌纤维生长和脂肪分子生成有关,过表达TMOD4可促进PPARγ、aP2、C/EBPα、C/EBPβ等成脂因子表达,促进3T3-L1脂肪细胞脂滴积累,但TMOD4过表达抑制C2C12肌细胞的增殖和分化,表明TMOD4在脂肪合成和骨骼肌发生之间起着开关作用[31]。
本研究中,EGR2、RARRES2、SFRP2在腹脂上调,可能促进腹膜脂肪细胞早期分化、增殖;TMOD4下调,提示S1阶段腹膜脂肪沉积的开关尚未开启、脂肪沉积以背部皮下脂肪为主,与S1组板油率最低的结果一致。
3.2 大型迪庆藏猪S2阶段背脂、腹脂差异的核心基因与脂质代谢
S2阶段,THBS1、PPARA、NRIP1、LPL在腹脂显著上调。THBS1编码一种脂肪来源的基质细胞蛋白,是肥胖、胰岛素抵抗和脂肪组织炎症反应的标志物[32],在人和小鼠内脏脂肪中的表达量显著高于皮下脂肪[33]。THBS1缺失小鼠,糖耐受量增加,胰岛素敏感性增加[34]。PPARA是脂肪酸氧化关键基因,通过促进脂肪酸β氧化第一限速酶ACOX1和长链脂肪酸β氧化限速酶CPT1表达而促进脂肪酸氧化和脂肪分解[35]。据研究,PPARA激活导致血脂水平降低,并从血浆中去除甘油三酯,导致高密度脂蛋白胆固醇水平升高[36]。NRIP1编码核受体相互作用蛋白1通过与核受体、转录因子和其他辅调控因子等相互作用而调控体内代谢、衰老等生理过程[37],在脂肪组织、肝和骨骼肌等代谢旺盛的组织中高表达[38]。NRIP1最早被确定为哺乳动物细胞雌激素受体的共抑制因子[39],进一步研究发现其可结合并调节过氧化物酶体增殖物激活受体、甲状腺激素受体和类维生素A受体活性[40],作为共激活因子参与甘油三酯的合成[41]。在间充质干细胞中敲除NRIP1,能延缓间充质干细胞衰老、抑制细胞凋亡、降低FABP4和PPARγ基因表达、抑制间充质干细胞成脂分化[42]。与野生型小鼠相比,敲除NRIP1基因小鼠,脂肪酸氧化和能量消耗相关基因(CPT1B、UCP1)表达上调,脂肪酸合成相关基因(ACC1、FAS、SCD1)和糖异生相关基因(PPARγ、C/EBPα、SREBP1C)表达下调,NRIP1基因敲除小鼠更消瘦,体内脂肪含量更低[43]。LPL编码甘油三酯水解的限速酶[44]。在实质细胞中首先合成的是非活性单体,在脂肪酶成熟因子LMF1作用下形成LPL活性二聚体,内皮膜蛋白GPIHBP1与LPL二聚体结合并将其运输至血浆中富含甘油三酯的脂蛋白上,在APCO-Ⅱ和APOA-Ⅴ作用下LPL水解活性被激活,将甘油三酯水解为游离脂肪酸和单酰甘油[45],水解产生的游离脂肪酸在脂肪酸转运蛋白CD36的作用下被脂肪细胞吸收、重新酯化,促进脂肪细胞甘油三酯积累[46]。
本研究中,THBS1、PPARA、NRIP1和LPL在S2阶段腹脂显著上调,协同作用,可能导致胰岛素抵抗、甘油三酯合成、腹膜脂肪组织胆固醇和脂质积累,与S2阶段板油率的增幅大于平均背膘厚和6-7肋间膘厚增幅的结果一致,表明大型迪庆藏猪的脂肪沉积在S2阶段已转入腹脂沉积为主,此阶段是腹脂大量沉积的阶段。
3.3 大型迪庆藏猪S3阶段背脂、腹脂差异的核心基因与脂质代谢
S3阶段,LRRK2、TSHR基因在腹脂显著上调,SHOX2、SOD3、STC2、HTRA1基因显著下调。LRRK2编码蛋白是一种具有激酶、GTP酶和蛋白质相互作用域的多功能蛋白[47],在HepG2细胞中过表达LRRK2,LRRK2通过上调CPT1A加速游离脂肪酸的β氧化而促进脂肪分解[48]。本研究中,LRRK2在腹脂上调,可能促进腹膜脂肪组织脂肪酸氧化分解、脂肪沉积减少,与Khan等[49]的研究结果一致。TSHR是前脂肪细胞分化为成熟脂肪细胞的关键基因,通过cAMP/PKA/PPARA通路激活成脂关键基因SREBP1c,促进甘油三酯积累[50]。在3T3-L1前脂肪细胞中敲除TSHR,成脂关键基因PPARγ表达降低,前脂肪细胞分化受阻[51]。过表达TSHR的小鼠更胖,肝和血清中甘油三酯积累更多[52]。TSHR缺失小鼠体温升高,耗氧量增加,能量消耗增加,表明TSHR可能通过减少能量消耗增加脂肪沉积。SHOX2是同源盒基因家族成员,通过与C/EBPα结合抑制C/EBPα与ADRB3启动子结合,抑制ADRB3表达从而抑制脂肪分解[53]。SOD3编码超氧化物歧化酶3。在人脂肪细胞中沉默SOD3,PPARγ、SREBP1c等成脂基因在脂肪细胞中的表达增加,甘油三酯积累增加,敲除SOD3的小鼠脂肪细胞增大[54]。在高脂饮食(HFD)的C57BL/6小鼠中过表达SOD3,动物体内高水平的SOD3活性上调了肝中CPT1α、CPT1β、PGC1α、PGC1β和UCP2等负责能量消耗的基因表达[55]。STC2编码分泌型糖蛋白,在人间充质干细胞中过表达STC2,显著下调PPARγ和FABP4表达进而抑制间充质干细胞的成脂分化,相反,敲除STC2可上调PPARγ和FABP4表达,细胞脂滴增大[56]。小鼠肝细胞过表达STC2,可促进STAT3磷酸化而激活STAT3信号通路,抑制SREBP1c表达,抑制脂肪合成,减少肝中甘油三酯积累[57]。HTRA1编码蛋白为胰蛋白酶家族成员,是间充质干细胞成骨分化和成脂分化的调节因子。在间充质干细胞中沉默HTRA1,促进成脂标记基因FABP4、PPARγ表达增加,诱导间充质干细胞分化为成熟脂肪细胞[58]。HTRA1还可通过上调MAP激酶信号通路,诱导脂肪形成的负调控因子MMP3表达从而抑制脂肪细胞脂滴形成[59]。
本研究中,TSHR在腹脂上调而SOD3、STC2和HTRA1下调,这4个基因协同,可能共同促进腹脂脂肪细胞分化、脂肪合成、甘油三酯积累和脂滴变大,LRRK2上调和SHOX2下调可能导致腹膜脂肪组织脂肪酸氧化分解、脂肪沉积减少;TSHR、SOD3、STC2和HTRA1可能协同调增腹膜脂质积累,而LRRK2和SHOX2基因协同调减腹脂沉积,最终减缓腹脂增速、防止机体腹脂过度增加而发生代谢功能障碍、降低炎症风险,与S2到S3板油率增幅(52.67%)比S1到S2增幅(133.55%)低的结果一致。
4 结 论
EGR2、THBS1、HTRA1等14个基因作为核心基因精细调控大型迪庆藏猪背脂与腹脂脂质代谢,10~40 kg:EGR2、RARRES2、SFRP2和TMOD4基因位于网络核心,促进脂肪细胞分化、增殖的基因在腹脂上调,脂肪合成的开关基因下调;40~80 kg:THBS1、PPARA、NRIP1和LPL基因位于网络核心,促进甘油三酯合成、胆固醇形成、脂质积累的基因在腹脂上调;80~120 kg:LRRK2、TSHR、SHOX2、SOD3、STC2和HTRA1基因位于网络核心,促进甘油三酯积累、脂肪酸氧化的基因在腹脂上调,抑制脂肪分解、脂肪合成、脂滴形成的基因下调。
