合成生物学技术在环境保护中的应用
2023-02-03高国辉马佳骏罗泽天
黄 磊, 高国辉, 马佳骏, 罗泽天, 齐 玉
(天津理工大学 化学化工学院, 天津 300384)
根据世界卫生组织最近的一份报告显示,环境风险因素导致约25%的人类死亡,相当于每年死亡1 260万人[1]。各种类型的环境污染物往往分布广泛,难以识别和定位,合成生物学技术有望为环境中的污染物监测和有效、有针对性地修复提供新的可能[2]。地球上人类的工业活动、石油泄漏、无节制地使用塑料以及电子废物等有害物的填埋都导致了各类污染物的大量释放,如多氯联苯(PCBs)、多环芳烃(PAHs)、挥发性有机物(VOCs)和多溴联苯(PBBs)等。持久性有机污染物(POPs)由于被固体颗粒吸附的速度比其溶解在水中的速度更快,因此可能通过有毒的农副产品进入我们的食物链和卫生系统[3]。另外,全球人口的不断增长,对粮食的需求不断提高,也强化了修复受污染场地的重要性[4]。合成生物学是将工程、科学和技术应用于编辑生物体的遗传物质,进而执行新的功能的一门学科[5]。这可以为应对当前环境挑战提供强有力的解决方案。规律间隔成簇短回文重复序列(Clustered Regularly Interspersed Short Palindromic Repeats,CRISPR)技术在基因工程细菌和古细菌的生物修复中得到了广泛应用,生物修复领域也出现了很多新的先进技术,包括生物膜工程、人工微生物群落的构建、基因驱动、酶和蛋白质工程等。生物修复中基因工程微生物的环境安全问题是这一领域研究的关键,包括基因工程微生物与本土微生物群落的基因水平相互作用、对非目标微生物群落的选择压力以及抗生素抗性基因的意外扩散等[6]。
1 基因工程中的合成生物学工具
建立一个基因工程系统需要一个合适的底盘细胞和基因编辑工具来控制和监测基因的重组。在合成生物学技术的帮助下,生物修复的最新趋势是应用非模式化生物作为宿主,利用CRISPR作为基因编辑工具,生物遗传电路作为工程化工具,来去除环境中的污染物[7]。根据代谢途径的可行性和实验室实验的便利性,微生物可以被分为模式和非模式生物两种类型,其中第一类是大多数合成生物学工作优先考虑的对象。然而,非模式生物由于其具有独特的基于代谢途径、碳源利用和对极端环境天然抗性的选择性污染物降解能力,近年来在生物修复研究中得到了广泛关注。尽管由于基因组GC含量高给非模式生物的基因改造带来了相当大的限制,但科学界通过发现和开发新的遗传组分 (如报告基因、穿梭载体、核糖体结合位点和启动子),成功地解决了一些物种的相关问题[8]。本文后续将讨论用于生物修复的合成生物学工具的具体类别,包括基因驱动和CRISPR工具,生物遗传电路,酶和蛋白质工程以及一些新的生物修复技术。
1.1 基因驱动和CRISPR工具
基因驱动是一种新的可用于大规模污染的生物修复策略,它可以使特定的优势基因在整个微生物群落或单个物种中扩散[9]。之前,石油衍生物污染地点的生物修复主要应用水平基因转移技术,主要是通过接种携带有效降解基因(alma、xylE、p450cam)的大肠埃希菌(Escherichiacoli)到被污染的沉积物中。因为没有外来物种干扰生态系统的风险,所以将降解基因转移到原位微生物群落中是可行的。这使得原位微生物群落获得了相关的降解基因,进而去除了石油烃类物质[10]。
有多种基因编辑技术可以作为人工分子剪刀在特定位点切割DNA,包括TALEN、ZFN和CRISPR。其中CRISPR技术应用于细菌系统是最有效和直接的。它最显著的优势是能够应用于多个位点,并且高效地实现常见的基因工程目的,包括插入、删除、替代和位点突变[11]。CRISPR工具包主要应用于假单胞菌(Pseudomonas)和大肠埃希菌等模式微生物。目前,其应用也已扩展到非模式生物,如橡胶红球菌 (Rhodococcusruber)TH、睾酮单胞菌(Comamonastestosteroni)和无色杆菌(Achromobactersp.)HZ01[6]。天津理工大学化学化工学院生物化工课题组(本课题组)也应用CRISPR技术对芽胞杆菌属(Bacillus)进行了改造,以期提高芽胞杆菌未来的应用潜力[12-13]。最近,又开发出多种基于CRISPR技术的极端微生物基因修饰系统,包括嗜热菌、嗜酸菌和嗜盐菌,这些系统可以在利用微生物进行生物修复研究中发挥关键作用[14]。CRISPR也被应用于产生物表面活性剂的微生物,以提升其降解芳香烃和农药的生物电动力学修复系统效率[15]。
CRISPR干扰(CRISPRi)这种工程学工具,可以通过下调不利基因的表达,调节相应的代谢通量来过表达所需的目的产物[16]。相反,CRISPR激活(CRISPRa)工具可以使生物体能够上调所需的目的基因[16]。例如,DeLorenzo等[17]报道了CRISPRi工具的应用,在非模式木质纤维素分解细菌浑浊红球菌(Rhodococcusopacus) PD630中构建了第一个靶向基因表达抑制系统。
总的来说,CRISPR在生物修复中的应用很广泛,但其在增强生物去除、抗逆性和优化生物降解途径方面的研究较少,值得未来进一步关注。CRISPR技术在生物修复中的早期应用之一是靶向基因的敲除和导入,这有助于研究人员验证特定基因的作用和功能。在Gallo等[18]最近的一项研究中,通过使用Thermo-Cas9工具进行靶向基因敲除证实了亚砷酸盐抗性基因(TtarsM)对嗜热细菌(Thermusthermophilus) HB27解毒系统的作用。这些靶向基因敲除方法也可以提高非模式生物中CRISPR工具的编辑效率,进一步为CRISPRi和CRISPRa以及多重基因组编辑进行代谢途径改造提供了可能[19]。
目前一些研究表明,应用CRISPR技术可以通过生物转化的方式将一些工业废物转化为高附加值产品。在针对木质纤维素的生物转化过程中,科学家引入了一种基于CRISPR的过表达木质素分解酶蛋白的工程技术[20]。另一个受益于CRISPR的生物转化领域是将CO、CO2和CH4(也称为C-1气体)等温室气体发酵生成各种附加值产品。许多产醋酸菌的大规模应用都受到其生长速度和一碳气体产量低的制约,最近CRISPR介导的基因编辑技术显示出克服这一限制的可能性。永达梭菌(Clostridiumljungdahlii)和石灰真杆菌(Eubacteriumlimosum)是两种主要进行CRISPR介导基因编辑的产乙酸菌[21]。例如,在永达梭菌中利用两个启动子(Pthl、ParaE)表达Cas9和sgRNA并删除4个基因(pta、adhE1、ctf和pyrE),这样既提升了菌株的生长速度也促进了C-1气体发酵生成乙酸和乙醇[22]。将CRISPRi工具箱应用于永达梭菌还可以增强温室气体发酵产丁酸的效率并减少相关碳源进入产乙醇支线途径的量[23]。
由于序列与目标位置相似,因此会导致在不需要的位置发生DNA切割,这种现象称为脱靶,脱靶也是CRISPR基因修饰广泛应用的主要挑战之一[24]。在开发基于CRISPR基因驱动的生物修复时要注意这部分情况,防止对自然生态系统产生不良影响。目前为止,为了确保CRISPR基因工程和基因驱动的安全性,全世界范围已经开发了许多相应策略(表1)。
包括国家图书馆在事业发展、资源建设、信息组织、服务创新、技术应用、管理体制改革以及国家数字图书馆建设等方面的历程与新的实践经验,并为其未来发展探讨思路、提供参考借鉴。

表1 针对工程物种的生物安全方法
1.2 遗传电路
遗传电路类似于普通电路,主要由以下几种分子基因设计和构建,包括报告基因、代谢基因、启动子、复制子和选择性标记。这些元件可以用来改变细胞行为,提高代谢产量或监测后续的代谢活动。构建合适的遗传电路最重要的是选择合适的遗传元件。例如,缺乏报告系统和可诱导的启动子会降低产乙酸工程菌利用空气污染物转化为附加值产品的能力。因此,对于遗传重组,表征新的和有效的遗传组分是至关重要的[20]。例如,耐重金属贪铜菌 (Cupriavidusmetallidurans) CH34是一种多用途化能自养微生物,可用于芳香族化合物的降解、废水生物发电和贵金属的生物矿化。开发高效的可诱导或组成型启动子有助于控制蛋白质生产和优化CRISPR编辑效率[26]。
遗传电路的应用也可以与CRISPRi相结合来建立转录逻辑门,通过电路输出结果来控制细胞行为,如糖分消耗等[26]。目前,在乙酸菌中已经成功实施这一策略,这样可以将代谢活动与环境变化联系起来,例如考察pH的改变与底物生长浓度变化的关系等[21]。
1.3 酶与蛋白质工程
复杂结构环境污染物的生物处理,如纸浆废水中的木质素、农药、芳香烃和塑料,都依赖于微生物降解酶的强大活性。然而,这些酶的自然产量不足,但可以通过遗传操纵增强酶的表达量[27]。目前,很多工具被用于基因工程生物修复系统酶的优化,包括理性、半理性设计和定向进化[20]。通过替换活性部位中的官能团对羧化酶进行合理的重组,可将一种剧毒的苯酚衍生物(4-羟基苯乙烯)的生物降解率提高40%,同时将立体异构选择性提高39倍[28]。另一方面,定向进化是一种基于随机突变的非结构性方法,已经发展出许多新功能,例如测定生物修复应用的真菌木质素分解酶的极端pH抗性[20]。在酵母细胞中建立甲基化合成功能的最好方法是在过氧化物酶体中定向表达蛋白质,这在甲基化微生物的生物碳捕获过程中尤其重要,特别是在消耗甲醇的酵母中,如解脂耶氏酵母(Yarrowialipolytica)[29]。 除上述方法之外,当具有污染物降解能力的基因在微生物宿主中表达时,重组DNA和异源表达技术也可以用于大规模酶的生产和纯化,通过定点诱变方法,底物的活性和范围得到了改善[30]。对不可培养环境样本的宏基因组分析也有助于研究人员发现编码具有特定功能的降解酶的新基因[30]。
1.4 优势突变的快速筛选与选择
采用实验室适应性驯化(ALE)技术,建立了一种新的快速鉴定有益突变体的筛选方法。这可以促进后续的逆向工程和构建高污染物耐受性降解菌株的发展。传统意义上,全基因组测序是在菌株逐步适应有毒物质或次生长条件后进行的,以揭示菌株的遗传变化[31]。然而,在ALE期间多重突变的出现使得定位突变基因变得困难且耗时。应用快速优势突变筛选与选择(RAMSES)方法,在指数生长期向培养物中加入突变的DNA会导致DNA通过等位基因的置换而掺入染色体中,然后在增加选择压力的情况下进行突变株的筛选,此时只有优势突变株才能生长[32]。一些科学家利用这种方法来表征木质素降解菌(Acinetobacterbaylyi) ADP1在暴露于高浓度阿魏酸盐(一种木质素衍生芳香化合物)后的有益突变。他们进一步用有益的突变(基因hcaE、hcaK)来改造细菌,以提高其对阿魏酸盐的耐受性[32](图1)。

图1 贝利不动杆菌ADP1中可能的芳香酸转运系统示意图[32]Fig.1 Schematic of possible aromatic acid transport systems in Acinetobacter baylyi ADP1[32]蓝色:转运体;绿色:孔道蛋白。虚线箭头表示多个步骤blue: transporters; green: porins. The dashed arrow refers to multiple steps
2 生物修复应用方面的最新成就
生物修复研究工作始于基本的原位修复技术,如生物强化、生物隔离、生物通风,以及非原位方法,如堆肥、土地耕作、生物堆填和生物反应器[8]。这些方法主要应用于一些未改造的生物体,进而减轻相应的环境污染程度。后续研究人员开始评估新的天然生物酶、分离不同的微生物,并且运用简单的基因编辑方法进行生物改造。目前采用合成生物学新技术(如生物膜工程及合成微生物群落)已经成功突破了生物修复屏障。
2.1 生物膜工程
生物膜工程是生物修复领域的一项最新发现,该技术旨在减弱有毒金属、烃类化合物及农药在自然界中的影响并进而克服天然生物膜的局限性。合成生物膜主要通过整合信号通路、代谢途径工程及胞外聚合物(EPS)重排设计完成[33]。生物膜的再生能力使其能够可持续利用,EPS组成的可控性使其能够满足各种生物修复的目的。生物浸出(从固体基质中溶解重金属)、金属腐蚀抑制及利用微生物燃料电池(MFC)处理废水是一些传统的生物修复应用,而生物膜工程可以对其做进一步的改进。例如,建立选择性汞解毒系统,其中汞检测器(MerR启动子)会在大肠埃希菌中组成型表达。然后,通过一种名为Curli的合成纳米纤维动态隔离游离的汞[34]。此外,通过用稀土结合标签修饰的Curli纤维,可以制备对稀有元素(Tb3+)具有选择性吸附能力的功能性生物膜[35]。这些研究说明了如何利用工程化生物膜从废水和固体废物中选择性地回收重金属。未来合成生物膜的环境研究方向包括更大规模的试验、响应电路的优化、将相关研究从蛋白质研究扩展到其他生物膜成分(如多糖和核酸)上[33]。
2.2 合成微生物群落
通过在人为选择的微生物群落中找到所需的优良性状,可以极大地提高生物修复水平[36]。合成微生物群落包括具有预期性质的特定菌株的共同培养,在这种设定中,每个微生物都可以部分参与降解途径。因此,可以避免单一菌株的过度工程化及其相关的代谢负担。Jia等[37]成功构建了一个双基因工程菌群落,其中大肠埃希菌HY1被接入4个表达菲双加氧酶的基因,而铜绿假单胞菌(Pseudomonasaeruginosa) PH2中导入一个高效的儿茶酚双加氧酶基因,使两株菌各自负责菲降解途径的一部分,从而共同降解目标污染物,这与单独培养相比,共培养体系有利于菌株的长期共存,对菲的去除效率更高。此外,了解微生物群落成员之间的合作效应可以为发现新的酶和生物降解途径开辟道路,从而实现更高效的污染物去除[38]。
如何保持微生物群落结构在生态学和进化上的稳定性是该领域急需解决的问题之一。最近Schlembach等[42]引入了各种在线和离线监测以及控制混合培养动态的测量技术。例如应用繁殖体策略来评估两个人工构建的细菌群落的可重复性及功能稳定性[43]。最近也有综述总结了用于工程化合成微生物群落的可用工具包,包括群体感应信号分子、诱导剂及共养相互作用[44]。
最近在共培养方法上改进的空间连接微生物群落(SLMC),为建立具有不相容生理需求的合成微生物群落提供了可能 (例如嗜酸菌与嗜碱菌、需氧菌与厌氧菌的共培养),而这是使用传统技术不可能实现的。SLMC涉及到的是工程化培养环境,而不是微生物菌株本身,并在互联模块内共同培养不同种类的微生物[45]。SLMC使不相容菌株之间的代谢物可以进行交换,并在选择微生物群落组成方面提供了更大的灵活性。然而,控制每个模块和通量交换中的群体大小仍具有挑战性,并且限制了该技术的进一步应用[45]。
2.3 土壤的生物修复
土壤修复的物理和化学途径包括土壤挖掘和溶剂清洗,而这可能会破坏土壤微生物菌群结构、养分循环和有机质储存,进而对土壤质量产生负面影响[46]。因此,生物修复作为一种低成本、省人力、省能源、无二次污染的治理方法推广就显得尤为重要。主要土壤污染物生物修复的最新趋势见表2。
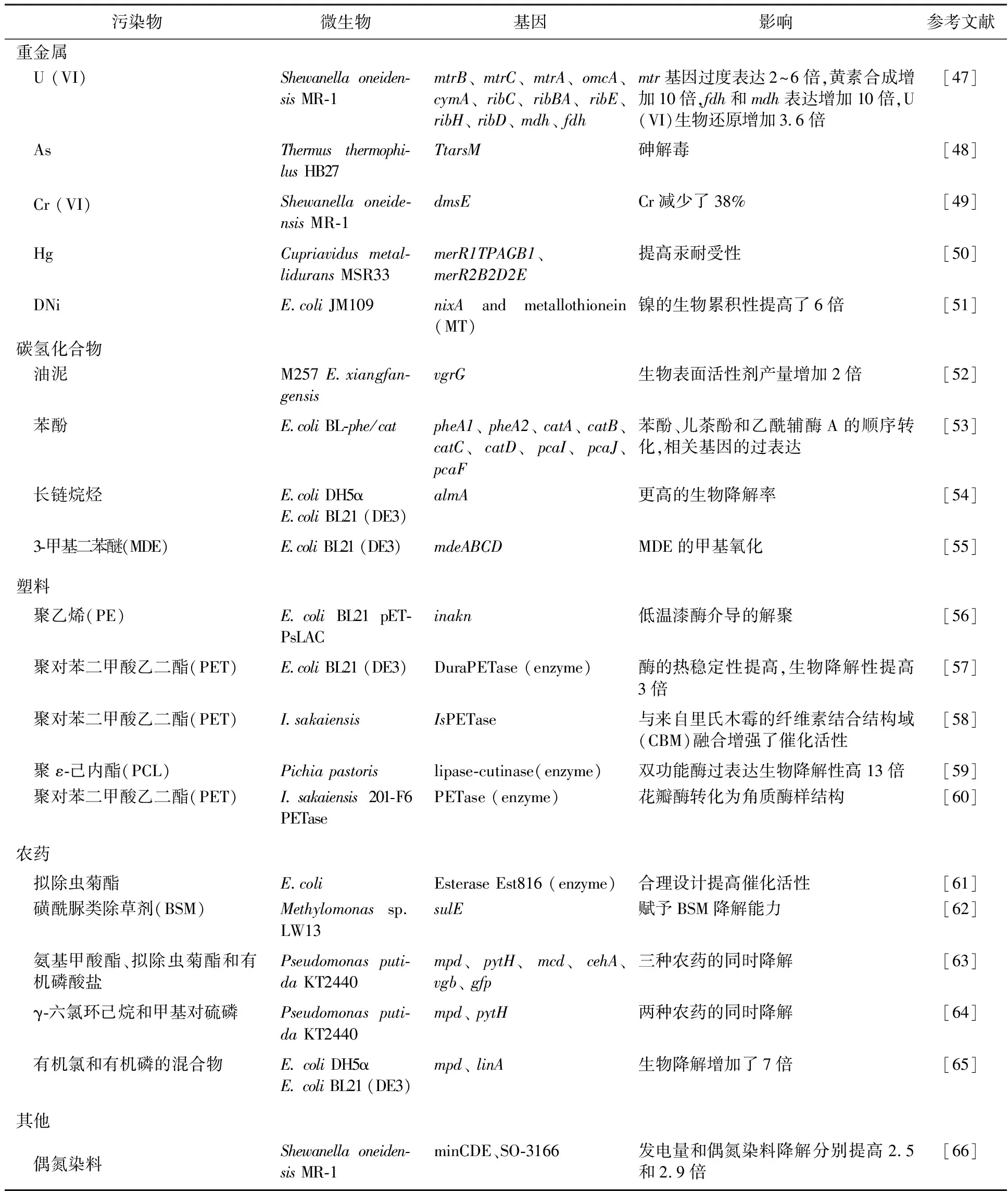
表2 用于土壤生物修复的基因工程微生物
2.3.1 重金属的去除 有多种方法可以减轻重金属对环境的威胁,其中可持续性、环境友好的选择包括生物淋滤(萃取和溶解)、生物吸附、生物还原(纳米颗粒生物合成)、生物矿化(沉淀和固定)、植物修复等。也有一些新型的方法,如绑定多肽和铁载体应用[67]。但所需时间较长而且金属去除率不足限制了这些工艺的大规模应用。因此,合成生物学家开始通过修饰活的有机体系统来应对这些挑战[68]。提高生物修复菌株对重金属的耐受性是提高处理速度和效率的常见做法,其中提高耐受性的方法之一是插入重金属抗性基因。Li等[69]在大肠埃希菌中表达豆梨中的植物螯合素合酶基因PcPCS1,使大肠埃希菌能耐受200 μmol/L的Hg2+,且能够在100 μmol/L的含Hg2+培养基中正常生长。相比于野生型大肠埃希菌只能积累0.49 mg/g的Hg2+,转PcPCS1基因的大肠埃希菌对Hg2+的积累量增加到了0.82 mg/g。最近也发现了几种提高微生物对铜耐受性的工程化策略[70]。而对酸硫杆菌属(Acidithiobacillusspp.)来说,寻找基因重组所需的合适遗传元件是非常重要的。一种已经确定了监测基因表达和调控方法的高效报告基因,可应用于所有酸性硫杆菌[71]。此前,研究人员曾应用遗传工具寻求其他类型的微生物底盘细胞,如异养菌、蓝藻和甲烷氧化菌[50]。合成生物学工具在金属的生物回收中的应用尚处于起步阶段。上述微生物过程的进一步改进可以为绿色有效的修复金属污染和从电子废物等危险固体废物中回收金属提供新的可能。
2.3.2 烃类化合物的降解 合成生物学对烃类化合物生物修复的影响越来越大。烃类化合物包括多环芳烃、石油及其衍生物、六氯环己烷(HCH)、芳香族染料、氯代烷烃、多氯联苯和有机卤化物[72]。通过转基因技术促进多环芳烃等化合物的生物降解策略包括:在基因表达调控的基础上,寻找相关生长与降解基因以及与降解相关的酶基因,提高生物催化剂的活性,并构建新的降解途径[73]。最近,合成生物学工具已成功用于操纵大肠埃希菌中的基因表达,并优化芳香族污染物的生物修复以及硝基烷烃氧化酶(NOE)在两个嗜热细菌中的同源和异源过表达[74]。另外,Wang等[75]在大肠埃希菌BL21中重建了苯酚完全生物降解的人工代谢途径,其中苯酚被苯酚羟化酶降解为儿茶酚,然后通过环裂解转化为粘康酸,最后在三羧酸(TCA)循环中彻底分解。本课题组目前主要以已筛选得到的苯系物高效降解菌为底盘细胞构建多环芳烃降解通路并提高其耐盐能力,使其能够应用于混合污染盐渍化土壤的生物修复[76-77]。能够降解聚氨酯(PU)的微生物包括假单胞菌、芽胞杆菌、不动杆菌、棒状杆菌(Corvnebacterium)等细菌和曲霉(Aspergillus)、拟青霉(Pestalotiopsis)、枝孢霉(Cladosporium)、镰刀菌(Fusarium)、青霉菌(Penicillium)等真菌,其中曲霉菌在PU解聚中显示出最强大的能力[78]。导致PU分解的微生物酶主要有水解酶(蛋白水解酶)、酯酶、尿酶和酰胺酶。
2.3.3 塑料的降解 塑料的微生物降解是一个酶促过程,目前已经分离筛选得到多种降解塑料的微生物,包括放线菌、藻类、细菌和真菌[79]。合成生物学中的不同方法如宏基因组学、克隆和计算方法等都已被用来增强假单胞菌属、埃希氏菌属和芽胞杆菌属物种在塑料酶促解聚中的能力[79]。塑料生物降解的大多数工程研究都集中在修饰编码有效降解酶基因上,假单胞菌是细菌宿主克隆有效降解酶方面研究最多的菌株之一[80]。然而,非细菌宿主最近也吸引了研究者的注意力。聚对苯二甲酸乙二醇酯因其具有较强的解聚性和难水解性,是近20年来生物修复研究者研究最多的塑料。一项新的研究报告表明通过在遗传宿主中表达阪崎氏菌(Sdeonellasakaisis)中的一个关键酶(PETase),实现了三角褐指藻(Phaeodactylumtricornutum)作为藻类遗传宿主成功在低成本生长条件下将聚对苯二甲酸乙二醇酯(PET)降解(图2a)[81]。最近,对角质酶的热稳定性和活性的研究促进了PET的降解,使其生物降解率超过了90% (比以前发现的酶高出33倍)[82]。可以通过分子对接和结合模式分析确定氨基酸残基的突变,从而提高酶的催化活性。为了解释来自垃圾填埋场裂解聚醚-聚氨酯(PE)-PU塑料的微生物群落的作用机制,Gaytán采用了一种新的结合物理和化学分析手段的元基因组方法(基于就近连接)(图2b)[83]。研究结果揭示了每个物种的降解基因和降解酶,为了解每个物种如何增强和促进塑料的生物降解提供了新的思路。最近,Carr等[84]也提供了在本土菌群中确定的聚对苯二甲酸乙二醇酯(PET)水解酶的清单。

图2 合成生物学技术对塑料的降解Fig.2 Degradation of plastics by synthetic biology techniquesa:AP_SP-PETase-FLAG蛋白示意图和PETase-GFP表达图;b:BP8细胞培养后附着在聚醚-聚氨酯-丙烯酸酯上的扫描电子显微镜图a:Schematic of AP_SP-PETase-FLAG protein, and PETase GFP expression diagram;b: Scanning electron microscopy image of BP8 cells attaching to olyether-polyurethane-acrylate after cultivation
2.3.4 农药的降解 近年来,人们应用了不同的策略来提升农药的生物修复效率,一般把重点放在人工选择微生物群落上,利用适应性基因丢失、代谢负荷分解、酶工程和群体感应来加强杀虫剂的去除。Li等[85]开发了一种合成微生物群落(PSC-1)来有效降解七种持久性农药:毒死蜱、吡虫啉、多菌灵、百菌清、λ-氯氟氰菊酯、β-氯氰菊酯和溴氰菊酯[86]。有机磷化合物(OPCs)是广泛分布的一类杀虫剂,在最近的一项研究中,设计了一种有机磷酸酯水解酶(OPH)来改善OPCs的降解。定点突变可以提高OPH水解酶的立体异构选择性。蛋白质融合、固定化和化学修饰等工程方法可以提高其催化性能、酶稳定性和底物亲和力[87]。在某些情况下,可以将一个完整的生物降解途径导入到新菌株并整合进染色体中。Zhao等[88]采用一种新的构建生物降解途径的策略,实现了γ-六氯环己烷(γ-HCH) (一种有机氯农药)和1,2,3-三氯丙烷(一种有毒溶剂)的完全去除[88]。
3 展 望
环境中的污染物类型广泛,往往难以识别和定位。开发新的、快速的、安全的并且具有成本效益的技术来消除它们是非常重要的。生物修复作为污染物去除技术已经非常成功,虽然一些微生物具有较好的降解能力,但由于污染物的持久性,仍然需要对这项技术进行进一步的优化改良。多年来,不同科学领域的进步带来了各种降解技术的改进。系统生物学、分子工具和多组学的知识是合成生物学的基础,它开创了生物修复的新时代。对经过基因和代谢优化的微生物进行分析、设计、构建和微调,可最大限度地降解污染物。
合成生物学技术的发展使得开发生物传感器来检测和监测污染物,了解微生物动力学来构建复合菌群以及创造新的代谢途径或增强酶促作用成为可能。另外,仍然有必要进行更多的现场实验,以支持在实验室获得的不同结果,并为工程化微生物进入环境建立必要的安全参数。生物修复的一个重要瓶颈是现有方法能够降解的污染物范围有限。此外,生物修复可能缓慢且低效,使其在某些应用中不切实际,而生物强化可通过将外源微生物引入到污染环境来帮助克服这些困难。宏基因组研究也有助于加速鉴定新酶和新的微生物。
尽管关键挑战有待解决,但合成生物学仍有很大可能解决环境问题和限制环境危害,同时将其转化为环境安全的物质。我们需要更加努力来加速从目前的传统方法向合成生物学方法的过渡。随着更多合成生物学方法的开发,期望设计更多的微生物来更有效地感知、量化和靶向特定的环境污染物。
