干旱胁迫下甘蓝型油菜籽粒含油量和蛋白质含量变化的全基因组关联分析
2023-02-01胡盛李阳阳唐章林李加纳曲存民刘列钊
胡盛,李阳阳,唐章林,李加纳,曲存民,刘列钊
干旱胁迫下甘蓝型油菜籽粒含油量和蛋白质含量变化的全基因组关联分析
胡盛,李阳阳,唐章林,李加纳,曲存民,刘列钊
西南大学农学与生物科技学院,重庆 400715
【】干旱是甘蓝型油菜生长发育过程中一种常见的非生物胁迫,严重影响了其产量和品质。联合全基因组关联分析和转录组差异表达分析,筛选干旱胁迫条件下影响甘蓝型油菜籽粒含油量和蛋白质含量变化的候选基因,为解释干旱胁迫下甘蓝型油菜籽粒含油量及蛋白质含量的变化提供理论基础。【】利用旱棚盆栽方式模拟干旱胁迫环境,使用183份甘蓝型油菜构成的自然群体,于2019和2020年在模拟干旱胁迫条件下收获2年的籽粒,并进行籽粒含油量以及蛋白质含量的测定,将得到的表型数据与60K芯片的基因型数据(包含34 103个SNP)进行全基因组关联分析,同时,结合相同处理下花后不同干旱时间段(30、40和50 d)的籽粒转录组差异基因数据,筛选共有基因,并利用甘蓝型油菜和拟南芥数据库注释信息、已报道的相关文献和转录组差异基因表达水平鉴定与干旱胁迫下甘蓝型油菜籽粒含油量及蛋白质含量变化相关的候选基因。【】通过对2年间籽粒含油量和蛋白质含量的分析,发现材料重复性较好,干旱胁迫造成甘蓝型油菜籽粒的含油量下降,蛋白质含量上升;全基因组关联分析得出的最佳模型主要为一般线性模型下的Q或naïve模型,共检测出38个显著关联位点(<1/31597或<1/31278),主要分布在A03(6个)、A04(8个)和C03(8个)染色体,各位点分别对含油量和蛋白质含量变化的贡献率均超过10%,其中含油量变化位点最大贡献率为23.97%,蛋白质含量变化位点最大贡献率为22.21%。通过整合全基因组关联分析和转录组分析,筛选出256个共有基因,根据基因注释和文献报道鉴定出与干旱胁迫下含油量及蛋白质含量变化相关的25个候选基因,其中包含转录因子(如bZIP transcription factor GBF6、TALE transcription factor ATH1、MYB-like Domain transcription factor MYBD、NAC transcription factor ANAC029和ERF transcription factor ERF111)、相关激酶(如与油脂相关的蛋白激酶CIPK9、水分胁迫响应激酶PIP5K1和代谢相关激酶PFK7)、相关蛋白(如与油脂相关转运蛋白ABCA9、与蛋白相关的存储蛋白CRU3、胁迫响应蛋白HUP26和M10、叶绿体蛋白DG238和CP12)等,涉及生长发育、光合反应、物质运输等多个生物学过程。【】鉴定出25个相关候选基因,可能响应干旱胁迫,并影响油菜籽粒蛋白质和含油量的积累。
甘蓝型油菜;干旱胁迫;含油量;蛋白质含量;候选基因
0 引言
【研究意义】干旱缺水是陆生植物经常遭受的一种环境胁迫,也是影响农林业生产的重要因素之一,特别是在中国干旱和半干旱地区[1]。据估计,全球约有40%的地区受到干旱的影响,而且还会随着气候的变化而加剧[2]。干旱作为非生物胁迫的一种,对作物生长发育的各个时期均有不同程度的危害[3],如种子萌发期受旱,容易造成发芽率低,出苗不齐[4];苗期受旱,光合性能下降,生长减缓[5];特别是花期受旱,普遍造成花粉活力弱,降低结实率和产量水平,如Yang等[6]发现水稻在花期受到干旱后会明显降低其籽粒产量,相关生理性状也会显著降低;Joshan等[7]发现油料作物红花在花期受到干旱胁迫后,其种子油脂成分含量会发生改变,软脂酸和硬脂酸等饱和脂肪酸显著增加,亚油酸含量减少,从而降低了油料品质。甘蓝型油菜作为中国重要油料作物之一,是中国食用植物油的主要来源。中国植物油自给率严重不足,而干旱胁迫又严重影响菜籽油的产量和品质,对中国食用油供给安全造成重大影响[8-9]。因此,模拟干旱胁迫环境,筛选耐旱种质,鉴定相关候选基因,对保障菜籽油产量和品质具有重要意义。【前人研究进展】基于高通量测序和DNA芯片分析技术的完善以及成本的降低,全基因组关联分析(genome-wide association study,GWAS)是目前研究复杂数量性状的有效方法,已在棉花[10]、大豆[11]、水稻[12]等诸多作物中广泛应用。甘蓝型油菜作为异源四倍体,遗传背景复杂,目前,利用关联分析挖掘出大量与产量、品质、抗逆等复杂数量性状相关的候选基因,为后续基因功能验证及等位基因利用提供依据。Xiao等[13]基于高通量重测序技术,对588份甘蓝型油菜种质的含油量进行全基因组关联分析,鉴定出17个显著SNP位点,并结合转录组数据,筛选出7个与含油量及蛋白质含量相关的候选基因。Tang等[14]对370份油菜籽粒12个性状进行测定,结合60K芯片数据进行全基因组关联分析,检测到7个与种子含油量和蛋白含质量相关的SNP,并在其中筛选到5个相关候选基因。Khanzada等[15]利用15% PEG6000模拟干旱处理,获得228份材料的耐逆指数和敏感指数,利用关联分析鉴定出与耐旱相关的85个候选基因。Zhang等[16]利用PEG6000模拟环境胁迫对140份甘蓝型油菜种质进行处理,全基因组关联分析鉴定出16个显著位点,并结合RNA测序结果联合筛选出79个候选基因,其中8个基因与拟南芥的抗旱基因同源相关。【本研究切入点】鉴于甘蓝型油菜异源四倍体复杂的遗传背景和种子发育复杂的生物学过程,籽粒性状的遗传研究一直是油菜遗传育种研究的重点和难点[14]。前期研究多数是对正常生长发育过程籽粒性状的遗传定位研究,而对干旱逆境胁迫下籽粒品质性状开展遗传定位和候选基因筛选的鉴定工作较少。【拟解决的关键问题】本研究以183份甘蓝型油菜作为分析群体,利用包含34 103个SNP标记的60K芯片数据,以干旱胁迫条件下2年油菜籽粒含油量以及蛋白含量变化为切入点进行全基因组关联分析,同时,结合花后不同干旱时段籽粒转录组数据,联合筛选出与干旱胁迫下甘蓝型油菜籽粒含油量及蛋白质含量变化相关的候选基因,为甘蓝型油菜耐旱育种研究提供依据。
1 材料与方法
1.1 供试材料的种植及处理
从国内外收集的183份甘蓝型油菜种质,包括地方性品种、育成材料和高代材料,其中,国内164份,主要来源于重庆、湖北、四川、江苏等地区,国外19份,来源于德国、加拿大、朝鲜等国家(电子附表1)。于2019年和2020年在重庆市西南大学油菜工程技术研究中心,以盆栽的方式种植在基地旱棚中,光周期同室外条件。每盆称取约6 kg混匀土(田间土﹕腐殖土=3﹕1),幼苗前期采取定量浇水的办法培养至四叶一心期,每个材料选择大小、长势一致的3盆进行干旱处理(drought stress,DS),3盆进行正常灌溉(well watering,WW),在抽薹、开花期间各补充一次水溶肥,等量施用。利用土壤水分测定仪(浙江托普仪器有限公司TZS-1K型)监测含水量,DS组在6%—10%,WW组保持在20%—24%,其他条件与大田生产完全一致。
1.2 籽粒性状的测定及处理
使用福斯(FOSS)近红外分析仪(NIRS DS2500)对所收获2年自交种籽粒进行品质测定,3次重复,分析含油量和蛋白质含量变化情况。利用IBM SPSS Statistics 25对表型数据进行统计学分析。
将相对应材料处理值与正常值(DS/WW)的比值作为该材料的耐旱指数,使用耐旱指数作为表型数据的指标,用于后续的关联分析。
1.3 全基因组关联分析
利用Illumina公司开发的油菜60k芯片对国内油菜育种单位收集的种质资源分析获得的基因型数据[17],按照Qu等[18]方法对标记进行质控,筛选并删除等位基因频率(allele frequency,AF)小于0.05,大于1.0,且SNP得率(call frequency)小于80%的标记,结合2年所获得的可用材料,分别保留31 597个SNP标记和31 278个SNP标记用于后续分析。
利用Tassel 5.2.80软件[19]对处理好的数据进行关联分析,分别在一般线性模型(general linear model,GLM)下进行PCA(主成分)、Q、naïve模型分析以及混合线性模型(mixed linear model,MLM)下的K、K+PCA、K+Q模型分析,利用R语言CMplot包对6个模型下的结果绘制Quantile-quantile(QQ)图,并分析比较观测值与期望值得出最佳模型,利用最佳模型绘制曼哈顿(Manhattan)图,显著性SNP位点筛选的阈值设定为“1/所使用的标记”。
1.4 差异基因分析
使用重庆市油菜工程技术中心提供的在花后不同时段干旱胁迫(30、40和50 d)的籽粒转录组数据[20]。转录组材料为油菜工程技术中心提供的重组自交系群体中6份含油量不同的株系,处理方式同1.1,取材时间定于花期后30、40和50 d,分别收取对照和处理植株的角果,于冰上小心剥取籽粒,将同一阶段的材料籽粒等量混匀,置于-80℃保存,送至北京百迈克生物科技有限公司进行测序。利用Cufflinks2.0.0软件[21]将干旱处理组和正常灌溉组的数据进行差异分析,筛选条件为差异倍数log2|(fold change)|≥1.7,错误发现率(false discovery rate,FDR)<0.01,得到各个比较组的差异基因。
1.5 候选基因鉴定
参考LD分析[22]的衰减距离(上下游500 kb),根据关联分析得到的显著SNP标记筛选候选区间内的所有基因,并联合转录组数据整理得到的差异基因,将同时筛选得到的初步定位相关基因。根据已公布的甘蓝型油菜基因组信息(“Darmor-Bzh”,BRAD (brassicadb.cn))以及拟南芥基因组信息(“Araport11”, TAIR-Home Page (arabidopsis.org)),进一步进行信息比对和功能注释,寻找注释信息中有关于干旱胁迫、含油量变化或蛋白质含量变化相关的基因,并查询相关已报道过的文献,最终确认筛选的甘蓝型油菜在干旱胁迫下响应籽粒含油量及蛋白质含量变化的候选基因。
2 结果
2.1 籽粒含油量及蛋白质含量分析
通过对2019年收获的162份可用材料和2020年收获的128份可用材料进行近红外数据分析(表1),与正常灌溉组(WW)相比,干旱处理组(DS)的含油量水平显著降低,蛋白质含量水平显著升高,2年的变化趋势一致,同时,变异系数结果均小于10,说明群体数据分布较为集中,趋势较好。相关性分析结果显示(表2),同一年份的各处理间相关性显著,而且含油量和蛋白质含量之间呈显著负相关,而不同年份各性状之间的相关性较弱。对耐旱指数分布作直方图(图1),发现2份低含油量高蛋白质含量材料(编号129和编号74)和2份高含油量低蛋白质材料(编号63和编号33)。
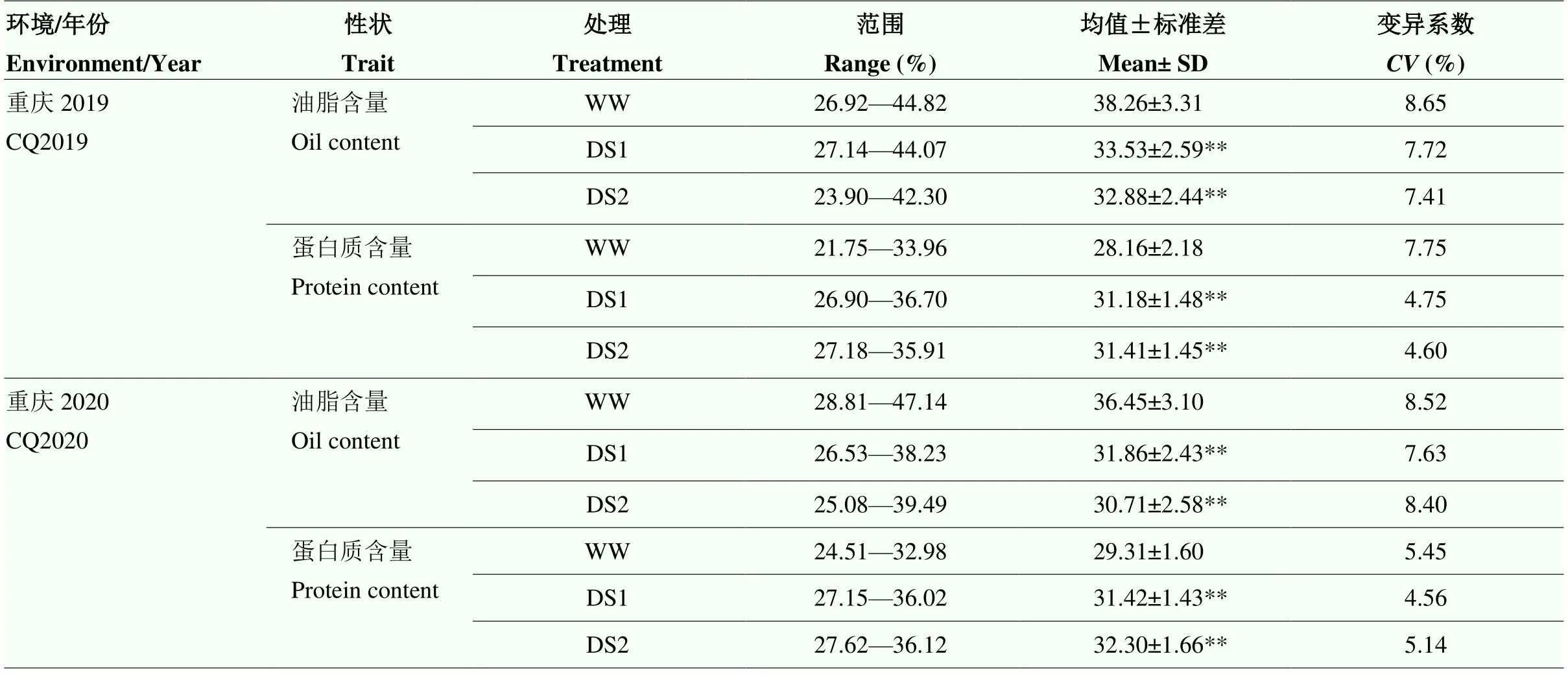
表1 2019及2020年干旱胁迫及正常处理下籽粒含油量和蛋白质含量统计分析
WW:正常灌溉;DS1、DS2分别指同年份处理的2个重复;**表示差异显著(p<0.01)。下同
WW: well watering; DS1 and DS2 refer to two repeats in the same year; ** represents the significant difference (p<0.01). The same as below
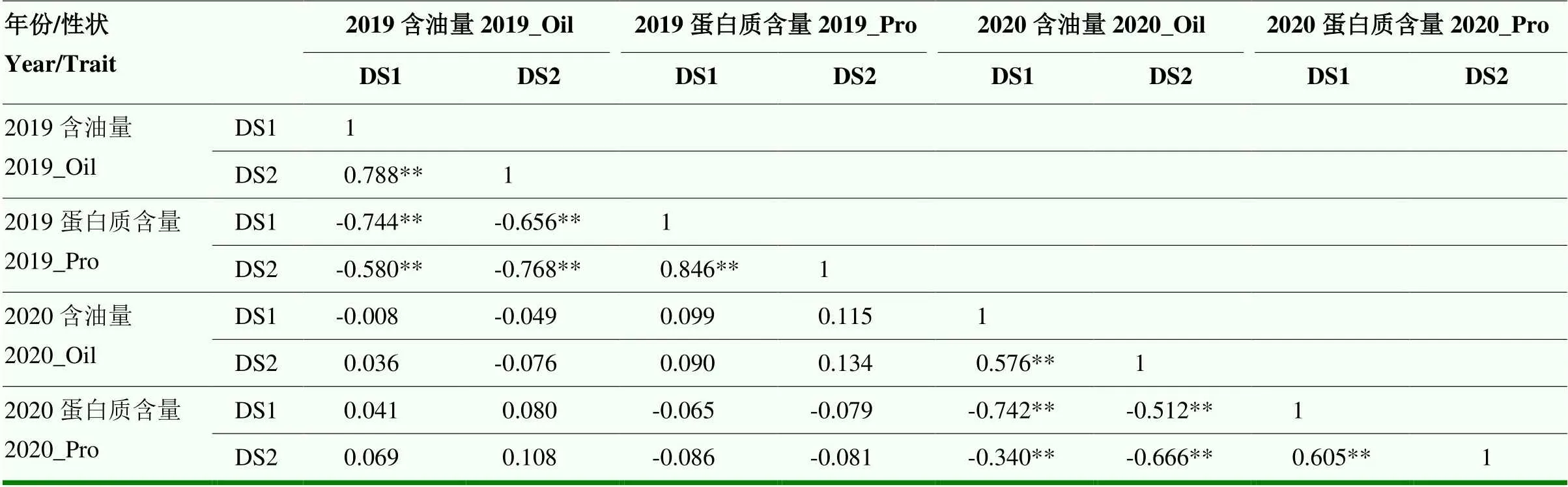
表2 2年各处理间籽粒蛋白质和含油量的相关性分析
2.2 全基因组关联分析
根据不同算法,将6种模型(PCA、Q、naïve、K、K+PCA、K+Q)下对含油量和蛋白质含量响应干旱胁迫变化关联分析得到的结果绘制QQ图,并对-log10()观察值与期望值的相关程度来选择对应的最佳模型,在最佳模型下,利用CQ2019的31 597个和CQ2020的31 278个SNP标记,进行Manhattan图的绘制,并以值小于定义阈值(1/31 597和1/31 278)的点作为显著关联SNP(图2)。
对含油量变化进行分析,在2019和2020年的4个处理中,共重复筛选出16个显著性SNP标记,蛋白质含量变化在2年4个处理下共重复筛选出22个显著性SNP标记(表3)。其中,位于C03染色体上的Bn-scaff_16755_1-p1427195标记在2019年处理二的含油量和蛋白质含量变化中重复筛选到,A03染色体上的Bn-A03- p21505530与Bn-A03-p21505363间仅167 bp,其余部分标记(Bn-scaff_18322_1-p2147199与Bn-scaff_18322_1-p2150739、Bn-A08-p18820218与Bn-A08-p18827698、Bn-A06-p5279490与Bn-A06- p5260549)之间距离为2—9 kb范围内。所有显著性标记对不同年份间的含油量以及蛋白质含量变化的贡献率均超过10%,部分达到20%以上,说明这些标记都可以考虑作为可靠显著性标记。
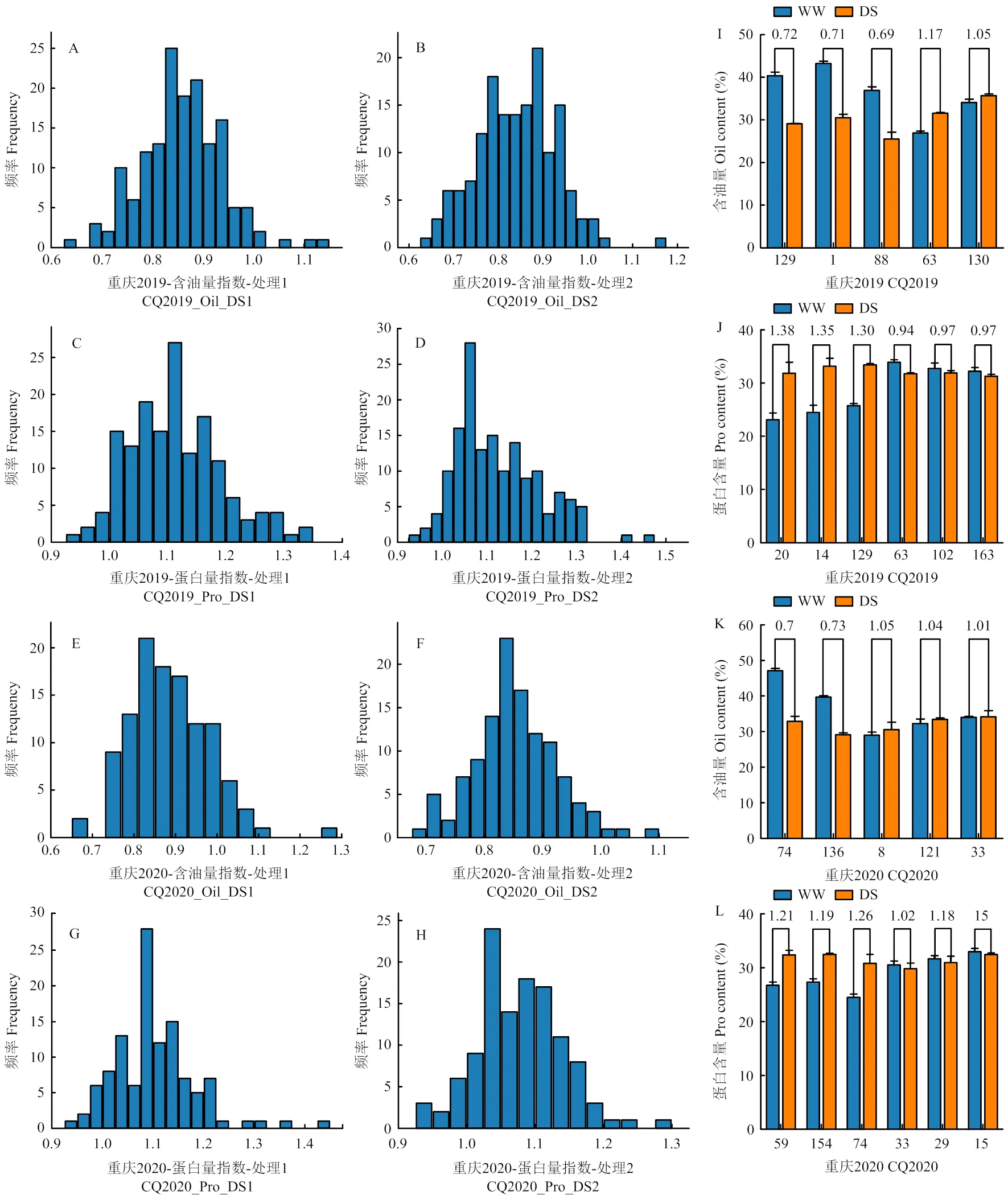
A—H:2019年和2020年含油量和蛋白含量耐旱指数频率分布图;I—L:2019年和2020年含油量和蛋白含量变化共有极端材料,蓝黄柱状图之间的数据表示该材料的耐旱指数
2.3 候选基因鉴定
根据GWAS筛选得出的表3数据,对应油菜基因物理位置信息,共找到3 681个基因,联合花后不同干旱时段籽粒转录组数据整理得出6 571个差异基因,筛选出256个共有相关基因(图3),参照油菜和拟南芥的基因组注释信息以及相关文献,从注释信息以及报道的文献中进一步筛选与含油量变化、蛋白质含量变化以及干旱胁迫相关的基因或拟南芥同源基因,参考转录组差异基因的表达水平变化,最终筛选出25个与干旱胁迫下甘蓝型油菜籽粒含油量和蛋白含量变化相关的主要候选基因(表4),这些基因主要包括编码一些与油脂以及蛋白相关的激酶和蛋白,功能涉及光合作用、物质储藏、胁迫响应等多个方面,其中包含一些重要转录因子,如、、、等。同时,根据该25个候选基因在转录组中的表达水平热图(图4),可以发现这些基因在不同时段的对照和处理中均有不同的表达,且在某一阶段内出现显著程度的表达差异,如、、等在干旱处理花后30 d就有显著表达差异;、、等在干旱处理花后40 d出现显著表达差异;、、等则在50 d出现显著差异,说明这些基因可能在不同的时间点对干旱处理条件下籽粒含油量和蛋白质含量变化起到一定的调控作用。
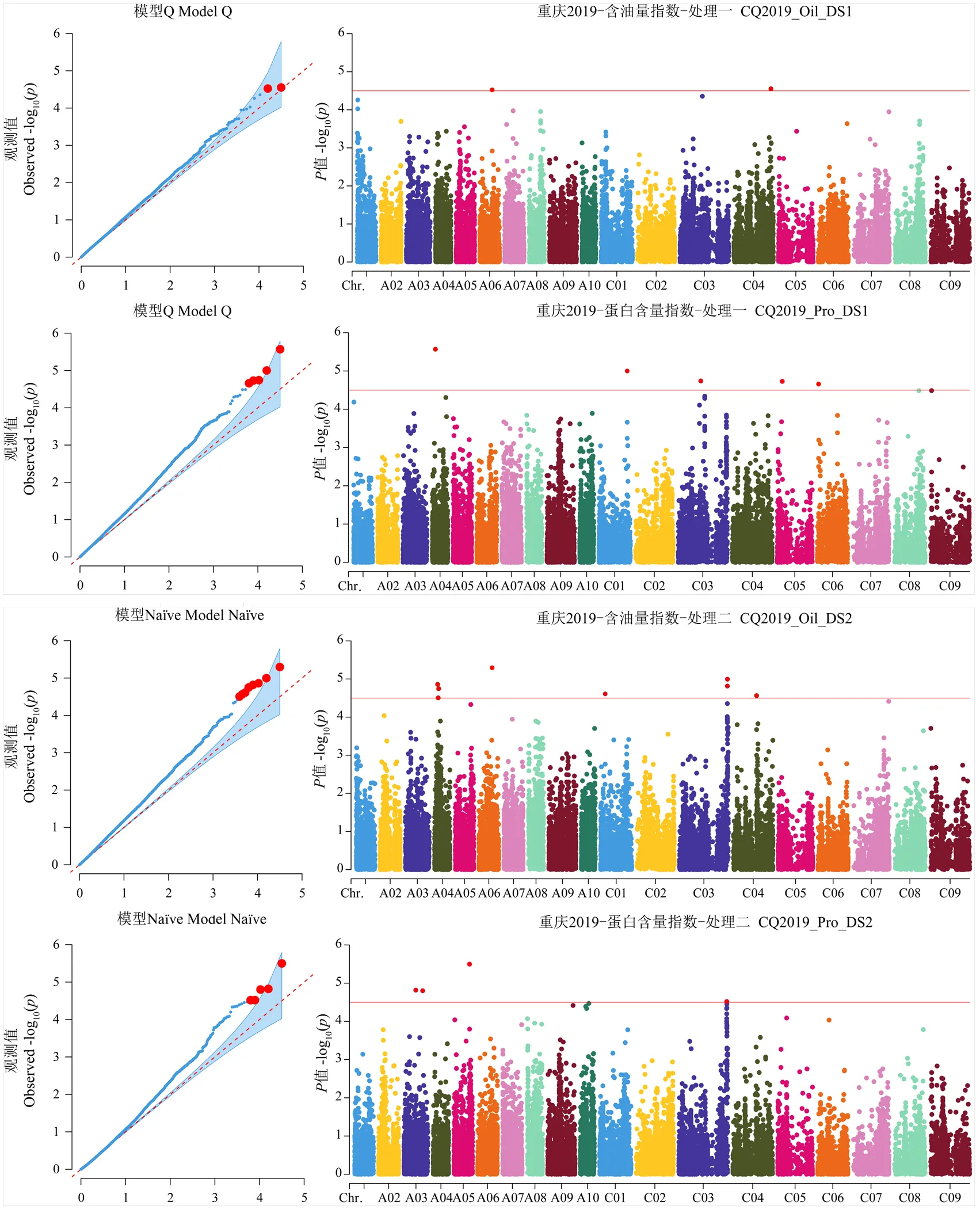
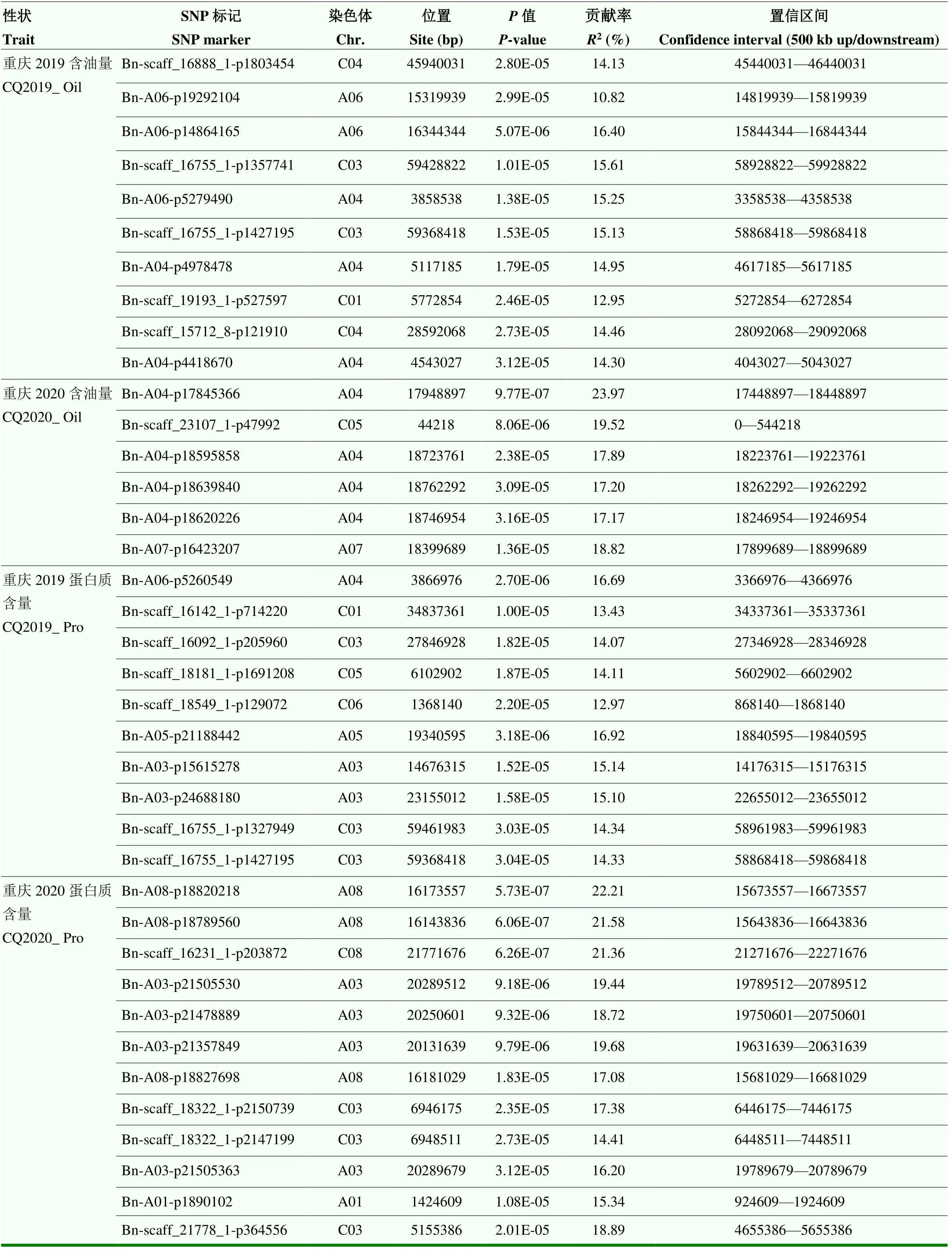
表3 干旱胁迫下油菜籽粒含油量和蛋白质含量变化显著关联的SNP位点

DAF30、DAF40、DAF50:分别代表花后30、40和50 d的差异基因集
3 讨论
3.1 GWAS和转录组联合分析
非生物胁迫影响作物的生长发育,特别是干旱胁迫,导致作物的产量和品质大幅降低。甘蓝型油菜作为中国主要的重要油料作物,有着不可或缺的价值地位[22-23]。随着气候的变化,干旱胁迫对甘蓝型油菜的生产造成了严重的影响[24-25]。因此,开展干旱胁迫条件下甘蓝型油菜含油量以及蛋白质含量变化相关性状的位点以及基因挖掘对增强甘蓝型油菜在干旱环境下耐受性、提高籽粒产量和品质具有重要意义。
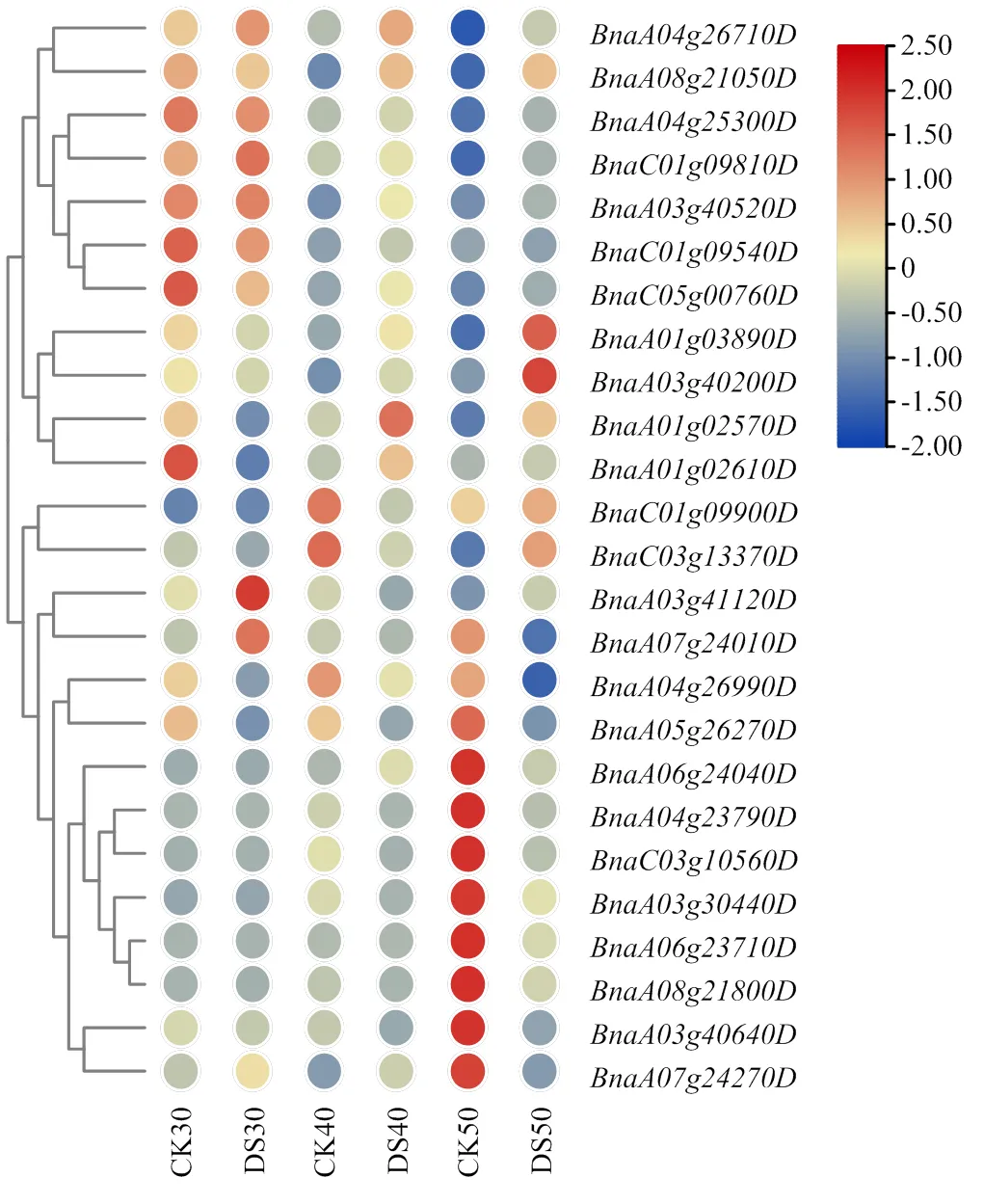
CK30、CK40、CK50:对照组花后30、40和50 d候选基因表达水平;DS30、DS40、DS50:干旱处理组花后30、40和50 d候选基因表达水平
Choudhury等[26]利用90K芯片数据对335份小麦材料进行抗病性全基因组关联分析,并在一般线性模型下的K模型中鉴定出4个重复位点,为小麦抗病育种提供依据;Zheng等[27]利用全基因组关联分析对水稻籽粒宽度变异程度进行研究,并在一般线性模型和混合线性模型下共同鉴定出7个候选位点,为候选基因筛选给出思路;Pace等[28]利用多态性标记对玉米384个自交系进行GWAS分析,分别在一般线性模型和混合线性模型下鉴定出263个和4个与根系发育性状相关的显著标记,并说明结合一般线性与混合线性2种模型的计算方式才是关联分析优选的方案。这些研究均将全基因组关联分析中的GLM和MLM的结果比较整合,并从中找出最相关的显著位点,极大地控制了假阳性的情况。本研究也利用2种模型用于关联分析,最终得到的最佳模型均在一般线性模型下产生,并得到了较多的显著SNP标记(38个)和较大的贡献率(10.82%— 23.97%)。另外,Liu等[29]对521份油菜籽粒含油量的全基因组关联分析中鉴定出50个显著相关位点,其中,Bn-A03-p21298611、Bn-A04-p18354325、Bn-A05- p21491115、Bn-scaff_16888_1-p1561387等位点与本研究中Bn-A03-p21357849、Bn-A04-p18595858、Bn-A05- p21188442、Bn-scaff_16888_1-p1803454位点存在重叠区间,且Bn-A03-p21357849和Bn-A05-p21188442位点是基于蛋白质含量变化检测,其原因可能说明含油量和蛋白质含量之间的协同变化可能通过相同位点实现;Fletcher等[30]对甘蓝型油菜的耐旱性进行QTL分析,鉴定出5条染色体上多个QTL区间,其中,A03中2个显著区间与本研究中Bn-A03-p15615278、Bn- A03-p21505530、Bn-A03-p21478889、Bn-A03-p21357849、Bn-A03-p21505363位点存在重叠区间。从抗旱性和含油量相关的分析中均可在本研究中鉴定出部分位点,为位点的可靠性提供了证据。除此之外,为了降低假阳性,研究还利用同等处理条件下的籽粒转录组数据,参考了干旱条件下花后30、40和50 d的差异表达基因结果,进一步了解候选基因的表达情况。通过整合GWAS和转录组差异分析结果,共找出256个共有基因,这大大缩小了筛选范围,同时根据注释信息和报道文献进一步从中筛选出25个可能与干旱胁迫下甘蓝型油菜籽粒含油量以及蛋白质含量变化相关的候选基因。
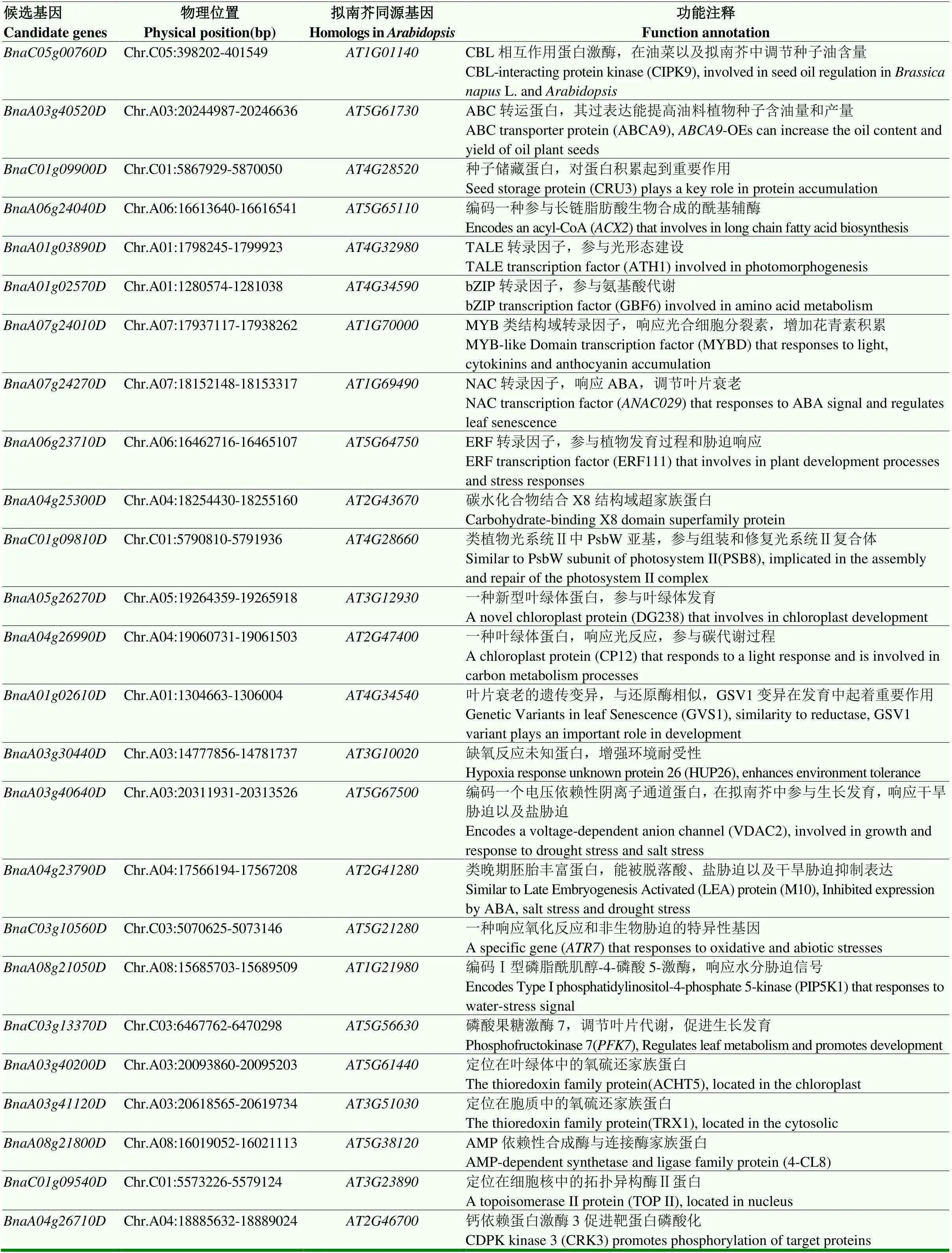
表4 候选基因鉴定
3.2 干旱胁迫下甘蓝型油菜籽粒成份变化候选基因分析
甘蓝型油菜籽粒是由多成份构成、多通路参与、多基因控制。在以前的一些研究中,Li等[20]对油菜籽粒油体的扫描电镜观察和含油量测定发现干旱胁迫会明显降低油菜籽粒中的油脂含量;油菜籽粒油脂含量的下降是由于环境中干旱和盐分的影响,这不仅会造成菜籽油的品质下降,还会增加其本身硫代葡萄糖苷和硬脂酸的含量[31-33];另外,Mohammadi等[34]发现干旱缺水的条件对作物的负面影响还存在于代谢方面,因为合成油的碳水化合物的减少以及体内油脂氧化反应的增加,造成生物体内的合成代谢活动减少,从而造成含油量方面的降低。本研究筛选出部分与含油量相关的基因,其中[35]负责编码与糖相关的蛋白激酶,能被外伤应激诱导表达,拟南芥中负调控脂质积累,油菜中该基因的过表达也会降低油脂的合成,并且在花后30 d的转录组的差异表达的基因中,该基因的表达显著下降,符合分析的结果;另外,还有一种编码ABC转运蛋白的基因[36],其过表达会增加亚麻荠种子的油脂含量,目前,在油菜中是否也存在这种功能还未得到具体的研究证实,在转录水平中,该基因在干旱处理花后40和50 d的表达出现上调,这说明该基因在协调含油量变化中起到一定的作用。除此之外,其他与环境胁迫(ERF111、M10、PIP5K1等)和氧化反应(如HUP26、ACHT5、TRX1等)相关的蛋白在本研究中也有被鉴定,共同解释干旱胁迫下油菜籽粒含油量降低的原因。
早期研究发现,油料作物中油脂含量和蛋白含量之间存在着负相关关系[37-38],这与本研究考察的籽粒性状一致。本研究鉴定出一种种子储存蛋白CRU3[39],是油菜籽粒中一种重要的12S球蛋白,主要为籽粒积累氮源,为油菜萌发提供能量。在表达水平上,该基因干旱处理花后30 d的表达水平未表现出明显变化,但在50 d表现出明显上调,说明种子的发育后期该基因可能对12S球蛋白的积累起到一定作用。这些也可以认为是籽粒含油量下降的协同原因之一。
ABA作为植物胁迫响应中常见的激素之一,参与调节植物生长发育的众多方面[40]。本研究鉴定出的部分基因也参与ABA的调控,如ANAC029[41],一类叶片衰老相关的转录因子,能被ABA诱导表达,加速衰老。除此之外,油脂生物合成也离不开光合产物的积累,Tang等[42]发现通过参与叶绿体的发育,调节光合作用,而且过表达该基因会增加种子含油量。转录组学[43]和蛋白质组学[44]研究说明不少差异结果富集的通路与光合作用相关,为其参与油脂的积累提供了证据。本研究中也有很多基因和蛋白涉及光合作用,参与叶绿体的发育或响应光信号或与光系统有关,如ATH1、PSB8、DG238、CP12等,这都为干旱胁迫下甘蓝型油菜籽粒含油量下降,蛋白质含量增加提供了一定的参考依据。
4 结论
干旱胁迫造成甘蓝型油菜含油量下降,蛋白质含量上升,2年籽粒干旱表型数据的全基因组关联分析鉴定出38个与干旱胁迫下甘蓝型油菜籽粒含油量与蛋白质含量变化显著关联的SNP位点,结合干旱处理下花后不同时段的籽粒转录组数据,共筛选出256个相关基因,利用甘蓝型油菜和拟南芥数据库的基因功能注释,参考已报道文献和差异基因
表达水平,进一步鉴定出可能在干旱胁迫条件下籽粒含油量及蛋白含量变化过程中起重要作用的25个候选基因。
[1] 王忠. 植物生理学. 北京: 中国农业出版社, 1999: 451-455.
Wang Z. Plant Physiology. BeiJing: China Agriculture Press, 1999: 451-455. (in chinese)
[2] Zhang X K, Lu G Y, Long W H, Zou X L, Li F, Nishio T. Recent progress in drought and salt tolerance studies incrops. Breeding Science, 2014, 64(1): 60-73.
[3] Dietz K J, Zörb C, Geilfus C M. Drought and crop yield. Plant Biology, 2021, 23(6): 881-893.
[4] Wijewardana C, Reddy K R, Krutz L J, Gao W, Bellaloui N. Drought stress has transgenerational effects on soybean seed germination and seedling vigor. PLoS One, 2019, 14(9): e0214977.
[5] Zhang Y B, Yang S L, Dao J M, Deng J, Shahzad A N, Fan X, Li R D, Quan Y J, Bukhari S A H, Zeng Z H. Drought- induced alterations in photosynthetic, ultrastructural and biochemical traits of contrasting sugarcane genotypes. PLoS One, 2020, 15(7): e0235845.
[6] Yang X L, Wang B F, Chen L, Li P, Cao C G. The different influences of drought stress at the flowering stage on rice physiological traits, grain yield, and quality. Scientific Reports, 2019, 9: 3742.
[7] Joshan Y, Sani B, Jabbari H, Mozafari H, Moaveni P. Effect of drought stress on oil content and fatty acids composition of some safflower genotypes. Plant Soil and Environment2019, 65(11): 563-567.
[8] HATZIG S V, NUPPENAU J N, SNOWDON R J, SCHIEßL S V. Drought stress has transgenerational effects on seeds and seedlings in winter oilseed rape (L.). BMC Plant Biology, 2018, 18(1): 297.
[9] Hu Q, Hua W, Yin Y, Zhang X, Liu L, Shi J, Zhao Y, Qin L, Chen C, Wang H. Rapeseed research and production in China. The Crop Journal, 2017, 5(2): 127-135.
[10] Li B Q, Chen L, Sun W N, Wu D, Wang M J, Yu Y, Chen G X, Yang W N, Lin Z, Zhang X L, DUAN L F, YANG X Y. Phenomics-based GWAS analysis reveals the genetic architecture for drought resistance in cotton. Plant Biotechnology Journal, 2020, 18(12): 2533-2544.
[11] Wen L W, Chang H X, Brown P J, Domier L L, Hartman G L. Genome-wide association and genomic prediction identifies soybean cyst nematode resistance in common bean including a syntenic region to soybean Rhg1 locus. Horticulture Research, 2019, 6: 9.
[12] Huang X H, Wei X H, Sang T, Zhao Q, Feng Q, Zhao Y, Li C Y, Zhu C R, Lu T T, Zhang Z W, LI M, FAN D L, GUO Y L, WANG A H, WANG L, DENG L W, LI W J, LU Y Q, WENG Q J, LIU K Y, HUANG T, ZHOU T Y, JING Y F, LI W, LIN Z, BUCKLER E S, QIAN Q, ZHANG Q F, LI J Y, HAN B. Genome-wide association studies of 14 agronomic traits in rice landraces. Nature Genetics, 2010, 42(11): 961-967.
[13] Xiao Z C, Zhang C, Tang F, Yang B, Zhang L Y, Liu J S, Huo Q, Wang S F, Li S T, Wei L J, DU H, QU C M, LU K, LI J N, LI N N. Identification of candidate genes controlling oil content by combination of genome-wide association and transcriptome analysis in the oilseed crop. Biotechnology for Biofuels, 2019, 12: 216.
[14] Tang M Q, Zhang Y, Liu Y, Tong C, Cheng X, Zhu W, Li Z, Huang J, Liu S. Mapping loci controlling fatty acid profiles, oil and protein content by genome-wide association study in. The Crop Journal, 2019, 7(2): 217-226.
[15] Khanzada H, Wassan G M, He H, Mason A S, Keerio A A, Khanzada S, Faheem M, Solangi A M, Zhou Q, Fu D, HUANG Y, RASHEED A. Differentially evolved drought stress indices determine the genetic variation ofat seedling traits by genome-wide association mapping. Journal of Advanced Research, 2020, 24: 447-461.
[16] Zhang J, Mason A S, Wu J, Liu S, Zhang X C, Luo T, Redden R, Batley J, Hu L Y, Yan G J. Identification of putative candidate genes for water stress tolerance in canola (). Frontiers in Plant Science, 2015, 6: 1058.
[17] Xu L P, Hu K N, Zhang Z Q, Guan C Y, Chen S, Hua W, Li J N, Wen J, Yi B, Shen J X, MA C Z, TU J X, FU T D. Genome-wide association study reveals the genetic architecture of flowering time in rapeseed (L.). DNA Research, 2015, 23(1): 43-52.
[18] Qu C M, Li S M, Duan X J, Fan J H, Jia L D, Zhao H Y, Lu K, Li J N, Xu X F, Wang R. Identification of candidate genes for seed glucosinolate content using association mapping inL.. Genes (Basel), 2015, 6: 1215-1229.
[19] Bradbury P J, Zhang Z W, Kroon D E, Casstevens T M, Ramdoss Y, Buckler E S. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics, 2007, 23(19): 2633-2635.
[20] Li Y, Zhang L, Hu S, Zhang J, Wang L, Ping X, Wang J, Li J, Lu K, Tang Z, Liu L. Transcriptome and proteome analyses of the molecular mechanisms underlying changes in oil storage under drought stress inL.. GCB Bioenergy, 2021, 13: 1071-1086.
[21] Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley D R, Pimentel H, Salzberg S L, Rinn J L, Pachter L. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature Protocols, 2012, 7(3): 562-578.
[22] Lu K, Wei L J, Li X L, Wang Y T, Wu J, Liu M, Zhang C, Chen Z Y, Xiao Z C, Jian H J, CHENG F, ZHANG K, DU H, CHENG X C, QU C M, QIAN W, LIU L Z, WANG R, ZOU Q Y, YING J M, XU X F, MEI J Q, LIANG Y, CHAI Y R, TANG Z L, WAN H F, NI Y, HE Y J, LIN N, FAN Y H, SUN W, LI N N, ZHOU G, ZHENG H K, WANG X W, PATERSON A H, LI J N. Whole-genome resequencing revealsorigin and genetic loci involved in its improvement. Nature Communications, 2019, 10: 1154.
[23] Raza A, Razzaq A, Mehmood S S, Hussain M A, SU W, HUANG H, Zaman Q U, ZHANG X K, CHENG Y, Hasanuzzaman M. Omics: The way forward to enhance abiotic stress tolerance inL.. GM Crops & Food, 2021, 12(1): 251-281.
[24] Batool M, El-Badri A M, Hassan M U, YANG H Y, WANG C Y, YAN Z K, KUAI J, BO W, Zhou G S. Drought stress in: Effects, tolerance mechanisms, and management strategies. Journal of Plant Growth Regulation, 2022: 1-25.
[25] Bianchetti G, Clouet V, Legeai F, Baron C, Gazengel K, Carrillo A, Manzanares-Dauleux M J, Buitink J, Nesi N. RNA sequencing data for responses to drought stress and/or clubroot infection in developing seeds of. Data in Brief, 2021, 38: 107392.
[26] Choudhury S, Larkin P, Xu R G, Hayden M, Forrest K, Meinke H, Hu H L, Zhou M X, Fan Y. Genome wide association study reveals novel QTL for barley yellow dwarf virus resistance in wheat. BMC Genomics, 2019, 20(1): 891.
[27] Zheng X M, Gong T, Ou H L, Xue D Y, Qiao W H, Wang J R, Liu S, Yang Q W, Olsen K M. Genome-wide association study of rice grain width variation. Genome, 2018, 61(4): 233-240.
[28] Pace J, Gardner C, Romay C, Ganapathysubramanian B, Lübberstedt T. Genome-wide association analysis of seedling root development in maize (L.). BMC Genomics, 2015, 16(1): 47.
[29] Liu S, Fan C C, Li J N, Cai G Q, Yang Q Y, Wu J, Yi X Q, Zhang C Y, Zhou Y M. A genome-wide association study reveals novel elite allelic variations in seed oil content of. Theoretical and Applied Genetics, 2016, 129(6): 1203-1215.
[30] Fletcher R S, Herrmann D, Mullen J L, Li Q F, Schrider D R, Price N, Lin J J, Grogan K, Kern A, McKay J K. Identification of polymorphisms associated with drought adaptation QTL inby resequencing. G3 (Bethesda), 2016, 6(4): 793-803.
[31] Sabagh E A, Hossain A, barutçular C, Islam M S, Ratnasekera D, Kumar N, Meena R S, Gharib H, Saneoka H, Teixeira d S J. Drought and salinity stress management for higher and sustainable canola (L.) production: A critical review. Australian Journal of Crop Science, 2019, 13: 88-97.
[32] Zahedi H, Tohidi Moghadam H R. Effect of drought stress on antioxidant enzymes activities with zeolite and selenium application in canola cultivars. Research on Crops, 2011, 12: 388-392.
[33] Zamani S, Nezami M T, Habibi D, Khorshidi m. Effect of quantitative and qualitative performance of four canola cultivars (L.) to salinity conditions. Advances in Environmental Biology, 2010, 4: 422-427.
[34] Mohammadi M, Ghassemi-Golezani K, Chaichi M R, Safikhani S. Seed oil accumulation and yield of safflower affected by water supply and harvest time. Agronomy Journal, 2018, 110(2): 586-593.
[35] Guo Y L, Huang Y, Gao J, Pu Y Y, Wang N, Shen W Y, Wen J, Yi B, Ma C Z, Tu J X, FU T D, ZOU J T, SHEN J X. CIPK9 is involved in seed oil regulation inL. and(L.) Heynh. Biotechnology for Biofuels, 2018, 11: 124.
[36] Cai G Q, Wang G L, Kim S C, Li J W, Zhou Y M, Wang X M. Increased expression of fatty acid and ABC transporters enhances seed oil production in camelina. Biotechnology for Biofuels, 2021, 14(1): 49.
[37] GRAMI B, STEFANSSON B R, BAKER R J. Genetics of protein and oil content in summer rape: heritability, number of effective factors, and correlations. Canadian Journal of Plant Science, 1977, 57(3): 937-943.
[38] Goffman F D, Alonso A P, Schwender J, Shachar-Hill Y, Ohlrogge J B. Light enables a very high efficiency of carbon storage in developing embryos of rapeseed. Plant Physiology, 2005, 138(4): 2269-2279.
[39] Kwak J S, Kim S I, Park S W, Song J T, Seo H S. E3 SUMO ligase AtSIZ1 regulates the cruciferin content ofseeds. Biochemical and Biophysical Research Communications, 2019, 519(4): 761-766.
[40] Xiong J L, Dai L L, Ma N, Zhang C L. Transcriptome and physiological analyses reveal that AM1 as an ABA-mimicking ligand improves drought resistance in. Plant Growth Regulation, 2018, 85(1): 73-90.
[41] Koeslin-Findeklee F, Rizi V S, Becker M A, Parra- Londono S, Arif M, Balazadeh S, Mueller-Roeber B, Kunze R, Horst W J. Transcriptomic analysis of nitrogen starvation- and cultivar-specific leaf senescence in winter oilseed rape (L.). Plant Science, 2015, 233: 174-185.
[42] Tang S, Peng F, Tang Q, Xia H, Yao X, Lu S, Guo L. BnaPPT1 is essential for chloroplast development and seed oil accumulation in. Journal of Advanced Research, 2022, 42: 29-40.
[43] Huang K L, Zhang M L, Ma G J, Wu H, Wu X M, Ren F, Li X B. Transcriptome profiling analysis reveals the role of silique in controlling seed oil content in. PLoS One, 2017, 12(6): e0179027.
[44] Zhou Z J, Lin B G, Tan J J, Hao P F, Hua S J, Deng Z P. Tandem mass tag-based quantitative proteomics reveals implication of a late embryogenesis abundant protein (BnLEA57) in seed oil accumulation inL.. Frontiers in Plant Science, 2022, 13: 907244.
Genome-Wide association Analysis of the Changes in oil content and protein content under drought Stress inL.
HU Sheng, LI YangYang, TANG ZhangLin, LI JiaNa, QU CunMin, LIU LieZhao
College of Agronomy and Biotechnology, Southwest University, Chongqing 400715
【】 Drought is one of the most adverse abiotic stresses that hinder growth and development ofL., threatening its yield and quality. In this study, we identified the candidate genes that influence oil and protein content changes according to the results of the genome wide association mapping (GWAS) and differentially expressed genes (DEGs) under drought stress in. 【】 A natural population comprising 183lines under drought stress across two years was genotyped with a Brassica 60K Illumina Infinium SNP array to perform GWAS. Combined with drought stressed seed transcriptome data from different treatment periods, the candidate genes related to the changes in oil and protein contents under drought stress were identified. 【】 The analysis showed that the best model for phenotypic data analysis in 2 years was mainly the Q or naïve model, and a total of 38 significant associated loci were detected (<1/31597 or<1/31278). By integrating GWAS and transcriptome results, 256 common genes were selected, and by comparing the database information ofand, 25 related candidate genes were identified. These genes, including transcription factors (e.g., bZIP transcription factor GBF6, TALE transcription factor ATH1, MYB-like Domain transcription factor MYBD, NAC transcription factorANAC029, ERF transcription factor ERF111), related kinases (e.g., PIP5K1, PFK7), related proteins (e.g., seed stores protein CRU3, chloroplast protein DG238 and CP12, stress-responsive proteins HUP26 and M10), are involved in multiple biological processes such as photosynthetic reaction, substance transport, and stress response. For instance, oil-related protein kinase CIPK9, ABCA9 transporter, storage-related protein CRU3, may provide basis for explaining the changes in oil and protein contents ofunder drought stress. 【】 The 25 identified candidate genes may affect the accumulation of protein and oil contents under drought stress during the seed development in.
L.; candidate genes; drought stress; oil content; protein content

2022-08-22;
2022-10-21
重庆市研究生科研创新项目(CYS21122)、国家自然科学基金(31971902,32001509)
胡盛,E-mail:916340758@qq.com。通信作者刘列钊,E-mail:liezhao@swu.edu.cn
(责任编辑 李莉)
