VNN1基因在急性髓系白血病中的表达及临床意义
2023-01-29吴八路梁雨星周芙玲
吴八路,梁雨星,周芙玲
(武汉大学中南医院血液内科,湖北 武汉 430000)
急性髓性白血病(acute myeloid leukemia,AML)是成人最常见的恶性髓性疾病[1]。据统计,2020年全球新发白血病约占新发肿瘤的2.5%,而死亡比例约为3.1%[2]。目前,AML患者总体生存率不足20%[3]。获得性染色体异常是AML最重要的独立预测因素[1,4]。然而,对于细胞遗传学正常的AML患者目前尚没有较好的风险预测因子[5]。因此,寻找新的生物标志物具有重要的临床意义。VNN1(Vanin-1)是一种糖基磷脂酰肌醇锚定表面分子[6]。它在多种疾病中发挥了重要作用[7]。而VNN1在AML中研究较少。本研究主要分析VNN1在AML中的表达和其对预后的影响,探索其临床价值,为进一步研究VNN1在AML中的作用机制提供理论基础。
1 资 料 与 方 法
1.1RNA测序数据和生物信息学分析 通过癌症基因图谱(the cancer genome atlas,TCGA)、组织特异性基因表达库(the genotype-tissue expression,GTEx)和高通量基因表达数据集(gene expression omnibus,GEO)检索并下载急性髓系白血病和正常对照者骨髓细胞的原始转录组测序数据(level 3,HTSeq-FPKM)和患者的临床信息。利用R 3.6.3软件工具提取VNN1基因的mRNA表达量,采用Wilcoxon秩和检验分析两组之间基因表达水平的差异。取AML患者骨髓细胞和(或)外周血单个核细胞中VNN1表达值的中位数,将AML患者划分为高表达组和低表达组,分析VNN1基因表达水平与患者临床特征之间的关系。
1.2VNN1基因表达与患者预后关系的分析 下载TCGA数据库中AML患者的生存随访数据,结合VNN1基因的表达水平,使用乘积限法(Kaplan-Meier,K-M)分析高表达组与低表达组患者总体生存率。采用二分类Logistics模型分析自变量VNN1表达对AML患者临床特征的影响。采用单因素和多因素Cox回归分析研究VNN1基因在AML中的预后价值。
1.3蛋白-蛋白相互作用(protein-protein interaction networks,PPI) 网络构建和功能富集分析String数据库(https://string-db.org/)是一个用于搜索已知蛋白质和预测蛋白质相互作用的开放数据库,该数据库可应用于2 031个物种,是目前覆盖物种最多,相互作用信息最大的一个数据库。本研究利用STRING 11.5在线数据库分析VNN1 PPI网络,探索其可能的分子机制。搜索时基因名称栏输入“VNN1”,物种类型选择“homo sapiens”,置信度选择中度置信“0.400”,截点值选择“不超过10个节点”。利用STRING 11.5在线分析工具对相互作用基因进行基因本体论(Gene Ontology,GO)功能及京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)的富集分析。
1.4免疫细胞浸润的相关性 分析通过单样本基因集富集分析 (single sample gene set enrichment analysis,ssGSEA) 来评估VNN1与免疫细胞浸润之间的相关性。共获得了24种浸润相关性免疫细胞。采用Spearman相关性分析方法分析了VNN1表达水平与24种免疫细胞富集评分之间的相关性。采用Wilcoxon秩和检验分析高、低VNN1表达组的富集分数。
1.5统计学方法 应用SPSS 24.0统计软件处理数据,计量资料采用t检验,计数资料采用χ2检验,Logistics回归分析临床病理特征与VNN1之间的关系。使用Kaplan-Meier模型评估TCGA中AML患者的预后因素。使用Cox比例风险模型进行单变量和多变量分析以估计VNN1表达对临床特征与总体生存期的影响。两组间生存率比较采用Lon-Rank检验,P<0.05为差异有统计学意义。
2 结 果
2.1VNN1基因在常见肿瘤中的表达 通过TCGA数据库,结合GTEx数据库,显示VNN1基因在不同类型肿瘤表达不一,如肾上腺皮质癌、乳腺癌、胆管癌、弥漫大B细胞淋巴瘤、肝细胞癌等肿瘤中都是低表达的,而在胶质瘤、结肠癌、胰腺癌、急性髓系白血病、卵巢癌、胃癌等肿瘤中则是高表达的,且在AML中显著高表达(3.306±1.991)(图1A、B)。多个不同的GEO数据集显示,VNN1在FMS样酪氨酸激酶3内部串联突变(FMS-like tyrosine kinease 3-internal tandem duplication,FLT3-ITD)的AML患者中表达水平显著高于未发生突变者(图1C~E)。
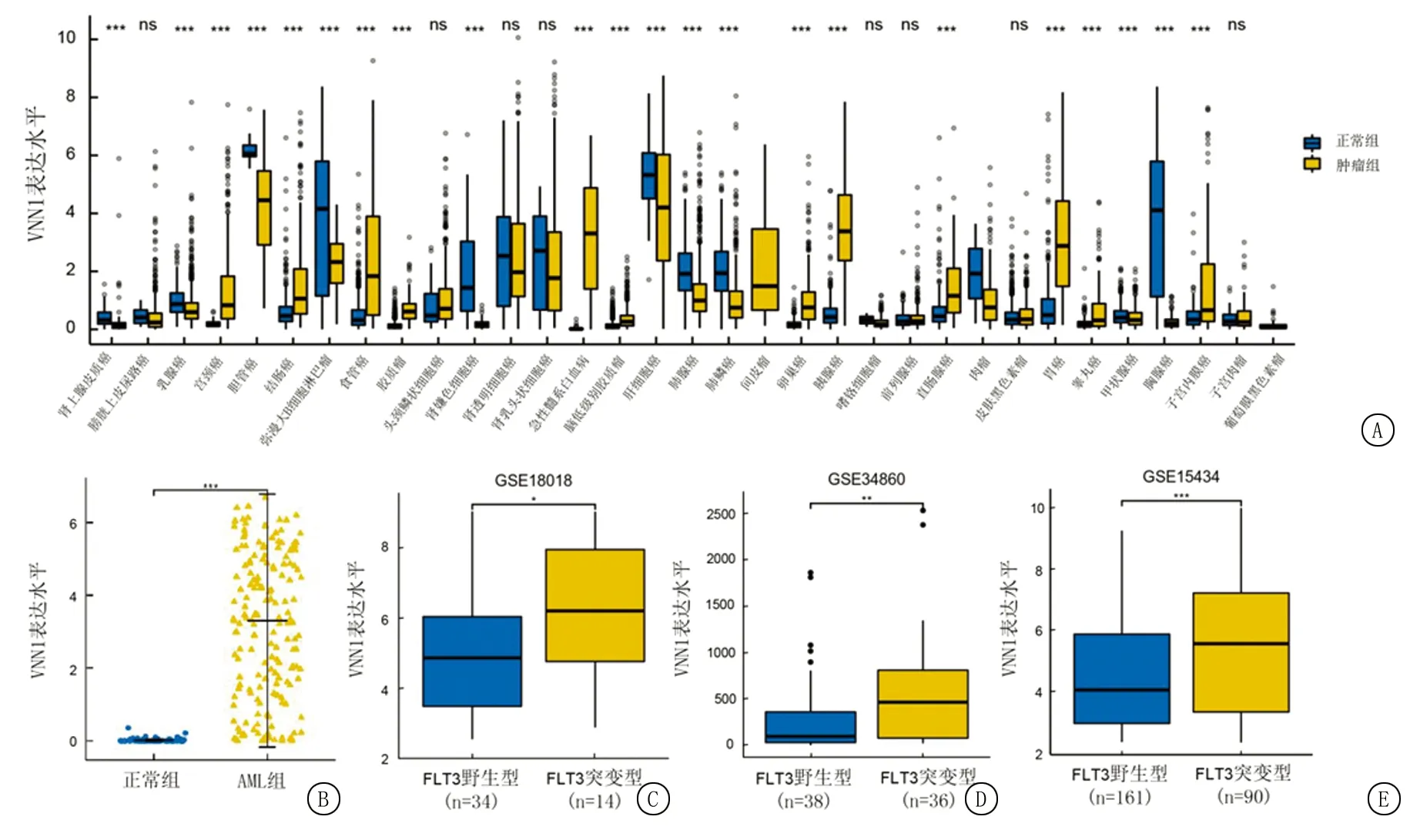
图1 VNN1在AML和正常对照组中的表达水平
2.2VNN1基因表达与AML临床特征的关系 根据TCGA数据库中VNN1表达的中位数,将AML患者分为高(n=76)和低(n=75)表达组。评估了VNN1的表达水平与AML患者临床病理特征之间的相关性。结果显示VNN1的表达与细胞遗传学(P=0.002)、FLT3突变(P=0.041)、核蛋白(nucleophosmin,NPM1)突变(P=0.008)、外周血白细胞计数(P=0.022)和总体生存期(overall survival,OS)(P=0.023)相关,见表1。采用二分类Logistic回归分析法进一步分析VNN1基因表达与AML临床特征的关系。以VNN1表达水平为因变量(低表达=0,高表达=1);以性别(女性=0,男性=1),年龄(≤60岁=0,>60岁=1),白细胞计数(<20×109/L=0,≥20×109/L=1),外周血原始细胞比例(<70%=0,≥70%=1),细胞遗传学风险(低分险=0,中+高风险=1),FLT3突变状态(野生型=0,突变型=1),大鼠肉瘤蛋白(rat sarcoma,RAS)突变状态(野生型=0,突变型=1),NPM1突变状态(野生型=0,突变型=1),VNN1表达(低表达=0,高表达=1)为自变量。结果显示,VNN1与细胞遗传学(P=0.001)、FLT3突变(P=0.042)、NPM1突变(P=0.005)显著相关,见表2。
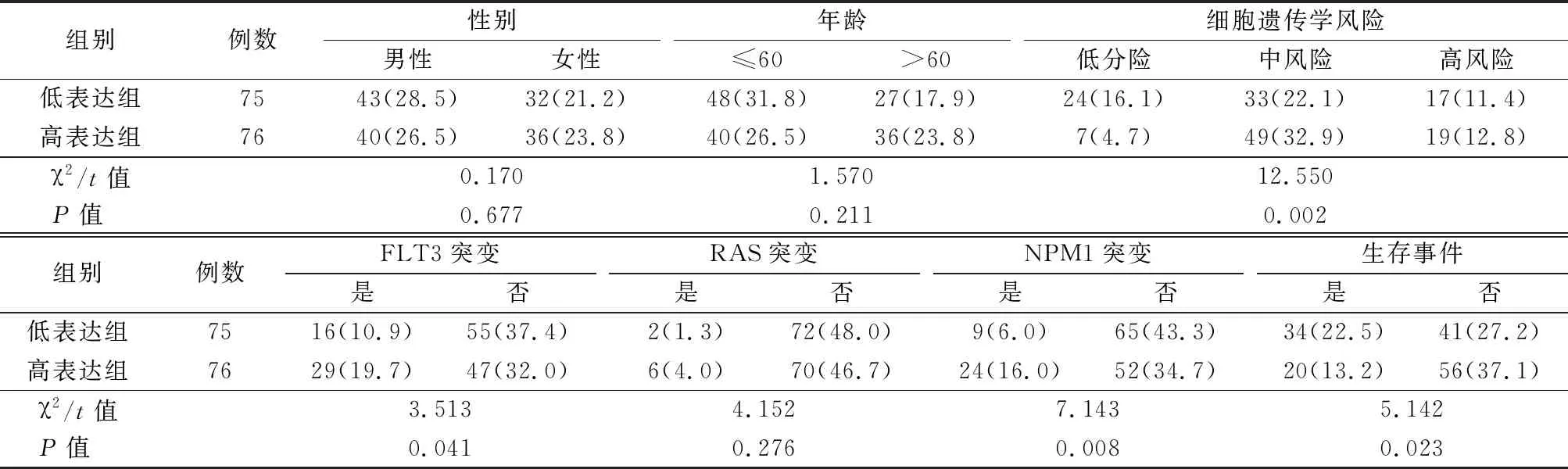
表1 VNN1在AML中的表达水平与临床特征的关系
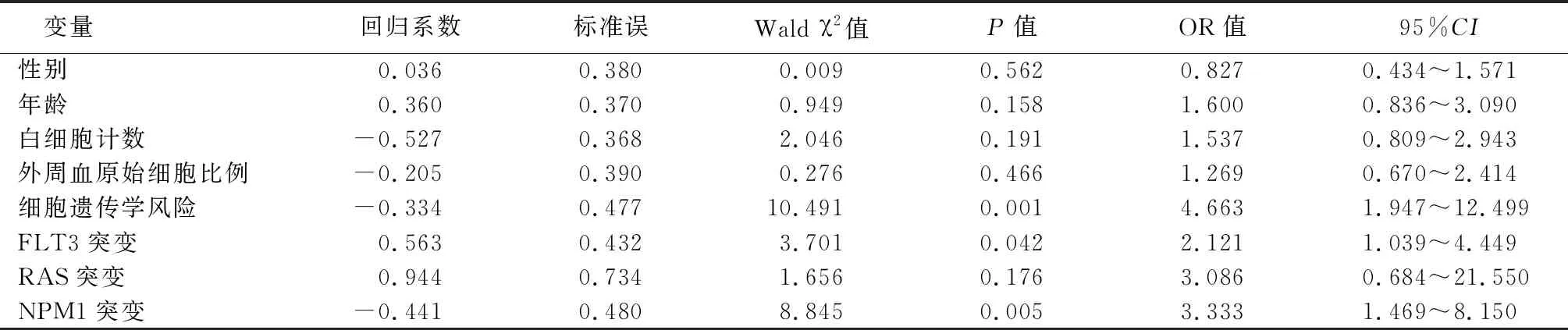
表2 Logistic回归分析VNN1表达与AML患者临床特征的相关性
2.3VNN1基因的表达水平在AML患者中的诊断及预后价值 通过受试者工作特征曲线(receiver operating characteristic curve,ROC)评估VNN1表达水平在鉴别AML和正常对照组准确性方面的价值。结果显示VNN1的预测能力有较高准确性(曲线下面积=0.969,置信区间=0.949~0.990)(图2A)。为了进一步了解VNN1表达水平是否影响AML患者的总体生存期,我们采用了Kaplan-Meier法对TCGA中AML患者的随访数据进行了生存分析。结果显示VNN1的表达水平对AML患者的总生存期有显著影响,高表达组患者的中位生存时间为10.1个月,而低表达组患者的中位生存时间为27.4个月,二者之间的差异有统计学意义(P<0.001)(图2B);多因素COX回归分析,以预后结局为因变量(生存=0,死亡=1);以性别(女性=0,男性=1),年龄(≤60岁=0,>60岁=1),白细胞计数(<20×109/L=0,≥20×109/L=1),骨髓原始细胞比例(<20%=0,≥20%=1),外周血原始细胞比例(<70%=0,≥70%=1),法美英分型(French-American-British,FAB)(M0+M1+M2=0,M4+M5=1),细胞遗传学风险(低分险=0,中+高风险=1),FLT3状态(野生型=0,突变型=1),RAS状态(野生型=0,突变型=1),NPM1状态(野生型=0,突变型=1),VNN1表达(低表达=0,高表达=1)为自变量。采用COX模型回归分析,结果显示高表达的VNN1是除了年龄、细胞遗传学之外又一个重要的独立预测因子。见表3。
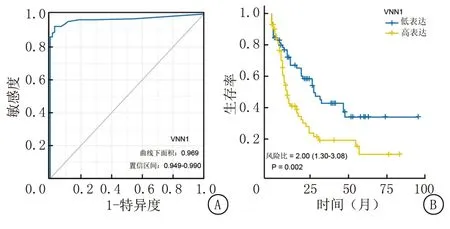
图2 VNN1的表达水平在AML患者诊断和预后方面的价值

表3 多因素COX回归分析 TCGA 中OS 和临床特征的关联
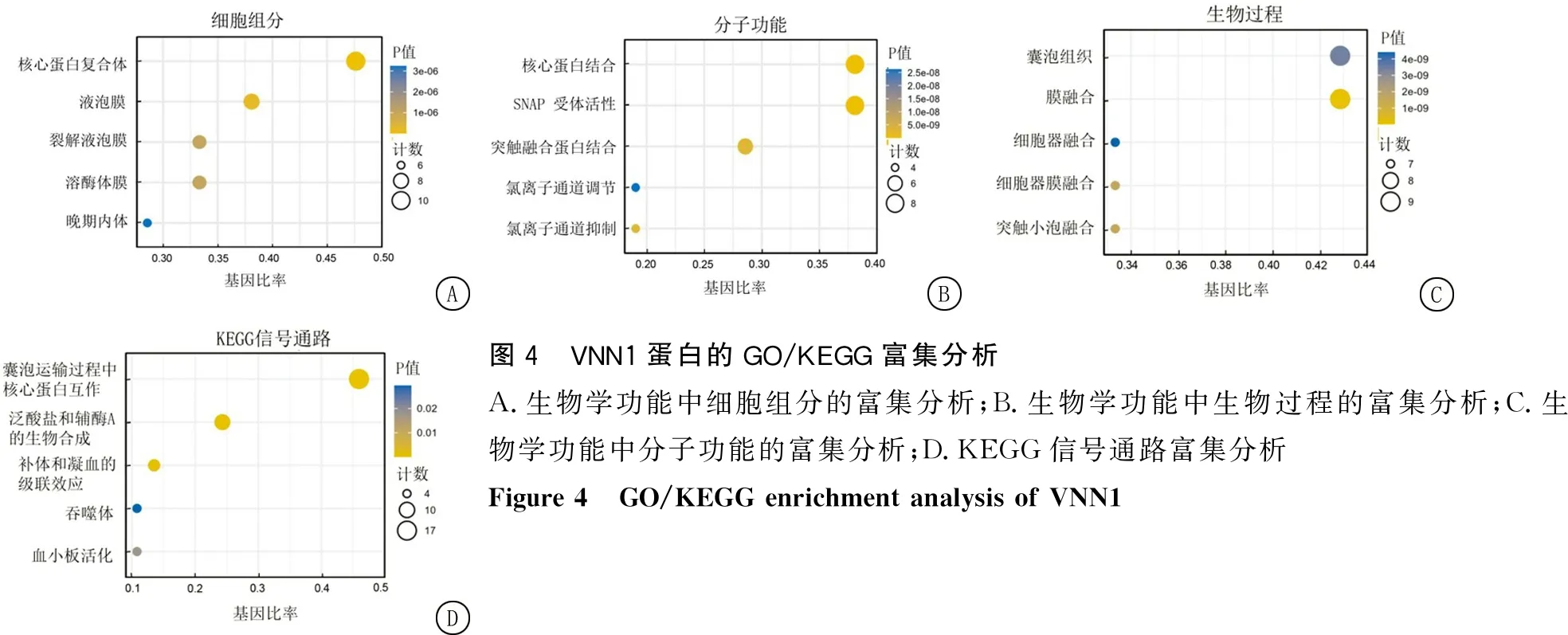
2.4与VNN1互作的蛋白网络 通过STRING数据库分析了与VNN1相互作用的蛋白网络,根据设定条件共得到互作蛋白富集的节点数11个,其中在PPI网络中与VNN1相互作用的蛋白有泛酸激酶1(pantothenate Kinase 1,PANK1)、泛酸激酶3(pantothenate Kinase 3,PANK3)、泛酸激酶2(pantothenate kinase 2,PANK2)、血管非炎性因子2(vascular non-inflammatory molecule 2,VNN2)、血管非炎症分子3(vascular non-inflammatory molecule 3,VNN3)、胶质连接素2(glia-derived nexin 2,SERPIN2)、微量胺相关受体2(trace amine-associated receptor 2,TAAR2)、微量胺相关受体5(trace amine-associated receptor 5,TAAR5)、微量胺相关受体1(trace amine-associated receptor 1,TAAR1)、突触融合蛋白7(syntaxin-7,STX7)(图3)。GO功能及KEGG途径的富集分析显示其主要参与的生物学过程有囊泡运输中的核心蛋白复合体相互作用、泛酸和辅酶A生物合成、补体和凝血级联反应过程、血小板活化等(图4)。

图3 VNN1蛋白-蛋白互作分析
2.5免疫细胞浸润分析免疫细胞浸润分析显示VNN1在AML中的表达水平与中性粒细胞、巨噬细胞、嗜酸粒细胞、活化的树突状细胞(activated dendritic cells,aDC)等富集评分显著正相关,而与自然杀伤细胞(natural killer cell,NK)、T细胞显著负相关(图5)。
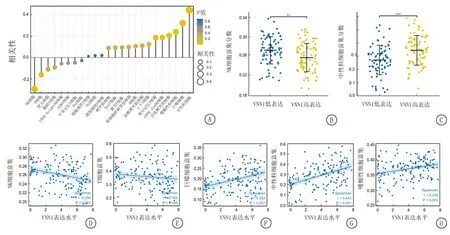
图5 VNN1的表达与AML微环境中免疫细胞浸润相关
3 讨 论
氧化应激是由于机体产生氧自由基的酶活性增加,去除氧自由基的酶活性降低,以及抗氧化剂水平不足造成的[8]。脂质是对氧化最敏感的分子,细胞膜脂质的氧化导致细胞膜弹性丧失,细胞脆性增加,细胞寿命缩短[9]。持续的氧化应激会诱发慢性炎症和许多慢性疾病,包括癌症[10]。一般而言,肿瘤的发生包括三个关键阶段:第一,以细胞DNA损伤和基因突变为标志的启动阶段;第二,以免疫监视效率降低和恶性细胞凋亡率下降为标志的促进阶段;第三,以突变加剧和免疫监视失效为标志的加剧阶段[11]。肿瘤发生的不同阶段几乎都有活性氧的参与。研究表明,氧化应激在AML的发生发展过程中同样发挥了重要作用[12]。VNN(vanins)是一类能将泛酰巯基乙胺水解为泛酸(也称维生素B5)和半胱胺的酶[6]。在人体中,已经发现了三种VNN,其中两种已证明具有酶活性(VNN1和 VNN2)。而VNN3的作用尚不清楚。目前研究认为VNN1的主要功能是参与泛酸循环,这是合成辅酶A(coenzyme A,CoA)的必要因素[13]。越来越多的研究表明,VNN1在急慢性炎症、代谢、氧化应激、肿瘤的发生发展中都发挥了重要的作用。例如,在急性和慢性炎症损伤模型中,敲除VNN1可减少微环境中的炎症并提高对全身氧化应激的抵抗力[14]。Lu等[15]研究发现血浆VNN1可以作为一种早期预测创伤患者发生败血症的重要因子。国外一项研究表明,高血压患者尿液中VNN1的水平可以提早预测患者发生高血压肾损害的风险[16]。机制研究方面,VNN1能够水解泛酸并产生维生素B5和半胱胺,半胱胺直接抑制谷胱甘肽合成途径中的关键酶γ-谷氨酰半胱氨酸合成酶,而谷胱甘肽是一种重要的抗氧化剂能够减少机体氧化应激[17]。另一方面,VNN1过表达也能直接诱导活性氧(reactive oxygen species,ROS)水平升高,这些因素可能协同导致组织氧化损伤[18]。Giessner等[19]发现,VNN1的致癌特性可能是由于在泛酸(维生素B5)和辅酶A的合成途径中产生了半胱胺,半胱胺直接抑制谷胱甘肽合成中的限速酶-谷氨酰半胱氨酸合成酶,而还原型谷胱甘肽的降低或缺乏会导致机体出现的氧化应激失去控制,并导致细胞中活性氧的增加,这可能会破坏DNA并激活致癌基因。目前,已有部分VNN1抑制剂被发现并已经开展临床应用研究[20]。
在这项研究中,通过生物信息学分析发现VNN1在大多数人类癌症存在差异表达,在不同的肿瘤中表达差异呈现不一致性。VNN1在AML中表达水平显著高于正常对照组,VNN1表达水平在诊断AML和正常对照组方面有着较高的价值。在多个不同的GEO数据集均显示FLT3突变的AML患者VNN1表达水平显著高于非突变组;ROC曲线结果显示VNN1高表达预示AML患者有着较短的生存期,是AML患者潜在的分子预后标志物。单因素和多因素COX回归分析均显示高表达的VNN1对于AML患者而言,是除了年龄、细胞遗传学之外的又一个重要独立预测因子。GO功能及KEGG信号通路的富集分析主要参与的生物学过程有囊泡运输中的核心蛋白复合体相互作用、泛酸和辅酶A生物合成、补体和凝血级联反应过程、血小板活化等。肿瘤免疫细胞浸润分析结果表明,VNN1的表达水平中性粒细胞、巨噬细胞、嗜酸粒细胞、活化的树突状细胞等富集评分显著正相关,而与NK细胞、T细胞显著负相关。研究表明,肿瘤相关免疫细胞主要包括巨噬细胞、T淋巴细胞、B淋巴细胞、自然杀伤细胞和肿瘤相关中性粒细胞(tumor-associatedneutrophils,TAN)等,它们介导了免疫抑制的肿瘤微环境,协助肿瘤细胞实现免疫逃逸,促进肿瘤的恶性发展[21-22]。这些结果提示,VNN1可能在AML肿瘤微环境免疫细胞的募集和调节中发挥了重要作用。
综上所述,本研究主要通过对在线数据库(TCGA、GTEx、GEO)中AML患者的VNN1基因信息进行深入挖掘和系统分析,发现VNN1基因在AML患者中高表达,且与AML患者不良预后存在密切关系,提示它可能在AML的发生发展过程中发挥了促癌作用。此外,我们通过VNN1相关蛋白互作网络,VNN1相关基因的主要生物学功能富集和参与的生物学过程以及肿瘤免疫细胞浸润等进行了预测和分析。总之,对于AML患者而言,VNN1可能是一种新的诊断和预后标志物,并可能为AML治疗策略的选择提供潜在价值。本研究也存在一定的局限性:研究结果均由生物信息学分析在线数据获取,尚需进一步的实验验证。
