基于IRAP标记的沙子空心李遗传多样性评价及指纹图谱构建
2023-01-19王贵李蕊蕊吴茂宏任菲宏王丽丽乔光
王贵,李蕊蕊,吴茂宏,任菲宏,王丽丽,乔光
1.贵州大学生命科学学院/山地植物资源保护与种质创新教育部重点实验室,贵阳 550025;2.贵州省铜仁市林业科学院,铜仁 554300;3.贵州省铜仁市农业科学院,铜仁 554300; 4.贵州省农业科学院,贵阳 550006
沙子空心李(Prunus salicinaLindl. ‘Shazikongx‑inli’),为蔷薇科李属植物、青皮黄肉李品种,是贵州省沿河土家族自治县栽培的地方特色水果,于2006年被批准为国家地理标志保护产品[1]。据沿河县县志记载,沙子空心李系1858年由外地引进栽培,属中国李的一类地方品种,由于沿河县独特的地理、气候和富含硒等多种微量元素的土壤条件适宜其生长,逐渐育成了这一优异地方李品种。目前沙子空心李主栽于沿河县南庄村沙子、中界等乡镇,投产种植面积已达30 km2,还未发现过野生资源。经过160多年的自然演变及人为选育栽培,沙子空心李逐渐形成品种群。近年来沿河县大面积发展沙子空心李,种苗生产混乱、接穗来源异质化,进一步促成沙子空心李品种的多样化[2]。通过笔者所在课题组走访调查发现,目前生产的沙子空心李在成熟时间及果实品质上存在差异。差异株系的存在为优异种质的挖掘提供了种质资源基础,但这些差异是仅由环境因素或栽培技术不同引起的,还是遗传信息发生不同程度的变异尚未明确。只有了解种质资源的遗传变异水平及亲缘关系,才能高效地选配亲本,培育出优异品系[3]。因此,有必要通过DNA分子标记技术从基因水平上对沿河县沙子空心李种质资源进行遗传多样性评价。
DNA分子标记技术,如RAPD、SSR及ISSR,已经广泛用于李研究的不同领域[4],但IRAP(inter-ret‑rotransposon amplified polymorphism,逆转座子位点扩增多态性)标记的应用研究还鲜有报道。IRAP是 Kalendar等[5]于1999年基于植物反转录转座子转座特性而建立的一种分子标记技术,具有高通量、基因组覆盖范围广、多态性丰富及异质性高等优点[6-7]。现已应用于樱桃[8]、梨[9]、葡萄[10]、火龙果[11-12]、梅[13]等亲缘关系鉴定及遗传多样性分析等研究。
本研究依据品种成熟期、果实品质、高产稳产这几个育种目标选择了92株具有优良性状的沙子空心李作为试验材料,采用IRAP标记技术,系统评价供试材料遗传多样性水平及亲缘关系,同时构建沙子空心李指纹图谱,旨在为沙子空心李优异种质的辅助育种、科学利用及保护等提供理论依据与应用参考。
1 材料与方法
1.1 试验材料
于2020年7月对贵州省沿河县南庄村等地进行实地走访调查,选取92株具有优良性状(高品质、高产稳产等)、健康的沙子空心李作为试验材料(表1)。于2022年4月取92份供试种质的幼嫩叶片,用于后续基因组DNA提取。
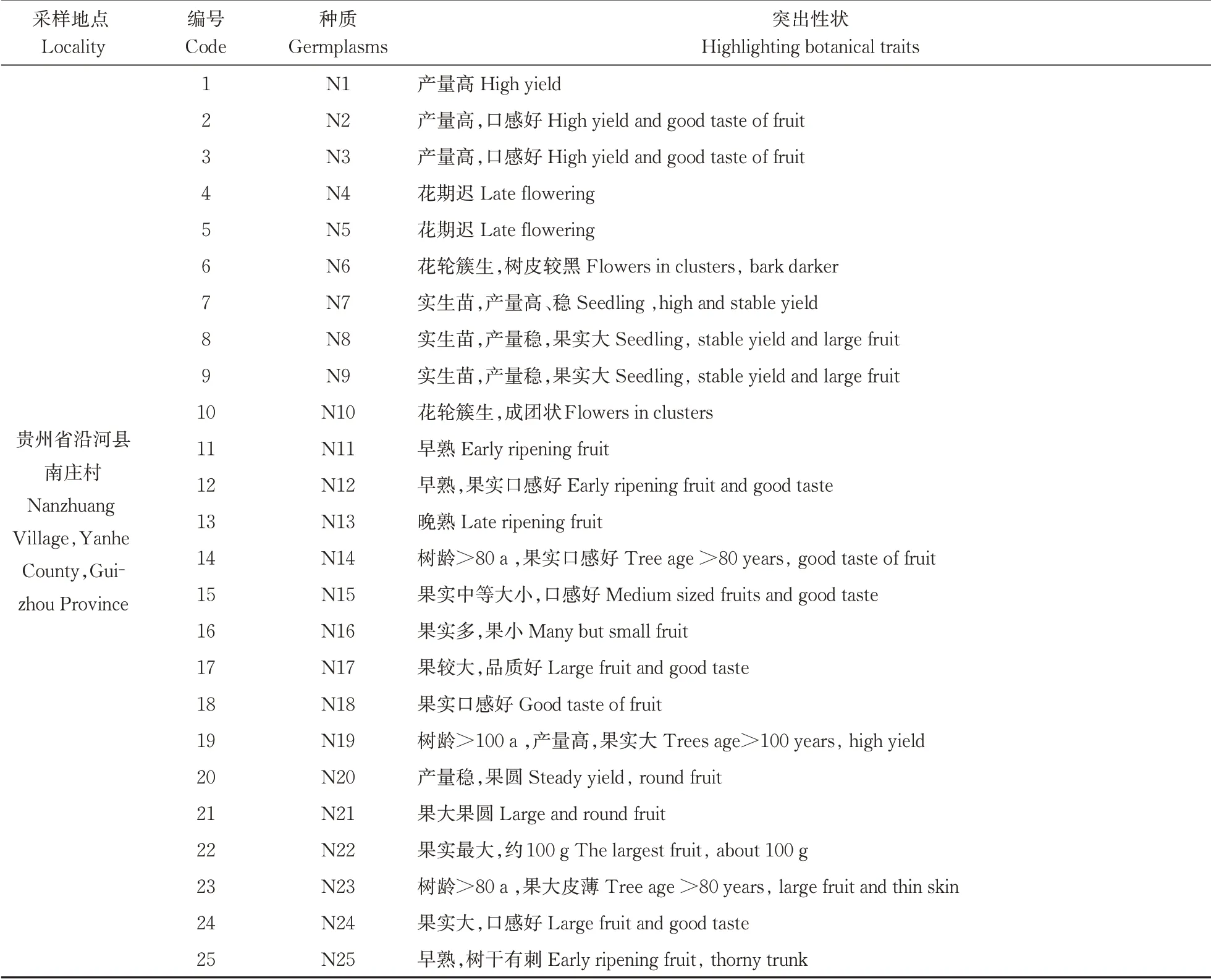
表1 92份沙子空心李种质材料Table 1 Ninety-two germplasm resources of Shazikongxinli for IRAP analysis

续表 1 Continued Table 1
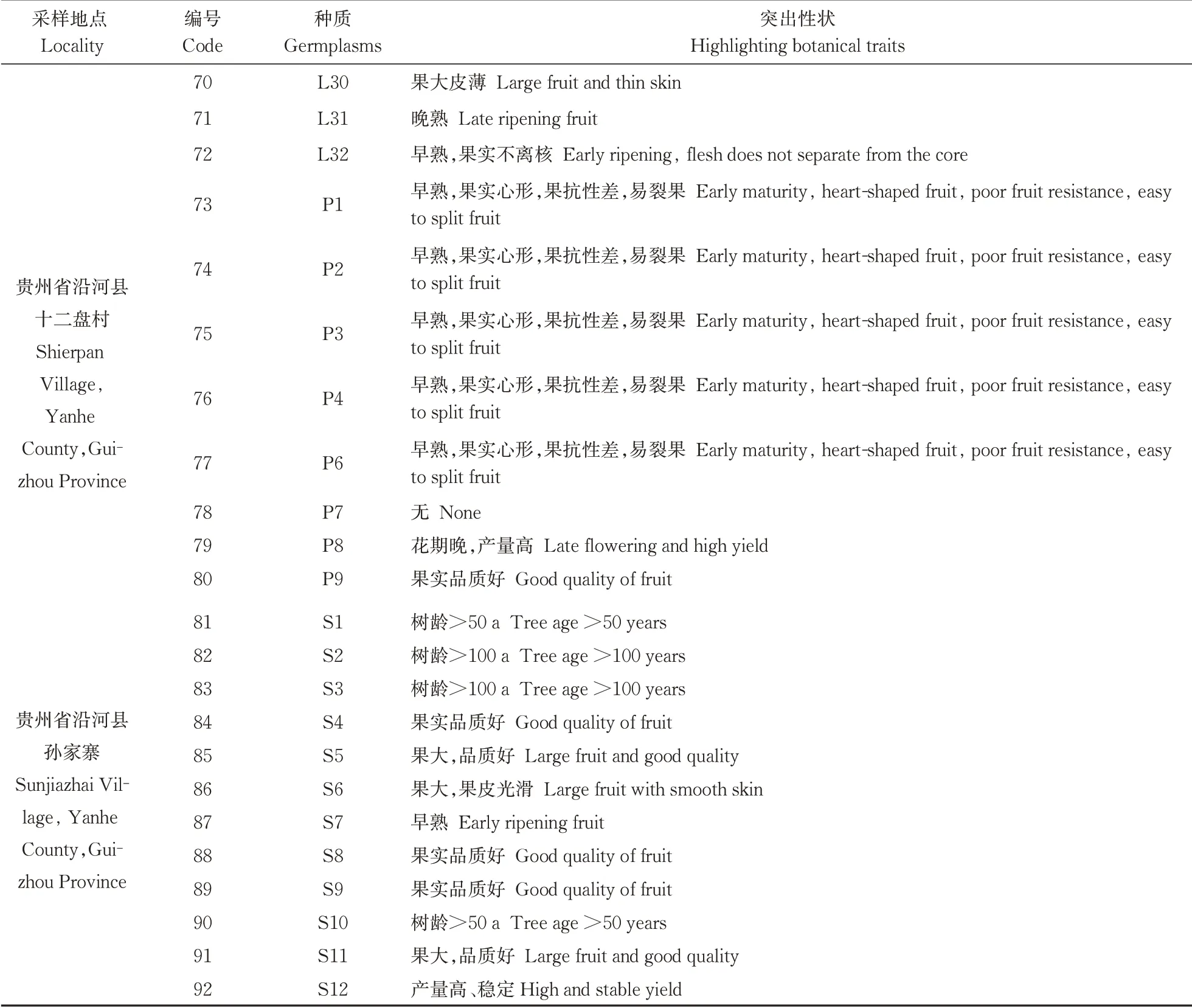
续表 1 Continued Table 1
1.2 基因组DNA提取
通过植物基因组DNA提取试剂盒DP320(北京,天根)提取92份供试材料幼嫩叶片DNA后,经1%琼脂糖凝胶电泳和紫外分光光度计检测质量及浓度后,将DNA稀释至约30 ng/μL,置于-20 ℃保存。
1.3 IRAP引物开发
通过Clustal W对李反转录转座子Ty1-copia和Ty3-gypsy的RT序 列(NCBI:K78998~K79023;K79024~K79041)进行多序列比对,获得保守区域,采用Primer 5.0程序以保守区域上游序列设计单向引物[14],共获得65条IRAP引物,由上海生工合成。
1.4 IRAP-PCR体系优化
设计L16( 43) 正交试验(表2)优化10 μL沙子空心李IRAP-PCR反应体系的主要影响因素Mix(赛默飞;0.4 mmol/LTaqDNA聚 合酶、0.4 mmol/L dNTP、4 mmol/L MgCl2)、IRAP引物和模板DNA的用量;PCR扩增程序为:94 ℃预变性4 min;94 ℃变性30 s,51~58 ℃退火30 s,72 ℃延伸1 min;72 ℃再延伸10 min;4 ℃保存;在此基础上对PCR循环次数设置3个梯度:30、35及40次。
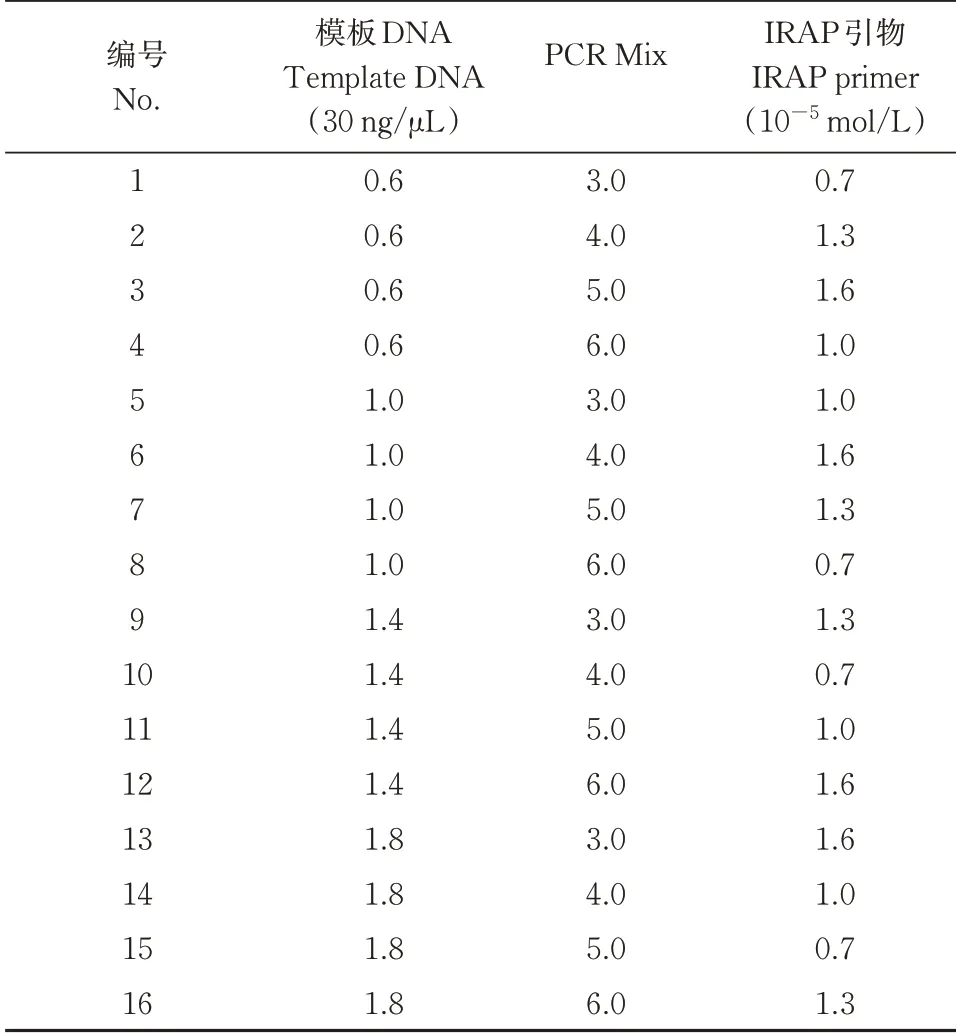
表2 L16( 43 ) 正交试验表Table 2 Table of L16(43) orthogonal experiments μL
1.5 IRAP引物筛选与PCR扩增
根据已优化的10 μL IRAP-PCR体系,随机选取8个供试种质DNA作为模板,利用筛选出来的IRAP引物对92份沙子空心李种质进行PCR扩增,每个引物重复2~3次,PCR产物在1×TAE缓冲液中用1.5%琼脂糖凝胶以6 V/cm电泳40 min后,于凝胶成像系统中观察并保存图像。
1.6 数据处理与分析
仅统计每个引物清晰明亮且可重复的条带,有和无条带分别以“1”和“0”记录,获得每个引物的数据矩阵,通过POPGEN 1.32[15]分析每个IRAP引物的等位基因数、有效等位基因数、Nei’s基因多样性指数及Shannon指数;利用NTSYS 2.10e软件[16]计算遗传相似性系数后,通过MEGA11采用邻接法(neighbor-joining)进行聚类,利用Evolview(http://evolgenius.info)美化聚类图。根据使用最少引物鉴定尽量多供试种质的原则,确定核心引物,直接以统计获得的0/1数据作为指纹代码,通过条形码和二维码生成器(http://qr-batch.com/)构建供试种质分子身份证。
2 结果与分析
2.1 PCR反应体系优化
PCR体系优化试验见图1,泳道1~16分别对应正交试验表(表2)中体系1~16,PCR循环次数依次为30、35、40。PCR结果显示,循环40次为最优。根据40次循环中电泳条带的数量、明暗及清晰度,16个PCR体系从优到差的排序为:7-12-11-16-3-4-8-2-6-14-15-10-5-9-1-13。通过对上述处理的各个因子比较可知,IRAP引物和Mix含量是影响条带多少及明暗程度的主要因素。体系1、5、9、13中Mix含量(3.0 μL)最少,条带均较少且不明亮;而体系2、6、10、14中Mix含量(4.0 μL)相同,体系10及14引物含量(0.7 μL及1.0 μL)相对较少,导致PCR结果相对较差;同理,体系8及15也是如此。综上,考虑经济成本等因素,确定PCR体系(10 μL)最优为体系7:10-5mol/L IRAP引 物1.3 μL,PCR Mix 5.0 μL、模板DNA(30 ng/μL)1.0 μL。PCR扩增程序为:94 ℃预变性4 min;94 ℃变性30 s,51~58 ℃退火30 s,72 ℃延伸1 min,共40次循环;72 ℃再延伸10 min;4 ℃保存。
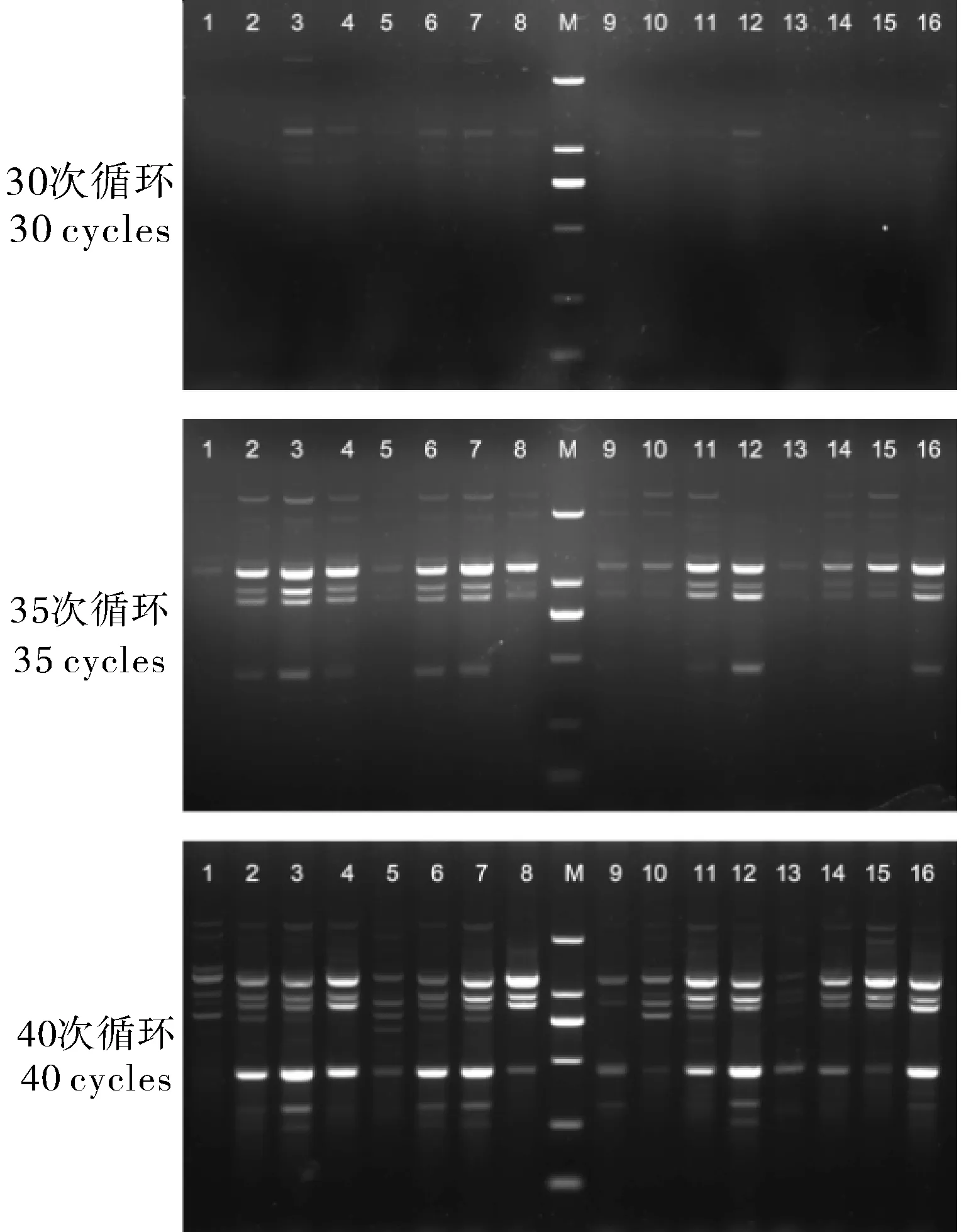
图1 10 μL IRAP-PCR体系优化结果Fig.1 Optimization results of 10 μL IRAP-PCR system
2.2 IRAP引物筛选
对设计的65条IRAP引物进行筛选,最终筛选出18条多态性良好、条带清晰且可重复性强的IRAP引物,且确定最佳退火温度为51~58 ℃(表3),部分引物筛选结果见图2。
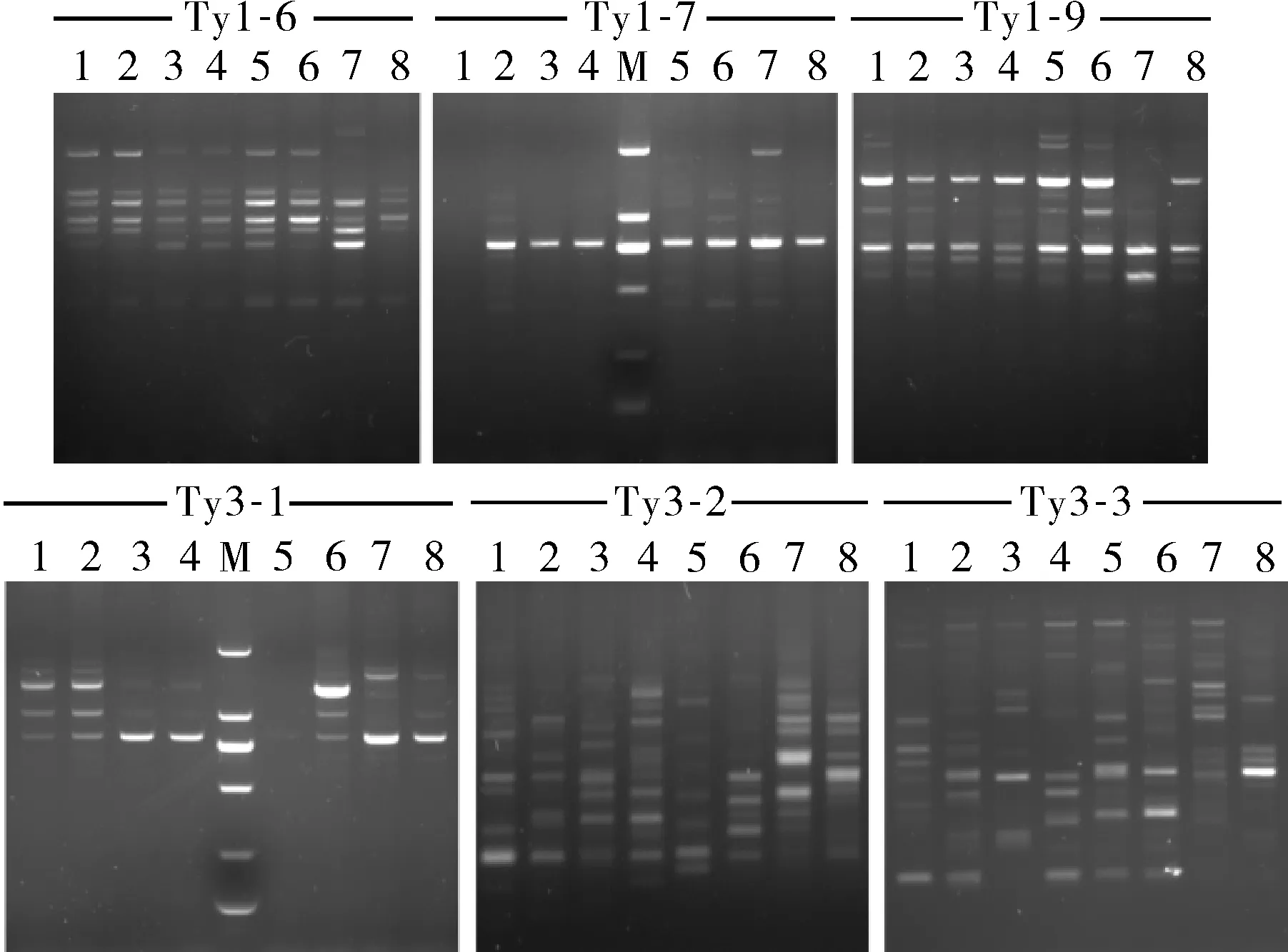
图2 6条IRAP引物筛选结果Fig.2 Screening results of 6 IRAP primers

表3 18条IRAP引物信息Table 3 Information of 18 IRAP primers
2.3 IRAP引物多态性分析
通过筛选出的18条IRAP引物对92份沙子空心李进行PCR分析,结果(表4)显示,18条IRAP引物共扩增出189个位点,其中多态性位点180个,平均多态性位点比率为95.24%。18条引物的有效扩增位点数4~17,平均每个引物扩增10.5个位点,其中引物Ty3-2扩增出的位点数目最多,共17个,而Ty3-14扩增位点数则最少,仅4个;另外,种质N39扩增出的位点数最多,为118个,种质N10扩增出的位点数则最少,共47个。18条IRAP引物中多态性位点比例达到100%的共有12条,表明这些引物具有较高的多态性,能够鉴别沙子空心李。引物Ty3-2及Ty3-6对92份沙子空心李种质PCR扩增结果见图3。
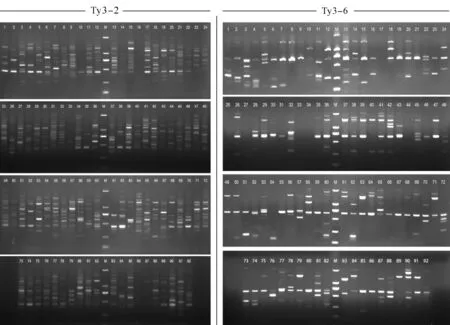
图3 引物Ty3-2及Ty3-6 PCR扩增结果Fig.3 PCR amplification results of Ty3-2 and Ty3-6
2.4 IRAP遗传多样性指数
通过Popgene 1.32对统计的0、1数据进行统计分析,结果(表4)显示,单个引物的等位基因数为1.667(Ty1-17)~2.000,平均等位基因数1.930;有效等位基因数为1.046(Ty1-2)~1.647(Ty1-18),均值1.426;Nei’s基因多样性指数变幅为0.041(Ty1-2)~0.379(Ty3-3),平均值0.261;Shannon信息指数变幅为0.089(Ty1-2)~0.563(Ty3-3),平均值0.405;4个遗传参数中,引物Ty1-7、Ty1-18及Ty3-3等各项值均相对较高,而引物Ty1-2及Ty1-3则相对偏低。
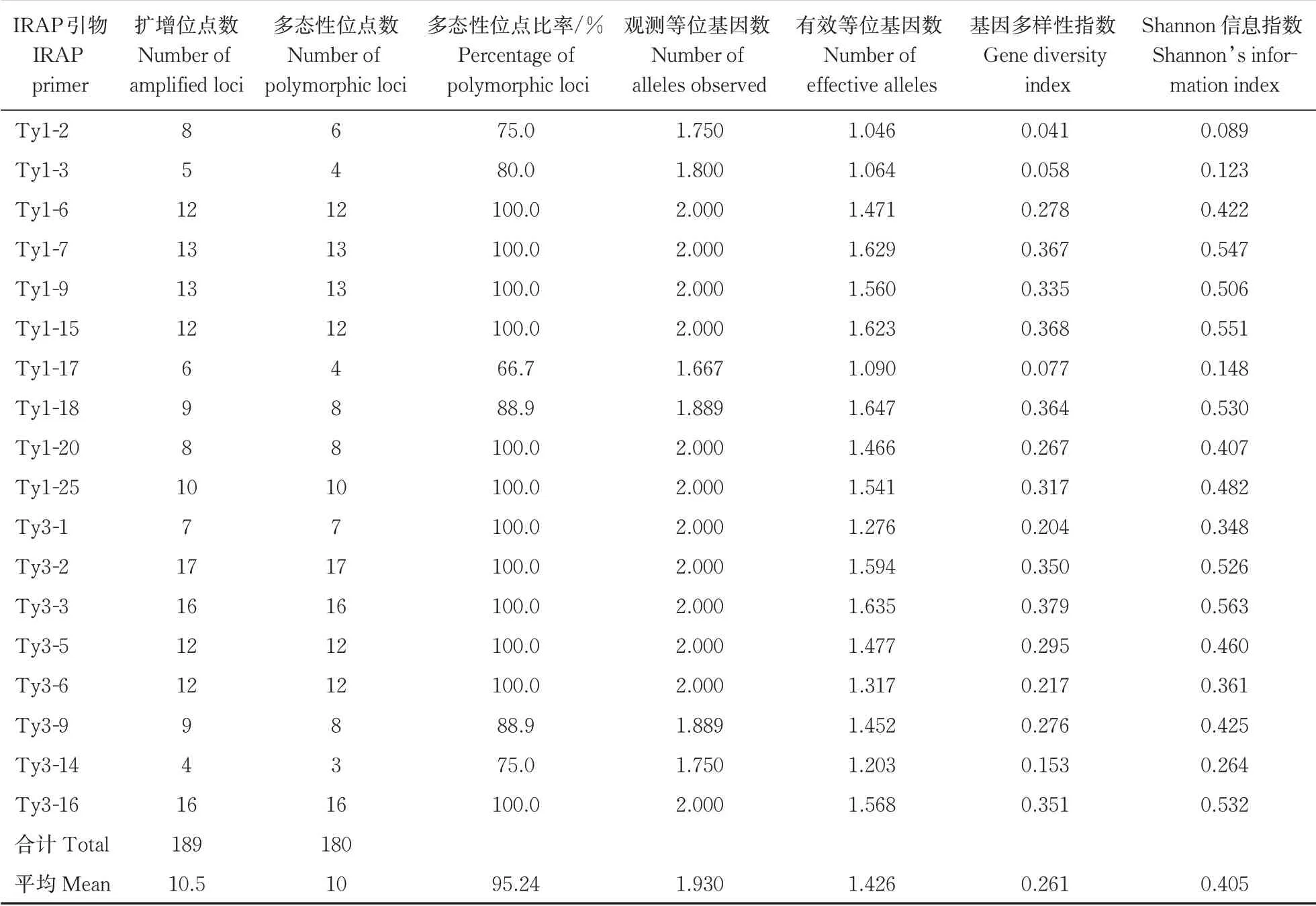
表4 IRAP引物扩增结果统计Table 4 Statistics of IRAP primer amplification results
2.5 沙子空心李种质聚类分析
利用NTSYS 2.10e软件计算供试种质的遗传相似性系数(DICE系数)。92份沙子空心李种质两两之间的DICE系数变幅为0.417~0.830,平均0.681。在遗传相似系数为0.645时,92份沙子空心李被聚类为4组(图4),聚类到第1组的沙子空心李数量最多,包含30份沙子空心李种质,第2组最少,共14个种质,第3组和第4组分别有22个及26个种质。92份沙子空心李种质采自4个种植区,然而聚类结果并不呈现出相应的规律性,这可能是因为采样区域地理间隔较小,栽培环境与技术差异较小,株系间基因交流频繁,因而聚类结果不规律。但根据聚类结果,92份沙子空心李种质在遗传相似系数为0.645时可被聚类为4组,这表明供试的沙子空心李种质可能来自4个不同品系。
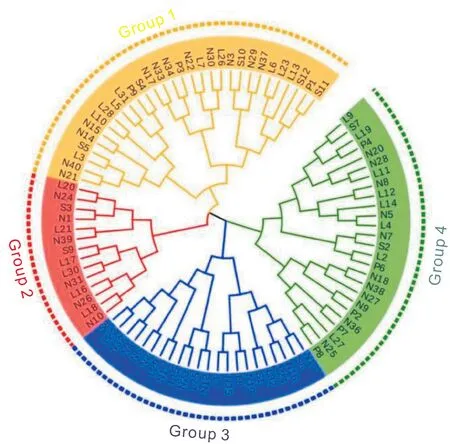
图4 92份沙子空心李聚类结果Fig.4 Dendrogram of 92 Shazikongxinli germplasms based on IRAP bands
2.6 分子指纹图谱构建
利用18条IRAP引物对92份沙子空心李进行PCR扩增,获得每个引物92个种质的0/1数据矩阵,这些0/1数据可作为供试空心李种质指纹图谱或分子身份证构建的直接依据。为达到利用最少的引物鉴定尽量多种质的目的,通过对有效等位基因数达到2.000的12条IRAP引物的种质鉴别情况进行统计(表5),最终确定核心引物为Ty3-2,其鉴别率最高(90.22%),共能鉴定出83个供试种质,另有9个沙子空心李种质(N10、N16、N35、N36、L26、L27、S2、S6、S7)无法鉴别出来,需与其他引物组合进行鉴别。引物Ty3-6与Ty3-2组成1组核心引物,可鉴别出所有种质,构建高效的指纹图谱。将表1中的4个采样地(沿河县南庄村、黎家寨、十二盘村及孙家寨)分别以数字1、2、3、4表示,同时12条IRAP引物依次以字母“A-L”表示(表5),直接以引物扩增出的0/1数值串构建指纹代码。以种质N1和L1为例,N1指纹代码为:1A01000000000110001I000000000101,第1位数字1代表采样地1(沿河县南庄村),A代表IRAP引 物Ty3-2,I表 示 引 物Ty3-6;L1指 纹 代 码 为:2A01010001010011001I011010000101,第1位数字2代表采样地2(沿河县黎家寨),最终利用每个种质的指纹代码,分别生成包含基本信息的条形码和二维码(图5)。
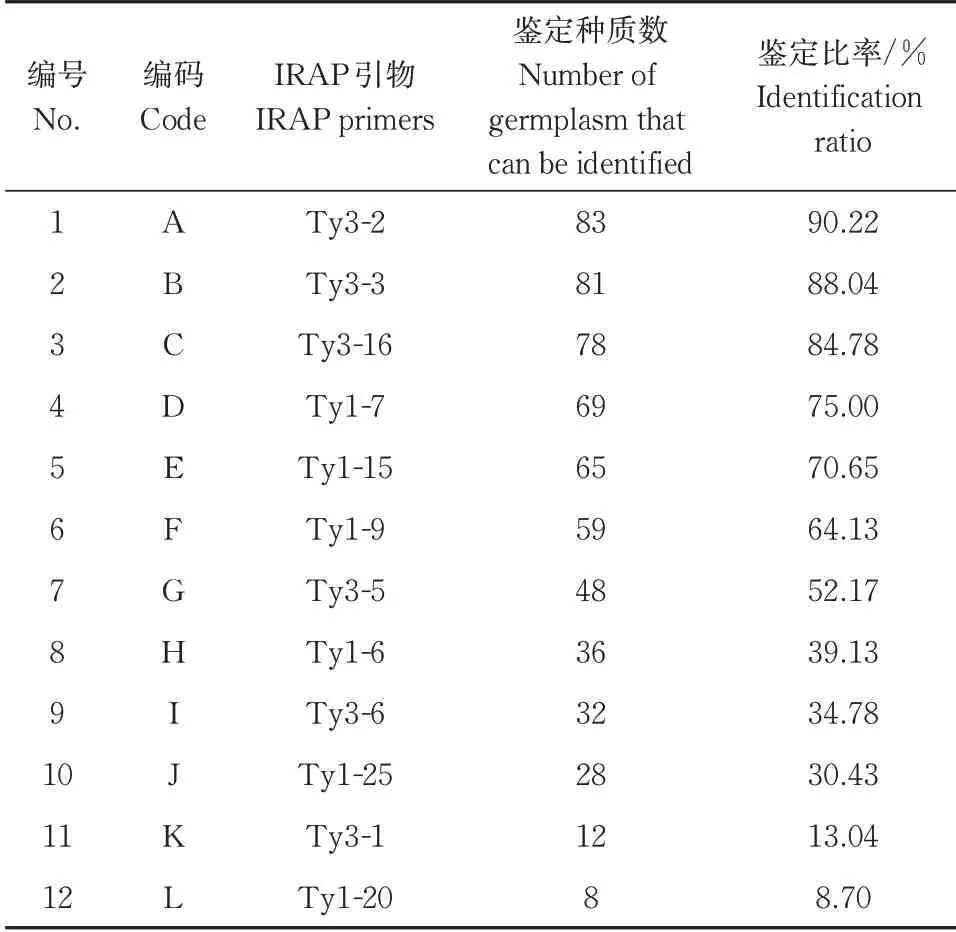
表5 IRAP引物鉴别供试种质情况Table 5 The number of germplasms that can be identi⁃fied by IRAP primers

图5 沙子空心李种质N1及L1分子身份证Fig.5 Prunus salicina Lindl. Shazikongxinli N1 and L1 molecular IDs
3 讨论
3.1 IRAP体系优化
本研究通过L16( 43) 正交试验确定10 μL沙子空心李IRAP-PCR反应的最优体系,经反复试验,认为Mix及IRAP引物含量是影响PCR结果优劣的主要影响因素。MIX和IRAP引物含量过低时,PCR扩增结果中条带均较少且较为暗淡,易造成试验误差,影响试验结果。但Mix和IRAP引物含量也不可太高,IRAP引物含量过高时,容易导致引物二聚体的产生,同时也会增加导致非特异扩增及错配的可能性[17],而Mix含量太高则会造成不必要的浪费,达不到经济高效的目的;另外,循环次数也是影响PCR扩增结果的重要因素,循环次数与PCR产物浓度成正比,次数太少易导致产物含量太少,而循环次数太多也会导致非特异性产物的产生,循环次数一般选择30~40次[18]。本试验对10 μL IRAP-PCR反应体系进行了优化,经济高效地保证了试验结果的准确性。
3.2 指纹图谱构建
目前构建指纹图谱的方法相对较多,有直接根据条带统计获得的“0/1”数据作为构建指纹的代码;还有从多个引物的扩增结果中选取稳定的特异条带组成“0/1”数据构建指纹代码;另外,还可将二进制的“0/1”数据转变为十进制的数字形成代码[18]。指纹图谱和分子身份证的构建对于种质鉴别具有重要意义。本研究确定引物Ty3-2及Ty3-6作为种质鉴定及指纹图谱构建的高效标记引物,并据此构建了供试种质的分子身份证。在构建的分子身份证中加入了种质的相关信息,包括栽培地、突出性状等信息,可为后续沙子空心李育成品种的分子身份证构建及种质知识产权的保护提供参考。
3.3 沙子空心李遗传多样性及聚类分析
遗传多样性能够反映出物种的遗传背景及育种潜力,是作物种内和种群之间育种过程的基础条件[19]。对沙子空心李种质进行遗传多样性分析,是种质资源鉴定、筛选的重要工作,也是培育优异种质的前提。李作为一种常见果树,对于其遗传多样性的研究相对较多。王红林等[20]通过SSR标记对16份贵州中晚熟李种质进行分析,平均Shannon多样性指数(I)为1.59,认为这些李种质具有丰富的遗传信息,这和魏潇等[21]利用22对SSR引物分析17份南方李品种的多样性结果类似(I=1.709)。左力辉等[22]也通过SSR标记检测了24份中国李品种的遗传多样性(H=0.227 3,I=0.358 6)。陈红等[23]则通过ISSR标记分析了48份李种质的多样性水平,平均Nei’s遗传多样性指数(H)为0.345,平均Shannon多样性指数(I)0.508。目前对单一李品种内的遗传多样性研究相对较少。Basilio等[24]以29份日本李核心种质为研究对象,通过11条ISSR引物评价其多样性,结果显示平均Nei’s遗传多样性指数(H)为0.15,平均Shannon多样性指数(I)0.27。这低于本研究通过18条IRAP引物对92份沙子空心李的分析结果(H=0.261,I=0.405)。其可能与本试验采用的分子标记检测能力不同,所收集的种质样本数量多等因素有关。另外,Guan等[25]通过9个IRAP标记检测了268份柿种间和种内的多样性水平(H=0.240,I=0.389),认为这些柿种质具有较为丰富的遗传信息。本研究验证了反转录转座子标记在李植物遗传多样性分析的适用性,显示IRAP标记即使在单一品种中也具有较高水平地检测变异的能力,这可能与反转录转座子在李中分布广泛、在种内也具有丰富的序列和插入多态性等特性有关。在逆境条件下,反转录转座子容易被激活产生新的插入突变,进行横向、纵向传递[26],因而相较于SSR、ISSR等标记用于种间系谱的研究,IRAP标记由于其标记性质,可能更适合用于栽培种内的变异检测[5],分析种内个体水平上的遗传变异情况及亲缘关系。
遗传相似性系数是判断物种间亲缘关系及遗传基础的标准之一[27]。本研究通过neighbor-joining法对92份供试沙子空心李进行聚类分析,在遗传相似系数为0.645时,供试种质被聚类为4类。在聚类结果中这些供试种质并不呈现出相应的地理分布规律性,这可能是因为采样的4个区域地理间隔较小,种苗来源途径基本相同,栽培环境及技术等相近,同时种质间交流频繁,渗入程度较大。本研究结果确定了供试种质基因水平上的亲缘关系,可为后续选育优异种质中高效选配亲本提供参考依据。
沙子空心李在生产上出现果实品质及成熟期不一致的现象,可能是在长期的栽培过程中,由地方品种的种性退化以及苗木繁育过程中产生的不同品系混杂引起的。本研究验证了沙子空心李种质在DNA水平上产生了较为明显的变异,排除了供试沙子空心李不同株系间在栽培表现上产生差异仅由环境因素或栽培技术不同而导致的情况,表明这些种质可作为优异品系培育的核心种质。后续可通过结合各个株系的优良性状包括抗逆能力、果实品质、产量等进行杂交选配,培育出更为优异的后代。如利用沙子空心李种质N22具有果实最大的突出品质,结合N27果实口感最好和N7等种质高产、稳定的优点进行杂交后连续回交繁育后代,实现沙子空心李的增产提质。
