5个棘胸蛙养殖群体微卫星遗传多样性分析
2023-01-19魏朝宇汪小冬魏秀英姚红艳陈敦学
魏朝宇,汪小冬,魏秀英,袁 鸿,姚红艳,陈敦学*
(1.贵州大学 动物科学学院,贵州 贵阳 550025; 2. 贵州大学 渔业资源与环境研究中心,贵州 贵阳 550025;3. 高原山地动物遗传育种与繁殖教育部重点实验室(贵州大学),贵州 贵阳 550025;4. 湖南农业大学 动物科学学院, 湖南 长沙 410128)
棘胸蛙Quasipaaspinosa又名石蛙、石鸡等,隶属两栖纲Amphibian无尾目Anura叉舌蛙科Dicroglossinae棘胸蛙属Quasipaa,主要分布在中国南方八省[1]。由于生态环境破坏和过度捕抓,棘胸蛙野生资源急剧减少,目前已被中国物种红色名录列为易危等级[2-3]。棘胸蛙蛙肉具有高蛋白和低脂肪的特点,被认为是药补、食补佳品,市场需求旺盛[4]。为了满足人们对蛙肉的需求,减少对野生资源的消耗,我国从20世纪80年代开始进行人工养殖,但主要采取“野外捕捞,就地繁养”模式,对繁殖群体缺乏遗传特征分析[2],容易造成近亲繁殖,导致棘胸蛙出现个头小、抗病性差等问题[5],因此,迫切需要对棘胸蛙养殖群体开展遗传多样性研究,评估棘胸蛙养殖群体的遗传现状,为提高养殖效益和选育棘胸蛙良种提供数据支撑。目前,养殖群体遗传多样性研究在鱼类中开展得较多,普遍认为养殖群体遗传多样性低于野生群体[6-9]。对棘胸蛙遗传多样性的研究依然很少,且主要集中在野生群体的研究,例如,刘南君等[10]采用RAPD技术分析贵州省宽阔水域棘胸蛙遗传多样性,认为贵州省棘胸蛙具有较高水平的遗传多样性和环境适应能力;王茂元等[11]采用GBS技术,证明福建南平棘胸蛙种群已表现出杂合度降低、遗传多样性下降等趋势;Zheng等[12]运用15个SSR位点对棘胸蛙2个群体进行分析,结果揭示棘胸蛙具有较高的多态性;Ye等[13]利用线粒体基因(12S rRNA 和16S rRNA)证明棘胸蛙具有很高的遗传多样性。
由于缺乏对养殖群体种质资源的研究,目前棘胸蛙养殖产业健康发展依旧存在隐患。选择合适的分子标记将有助于我们了解物种的遗传特征,进而推动种质资源开发与创新利用[14]。微卫星又称简单重复序列,主要由高突变的核心序列和保守的侧翼序列组成,具有数量多、分布广泛且均匀、杂合率高、重复性好且数据易统计等优点[15],被广泛应用于水产动物种质资源评估、群体遗传多样性分析、亲缘关系鉴定等方面[12,16]。截止2021年7月,NCBI数据库公布的棘胸蛙SSR位点仅31个,远不足以评价棘胸蛙遗传多样性。因此,本研究通过转录组筛选棘胸蛙9对有效SSR位点,并依托9个位点对5个养殖群体棘胸蛙(148个样本)进行遗传多样性分析,阐明棘胸蛙养殖群体的遗传现状,以期为棘胸蛙种质资源开发利用和良种培育提供基础数据支撑。
1 材料与方法
1.1 样本采集
实验所用棘胸蛙蝌蚪采自江西明月山石蛙养殖股份有限公司(江西群体30尾)、浙江双吉山养殖有限公司(浙江群体30尾)、广西贺州全宇石蛙养殖专业合作社(广西群体28尾)、广东清远市盈信农业有限公司(广东群体30尾)、福建德化双全农业有限公司(福建群体30尾),共计148尾。样本用无水乙醇浸泡,-20 ℃保存备用。
1.2 基因组DNA提取
取少量的肌肉,用基因组DNA试剂盒(北京天根生化科技有限公司)并参照说明书进行DNA提取,提取后的DNA用0.8%琼脂糖凝胶电泳检测其完整性,并于-20 ℃保存备用。
1.3 微卫星DNA引物设计与合成
用转录组开发棘胸蛙潜在的SSR位点,选择SSR两端序列长度≥50 bp且碱基重复类型为2碱基重复与3碱基重复的序列,primier 5.0进行引物设计;随机挑选50对引物进行验证,以浙江(ZJ)群体棘胸蛙基因组DNA为模板扩增,以产生稳定扩增条带且条带大小与预期一致为筛选依据。共筛选到15对引物,进而将扩增产物用 8% 非变性聚丙烯酰胺电泳和硝酸银染色观察,根据条带稳定情况,筛选出9对引物(见表1)。
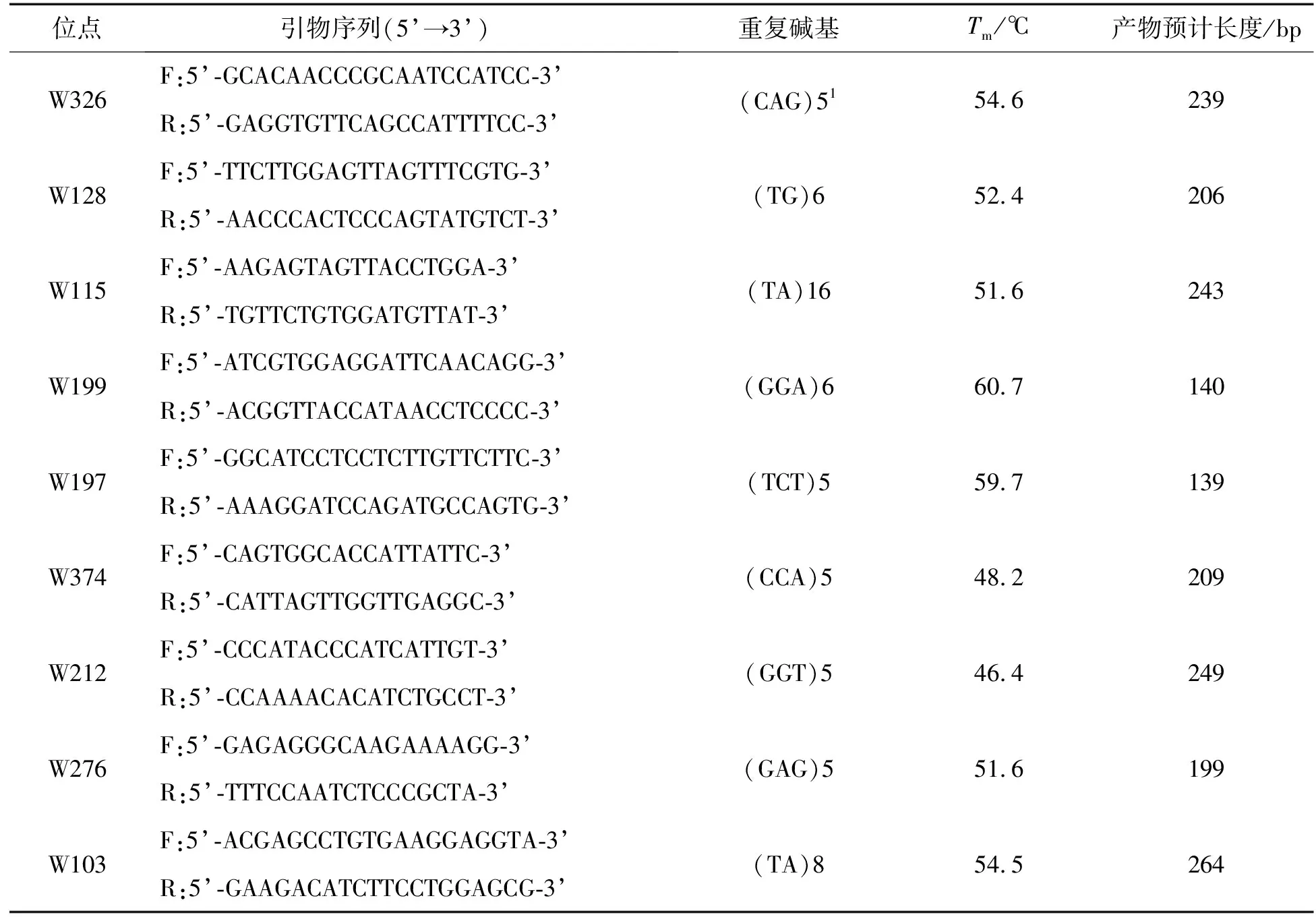
表1 9对微卫星位点信息Tab. 1 Information of 9 microsatellite loci
1.4 PCR扩增
将筛选得到的9对引物分别在148个样品中进行PCR扩增,PCR反应体系为25 μL:2×Tap PCR Master Mix(MT201,博迈德生物,中国)12.5 μL,上下游引物各1 μL,DNA模板1 μL,ddH2O 9.5 μL。PCR反应程序为:94 ℃预变性 5 min;94 ℃变性30 s,引物在各自合适的退火温度下退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min。扩增产物用2%琼脂糖凝胶检测扩增效果。
1.5 聚丙烯酰胺凝胶电泳并银染
扩增产物经8%非变性聚丙烯酰胺凝胶分离,产物上样量为2 μL,内外电泳槽各加入1×TBE电泳缓冲液,设置电压180 V,电流150 mA,电泳5 h后,硝酸银染色并观察其条带情况。
1.6 数据分析
用Popgen32软件计算各位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、近交系数(Fis)、遗传分化系数(Fst)及Hardy-Weinberg平衡检验。用Cervus计算各位点的多态信息含量(PIC)。基于群体间的Nei’s遗传距离使用MEGA构建UPGMA系统进化树。
2 结果与分析
2.1 微卫星位点的多态性
9对引物在棘胸蛙5个养殖群体中共检测到46个等位基因,详见表2。各位点等位基因数为4~9个,平均为5.111个。其中,引物W115检测到的等位基因数最多,为9个。各位点的有效等位基因数介于1.731~5.291,均值为2.702。观测杂合度在0.007~0.322,平均值为0.182。期望杂合度介于0.423~0.815,平均值为0.589。9对引物的多态信息含量介于0.392~0.789,平均值为0.536。其中,引物W128、W115、W212和W103表现为高度多态位点,其余为中度多态性。

表2 9个位点的遗传多样性参数Tab. 2 Genetic diversity indices of 9 microsatellite loci in five cultured populations
从群体内近交系数(Fis)和遗传分化系数(Fst)来看,近交系数在引物W276(-0.147)表现为负值,其余均为正值,平均值为0.523。遗传分化系数在0.070~0.717,平均值为0.332。位点W326的遗传分化系数为中度分化(0.05~0.15),其余位点均为高度分化。平均总遗传分化系数为0.332(遗传分化系数大于0.15),属于高度分化。
2.2 棘胸蛙群体的遗传多样性
分析9个SSR位点在5个养殖群体中的多态性,结果见表3,发现5个群体的平均等位基因数(Na)为2.889~3.444,其中广东群体最少,江西群体最多。有效等位基因数(Ne)平均为1.737~2.131,江西群体最少,广西群体最多,5个养殖群体的总体平均值为1.945。5个群体 PIC值平均在0.312~0.400,平均值为0.355,其中江西群体最低,浙江群体最高。5个养殖群体的观测杂合度(Ho)和期望杂合度(He)分别为0.130~0.230和0.353~0.468。其中浙江群体的Ho和He均达到最大值,而观测杂合度和期望杂合度的最小值分别出现在福建群体和江西群体(表3)。

表3 5个群体在9个微卫星位点的遗传多样性参数Tab. 3 Genetic diversity indices of 9 microsatellite loci in different cultured populations
2.3 群体间遗传相似度和聚类分析
不同养殖群体棘胸蛙的遗传相似度和遗传距离存在较大差异,其中:广东和广西遗传相似度最高(0.819),遗传距离最近(0.200);福建和江西的遗传相似度最低(0.441),遗传距离最远(0.819)(表4)。利用MEGA软件构建UPGMA系统发育进化树,结果显示,广东群体和广西群体聚为一支,而后再与福建群体聚在一起,而浙江和江西群体聚为一支,具有明显的地理区系(图1)。

图1 基于Nei’s无偏遗传距离构建的5个棘胸蛙养殖群体UPGMA树Fig. 1 UPGMA dendrogram of five cultured populations of Q. spinosa based on Nei’s
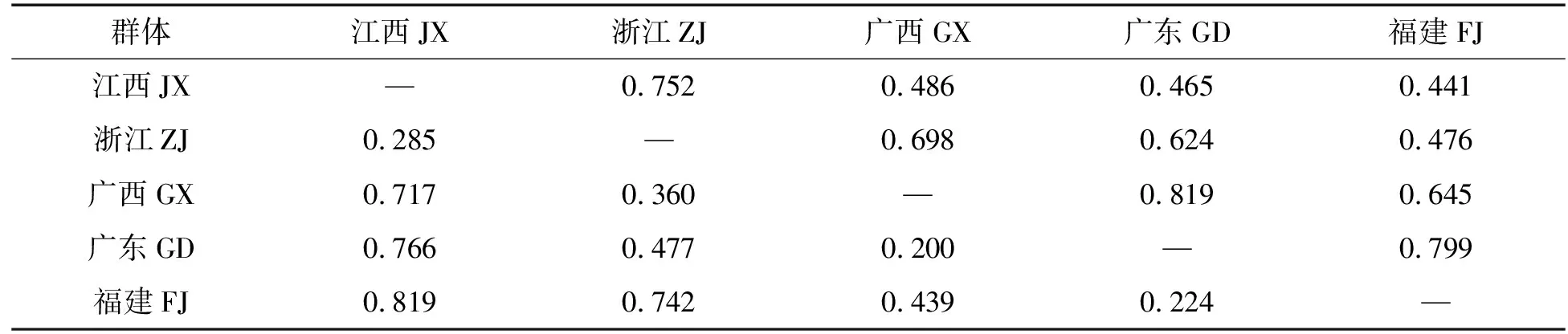
表4 5个棘胸蛙养殖群体的遗传相似度和遗传距离Tab. 4 Genetic distance genetic similarity of 5 cultured populations of Q. spinosa
进一步利用AMOVA软件进行群体内和群体间的遗传变异分析,结果显示,有70.95%的遗传变异来自群体内,而群体间的变异为29.05%,群体内的变异大于群体间的变异(表5)。
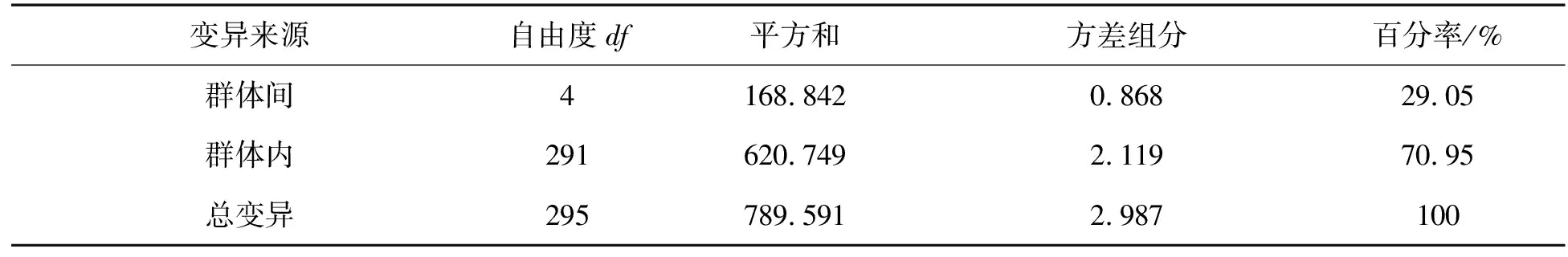
表5 5个棘胸蛙群体的方差分析Tab. 5 AMOVA analysis among five Q. spinosa populations
2.4 Hardy-Weinberg平衡分析
利用Popgen软件,经Hardy-Weinberg平衡(HWE平衡)的卡方检验发现,在棘胸蛙5个养殖群体,9个多态性SSR位点组合而来的45个位点中,大部分都偏离了平衡状态,呈现极显著偏离 HWE 平衡(30个位点),仅有11个位点未偏离HWE 平衡,另有4个位点为单倍性位点,未进行HWE 平衡分析(表6)。江西、浙江、广西、广东和福建5个养殖群体中,分别有5、8、8、5、4个位点偏离了HWE 平衡。此外,有2对引物(W326和W115)在5个养殖群体中均偏离HWE 平衡。
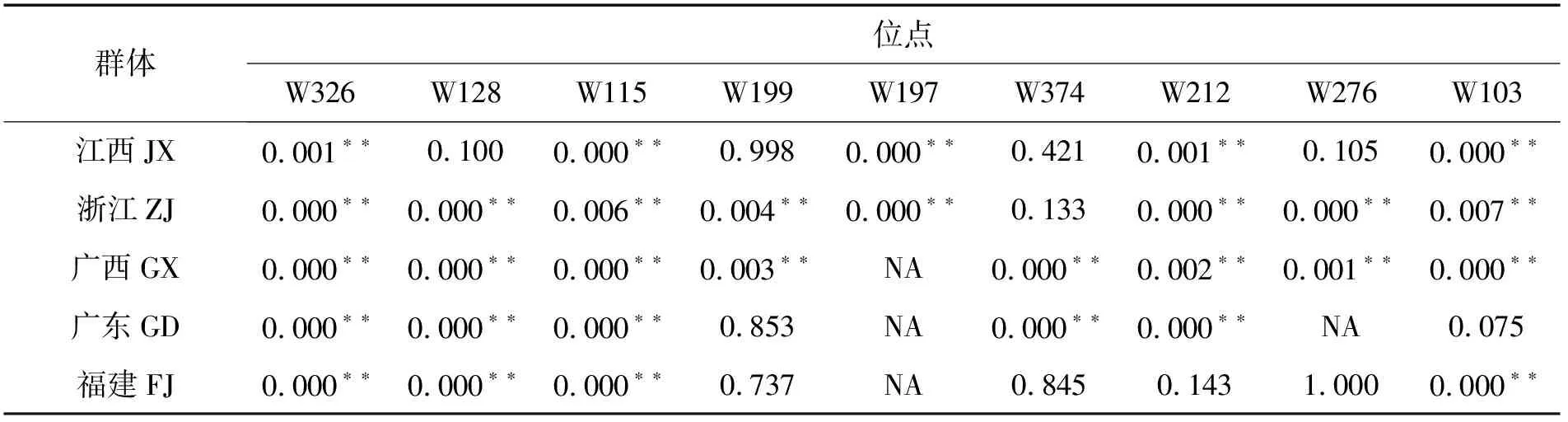
表6 5个群体在9个微卫星位点的Hardy-Weinberg平衡分析Tab. 6 Hardy-Weinberg equilibrium of 5 populations at 9 microsatellite loci
3 讨论
3.1 多态信息含量及基因杂合度分析
多态性信息含量是描述遗传多样性的指标,标志着群体对环境变化的适应能力和生存能力[17-18]。一般认为,多态性信息含量大于0.5时,该位点表现为高度多态性;当多态性信息含量介于0.25与0.5之间时,为中度多态性;而当多态性信息含量小于0.25时,则表现为低多态性[19],不适合直接作为育种材料。本文选取的9个SSR位点中,多态信息含量平均值为0.536,其中有5个位点属于中度多态性,4个位点属于高度多态性,据此我们认为5个棘胸蛙养殖群体整体呈现中度偏高的多态性。本文结果与课题组利用SSR长度多态性分析研究的结果[20 ]一致。Zheng等[12](微卫星标记)和刘南君等[10](RAPD分析)均认为棘胸蛙具有较高的遗传多样性,由于实验群体为人工养殖群体,因人为干扰、近亲繁殖等,导致多态性下降。类似现象在其他物种中也有报道,例如斑点叉尾鮰Ictaluruspunctatus[21]、斑节对虾Penaeusmonodon[22]、许氏平鲉Sebastesschlegeli[23]和金边鲤Cyprinuscarpiovar. Jinbian[24]等。
基因杂合度是衡量群体遗传变异的最适参数[25-26],杂合度可以分为观测杂合度和期望杂合度,当观测杂合度与期望杂合度值相近时,表明外部环境选择及近交等因素对该物种遗传变异影响较小,此时群体内处于遗传平衡状态[27]。本研究分析5个群体的期望杂合度和观测杂合度,发现期望杂合度(0.589)远远高于观测杂合度(0.182);进一步分析每个群体间的杂合度,发现各个群体内部的期望杂合度均高于观测杂合度,其中以福建群体的期望杂合度与观测杂合度差别最大,达到2.88倍。类似现象在很多人工养殖(选育)群体中均有发现,例如中华绒螯蟹[8]、黄颡鱼[9]、鳙鱼[28]、大口黑鲈[29]等,可能是由于人工养殖群体近亲繁殖普遍存在而使群体发生纯合反应,导致杂合度降低,物种对环境变化适应性减低[30]。因此,在人工养殖棘胸蛙的过程中,应尽量扩大选育范围,避免近亲繁殖,同时,建议定期从外地良种场引进良种作为繁殖亲本,以提高亲本遗传多样性。
3.2 遗传距离与遗传分化系数相关性分析
遗传距离是衡量群体间遗传关系的指标,一般来说群体遗传距离越近,遗传相似度越大,则亲缘关系越近[31-32],并认为遗传相似度大于0.5时,此时群体间的亲缘关系为一级亲缘关系[22]。本实验中,广西和江西群体、广东和江西群体、福建和江西群体、福建与浙江群体的遗相似度较低(小于0.5),可能与群体的地理位置有关;进一步构建群体遗传的UPGMA进化树,发现江西与广东、广西、福建群体遗传距离较远,同时福建与浙江群体遗传距离也较远,聚类分析结果与地理位置分布相吻合。
基因遗传分化系数(Fst)是衡量群体间遗传分化程度的重要参数。Balloux等[32]认为,当群体间Fst值大于0.25时,表示群体间的遗传分化极大。在我们选取的5个养殖群体中,平均Fst值达到0.332,显示棘胸蛙的5个养殖群体已经呈现较大的遗传分化。同时进行的分子方差分析结果显示:棘胸蛙遗传变异,大部分来自群体内个体之间,来自群体间的仅占29.05%。
3.3 HWE平衡分析
HWE平衡检验被广泛应用于分析群体遗传进化、结构特征[33]以及定位重要功能基因等方面[34]。在本文构建的45个SSR位点中,有30个位点极显著地偏离了HWE平衡,偏离率达到66.67%,远远高于鳜的野生群体(21.67%)[35]。相较而言,养殖群体的HWE平衡偏离率往往比较高,例如军曹鱼Rachycentroncanadum偏离率达到53.13%[36]、克氏原螯虾Procambarusclarkii偏离率达到82.5%[37]、红罗非鱼偏离率达到90%[31]、虾夷扇贝Mizuhopectenyessoensis偏离率达到77.5%[38]等,可能是由于人工养殖过程中,奠基者效益以及非随机交配较严重,导致群体中纯合子增加,杂合子缺失,使养殖群体的遗传结构遭到破坏。本文进一步分析5个养殖群体的偏离情况,发现浙江和广西群体HWE平衡偏离率较高,人工选择力度较低。
4 结论
本文利用9对引物在5个棘胸蛙养殖群体中进行遗传多样性分析,结果显示:棘胸蛙5个养殖群体呈现中度偏高的遗传多样性,群体间呈现高度分化,而群体内部则出现遗传多样性降低的现象,且严重偏离HWE 平衡,推测可能是瓶颈效益和奠基者效益导致了近亲繁殖现象。因此,建议在人工养殖棘胸蛙的过程中,应尽量扩大选育范围,避免近亲繁殖,同时,定期从外地良种场引进良种作为繁殖亲本,提高亲本遗传多样性。
