甘蔗割手密种NAC转录因子ATAF亚家族鉴定及栽培品种ScNAC2基因的功能分析
2023-01-12王恒波吴明星蒋钟莉林容潇郭晋隆阙友雄
王恒波 张 畅 吴明星 李 湘 蒋钟莉 林容潇 郭晋隆 阙友雄
甘蔗割手密种NAC转录因子ATAF亚家族鉴定及栽培品种基因的功能分析
王恒波**张 畅**吴明星 李 湘 蒋钟莉 林容潇 郭晋隆 阙友雄*
福建农林大学农业 / 农村部福建甘蔗生物学与遗传育种重点实验室 / 国家甘蔗工程技术研究中心, 福建福州 350002
NAC (NAM, ATAF和CUC)是陆生植物特有的转录因子家族, 包含18个亚家族, 其中ATAF亚家族成员广泛参与生物和非生物胁迫应答过程。本研究基于甘蔗割手密种基因组数据和栽培品种ROC22的cDNA文库, 首先, 通过比较基因组学方法, 对ATAF亚家族成员进行鉴定、蛋白多序列比对、系统进化分析及启动子区域顺式作用元件预测; 其次, 从甘蔗栽培品种克隆获得割手密种ATAF亚家族成员的同源基因, 在生物信息学分析基础上, 采用qRT-PCR技术分析该基因的组织特异性表达模式, 及其在不同外源胁迫下的表达特性; 最后, 对基因的编码蛋白进行亚细胞定位和转录激活活性试验。结果表明, 一共鉴定到6个ATAF亚家族成员, 开放读阅读框在889~1017 bp之间, 相对分子量介于32.067~35.819 kD之间, 理论等电点分布在5.09~8.92之间, 所有成员的编码蛋白被预测均定位于细胞核上。这些基因的Ka/Ks比值均小于1, 表明纯化选择在进化过程中起重要作用。蛋白的氨基酸序列比对显示, ATAF亚家族成员都含NAM保守结构域(由I、II、III、IV和V亚结构域组成)。系统进化分析揭示, 禾本科的甘蔗、高粱、玉米与水稻亚家族成员都聚类在一起, 表明有较近的亲缘关系, 同时拟南芥、水稻、玉米和高粱等40个ATAF亚家族成员分为两个组(Group A和Group B), 其中玉米ATAF亚家族发生明显的基因扩增现象。此外, ATAF亚家族成员启动子区域均包含响应低温、干旱和激素等逆境胁迫的顺式作用元件, 推测其参与多种生物和非生物胁迫的应答过程。进一步, 从甘蔗栽培品种ROC22中克隆获得基因的cDNA全长序列(GenBank登录号为OL982539), 其开放读码框为891 bp, 编码296个氨基酸残基; 该基因的编码蛋白与割手密种ATAF亚家族Group B 中SsNAC2蛋白的氨基酸序列相似性为97.99%。qRT-PCR表达分析结果表明,基因在甘蔗不同组织中组成型表达, 在蔗叶和蔗皮中表达量高于蔗肉、蔗芽和蔗根; 在水杨酸和茉莉酸甲酯胁迫下, 表达量显著下调; 在脱落酸、4℃低温和氯化钠胁迫下,基因的表达呈现由低到高的模式, 且差异达显著水平。亚细胞定位结果显示, ScNAC2-GFP融合蛋白定位在细胞核上。转录激活活性试验表明, ScNAC2蛋白不具有转录自激活活性。以上结果为深入解析甘蔗NAC-ATAF亚家族成员在响应生物和非生物胁迫应答中的生物学功能奠定了基础, 为甘蔗抗性分子育种提供潜在的基因资源。
甘蔗; 转录因子; NAC基因家族; 生物和非生物胁迫; 表达模式
甘蔗是世界上重要的糖料作物和能源作物, 我国甘蔗种植面积位居世界第三, 约为140.58万公顷(2018 FAO)。2021年, 我国甘蔗种植面积为111.5万公顷, 蔗茎产量大约7389万吨, 产糖量达945万吨, 对我国能源战略和国民经济发展起到重要作用。20世纪90年代以来, 甘蔗主产区逐步由湿润多雨、肥沃的东南地区(福建、广东、海南), 西移到气候较为干旱和土壤较为贫瘠的西部地区(特别是云南、广西等地)。提高甘蔗的抗逆性尤其是抗旱、抗寒和抗病性是保证甘蔗产业持续快速健康发展的重要前提和基础。因此, 若能直接从甘蔗自身挖掘和鉴定抗性基因并对其进行功能解析, 将为基于分子育种技术定向改良和培育抗性优良的甘蔗新品种积累重要理论基础, 并提供潜在的有育种利用价值的候选基因。
NAC (NAM、ATAF和CUC)是仅存在于陆生植物的转录因子家族, 也是数量最多的转录因子家族之一[1-2]。该蛋白家族分为2个大类(I和II), 分别包含14个和4个亚家族[2], 且同一亚家族内的不同成员之间具有类似的功能。NAC家族是以矮牵牛(No Apical Meristem)基因, 拟南芥的1/2 (activation factor, ATAF)基因和(cup-shaped cotyledon)基因的首字母缩写命名[3]。其中,基因影响顶端分生组织的形成与分化, 该基因编码蛋白的N端含约150个保守的氨基酸序列, 也称为NAM结构域, 可再分为I、II、III、IV和V亚结构域, 而C端序列则显示出高度分化的特征[3]。基因则通过与(PIN-FORMED1)和(PINOID)基因的互作, 调控拟南芥种子萌发过程中组织边界的产生和子叶的发育[4], 并且和的双突变体植株中雄蕊、萼片和子叶呈现愈合状态, 无法形成茎顶端分生组织[5]。和基因是最早从拟南芥中鉴定的NAC家族成员,因其能够激活酵母的花椰菜花叶病毒(, CaMV) 35S启动子[6]。研究发现, 多数陆生植物中与基因同源的NAC基因受多种非生物和生物胁迫[7-8], 因此, 将其命名为ATAF亚家族。ATAF亚家族蛋白在N端的II和V亚结构域中有高度保守基序, 分别为D-L-Y-K-X-D-P-W-X-L-P和LDDWVLCR[I/L]YNKK; 在C端的转录调控区域中也有相对特异基序EVQS[E/x]PK[W/I][2], 这些基序决定NAC转录因子与靶标蛋白的相互作用。同时, C端的多样性基序也导致NAC蛋白执行不同的生物学功能[9]。
前人研究表明, 该转录因子家族参与调控多个植物营养器官、生殖器官、细胞次生壁、侧根、叶片衰老等的生长发育, 并积极响应生物和非生物的胁迫应答[9-11]。在水稻中, 一方面, ATAF亚家族成员受到白叶枯病菌、低温、干旱和高盐胁迫的诱导表达; 另一方面, 过表达基因导致转基因水稻的生长缓慢、产量降低, 但对干旱、高盐、低温及叶枯病、稻瘟病表现出较强的抗性[12]。在大麦中, 沉默基因的转基因植株表皮细胞降低对白粉病菌的抗性, 而过表达基因的转基因株系中表皮细胞对白粉病菌的抗性增强[8]。在逆境胁迫下, 过表达大豆基因的转基因烟草能引起叶片中丙二醛含量明显升高, 活性氧(reactive oxygen species, ROS)代谢途径相关基因下调表达, 且对低温、高盐和干旱胁迫表现出较高的敏感性, 表明参与调控植株ROS信号通路, 是低温、高盐和干旱胁迫的负调控因子[13]。在水杨酸和茉莉酸处理下, 灰葡萄球菌或丁香假单胞杆菌侵染后, 拟南芥基因下调表达[14]。研究发现, 过表达基因的转基因拟南芥对于真菌和细菌性病害表现出感病表型, 如植株矮小、根长变短[14-15]。同时, 该转基因植株对脱落酸和高盐胁迫呈现较高的敏感性[15]。Lu等[16]等发现, 在干旱和脱落酸处理下, 拟南芥基因被诱导表达; 在干旱胁迫处理下, 拟南芥突变体和植株的恢复率是野生型植株的7倍, 该研究还揭示基因负调控干旱胁迫下相关基因的表达[16]。进一步研究显示, 拟南芥突变体对非宿主病原菌的渗透抗性降低, 推测ATAF1是一种新的调节ABA信号通路的衰减剂, 能够通过对ABA通路的关键基因的下调表达, 降低植株的渗透抗性[17]。Delessert等[7]发现, 在损伤、茉莉酸甲酯和水杨酸的胁迫下, 拟南芥基因呈现诱导表达模式。而在脱落酸的胁迫下,基因的表达量没有改变, 过表达基因的转基因植株病程相关基因表达量降低, 对镰刀霉菌()的抗性减弱, 揭示负向调控植物病害抗性相关基因表达。以上研究结果表明, 不同物种ATAF亚家族成员广泛参与植物对生物和非生物胁迫的响应。
在甘蔗中, Nogueira等[18]克隆获得ATAF亚家族基因在4℃低温处理下, 该基因被诱导表达, 而在12℃处理下表达量为0, 表明基因能够参与甘蔗对极端低温胁迫的响应。此外, 从甘蔗克隆获得的NAC家族成员基因, 在高盐、干旱和脱落酸胁迫下呈现诱导表达模式, 异源过表达烟草显示出衰老相关表型[19]。根据以上研究结果, 并结合课题组前期对甘蔗割手密种NAC家族成员的转录组分析, 我们获得了ATAF亚家族成员在甘蔗中的表达特性(另文发表), 推测该亚家族成员除了在蔗叶、蔗茎的生长发育过程中执行重要的生物学功能外, 对生物和非生物的逆境胁迫也起到潜在的调控作用。因此, 本研究中, 我们拟首先结合甘蔗割手密种基因组数据, 鉴定ATAF亚家族成员, 并对其进行系统进化分析、多序列比对和启动子区域的顺式作用元件预测等生物信息学分析; 其次, 克隆获得甘蔗栽培品种ROC22中ATAF亚家族的同源基因, 对其进行组织特异性和不同外源激素及低温、高盐胁迫下的表达模式分析; 最后, 进行亚细胞定位和转录激活活性验证等功能验证。研究结果有望为深入解析甘蔗ATAF亚家族成员的生物学功能奠定良好的基础, 同时为利用基因工程技术改良甘蔗品种的抗性提供候选基因资源。
1 材料与方法
1.1 甘蔗割手密种NAC-ATAF亚家族的鉴定及生物信息学分析
根据NAC家族蛋白都具有NAM结构域特征, 先从Pfam (http://pfam.xfam.org/) 数据库中下载NAM.hmm (PF02365.15)种子序列文件, 利用hmmsearch HMM模型在不同物种基因组数据库中BLAST, 得到NAC蛋白, 再利用NCBI (The National Center for Biotechnology Information) Conserved Domain (https://www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb.cgi)和SMART (http://smart.embl.de/)数据库分析保守结构域。通过与105个拟南芥NAC家族成员进行系统进化树聚类分析, 并依据拟南芥中建立的18个亚家族分类方法[2], 本研究在甘蔗割手密()基因组中鉴定到6个ATAF亚家族成员, 命名为SsNAC1~SsNAC6。甘蔗割手密种基因组数据库来源(http://sugarcane.zhangjisenlab. cn/sgd/html/download. html)。采用ExPASy网站分析理化性质。基于WOLFPSORT网站(https://wolfpsort. hgc.jp/)进行亚细胞定位预测; 基于cNLS Mapper (http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)网站进行核定位信号预测。利用Prabi软件(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_hnn.html)预测蛋白的二级结构。采用Meme suite在线网站 (https://meme-suite.org/meme/ tools/meme)预测Motif基序。运用MEGA7软件的最大似然法(maximum likelihood, ML), 采用多个不同物种(拟南芥、水稻、玉米、高粱和甘蔗)NAC-ATAF亚家族的氨基酸序列构建系统进化树[2,20-21], 并将Bootstrap 设置为1000, Poisson model, 其他为默认参数。利用TBtools软件对保守基序分析和MEGA7聚类结果合并及顺式作用元件进行绘图[22]。依据基因起始密码子ATG上游2000 bp为启动子区域, 利用plantCARE网站预测顺式作用元件。运用DNAMAN软件对其进行蛋白的氨基酸序列比对分析。使用Adobe Illustrator CS6软件处理图片。
1.2 甘蔗栽培品种NAC转录因子ATAF亚家族成员基因的克隆和序列分析
基于甘蔗栽培品种的PacBio三代全长转录组测序注释基因文库, 利用甘蔗割手密种ATAF亚家族6个成员CDS (coding sequence)为查询序列, 对其进行本地BLAST序列比对, 从中筛选到1条相似度高的同源基因序列(c95048f2p01259), 以此为模板, 利用Primer Premier 5.0设计克隆引物(表1), 从我国栽培面积最大的甘蔗栽培品种ROC22中克隆其同源基因。进一步以甘蔗栽培品种ROC22的不同组织(根、茎、叶)混合cDNA为模板, PCR扩增, 将符合预期目标基因片段的PCR产物进行胶回收、连接、转化、克隆和测序, 将测序结果正确的基因命名为, 并利用NCBI-ORF finder分析基因的开放读码框(https://www.ncbi.nlm.nih.gov/orffinder/)。
1.3 甘蔗ScNAC2基因在不同组织和激素及非生物胁迫下表达模式分析
1.3.1 甘蔗的处理和取样 对甘蔗栽培品种ROC22蔗茎切段, 催芽处理后, 温室(30℃, 60% RH)下培养20 d获得无性繁殖苗。选择生长状态良好, 且高度和长势相对一致的幼苗作为试验材料。4℃低温和250 mmol L–1氯化钠(sodium chloride, NaCl)的处理后, 分别采集0、24、48、72 h和0、12、24、48、72 h处理叶片样品[23]。同时, 参照Liu等[24]方法, 用0.1 mmol L-1脱落酸(abscisic acid, ABA)喷施无性繁殖苗的叶片, 采集处理 0、3、6和24 h的叶片样品; 用5 mmol L–1水杨酸(salicylic acid, SA)和0.1 mmol L–1茉莉酸甲酯(methyl jasmonate, MeJA)喷施无性繁殖苗的叶片, 采集处理0、3、12和24 h[25]。此外, 选取长势一致的甘蔗栽培品种ROC22成熟期植株, 连根挖出, 冲洗干净后, 选取幼嫩的根、+1叶、蔗芽、蔗皮和蔗肉组织, 用于目标基因的组织特异性表达分析[24]。以上样品液氮冷冻后保存-80℃冰箱。每3株为1次生物学重复, 每个样品设置3次生物学重复。
1.3.2 实时荧光定量PCR检测基因的表达量 利用南京诺唯赞生物科技股份有限公司的RNA-easy Isolation Reagent试剂提取甘蔗不同组织及不同外源胁迫样品的RNA。按照上海翌圣生物科技股份有限公司Hifair II 1st Strand cDNA Synthesis Kit的操作说明, 将RNA反转录合成cDNA第1链, 以此为定量PCR模板。甘蔗甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase, GAPDH)为内参基因[26], 采用Primer premier 5.0软件设计基因的定量PCR引物(表1)。按照南京诺唯赞生物科技股份有限公司的ChamQ Universal SYBR qPCR Master Mix说明书配置体系, 在ABI Quant Studio3荧光定量PCR仪上进行qRT-PCR扩增, 3个技术重复, 利用2–ΔΔCt法进行数据处理和分析[27], 并用GraphPad Prism软件进行显著性分析, 最后运用Microsoft Excel 2016作图。
1.4 亚细胞定位分析
根据基因序列设计入门载体引物(表1), 进行PCR扩增, 将纯化的PCR产物按照Invitrogen公司的Gateway BP Clonase II Enzyme Mix试剂连接到入门载体pDONR207; 将测序验证正确的pDONR207-质粒, 再使用Invitrogen公司的Gateway LR Clonase II Enzyme Mix试剂连接至亚细胞定位载体pFAST-R05-GFP, 经测序验证后获得重组质粒pFAST-R05--GFP。将重组质粒和空载质粒分别转化农杆菌GV3101[28], 根据Liu等[28]方法将活化后的重组载体和空载体的农杆菌GV3101菌液针刺接种3~4叶龄的本氏烟()叶片, 置于28℃培养箱中(光照16 h/黑暗8 h), 连续培养48 h[28], 采用Leica TCS SP8激光共聚焦显微镜(莱卡, 德国)对其照相。
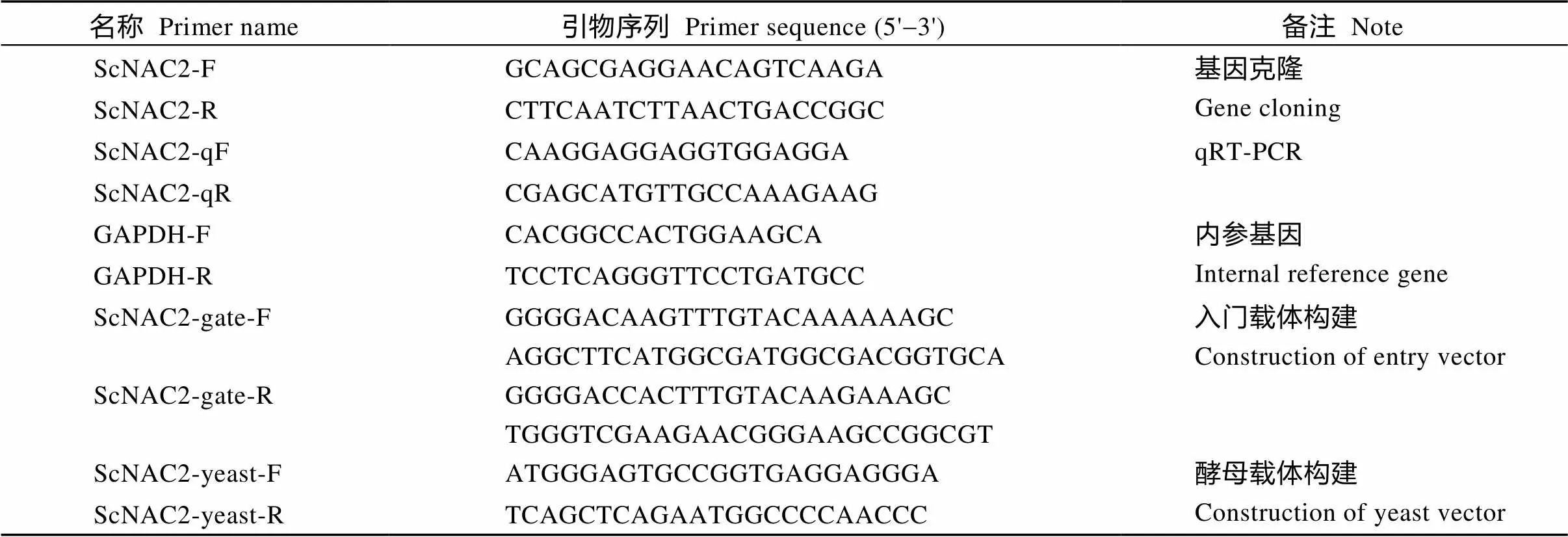
表1 本研究所用引物序列
1.5 转录自激活活性验证
基于Primer Premier 5.0软件设计引物用于酵母载体构建(表1), 通过宝日医生物技术(北京)有限公司的In-Fusion PCR Cloning Kit试剂盒, 将甘蔗基因的ORF (Open reading frame)序列融合到pGBKT7载体上的GAL4 DNA结合域, 构建pGBKT7-ScNAC2重组表达载体。针对pGBKT7-53+ pGADT7-T (阳性对照)、pGBKT7 (阴性对照)、pGBKT7-ScNAC2 (重组载体), 采用PEG/LiAc (polyethylene glycol/lithium acetate)法转化至Y2H酵母感受态细胞, 涂布于SD/-Trp的平板上, 30℃培养2~3 d后观察酵母生长情况。随机挑取2~3 mm的酵母单菌落于1 mL 0.9%的生理盐水重悬菌体, 吸取5 μL悬浮的菌液, 打点于SD/-Trp、SD/-Trp-His/X--gal、SD/-Trp-His-Ade/X--gal平板上, 30℃培养, 采用Leica M125 C体视显微镜(莱卡, 德国)照相和观察菌落的生长情况。
2 结果与分析
2.1 甘蔗割手密种NAC-ATAF亚家族基因的鉴定及理化性质分析
通过序列比对和分析, 共筛选到151个家族成员的蛋白序列, 与拟南芥的105个NAC家族成员聚类后, 发现其中6个成员的蛋白序列属于NAC- ATAF亚家族(表2)。理化性质分析表明, 所有亚家族成员的氨基酸数量分布在295~376 aa之间, 与高粱直系同源基因编码的氨基酸数量基本一致, 其开放读阅读框在889~1017 bp之间, 相对分子量介于32.067~35.819 kD之间, 其理论等电点分布5.09~ 8.92之间, 有3个成员的等电点大于7, 是碱性蛋白, 另外3个蛋白的等电点小于7, 是酸性蛋白, 同时它们的平均疏水性预测指标为负值, 表明都是亲水性蛋白。除SsNAC1的不稳定系数小于40外, 其他5个成员都大于40, 说明该亚家族大部分成员都是亲水性稳定蛋白。另外, 核定位区间和亚细胞定位预测结果表明所有亚家族成员均定位于细胞核上。
以高粱和甘蔗割手密种NAC-ATAF亚家族直系同源基因对为目标, 来估算基因进化过程中受到的选择压力, 当Ka/Ks > 1, 表明基因受正选择压力, Ka/Ks < 1, 表明基因受纯化选择压力。从表2中可以看出, 除外, 其他5个基因对的Ka/Ks都小于0.3, 表明纯化选择在NAC-ATAF亚家族的进化中发挥主导作用, 可能预示着该亚家族基因比较保守且有重要的生物学功能。
2.2 甘蔗割手密种NAC-ATAF亚家族蛋白的序列比对和系统进化分析
本研究将甘蔗割手密种的6个ATAF亚家族成员、甘蔗热带种的1个成员(SsNAC23)及拟南芥的4个ATAF亚家族成员(ATAF1、ANAC032、ATAF2和ANAC102), 进行蛋白的氨基酸序列比对(图1)。结果显示, 所有蛋白都符合NAC转录因子特异的ATAF亚结构域特征, N端蛋白序列都高度保守。同时, 在III亚结构域上有类似于WRKY的保守序列WKATGAD的氨基酸残基。值得注意的是, 来自甘蔗热带种的SsNAC23与割手密种的SsNAC4成员仅在7个氨基酸残基上存在差异, 相似性高达97.64%, 这从侧面佐证了热带种和割手密种亲缘关系较近, 由甘蔗属的共同祖先种分化而来。
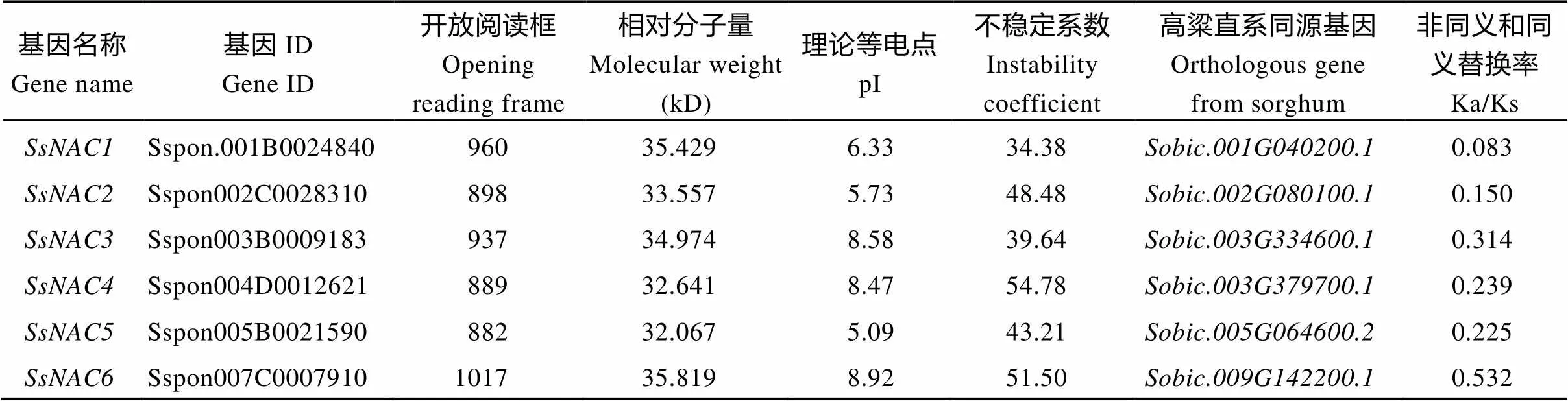
表2 甘蔗割手密种SsNAC-ATAF成员的理化性质分析

图1 甘蔗和拟南芥NAC-ATAF亚家族蛋白的序列比对
基于上述的方法, 本研究鉴定到其他物种的ATAF亚家族, 如玉米(13个)、水稻(6个)和高粱(6个), 并结合文献报道的其他物种ATAF亚家族成员, 比如: 大豆() (AAX85979.1, GmNAC2)、石兰属梭梭() (AIS71992.1, HaNAC1)、芒属南荻() (AIS74872.1, MlNAC5)、甘蔗热带种() (AAW62955.1, SsNAC23)、大麦() (HORVU1Hr1G063740.1, HvNAC6),共5个ATAF亚家族基因, 合计40个成员。利用MEGA7软件将其构建系统进化树, 40个成员可分为2个组(Group A和Group B) (图2), Group A的一部分成员有来自双子叶植物的拟南芥4个、大豆1个、梭梭1个, 并聚类为相对独立分支; 另一部分成员有单子叶植物的南荻1个、甘蔗热带种1个、大麦1个、水稻3个, 高粱3个、割手密种3个和玉米7个被聚类一起, 该组也是成员数量最多的组, 共有25个成员; Group B有19个成员均来自禾本科植物, 除来自玉米的9个成员外, 其他禾本科物种(高粱、玉米、水稻和甘蔗)均只有3个成员, 且这些物种基本都是成组出现, 尤其是高粱和甘蔗基本都是成对出现, 不仅表明二者具有较近的亲缘关系, 而且也预示其生物学功能相近。同时, 本研究发现, 玉米ATAF亚家族成员数量多于甘蔗、高粱和水稻, 表明玉米在演化过程中ATAF亚家族成员出现明显的扩张现象。系统进化关系进一步证实单子叶植物(水稻、玉米、高粱和甘蔗)亲缘关系要近于双子叶植物(拟南芥、大豆)。
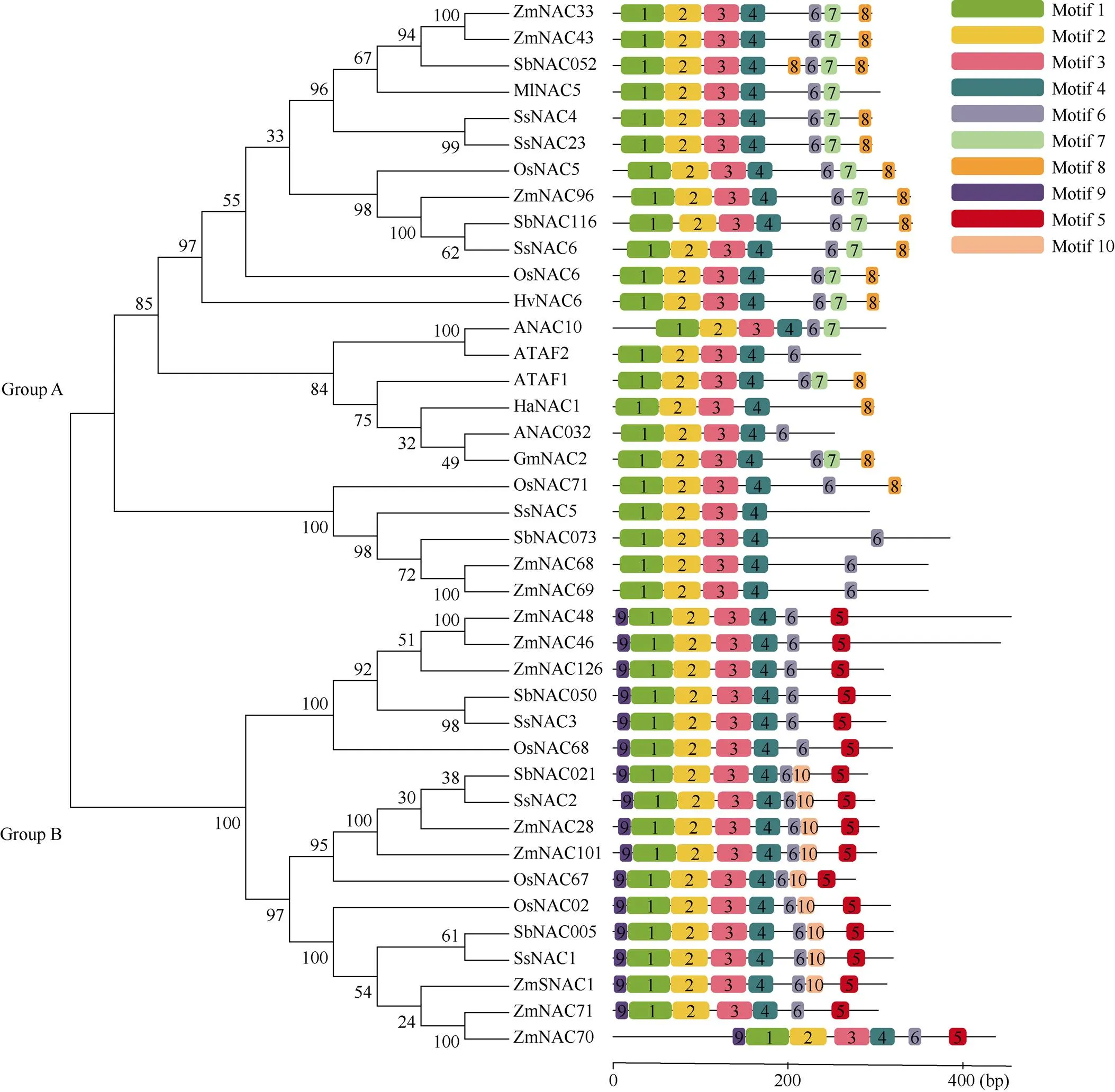
图2 NAC-ATAF的系统进化树的构建与保守基序的预测
NAC-ATAF成员分别来自: 玉米: ZmNAC; 拟南芥: ANAC或ATAF1/2; 水稻: OsNAC; 高粱: SbNAC; 大豆: AAX85979.1, GmNAC; 石兰属梭梭: AIS71992.1, HaNAC1; 芒属南荻: AIS74872.1, MlNAC5; 甘蔗热带种: AAW62955.1, SsNAC23; 甘蔗栽培品种: ScNAC2; 大麦: HORVU1Hr1G063740.1, HvNAC6。
These NAC-ATAF members are from different species. ZmNAC:; ANAC or ATAF1/2:;: OsNAC; SbNAC:; AAX85979.1, GmNAC:; AIS71992.1, HaNAC1:; AIS74872.1, MlNAC5:; AAW62955.1, SsNAC23:; ScNAC2:hybrid; HORVU1Hr1G063740.1, HvNAC6:.
从亚家族蛋白的保守结构域显示(图2), 同一个分组的不同物种成员具有相似的保守基序, 蛋白序列的N端(1~200 aa)相对保守, 而C端分化比较明显(200~400 aa)。motif 1~motif 4出现在所有家族成员中, 同组成员存在更多相同基序, 如除双子叶植物4个成员(ATAF2、ANAC102、HaNAC1和ANAC032)外, 第1组的其他成员都有motif 7和motif 8; 又如motif5和motif9只出现在第2组上, 且大部分成员有motif 10。我们推测, 在物种演化过程中, NAC- ATAF不同组成员由于在保守基序存在明显分化, 可能导致其在功能上也发生着分化。
2.3 甘蔗ATAF亚家族的顺式作用元件预测
探索和分析位于启动子区域的顺式作用元件, 对于了解特定逆境下基因的组织特异性或应激反应具有重要作用。基于比较基因组学和生物信息学方法,本研究对6个割手密种ATAF亚家族成员的启动子区域进行顺式作用元件预测, 除去一般性转录调控元件和功能未知元件外, 共检测到305个元件, 分为激素响应元件数量最多有108个, 分布在所有亚家族成员中, 非生物响应元件有66个、转录调控相关元件有63个及光响应元件有68个(图3)。其中, SsNAC2和SsNAC4的响应元件数量较多, 分别有74个和65个, 其他成员响应元件范围分布31~48之间。在激素响应元件中, 所有成员都含有ABA和MeJA的诱导元件, 且二者占比为91.0%, 但响应SA和吲哚-3-乙酸(Indole-3-acetic acid, IAA)元件数量较少。在非生物胁迫响应元件中, 大部分成员都涉及低温、机械损伤和厌氧诱导响应元件, 且数量相对较多, 而抗旱和赤霉素响应元件数量较少。在转录调控相关响应元件中,每个成员启动子区域的响应干旱和耐盐的MYB (v-myb avian myeloblastosis viral oncogene homolog)、MYB like及MYC (myelocytomatosis)响应元件出现数量最多, 其次是与WRKY (WRKYGQK) 转录因子相关的作用元件。而光响应元件中, 除了G-box motif是每个成员都具有的诱导元件外, 其他元件在每个成员上分布相对较少, 也呈不均匀分布。
2.4 甘蔗栽培品种ScNAC2基因的克隆与生物信息分析
现有文献报道ATAF亚族成员相关功能的分析都是来自ATAF Group A的成员[8,13,18,29-31], 但迄今尚未见Group B组成员的分析。为探讨ATAF亚家族成员Group B执行的生物学功能, 本研究以甘蔗割手密种基因开放阅读框序列比对甘蔗栽培品种的PacBio三代全长转录组测序注释基因文库,获得一条相似性高的全长转录本序列, 并以此序列为模板, 设计引物, 克隆甘蔗栽培品种ROC22中的同源基因。利用RT-PCR技术, 克隆获得同源基因(图4), 开放读码框长度分别891 bp, 编码296氨基酸, 与SsNAC2蛋白的氨基酸序列相似性高达97.99%, 仅有6个氨基酸残基的差异, 且与割手密种ATAF亚家族的其他蛋白序列相似性为55.73%~71.05%, 表明所克隆的甘蔗栽培品种基因可能来源于割手密种。保守结构域预测显示, ScNAC2蛋白具有典型和完整的NAM结构域。核定位区间和亚细胞定位预测表明ScNAC2蛋白定位于细胞核上。该蛋白的蛋白二级结构主要由α螺旋(29.19%)、β折叠(11.41%)和无规则卷曲(59.40%)组成, 其分子量为34.995 kD, 理论等电点pI为6.20。针对ScNAC2蛋白序列, 利用NCBI数据库进行相似性比对, 结果发现高粱XP_002459566.1蛋白序列与它相似性最高(94%), 这也暗示甘蔗与高粱具有较近的亲缘关系, 且ScNAC2在物种进化过程中具有较高保守性。
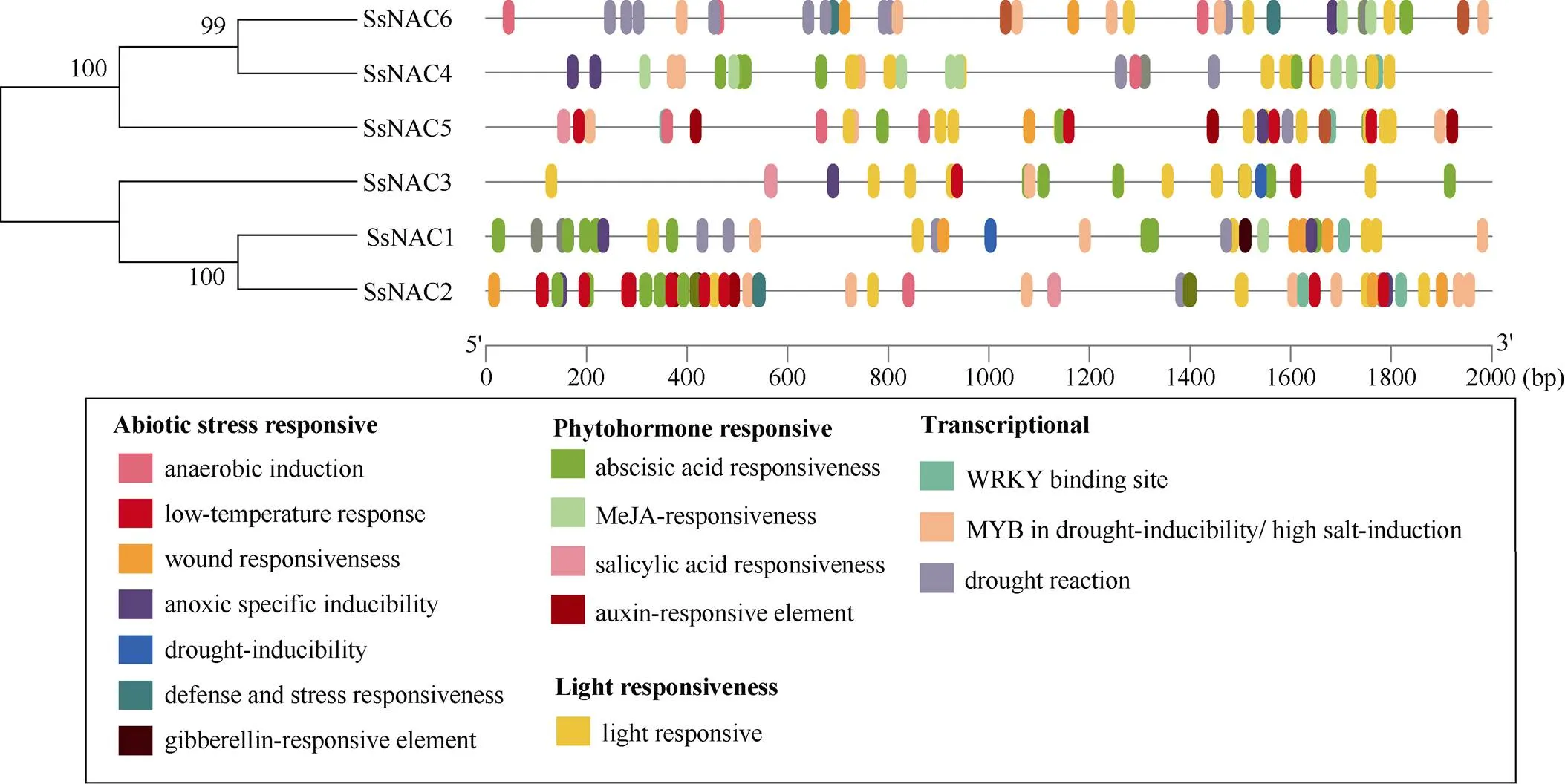
图3 顺式作用元件预测
2.5 甘蔗ScNAC2基因的组织特异性表达分析
qRT-PCR结果表明, 与蔗根相比,基因在蔗叶和蔗皮的表达量最高(图5), 分别是蔗根的9.7倍和8.0倍; 而蔗肉、蔗根和蔗芽中表达量较低, 且三者表达量没有显著性差异。因此,基因在甘蔗栽培品种ROC22中组成型表达, 在蔗叶和蔗皮中呈现高表达。

图4 ScNAC2的核酸序列及推导的氨基酸序列
下画线部分: ScNAC2蛋白的NAM保守结构域。*: 代表终止密码子。
Underlined: the NAM conserved domain of ScNAC2 protein. *: stop codon.
2.6 甘蔗ScNAC2基因在不同逆境胁迫下的表达模式分析
在SA、MeJA、ABA、高盐和低温的胁迫处理下,基因表现出不同表达模式(图6)。在SA处理下,基因的表达量呈现由高到低表达趋势; 在MeJA处理下,基因的表达量在0~12 h呈现由高到低表达趋势, 并在24 h时又恢复到对照水平。在ABA处理下,基因表达量呈现由低升高趋势, 24 h时基因的表达量达最高值,为对照的5.39倍。在NaCl胁迫下,基因表达量在24 h内表现响应胁迫, 且表达量由低到高, 接近对照的2.83倍, 但在24~72 h间基因表达量逐渐下降对照水平。在4℃低温胁迫下,基因的表达量在0~72 h内表现出响应胁迫的诱导表达模式, 第72 小时的表达量最高, 为对照的6.97倍, 且差异显著。表明,基因不同程度地受SA、MeJA、ABA、高盐和低温胁迫的调节。我们推测, 该基因在SA、MeJA胁迫下相对表达量低于ABA、高盐和低温胁迫, 可能是由于该基因对非生物胁迫具有较高的敏感性, 主要参与响应非生物胁迫的防御反应。
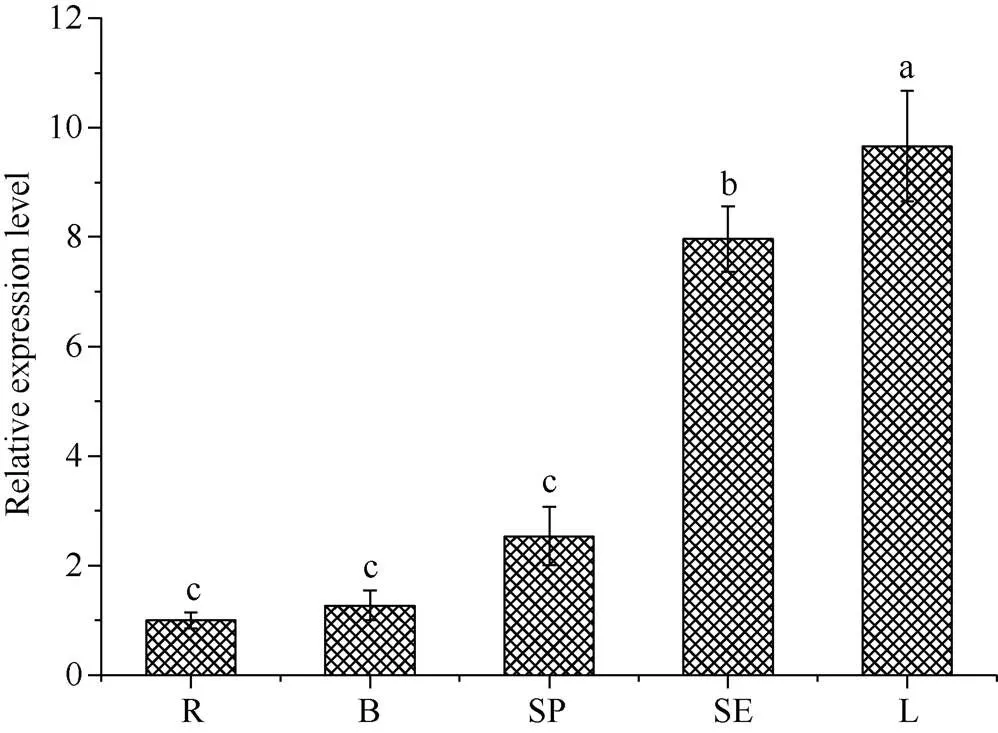
图5 ScNAC2基因在甘蔗不同组织的表达量
R: 蔗根; B: 蔗芽; SP: 蔗肉; SE: 蔗皮; L: 蔗叶。误差线为每组处理的标准误差(= 3)。不同小写字母表示表示5%水平下差异的显著性。
R: root; B: bud; SP: stem pith; SE: stem epidermis; L: leaf. The error bar represents the standard error of each group treatment (= 3). Different lowercase letters indicate significant differences at the 5% probability level.
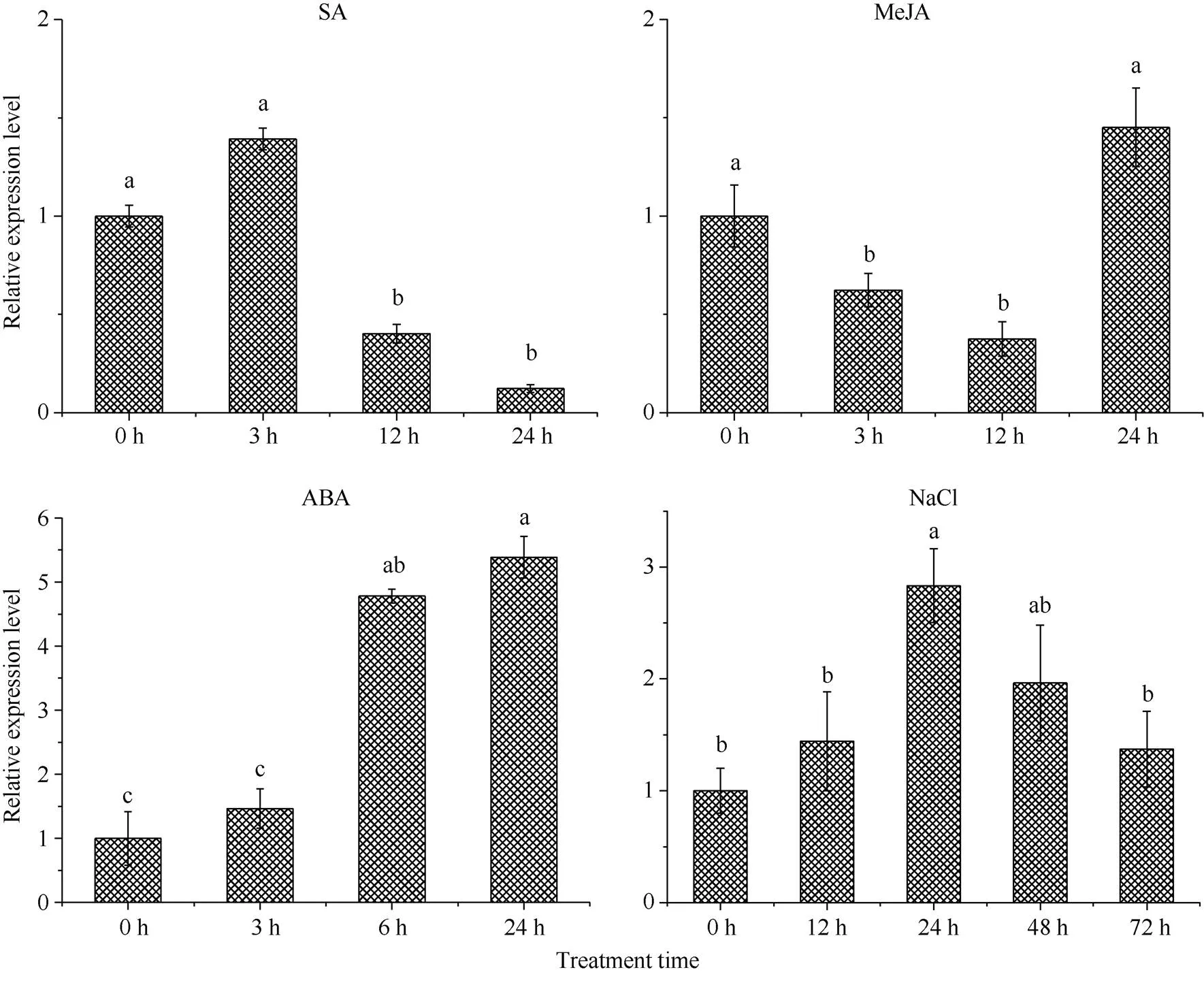
(图6)
基因在水杨酸、茉莉酸甲酯、脱落酸、氯化钠和4℃低温处理下的表达量。纵坐标代表基因的表达量; 横坐标代表不同处理时间。误差线为每组处理的标准误差(= 3)。不同小写字母表示表示5%水平下差异的显著性。
The relative expression level ofgene under SA, MeJA, ABA, sodium chloride, and 4℃ cold temperatures. Y-axis represents the relative expression level ofgene; X-axis represents the different treatment times. The error bar represents the standard error of each group treatment (= 3). Different lowercase letter indicates the significant differences at the 5% probability level.
2.7 蛋白的亚细胞定位
构建瞬时表达载体pFAST-R05-ScNAC2-GFP融合载体, 转化农杆菌GV3101后针刺接种、侵染、共培养烟草叶片, 通过融合蛋白的绿色荧光显示目的蛋白的亚细胞定位。从图7可以得出, 具有GFP的空白载体对照烟草叶肉细胞的质膜、细胞质和细胞核中均没有绿色荧光, 也没有细胞器特异性分布。融合载体表达蛋白仅在细胞核有绿色荧光, 表明其主要定位细胞核, 该结果与采用WOLFPSORT预测的亚细胞定位结果相吻合。
2.8 转录激活活性验证
利用酵母试验研究ScNAC2蛋白的自激活转录活性。由图8可知, 在SD-Trp培养基上, 重组表达载体pGBKT7-ScNAC2、阴性对照pGBKT7和阳性对照pGADT7-TpGBKT7-P53都呈现出白色菌落, 表明基因成功转化酵母Y2H Gold细胞, 且可以激活下游色氨酸基因表达, 在色氨酸缺陷型培养基正常生长。在SD-Trp-His+X--gal+AbA培养基上, 阴性对照没有出现菌落, 重组表达载体为白色菌落, 而阳性对照表现出蓝色菌落, 表明后两者激活组氨酸基因表达, 但只有阳性对照激活编码半乳糖苷酶已基因表达。在SD-Trp-His-Ade+X--gal+ AbA培养基上, 只有阳性对照正常生长并显示蓝色菌落, 重组表达载体和阴性对照都没有菌落, 表明ScNAC2蛋白没有转录激活活性。
3 讨论
NAC作为陆生植物特有的转录因子基因家族, 主要参与调控植物生长发育过程, 如花器官分化、侧根形成、细胞次生壁形成及植株衰老等[9-11]。该转录因子不同亚家族成员之间在保守结构域上的差异导致了功能的多样性, 其中ATAF亚家族对于植物参与逆境胁迫的防御反应起到重要功能[11]。NAC- ATAF亚家族已经在多个物种中被鉴定, 比如在拟南芥中有4个成员、高粱中有6个、玉米中有12个、水稻中有6个[6,20-21]。本研究在甘蔗割手密种基因组中鉴定到6个ATAF亚家族成员, 与高粱、水稻相比, 三者在数目上差异不大。前人研究表明, 高粱、甘蔗与玉米在1200~1500万年前由共同祖先分化而来, 其中甘蔗与高粱在800~900万年前有共同祖先[32], 而玉米的祖先种在5~20万年前经历一次全基因组复制事件[33], 同时甘蔗割手密种的祖先种最少经历2次全基因组复制事件形成同源八倍体[34], 据此我们推测甘蔗割手密种的2次全基因组复制事件对NAC-ATAF亚家族成员的数量没有影响, 而玉米的NAC-ATAF亚家族成员数量的扩增可能来自于玉米特有的全基因组加倍事件。核定位区间和亚细胞定位预测结果表明所有ATAF亚家族成员均定位于细胞核上, 这与已报道NAC转录因子的定位结果一致[18-20,35]。本研究通过烟草亚细胞定位发现, ScNAC2是一个定位于细胞核的蛋白, 这与生物信息预测和已报道的亚细胞定位结果相一致[18-19,29,31,35-36]。以高粱和割手密种NAC-ATAF亚家族成员计算Ka/ Ks比值, 结果显示所有Ka/Ks < 1, 其中有5个基因对Ka/Ks<0.32, 表明其经历较强的纯化选择压力, 进一步揭示该亚家族基因比较保守且可能执行一些重要的生物学功能。
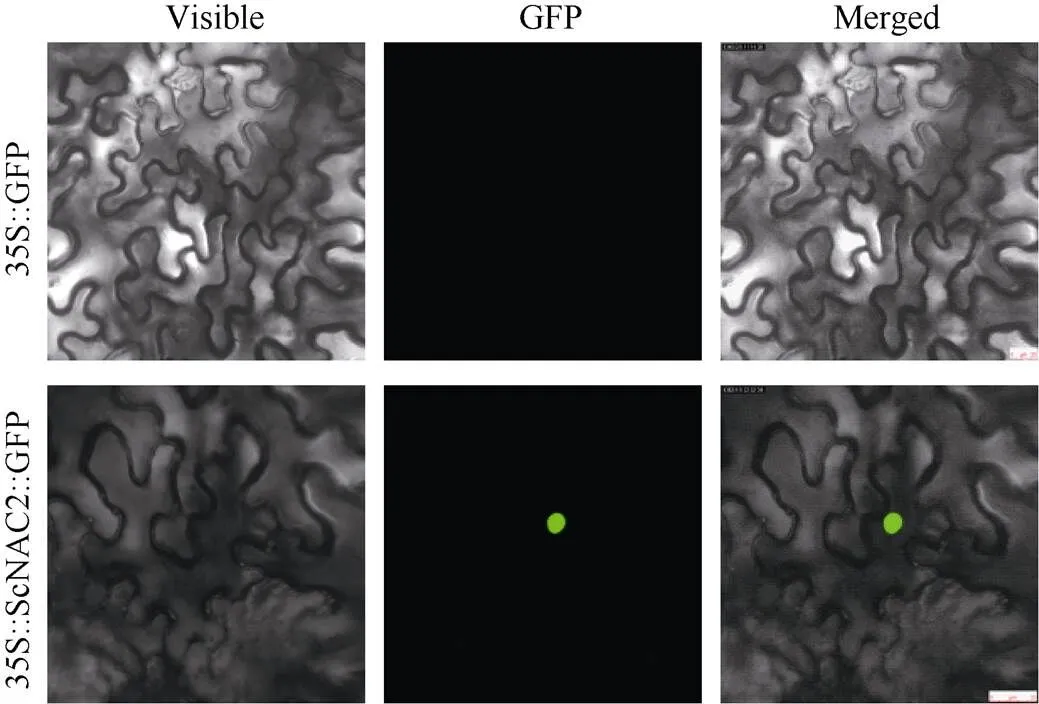
图7 ScNAC2蛋白的亚细胞定位
该结果包含明场、绿色荧光、叠加场3个视野下拍摄的照片。35S::GFP 代表空载的GV3101菌液注射本氏烟叶片的结果; 35S::ScNAC2::GFP重组载体的GV3101菌液注射本氏烟叶片的结果。
The results contained photographs taken in three fields of view, visual field, green fluorescence, and merged field. 35S::GFP: GV3101 bacterial solution injection inleaf by empty vector; 35S::ScNAC2::GFP: GV3101 bacterial solution injection inleaf by recombinant vector.

图8 ScNAC2转录激活活性验证
SD/-Trp: 色氨酸营养缺陷型平板培养基; SD/-Trp-His (+X--Gal+AbA): 色氨酸+组氨酸营养缺陷型平板培养基(添加5-溴-4-氯-3-吲哚--D-半乳糖苷和金担子素A); SD/-Trp-His-Ade (+X--Gal+AbA): 色氨酸+组氨酸+腺嘌呤营养缺陷型平板培养基(添加5-溴-4-氯-3-吲哚--D-半乳糖苷和金担子素A)。
SD/-Trp: the synthetic dropout medium without tryptophan plate; SD/-Trp-His (+X--Gal+AbA): the synthetic dropout medium without tryptophan and histidine plate (add 5-Bromo-4-chloro-3-indolyl-alpha-D-galactopyranoside and Aureobasidin A); SD/-Trp-His-Ade (+X-- Gal+AbA): the synthetic dropout medium without tryptophan, histidine, and adenine plate (add 5-Bromo-4-chloro-3-indolyl-alpha-D-galactopyranoside and Aureobasidin A).
现代甘蔗栽培品种是由热带种和割手密种进行种间杂交, 并与热带种多次回交而来[37]。甘蔗割手密种是抗旱、抗寒和抗病等优异基因资源的重要供体, 且是甘蔗属目前唯一完成基因组测序组装到染色体水平的同源多倍体[34]。因此, 在鉴定和挖掘甘蔗割手密种中NAC家族基因的基础上, 克隆获得栽培品种中同源基因并对其参与或介导的生物学功能进行研究, 有望为提高甘蔗栽培品种的抗逆性和适应性提供参考和借鉴。植物NAC家族蛋白的N端序列具有高度保守、约有150~160个氨基酸残基组成NAM结构域, NAM可划分为5个亚结构域[2], 分别为I、II、III、IV和V, 其中III和IV 2个亚结构域中含有核定位信号, 可能参与核定位及识别启动子上特定顺式调控元件。然而, NAC家族蛋白的C端为转录调控区, 保守性较低, 存在生物学功能的分化[38], 主要发挥调节NAC转录因子与各种靶蛋白的相互作用[9]。因此, 我们课题组从甘蔗割手密种中鉴定到6个ATAF亚家族成员和克隆获得甘蔗栽培品种基因, 且都符合NAC蛋白的保守结构域特征, 属于NAC-ATAF亚家族成员。同时, 栽培品种ScNAC2与割手密种SsNAC2成员具有较高的蛋白序列相似性(97.99%), 属于ATAF亚家族的Group B。
前人研究表明, NAC-ATAF亚家族成员在植物的不同组织器官中组成型表达, 且不同组织中存在表达量的差异。比如: 梭梭的ATAF亚族的基因组成型表达, 且在茎和叶中的表达量明显高于根和种子[29]; 又如, 玉米的基因在不同组织中组成型表达, 且在叶片中的表达量最高, 在叶耳和花丝中表达量最低[25]。本研究中, 我们克隆获得的基因在甘蔗所有组织中都有表达, 尤其在蔗叶和蔗茎中的表达量高, 蔗根、蔗芽和蔗肉中的表达量低, 这与其他物种ATAF亚家族基因, 如[39]、和[36]的表达模式相吻合, 表明该基因是组成型表达, 对于甘蔗生长发育起到重要的作用, 推测其首要扮演的功能与NAC家族最主要功能基本一致, 即参与植物营养、生殖器官发育和促进细胞次生壁的形成。
当植物受到细菌、真菌和病毒等病原微生物侵染时, 可快速启动防御反应信号通路, 包括水杨酸和茉莉酸甲酯及乙烯等抗病信号, 它们通过诱导抗性基因的表达或介导信号传递途径等参与植物抗病防御反应[40]。SA能够抑制过氧化氢酶活性, 促进H2O2的积累和木质素的合成, 进而抑制病原菌的侵染[41]。已有研究表明, 对紫花苜蓿喷施SA 12 h 后, 诱导其NAC亚家族基因表达, 提高细胞壁木质素和纤维素含量, 增加对病原菌的抗性[42]。在SA和JA处理拟南芥植株后,基因表达量显著受到抑制, 过表达转基因植株对丁香假单胞菌番茄致病变种的抗性减弱[14], 反之, 拟南芥植株在接种丁香假单胞菌番茄致病变种后, 促进了防御相关基因(pathogenesis-related protein, PR)、和(plant defensing 1.2, PDF)的上调表达[14]。在损伤、MeJA和SA的胁迫下,基因受到诱导表达, 而过表达和基因的转基因植株都对致病菌的抗性减弱[7], 这些研究结果表明, 尽管和基因在逆境胁迫过程中表达趋势不同, 但在病害防御上都具有负反馈调节作用。在SA和MeJA处理玉米后,基因也是显著下调表达[30]; 在MeJA处理水稻后,和基因被诱导表达[31,43], 水稻OsNAC5转录因子能直接激活肉桂酰辅酶a还原酶(OsCCR10), 通过调节木质素积累, 提高植物耐旱性[44]。在本研究中, 甘蔗基因的表达量, 在SA和MeJA胁迫后显著下调表达, 这与拟南芥的和玉米的基因表达模式一致[14,30], 但与拟南芥的和水稻的、基因表达模式不同[7,31,43]。预测其可能参与病原菌抗性相关, 但鉴于该基因与其他NAC家族成员表达模式不同, 推测不同亚族成员参与的生物学功能有所差异。
植物非生物胁迫主要是指干旱、高盐和低温等逆境胁迫。在受到非生物胁迫后, 植物感知相应的信号, 通过ABA、GA、乙烯和生长素等信号途径传递, 胁迫应答相关因子接收后, 启动胁迫应答基因的表达, 激活抗逆反应并响应逆境胁迫[11]。在高盐、低温和ABA胁迫下, 水稻基因被诱导表达[12]; 在高盐和ABA胁迫下, 玉米基因的表达受到诱导, 并在启动子区域发现多个激素响应元件, 同时转基因拟南芥在ABA胁迫下的发芽率高于对照组[36]。在干旱和正常胁迫下, 过表达梭梭基因的转基因拟南芥植株在干旱胁迫下脯氨酸、IAA和ABA含量明显提高[35]。过表达南荻基因的转基因拟南芥, 通过ABA信号通路增强转基因植株对于低温的响应[29]; 在拟南芥中, ABA植物激素生物合成关键基因的启动子上有ATAF1的结合位点, ChIP (chromatin- immunoprecipitation)和共表达定量分析都证明ATAF1与NCED3启动子的结合能够调控ABA的生物合成[45]。本研究中, 甘蔗的表达量在ABA、高盐和低温胁迫下, 都出现显著上调表达, 这与其他植物ATAF亚家族成员的表达模式基本一致[8,12,29,36], 推测其参与ABA信号通路的逆境胁迫防御反应[38], 在植物适应逆境胁迫中起到重要作用。综上所述, 甘蔗基因广泛参与甘蔗响应生物胁迫和非生物胁迫(低温、高盐)的作用, 但尚需遗传转化试验验证其确切的生物学功能。
转录因子的主要作用是结合在靶标基因的启动子部位与RNA聚合酶形成转录起始复合体因子, 进而激活或抑制下游靶标基因的表达[46]。因此, 通过验证含有重组表达载体的酵母能否在筛选培养基上正常生长, 可以揭示转录因子是否具有转录激活活性。本研究中, 我们通过酵母试验发现在筛选SD- Trp-His+X--gal+AbA培养基上, 转化重组表达载体的酵母都能正常生长, 且表现为白色菌落, 表明重组表达载体蛋白能激活组氨酸和(auranofin)基因的表达, 具有转录激活活性, 但不能引起底物X--gal变蓝色, 这提示没有激活半乳糖苷酶基因表达。然而, 在SD-Trp-His-Ade+X--gal培养基上, 重组表达载体的酵母不能激活腺嘌呤的表达, 无法正常生长。因此, 以上结果证明了甘蔗ScNAC2蛋白在酵母细胞中没有转录激活活性。前人研究结果表明, 拟南芥NAC转录因子(secondary wall- associated NAC domain protein)能结合到MYB46启动子上, 转录激活茎秆的基因表达, 调控纤维分和导管中次生细胞壁合成相关基因的表达[47]。当茎秆基因表达被抑制后, 导致纤维分和导管的次生细胞壁含量减少; 而过表达基因可激活纤维素、木质素的生物合成通路, 引起细胞内次生细胞壁的异常沉积[47]。此外, 拟南芥VND7 (vascular-related NAC-domain)作为木质部形成的主要调控因子, 与NAC家族成员VNI1 (VND-interacting1)和VNI2相互作用, 其中VNI2作为转录抑制子, 主要在根和茎的木质部及韧皮部表达, 并通过与VND7的互作调控木质部细胞生长发育[48]。鉴于甘蔗ScNAC2蛋白在酵母细胞中没有转录激活活性, 进一步的研究不仅可以在缺陷型培养基上筛选酵母双杂交文库, 获得与ScNAC2的互作蛋白, 还能对ScNAC2调控的下游靶基因的转录表达情况进行分析。后续, 我们将在筛选获得甘蔗ScNAC2互作蛋白的基础上, 探讨他们之间的互作是如何调控下游靶标基因的转录表达, 最终解析甘蔗NAC-ATAF亚家族成员在响应生物和非生物胁迫应答中的生物学功能。
4 结论
本研究首先获得6个甘蔗割手密种NAC-ATAF亚家族基因成员, 并对其理化性质、系统进化、蛋白的氨基酸基序等进行分析, 并从ATAF亚家族基因启动子区域预测到多个与生物和非生物胁迫相关的顺式作用元件。其次, 本研究从甘蔗栽培品种ROC22中克隆到与割手密种的同源基因, 了解了该基因在甘蔗不同组织中是组成型表达, 尤其是在蔗叶和蔗皮中表达量高。再次, 发现该基因在ABA、高盐和4℃低温的胁迫下被诱导表达, 而受SA和MeJA胁迫下表达量受到抑制, 推测甘蔗ScNAC2基因积极响应生物和非生物胁迫。最后, 亚细胞定位结果显示该蛋白定位于细胞核上, 酵母自激活活性试验表明, ScNAC2基因编码的蛋白无转录自激活活性, 可通过酵母双杂交试验, 筛选互作蛋白。本文为深入鉴定甘蔗NAC-ATAF亚家族成员在响应生物和非生物胁迫应答中的生物学功能奠定了基础, 并为甘蔗抗性分子育种提供了潜在的基因资源。
[1] Duval M, Hsieh T F, Kim S Y, Thomas T L. Molecular characterization of AtNAM: a member of theNAC domain superfamily.,2002, 50: 237–248.
[2] Ooka H, Satoh K, Doi K, Nagata T, Otomo Y, Murakami K, Matsubara K, Osato N, Kawai J, Carninci P, Hayashizaki Y, Suzuki K, Kojima K, Takahara Y, Yamamoto K, Kikuchi S. Comprehensive analysis of NAC family genes inand.,2003, 10: 239–247.
[3] Souer E, van Houwelingen A, Kloos D, Mol J, Koes R. The no apical meristem gene of petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries.,1996, 85: 159–170.
[4] Takada S, Hibara K, Ishida T, Tasaka M. Thegene ofregulates shoot apical meristem formation., 2001, 128: 1127–1135.
[5] Aida M, Ishida T, Tasaka M. Shoot apical meristem and cotyledon formation duringembryogenesis: interaction among theandgenes., 1999, 126: 1563–1570.
[6] Christianson J A, Dennis E S, Llewellyn D J, Wilson I W. ATAF NAC transcription factors: regulators of plant stress signaling.,2010, 5: 428–432.
[7] Delessert C, Kazan K, Wilson I W, Van Der Straeten D, Manners J, Dennis E S, Dolferus R. The transcription factorrepresses the expression of pathogenesis-related genes in., 2005, 43: 745–757.
[8] Jensen M K, Rung J H, Gregersen P L, Gjetting T, Fuglsang A T, Hansen M, Joehnk N, Lyngkjaer M F, Collinge D B. Thetranscription factor: a positive regulator of penetration resistance in barley and Arabidopsis., 2007, 65: 137–150.
[9] 马雪祺, 阴艳红, 冯婧娴, 陈万生, 孙连娜, 肖莹. 植物NAC转录因子研究进展. 植物生理学报,2021, 57: 2225–2234.
Ma X Q, Yin Y H, Feng J X, Chen W S, Sun L N, Xiao Y. Research progress of NAC transcription factors in plant., 2021, 57: 2225–2234 (in Chinese with English abstract).
[10] Olsen A N, Ernst H A, Leggio L L, Skriver K. DNA-binding specificity and molecular functions of NAC transcription factors.,2005, 169: 785–797.
[11] 李桂玲, 李思云, 刘卫群. 转录因子NAC及其在植物生长发育中的作用. 分子植物育种2019, 17: 811-826.
Li G L, Li S Y, Liu W Q. Transcription factor NAC and its role in plant growth and development., 2019, 17: 811–826 (in Chinese with English abstract).
[12] Nakashima K, Tran L S, Van Nguyen D, Fujita M, Maruyama K, Todaka D, Ito Y, Hayashi N, Shinozaki K, Yamaguchi-Shinozaki K. Functional analysis of a NAC-type transcription factorinvolved in abiotic and biotic stress-responsive gene expression in rice.,2007, 51: 617–630.
[13] Jin H, Huang F, Cheng H, Song H, Yu D. Overexpression of thegene, an NAC transcription factor, reduces abiotic stress tolerance in tobacco.,2013, 31: 435–442.
[14] Wang X, Basnayake B M, Zhang H, Li G, Li W, Virk N, Mengiste T, Song F. The, a NAC transcription factor, is a negative regulator of defense responses against necrotrophic fungal and bacterial pathogens.,2009, 22: 1227–1238.
[15] Wu Y, Deng Z, Lai J, Zhang Y, Yang C, Yin B, Zhao Q, Zhang L, Li Y, Yang C, Xie Q. Dual function ofATAF1 in abiotic and biotic stress responses.,2009, 19: 1279–1290.
[16] Lu P L, Chen N Z, An R, Su Z, Qi B S, Ren F, Chen J, Wang X C. A novel drought-inducible gene, ATAF1, encodes a NAC family protein that negatively regulates the expression of stress-responsive genes in.,2007, 63: 289–305.
[17] Jensen M K, Hagedorn P H, De Torres-Zabala M, Grant M R, Rung J H, Collinge D B, Lyngkjaer M F. Transcriptional regulation by an NAC (NAM-ATAF1, 2-CUC2) transcription factor attenuates ABA signalling for efficient basal defence towardsf. sp.in.,2008, 56: 867–880.
[18] Nogueira F T S, Schlögl P S, Camargo S R, Fernandez J H, De Rosa V E, Pompermayer P, Arruda P. SsNAC23, a member of the NAC domain protein family, is associated with cold, herbivory and water stress in sugarcane., 2005, 169: 93–106.
[19] Carrillo-Bermejo E A, Gamboa-Tuz S D, Pereira-Santana A, Keb-Llanes M A, Castaño E, Figueroa-Yañez L J, Rodriguez-Zapata L C. Thegene from sugarcane () encodes a senescence-associated NAC transcription factor involved in response to osmotic and salt stress., 2020, 133: 897–909.
[20] Peng X, Zhao Y, Li X, Wu M, Chai W, Sheng L, Wang Y, Dong Q, Jiang H, Cheng B. Genomewide identification, classification and analysis of NAC type gene family in maize.,2015, 94: 377–390.
[21] Kadier Y, Zu Y Y, Dai Q M, Song G, Lin S W, Sun Q P, Pan J B, Lu M. Genome-wide identification, classification and expression analysis of NAC family of genes in sorghum [(L.) Moench].,2017, 83: 301–312.
[22] Chen C, Chen H, Zhang Y, Thomas H R, Frank M H, He Y, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020, 13: 1194–1202.
[23] Feng M, Yu Q, Chen Y, Fu Z, Xu L, Guo J., a metallothionein-like gene from sugarcane, enhances freezing tolerance intransgenic plants.,2022, 194: 104750.
[24] Liu F, Huang N, Wang L, Ling H, Sun T, Ahmad W, Muhammad K, Guo J, Xu L, Gao S, Que Y, Su Y. A novel l-ascorbate peroxidase 6 gene,, plays an important role in the regulation of response to biotic and abiotic stresses in sugarcane.,2018, 8: 2262–2262.
[25] 苏亚春, 李聪娜, 苏炜华, 尤垂淮, 岑光莉, 张畅, 任永娟, 阙友雄. 甘蔗割手密种类甜蛋白家族鉴定及栽培种同源基因功能分析. 作物学报, 2021, 47: 1275–1296.
Su Y C, Li C N, Su W H, You C H, Cen G L, Zhang C, Ren Y J, Que Y X. Identification of thaumatin-like protein family inand functional analysis of its homologous gene in sugarcane cultivar., 2021, 47: 1275–1296 (in Chinese with English abstract).
[26] Xue B, Guo J, Que Y, Fu Z, Wu L, Xu L. Selection of suitable endogenous reference genes for relative copy number detection in sugarcane.,2014, 15: 8846–8862.
[27] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(–Delta Delta C(T)) Method.,2001, 25: 402–408.
[28] 黄宁, 惠乾龙, 方振名, 李姗姗, 凌辉, 阙友雄, 袁照年. 甘蔗β-胡萝卜素异构酶基因家族的鉴定、定位和表达分析. 作物学报, 2021, 47: 882–893.
Huang N, Hui Q , Fang Z M, Li S S, Ling H, Que Y X, Yuan Z N. Identification, localization and expression analysis of beta-carotene isomerase gene family in sugarcane., 2021, 47: 882–893 (in Chinese with English abstract).
[29] Yang X, Wang X, Ji L, Yi Z, Fu C, Ran J, Hu R, Zhou G. Overexpression of aNAC geneconfers enhanced drought and cold tolerance in.,2015, 34: 943–958.
[30] Lu M, Ying S, Zhang D F, Shi Y S, Song Y C, Wang T Y, Li Y. A maize stress-responsive NAC transcription factor,, confers enhanced tolerance to dehydration in transgenic.,2012, 31: 1701–1711.
[31] Ohnishi T, Sugahara S, Yamada T, Kikuchi K, Yoshiba Y, Hirano H Y, Tsutsumi N., a member of the NAC gene family, is induced by various stresses in rice.,2005, 80: 135–139.
[32] Jannoo N, Grivet L, Chantret N, Garsmeur O, Glaszmann J C, Arruda P, D’Hont A. Orthologous comparison in a gene-rich region among grasses reveals stability in the sugarcane polyploid genome.,2007, 50: 574–585.
[33] Hufford M B, Seetharam A S, Woodhouse M R, Chougule K M, Dawe R K.assembly, annotation, and comparative analysis of 26 diverse maize genomes.,2021, 373: 655–662.
[34] Zhang J, Zhang X, Tang H, Zhang Q, Hua X, Ma X, Zhu F, Jones T, Zhu X, Bowers J, Wai C M, Zheng C, Shi Y, Chen S, Xu X, Yue J, Nelson D R, Huang L, Li Z, Xu H, Zhou D, Wang Y, Hu W, Lin J, Deng Y, Pandey N, Mancini M, Zerpa D, Nguyen J K, Wang L, Yu L, Xin Y, Ge L, Arro J, Han J O, Chakrabarty S, Pushko M, Zhang W, Ma Y, Ma P, Lv M, Chen F, Zheng G, Xu J, Yang Z, Deng F, Chen X, Liao Z, Zhang X, Lin Z, Lin H, Yan H, Kuang Z, Zhong W, Liang P, Wang G, Yuan Y, Shi J, Hou J, Lin J, Jin J, Cao P, Shen Q, Jiang Q, Zhou P, Ma Y, Zhang X, Xu R, Liu J, Zhou Y, Jia H, Ma Q, Qi R, Zhang Z, Fang J, Fang H, Song J, Wang M, Dong G, Wang G, Chen Z, Ma T, Liu H, Dhungana S R, Huss S E, Yang X, Sharma A, Trujillo J H, Martinez M C, Hudson M, Riascos J J, Schuler M, Chen L-Q, Braun D M, Li L, Yu Q, Wang J, Wang K, Schatz M C, Heckerman D, Van Sluys M-A, Souza G M, Moore P H, Sankoff D, VanBuren R, Paterson A H, Nagai C, Ming R. Allele-defined genome of the autopolyploid sugarcaneL., 2018, 50: 1565–1573.
[35] Gong L, Zhang H, Liu X, Gan X, Nie F, Yang W, Zhang L, Chen Y, Song Y, Zhang H. Ectopic expression of, an ATAF transcription factor from, improves growth and drought tolerance in transgenic.,2020, 151: 535–544.
[36] D'Hont A, Grivet L, Feldmann P, Glaszmann J C, Rao S, Berding N. Characterisation of the double genome structure of modern sugarcane cultivars (spp.) by molecular cytogenetics.,1996, 250: 405–413.
[37] 张欢, 杨乃科, 商丽丽, 高晓茹, 刘庆昌, 翟红, 高少培, 何绍贞. 甘薯抗旱相关基因的克隆与功能分析. 作物学报, 2020, 46: 1649–1658.
Zhang H, Yagn N K, Shang L L, Gao X R, Liu Q C, Zhai H, Gao S P, He S Z. Cloning and functional analysis of a drought tolerance-related genein sweet potato., 2020, 46: 1649–1658 (in Chinese with English abstract).
[38] Hong Y, Zhang H, Huang L, Li D, Song F. Overexpression of a stress-responsive NAC transcription factor gene ONAC022 improves drought and salt tolerance in rice., 2016, 7: 4.
[39] Liu W, Zhao B G, Chao Q, Wang B, Zhang Q, Zhang C, Li S, Jin F, Yang D, Li X. Function analysis of ZmNAC33, a positive regulator in drought stress response in., 2019, 145: 174–183.
[40] Vlot A C, Dempsey D M A, Klessig D F. Salicylic Acid, a multifaceted hormone to combat disease.,2009, 47: 177–206.
[41] 龙亚芹, 王万东, 王美存, 陈于福, 解德宏, 陈华蕊, 俞艳春, 尼章光. 水杨酸(SA)诱导植物对病虫害产生抗性及作用机制研究. 热带农业科学,2009, 29(12): 46–50.
Long Y Q, Wang W D, Wang M C, Chen Y F, Xie D H, Chen H R, Yu Y C, Ni Z G. Salicylic acid induced resistance of plants against insects and diseases and its interaction mechanism., 2009, 29(12): 46–50 (in Chinese with English abstract).
[42] 蒋旭, 崔会婷, 王珍, 张铁军, 龙瑞才, 杨青川, 康俊梅. 紫花苜蓿的克隆及对木质素与纤维素合成的功能分析. 中国农业科学,2020, 53: 3818–3832.
Jiang X, Cui H T, Wang Z, Zhang T J, Long R C, Yang Q C, Kang J M. Cloning and function analysis ofin lignin and cellulose biosynthesis pathway from alfalfa.,2020, 53: 3818–3832 (in Chinese with English abstract).
[43] Takasaki H, Maruyama K, Kidokoro S, Ito Y, Fujita Y, Shinozaki K, Yamaguchi-Shinozaki K, Nakashima K. The abiotic stress-responsive NAC-type transcription factorregulates stress-inducible genes and stress tolerance in rice.,2010, 284: 173–183.
[44] Bang S W, Choi S, Jin X, Jung S E, Choi J W, Seo J S. Transcriptional activation of rice CINNAMOYL-CoA REDUCTASE 10 by OsNAC5, contributes to drought tolerance by modulating lignin accumulation in roots., 2022, 20: 736–747
[45] Jensen M K, Lindemose S, Masi F D, Reimer J J, Nielsen M, Perera V, Workman C T, Turck F, Grant M R, Mundy J, Petersen M, Skriver K.ATAF1 transcription factor directly regulates abscisic acid biosynthetic genein., 2013, 3: 321–327.
[46] 张艳馥, 沙伟. 转录因子概述. 生物学教学, 2009, 34(10): 7–8.
Zhang F X, Sha W. Overview of transcription factors.,2009, 34(10): 7–8 (in Chinese with English abstract).
[47] Zhong R, Richardson E A, Ye Z H. The MYB46 transcription factor is a direct target of SND1 and regulates secondary wall biosynthesis in.,2007, 19: 2776–2792.
[48] Yamaguchi M, Ohtani M, Mitsuda N, Kubo M, Ohme-Takagi M, Fukuda H, Demura T., a NAC domain transcription factor, negatively regulates xylem vessel formation in., 2010, 22: 1249–1263.
Genome-wide identification of NAC transcription factors ATAF subfamily inand functional analysis of its homologous genein sugarcane cultivar
WANG Heng-Bo**, ZHANG Chang**, WU Ming-Xing, LI Xiang, JIANG Zhong-Li, LIN Rong-Xiao, GUO Jin-Long, and QUE You-Xiong*
Key Laboratory of Sugarcane Biology and Genetic Breeding (Fujian), Ministry of Agriculture and Rural Affairs / Fujian Agriculture and Forestry University / National sugarcane Engineering Technology Research, Fuzhou 350002, Fujian, China
NAC (NAM, ATAF, and CUC) is a family of transcription factors unique to terrestrial plants, including 18 subfamilies, of which ATAF subfamily members are mainly involved in the response processes of biotic and abiotic stresses, such as salicylic acid (SA), methyl jasmonate acid (MeJA), abscisic acid (ABA), pathogenic bacteria, mechanical damage, low temperature, and sodium chloride (NaCl). The data were from the genomic database ofand the cDNA library of a sugarcane cultivar ROC22. Firstly, the ATAF subfamily members inwere identified and analyzed for their protein multiple sequence alignment, phylogenetic tree construction, and promoter region cis-acting element prediction using comparative genomics methods and various bioinformatics methods. Secondly, one homologous gene of the ATAF subfamily SsNAC2,, was cloned from a prevalent sugarcane cultivar ROC22 in China. The qRT-PCR was used to detect the tissue-specific expression pattern and the relative expression levels ofgene under different exogenous stresses. Finally, the subcellular localization and the transactivation analysis of ScNAC2 protein were performed. The results showed that six members of the ATAF subfamily were identified with the open read reading frames between 889 bp and 1017 bp, relative molecular weights between 32.067 and 35.819 kD, the theoretical isoelectric points from 5.09 to 8.92, and the proteins of all members were predicted to localize on the nucleus. In addition, the Ka/Ks ratios of six gene pairs were all less than 1, indicating that purification selection played an important role during evolution. The amino acid sequence alignment indicated that all members of the ATAF subfamily contained the NAM conserved domains, consisting of I, II, III, IV, and V subdomains. Phylogenetic analysis revealed that the members from sugarcane, sorghum, maize, and rice, that belonged towere clustered together, indicating that they had a close evolutionary relationship. Forty members of the ATAF subfamily from, rice, maize, and sorghum were divided into two groups (Group A and Group B), in which the subfamily members of maize had obvious gene expansion. Furthermore, the promoter regions of ATAF subfamily members all contained-acting elements that responded to stresses such as low temperature, drought, and hormones, and we thus speculated that they were involved in the response processes of a variety of biotic and abiotic stresses. Furthermore, the full-length cDNA sequence of thegene (GenBank accession number: OL982539) was cloned from the sugarcane cultivar ROC22, with an open reading frame of 891 bp and encoding 296 amino acid residues. The similarity of amino acid sequence between ScNAC2 and SsNAC2 proteins both from ATAF subfamily Group B was 97.99%. The qRT-PCR showed that thegene was constitutively expressed in different tissues of sugarcane, and its expression level in sugarcane leaves and stem epidermis was higher than that in stem piths, buds, and roots. Besides, the relative expression level ofgene was significantly down-regulated under SA and MeJA stresses, however, it showed an expression pattern from low to high and varied to significant levels under the stress of ABA, 4℃, and NaCl. Subcellular localization revealed that the ScNAC2-GFP fusion protein was localized in the cell nucleus ofleaves. Furthermore, the transactivation experiment showed that ScNAC2 protein did not have the transcriptional self-activation activity. The above results established the foundation for identifying the biological functions of sugarcane NAC-ATAF subfamily members in response to biotic and abiotic stresses and provided potential genetic resources for sugarcane resistance molecular breeding.
sugarcane; transcription factor; NAC gene family; biotic and abiotic stress; the relative expression pattern
10.3724/SP.J.1006.2023.24005
本研究由国家重点研发计划项目(2018YFD1000503), 福建省自然科学基金项目(2022J01160)和财政部和农业农村部国家现代农业产业技术体系建设专项(糖料,CARS-17)资助。
This study was supported by the National Key Research and Development Program of China (2018YFD1000503), the Natural Science Foundation of Fujian Province, China, and the China Agriculture Research System of MOF and MARA (Sugar Crop, CARS-17).
通信作者(Corresponding author):阙友雄, E-mail: queyouxiong@126.com
**同等贡献(Contributed equally to this work)
王恒波, E-mail: wanghengbo_0354@126.com; 张畅, E-mail: 1223134902@qq.com
2022-01-04;
2022-03-25;
2022-04-20.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220418.1843.040.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
