大豆GmPIN2家族基因调控根系发育功能初探
2023-01-12柯美玉陈志威
梁 政 柯美玉 陈志威 陈 栩 高 震
大豆GmPIN2家族基因调控根系发育功能初探
梁 政1,3柯美玉1,3陈志威2陈 栩3,*高 震3,*
1福建农林大学生命科学学院, 福建福州 350002;2福建农林大学资源与环境学院, 福建福州 350002;3福建农林大学海峡联合研究院园艺植物生物学及代谢组学研究中心, 福建福州 350002
生长素作为重要的植物激素之一。生长素的转运参与了植物各组织器官发育调控。在拟南芥中, 生长素转运调控主要由PIN家族蛋白所介导, 其中拟南芥AtPIN2主要通过介导生长素向基运输调控根的向重力性。大豆中PIN2家族蛋白及其功能研究尚未有报道。本研究通过构建系统进化树和蛋白质结构域分析发现, 大豆和为的同源基因。组织表达分析研究发现,和在根、根瘤原基和根瘤等组织中高表达。和在根部主要表达在根尖表皮和外部皮层细胞, 在根瘤中、均定位于根瘤基部维管束区, 此外,定位在根瘤顶部表皮及外皮层。利用基因编辑技术CRISPR/Cas9 (Clustered Regularly Interspersed Short Palindromic Repeats/CRISPR associated 9)同时敲除和后发现,突变体根具有明显的重力缺失表型。与野生型相比,和的根面积和侧根长度显著降低,侧根夹角显著上升, 而侧根夹角不变。综上所述,和通过介导生长素向基运输对大豆根型调控具有重要作用。本研究为深入探究大豆PIN蛋白介导生长素极性运输在根形态建成的作用机制奠定了一定研究基础。
大豆; 生长素; PIN蛋白; 根
生长素(主要形式为吲哚-3-乙酸)是最早发现的植物激素, 通过合成、信号转导、运输和代谢等途径共同影响植物对生长素的响应, 从而影响植物发育的多个方面, 如: 细胞分裂、伸长和分化、胚胎发育、根向性、顶端优势和开花等[1-2]。生长素主要通过调控植物组织中生长素浓度梯度调控植物生长发育, 而这种梯度主要是由生长素合成和极性运输(polar auxin transport, PAT)进行维持[3]。
生长素极性运输主要由生长素输入载体和输出载体调控, 前者主要是AUX1/LAX蛋白家族(AUXIN1/ LIKE-AUX1), 后者主要是PIN蛋白家族(PIN- FORMED)和ABCB/PGP蛋白家族(ATP-BINDING CASSETTE B/P-GLYCOPROTEIN)[4]。其中, PIN蛋白可以定位在质膜介导生长素流向, 使生长素不对称分布, 从而调节植物器官建成, 此外PIN蛋白也可以定位在内质网膜上进行细胞内调节生长素稳态[4]。目前, 已经鉴定多种植物具有不同数目的PIN蛋白, 例如: 拟南芥、玉米、水稻、高粱分别有8、12、12、11个PIN蛋白[5]。在器官形成过程中, PIN家族蛋白通过介导生长素流动方向和生长素的局部浓度梯度参与调控植物器官发育过程[6]。
根系作为植物获取养分的重要器官, 其形态及发育过程受到了生长素严格的调控[7-9]。在拟南芥中, PIN蛋白家族极性定位调节生长素流向及浓度梯度进而调控根形态的各个方面, 如根长、侧根发育、根毛发育和向重力性等[5]。在拟南芥中,主要介导生长素向基运输调控根的向重力性。和野生型相比,和单突变体的根长和根分生组织大小减少, 而、和单突变体在静止中心和根冠出现分裂缺陷[10]。在拟南芥侧根原基发育过程中,、、、、和以特定的部分重叠模式表达[11]。和协同调节生长素在根两侧的极性运输和分布, 参与根系重力形态建成[12]。在经济作物大豆中, 与拟南芥AtPIN1蛋白同源的GmPIN1蛋白家族特异在根瘤原基发生区域表达,三突变体根瘤原基细胞分裂异常,过表达株系根瘤原基发育受到抑制, 揭示GmPIN1蛋白家族介导生长素向顶运输调控根瘤原基的形成, 此外, GmPIN9d蛋白在根和根瘤之间的维管束积累, 并与GmPIN1协同作用在维管束内向顶运输生长素, 调控生长素极性运输以促进根瘤增大[13]。在蒺藜苜蓿中,是唯一一个在根瘤中表达水平高于根尖的基因。这一结果表明, MtPIN9转运蛋白可能参与蒺藜苜蓿根瘤的形成。此外, 拟南芥同一分支的同源基因在根瘤中表达量较高。该分支的MtPIN蛋白定位于内质网, 这可能表明蒺藜苜蓿根瘤的发育和分生组织活性的维持与生长素内稳态有关[14]。与拟南芥同源的可以在数小时内被结瘤因子诱导表达, 但在成熟根瘤中低水平表达, 用RNAi (RNA interference)技术降低表达量, 发现与对照组相比,的根瘤数目显著减少[15-17]。百脉根LjPIN蛋白家族介导的生长素极性运输对于维持根瘤和根之间的维管束连接的功能是必不可少的[18]。在一些禾本科作物中,介导的生长素极性运输调节根的生长发育能够影响粮食产量等。降低水稻的表达能显著抑制不定根的形成, 而双突变体表型表现为短根, 根毛变长以及向重力性缺失[19-20]。在玉米中, 与拟南芥同源的家族共有4个成员, 其中过表达株系地下部分根系发达, 具有较长的胚根, 短而密集的侧根[21]。
由此可见, PIN蛋白家族在根和根瘤发育过程中起到重要调控作用。而大豆中GmPIN2家族蛋白及其功能研究尚未有报道。本研究旨在探究大豆GmPIN2蛋白家族介导生长素向基运输对大豆根部形态建成的影响。
1 材料与方法
1.1 试验材料
大豆品种为Williams 82 (Wm82), 根瘤菌菌株为BXYD3。大豆种子在蛭石中萌发, 生长在光照14 h/黑暗10 h, 25℃恒温的温室中, 瞬时转化后的大豆苗在低氮营养液中培养。低氮营养液包括: KNO3、NH4NO3、Ca(NO3)2·4H2O、(NH4)2SO4(15∶4∶12∶3)、1.2 mmol L–1CaCl2、1.05 mmol L–1K2SO4、0.5 mmol L–1MgSO4·7H2O、25 mmol L–1MgCl2、2.5 mmol L–1NaB4O7·10H2O、0.5 mmol L–1MnSO4·H2O、1.5 mmol L–1ZnSO4·7H2O、0.5 mmol L–1CuSO4·5H2O、0.15 mmol L–1(NH4)6Mo7O24·4H2O、40 mmol L–1Fe-Na-EDTA、250 mmol L–1KH2PO4。
根瘤菌培养基是酵母甘露醇琼脂(YMA)培养基, 培养基有10 g L–1甘露醇、0.2 g L–1MgSO4·7 H2O、0.1 g L–1NaCl、3 g L–1酵母提取物、0.25 g L–1K2HPO4和0.25 g L–1KH2PO4组成。
1.2 试验方法
1.2.1 生物信息学分析 在植物基因组网站Phytozome (http://www.phytozome.org/index.php)数据库中获得拟南芥和大豆的PIN蛋白家族的氨基酸序列, 根据软件MEGA5.1采用邻近算法(Neighbor- Joining, Bootstrap检验1000次)生成拟南芥和大豆PIN蛋白的系统进化树。根据在植物基因组网站Phytozome数据库中获得拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2的氨基酸序列, 根据motif分析网站(https://meme-suite.org/meme/index.html)生成拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白序列比对结果, 并使用BioEdit软件对拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白进行同源性比对。根据拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米的基因家族的基因组DNA和CDS序列, 利用基因结构显示系统Gene Structure Display Server (GSDS)[22](http://gsds.cbi. pku.edu.cn/)绘制基因结构图, 通过网站PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/ html/)和TBtools软件[23]分析基因家族上游2000 bp序列。
1.2.2 植物RNA的提取及反转录 取大豆根、子叶、下胚轴、叶片、茎、花、种子和根瘤各约5 g, 放入液氮预冷的研钵中磨碎至粉末状, 移到RNA-free 2.0 mL离心管中, 根据艾科瑞生物RNA提取试剂盒说明书, 加入1.0 mL RNAex并混合均匀。室温静置5 min, 12,000´, 4℃离心5 min, 小心吸取上清液, 加入新的RNA-Free 2.0 mL离心管中, 再加入200 μL氯仿, 充分混匀, 室温静置5 min。12,000´, 4℃离心15 min, 小心吸取上清液转移至另一只新的RNA-Free 1.5 mL离心管中, 加入500 μL异丙醇, 充分混匀, 室温静置10 min。12,000´, 4℃离心10 min, 离心完成后弃上清, 加入80% (-20℃)预冷的乙醇, 清洗RNA和离心管壁, 7500´, 4℃离心5 min, 小心弃上清, 打开离心管盖, 放入超净台将管内残留的乙醇吹干。吹干后加入RNA-Free灭菌水溶解RNA。用分光光度计(IMPLEN, 美国)测定RNA浓度, 然后参照上海近岸蛋白质有限责任公司反转录试剂盒说明书, 合成cDNA第1链, 反应结束后, 放入-20℃冰箱保存备用。
1.2.3 实时荧光定量PCR 用NCBI Primer Blast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计和的定量PCR引物(表1); 大豆管家基因作为内参(表1)。将所得第1链稀释5倍做为定量PCR反应模板; 定量PCR反应体系: 7.5 μL 2× Novo Start SYBR qPCR SuperMix Plus、正向引物和反向引物各0.3 μL、0.7 μL模板、6.2 μL RNA-Free H2O。反应程序: 94℃预变性3 min; 94℃ 5 s, 60℃ 30 s, 79℃ 5 s, 40个循环。反应结束后用实时荧光定量PCR仪(CFX96 Touch实时定量PCR系统, Bio-Rad, 美国)携带的计算每个样品的表达量。相对表达量为目的基因的表达量与管家基因表达量的比值。

表1 引物序列
1.2.4 大豆和基因启动子融合报告基因GUS载体的构建 从Phytozome数据库中获得大豆(Glyma.13G101900)和(Glyma.17G057300)的基因组序列, 根据基因组序列设计和启动子的引物(表1)。以大豆Williams 82基因组DNA为模板, 使用高保真聚合酶GXL分别扩增和基因的启动子2000 bp序列, 采用Gateway BP克隆酶II混合物将启动子序列克隆到入门载体pDONR221上。经过测序验证正确后, 使用Gateway LR克隆酶II酶混合物将启动子片段克隆到目的载体pGWB633中, 构建获得pGmPIN2a/b::GUS最终载体。
1.2.5双突变体的构建 通过NCBI网站下载GmPIN2a和GmPIN2b的蛋白质编码序列(coding sequence, CDS), 利用华南农业大学刘耀光课题组开发的基因组编辑工具软件包CRISPR-GE (http://skl. scau.edu.cn/home/)预测sgRNA[24], 根据上述和的CDS序列, 以大豆栽培品种Williams 82基因组序列(Wm82.a2.v1)为参考基因组, 设计1条sgRNA序列: 5′-TGG-3′ (下画线部分为sgRNA靶点序列), 根据Bai等报道的CRISPR/Cas9载体构建方法进行sgRNA克隆[25]。设计1对引物, 分别为pCas9-F和pCas9-R (表1), 用以验证基因编辑载体是否转入植株中。
1.2.6和的组织定位分析
本研究主要分析和在大豆根部, 根瘤原基和根瘤中的分布。取根瘤菌侵染前的根部, 根瘤菌侵染后第6天的根瘤原基, 根瘤菌侵染后第14天的成熟根瘤, 使用7%的琼脂糖凝胶(7 g琼脂糖凝胶, 100 mL超纯水)对样品进行包埋, 使用振荡切片机对包埋的样品进行切片, 厚度为80 μm。切好的样品浸泡在配置好的GUS染色液(50 mmol L–1磷酸钠缓冲液液(pH 7.0), 0.1% (v/v) Triton X-100, 0.1 mmol L–1K3Fe(CN)6, 0.1 mmol L–1K4[Fe(CN)6]·3H2O, 1 mg mL–1X-Gluc (5-溴-4-氯-3-吲哚-β-D-葡萄糖醛酸环己胺盐), 1% (v/v)二甲基甲酰胺)中, 37℃条件下, 染色1~4 h, 用体积分数为75%的乙醇进行脱色, 透明液(80 g三氯乙醛水合物, 21.3 mL水, 8.7 mL 100%丙三醇)制片, NI-U研究级正置显微镜(DIC)进行拍摄和收集图片。
1.2.7 毛状根转化和土培结瘤 采用瞬时转化体系将载体pGmPIN2a::GUS和pGmPIN2b:: GUS转入大豆植株中。选择萌发5 d的大豆幼苗, 将携带载体的农杆菌菌株K599注入到大豆幼苗的子叶近端下胚轴。然后将大豆苗放置到低氮营养液中进行培养。水培2周后, 毛状根根长10 cm左右时, 剪去主根并筛选阳性根, 实验使用的最终载体pGWB633带有GUS (β-glucuronidase, β-葡萄糖苷酸酶)报告基因工作元件, 筛选阳性根时, 剪毛根根尖约1 cm, 放入装有GUS染液的离心管中, 做好标记, 用锡箔纸包裹。然后放入37℃恒温培养箱1~2 h, 取出离心管, 观察毛根是否变蓝, 如果变蓝, 那么该毛根为阳性根。阳性根筛选完成后将植株转移至蛭石中土培进行根瘤菌接种。
1.2.8 野生型、和双突变体表型分析 将野生型、和双突变体株系的种子在蛭石中萌发, 生长7 d, 待双突变体根部出现向重力性缺失, 用相机进行照相。将野生型、和双突变体株系的种子在装有蛭石的夹板中萌发, 生长7 d, 取出野生型、和双突变体, 用扫描仪扫描植株根系, 并用ImageJ软件统计各植株根系的根长、侧根长、侧根夹角、根面积、侧根数目。每次统计重复7株植株。
2 结果与分析
2.1 大豆PIN的系统发育分析
由于大豆为古四倍体, 而大豆基因组中具有23个GmPIN蛋白, 为确定大豆GmPIN2蛋白家族成员, 本研究构建了大豆和拟南芥的PIN蛋白家族进行系统进化树(图1)。进化树结果显示, 拟南芥和大豆PIN蛋白家族在进化树上可以分为7个分支。拟南芥AtPIN2蛋白与大豆GmPIN蛋白家族2个成员聚在一个分支上。因此, 本研究将其分别命名为 GmPIN2a和GmPIN2b。
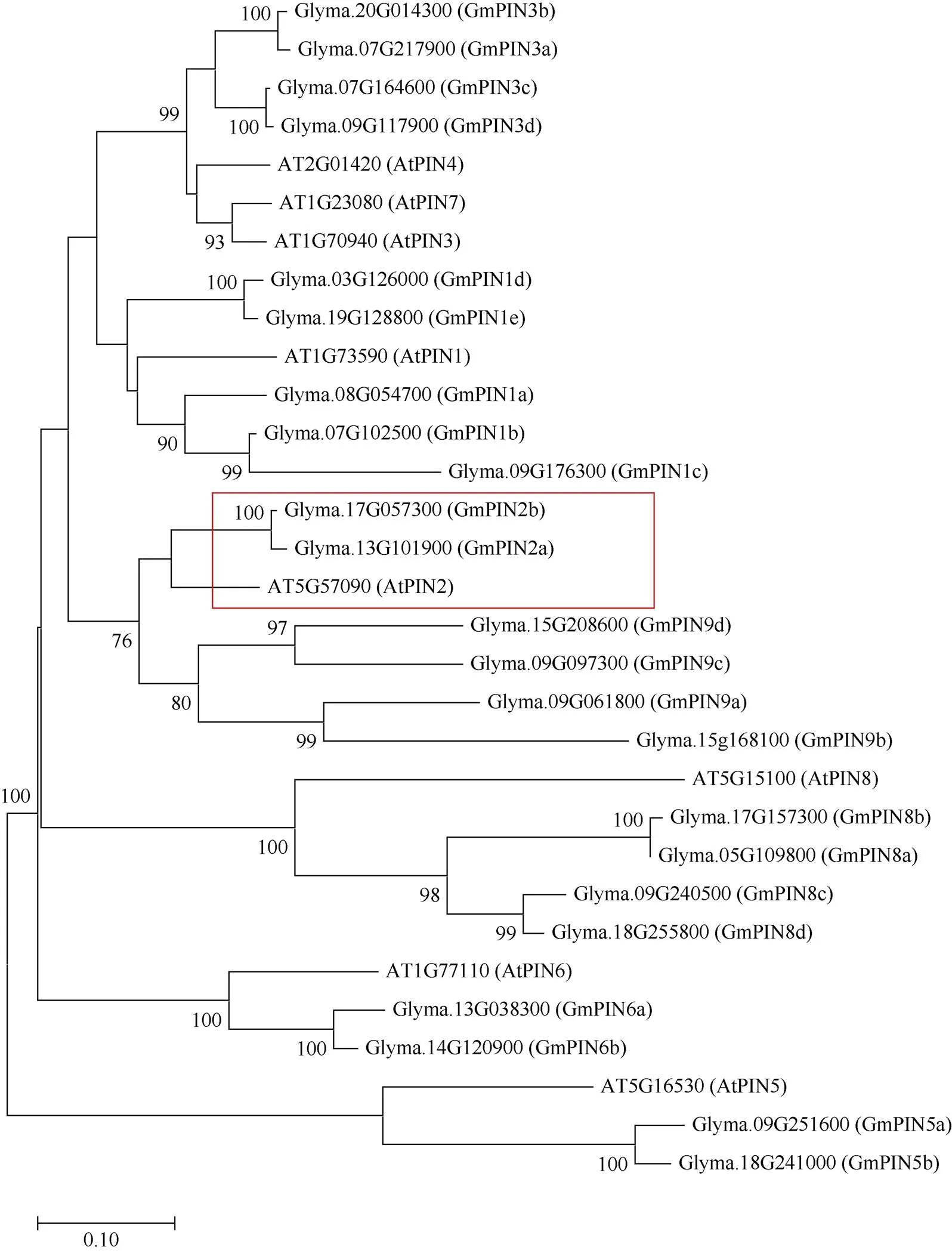
图1 大豆GmPIN和拟南芥AtPIN蛋白家族系统进化树
红色框表示大豆GmPIN2和拟南芥AtPIN2。
The red box represents soybean GmPIN2 andAtPIN2.
2.2 拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白的生物信息学分析
为探索不同植物PIN2蛋白之间的核酸及蛋白序列保守性。本研究选取了双子叶植物(拟南芥、大豆、蒺藜苜蓿、百脉根)和单子叶植物(水稻、玉米) 6种植物共8个PIN2核酸及蛋白序列进行分析。基因结构显示: 拟南芥具有8个内含子, 大豆和均具有5个内含子, 蒺藜苜蓿和分别含有5个和4个内含子, 百脉根具有5个内含子, 而水稻和玉米均具有6个内含子(图2-A)。
本研究对拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米基因上游2000 bp序列进行分析(图2-B)发现, 大部分基因上游启动子区域除了包含大量的顺式作用元件CAAT-box和TATA-box, 还存在以下几种元件: (1) 与激素调控相关的顺式作用元件, 如: 生长素应答元件TGA-element, 茉莉酸甲酯(MeJA)反应元件TGACG-box, 水杨酸反应元件TCA-element, 脱落酸反应元件ABRE, 赤霉素反应元件P-box; (2) 与光响应元件G-box; (3) 与生长发育相关元件: 分生组织调控元件CAT-box, 与胚乳发育相关元件CGN4-motif。对拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白的氨基酸序列进行同源比对(表2)发现, GmPIN2a和GmPIN2b氨基酸序列相似性为96.8%, GmPIN2a和AtPIN2、MtPIN2a、MtPIN2b、LjPIN2、OsPIN2、ZmPIN2氨基酸序列相似性分别为76.5%、85.1%、68.3%、90.7%、63.9%、61.9%, GmPIN2b和AtPIN2、MtPIN2a、MtPIN2b、LjPIN2、OsPIN2、ZmPIN2氨基酸序列相似性分别为77.2%、85.5%、68.8%、90.8%、63.5%、61.9%。对大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白氨基酸序列进行多序列比对(图2-C)发现, 单子叶植物PIN2蛋白相较于双子叶植物PIN2蛋白缺失部分基序, 表明PIN2蛋白在双子叶植物和单子叶植物进化过程中在蛋白序列产生部分分化。

表2 拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白家族同源比对

(图2)
A: 拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白家族基因结构。B: 拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白家族基因启动子区域分析。C: 拟南芥、大豆、蒺藜苜蓿、百脉根、水稻和玉米PIN2蛋白家族蛋白序列比对。AT5G57090: AtPIN2; Glyma.13G101900: GmPIN2a; Glyma.17G057300: GmPIN2b; LOC_Os06g44970: OsPIN2; Medtr4g127100: MtPIN2a; Medtr4g127090: MtPIN2b; Lj4g3v2139970: LjPIN2; Zm00001d046893_P001: ZmPIN2。
A: gene structure of the PIN2 protein family in,,,,,and. B: analysis of gene promoter regions of the PIN2 protein family in,,,,,and. C: protein sequence alignment of the PIN2 protein family in,,,,,and. AT5G57090: AtPIN2; Glyma.13G101900: GmPIN2a; Glyma.17G057300: GmPIN2b; LOC_Os06g44970: OsPIN2; Medtr4g127100: MtPIN2a; Medtr4g127090: MtPIN2b; Lj4g3v2139970: LjPIN2; Zm00001d046893_ P001: ZmPIN2.
2.3 大豆GmPIN2基因组织表达模式分析
为研究大豆基因在大豆各组织中表达模式, 对大豆和基因在根、子叶、下胚轴、叶片、茎、花、种子和根瘤8个组织中的表达量进行检测(图3-A)。结果显示,和具有明显的组织特异性。相对于其他组织,和在大豆根部高表达, 且根部的表达量高于。在子叶、下胚轴、叶片、茎、花、种子和根瘤中,表达量较低但均高于的表达量。
为进一步确认和在地下部分的组织定位。分别构建和编码区域上游约2000 bp启动子序列融合报告基因GUS载体, 并通过瞬时毛根转化将其导入大豆幼苗中。经过组织染色显示,和主要定位在大豆根部表皮和外皮层, 根瘤原基基部的维管束, 成熟根瘤维管束,在根瘤顶部皮层表达(图3-B)。表明大豆GmPIN2蛋白家族成员可能主要调控大豆地下部生长素的转运功能。
2.4 Gmpin2ab-L1和Gmpin2ab-L2双突变体构建
因为大豆GmPIN2a和GmPIN2b氨基酸序列相似性高达96.8%, 且蛋白基序高度相似, 所以本研究利用CRISPR/Cas9基因编辑技术同时敲除和构建双突变体。本研究中设计1条sgRNA, 可同时靶向和的第一个外显子区域, 命名为sgRNA- GmPIN2ab (图4-A)。
T0代稳转苗经过Basta除草剂筛选后获得阳性植株, 再利用载体上Cas9的特异性引物pCas9-F/R进行T0代阳性植株检测, 扩增产物868 bp的片段为阳性转基因植株(图4-B)。利用QRT检测野生型、、和中大豆基因家族表达量发现, 野生型和中基因表达量没有显著性差异, 野生型和、中、基因表达量均具有显著性差异, 而中表达量是野生型的3倍(图4-C)。在T2代进行纯合突变体的筛选, 由测序碱基序列比对结果可知(图4-D),和基因共产生2个位点的突变, 从而获得2种不同突变类型的纯合突变体:(+1 bp)和(-1 bp)。在中,和基因第282位碱基和第283位碱基中间插入了1个碱基A, 使得原来第97位编码丝氨酸(Ser)的密码子AGC变为终止密码子TAG, 导致蛋白翻译提前终止。而在中,和基因第282位碱基A缺失, 使得原来第98位编码亮氨酸(Leu)的密码子CTA变为终止密码子TAG, 导致蛋白翻译提前终止。与、野生型蛋白序列相比,和突变类型均为移码突变并导致蛋白翻译过程提前终止(图4-E)。
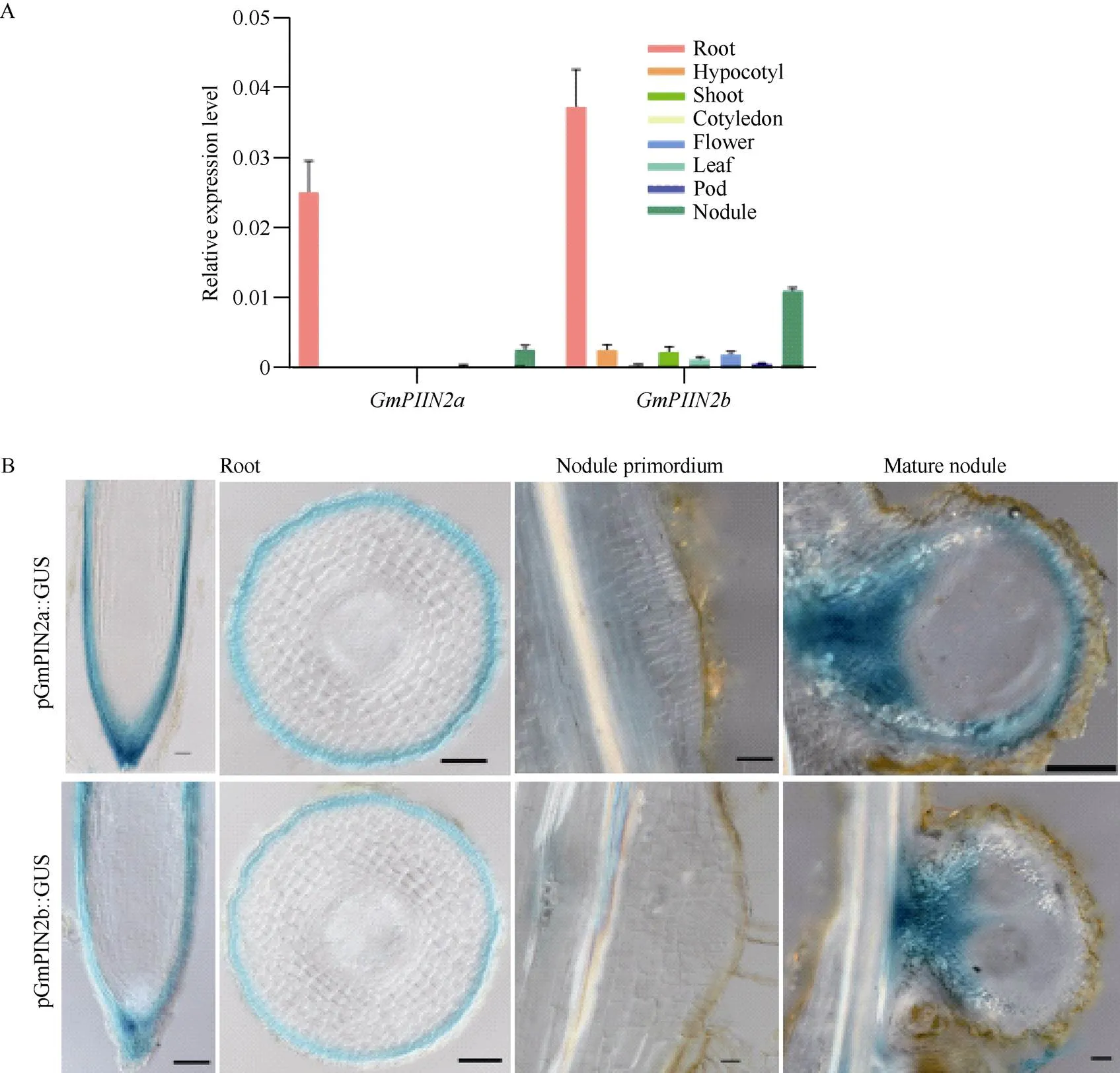
图3 GmPIN2a和GmPIN2b基因组织表达模式分析和组织化学定位
A:和基因组织表达模式; B:和在根和根瘤中的组织化学定位, 标尺为100 μm。
A: the relative expression pattern ofandgenes; B: histochemical localization ofandin root and nodule. Bars: 100 μm.

(图4)
A:和基因结构及sgRNA靶向位点示意图。黑色箭头代表sgRNA位置, 黑色框代表外显子, 红色框代表5′UTR和3′UTR, 黑色线条代表内含子。B:和中基因编辑载体的验证。白色数字从左往右分别表示: 野生型、、和。C: 野生型、、和中大豆家族表达量检测。值由假设方差相等的双尾检验确定, **表示与野生型在< 0.01水平差异显著; ***表示与野生型在< 0.001水平差异显著; ****表示与野生型在< 0.0001水平差异显著。ns: 没有显著差异。D:和纯合突变体序列对比。绿色字母代表sgRNA靶向序列, 红色字母, 红色短线代表增添或缺失的密码子。E: 野生型,和纯合突变体蛋白序列。AA代表氨基酸(amino acid), 黑色箭头表示移码突变起始位点, 紫色框表示移码突变蛋白序列, 数字代表移码的氨基酸数量。
A: the gene structure ofandand target sites of designed sgRNAs. The black arrow represents the sgRNA position, black boxes represent exons, red boxes represent 5′UTR and 3′UTR and black lines represent introns. B: the validation of gene editing vectors inand. White numbers from left to right indicate the wild type,GmPIN2b,,and. C: the relative expression level offamily in wild type,,,and.-values are determined by a two-tailed Student’s-test assuming equal variances, ** indicates that,significantly different from wild type at< 0.01; *** indicates that::significantly different from wild type at< 0.001; **** indicates that,significantly different from wild type at< 0.0001. ns: not significant. D: the sequence alignment ofand.Green letters represent sgRNA targeting sequences; red letters and red dash lines represent added or missing codons. E: the protein sequence of WT,andhomogenous mutants. AA represents amino acid. The black arrow represents the positions of frameshift mutations. The purple box represents the frame-shifting mutant protein sequence. The number represents the number of frameshift amino acids.
2.5 GmPIN2对大豆根系发育调控分析
为进一步研究对大豆根系形态的调控, 本研究同时构建了大豆过表达稳转植株。将野生型、、、在蛭石中萌发7 d, 发现和突变体根部向地上生长, 出现典型的根向重力性缺失表型(图5-A), 这与拟南芥突变体中根部向重力性缺失表型一致[26]。表明大豆调控了大豆根部向重力性。
此外, 通过对突变体, 过表达与野生型7 d幼苗根系扫描图像分析, 突变体较野生型主根弯曲, 侧根表现出不同程度地向上生长趋势(图5-B)。因此, 在调控根向重力性方面, 大豆GmPIN2蛋白家族和拟南芥AtPIN2具有高度功能保守性。通过统计野生型、、和的根长、侧根数、侧根夹角、侧根长、根面积发现,双突变体和过表达植株的主根根长以及侧根数目没有显著性差异。而侧根夹角仅在双突变体中显著上升, 在过表达株系中未有显著变化。双突变体和过表达植株的侧根根长和根面积则较野生型均显著降低。表明缺失或过量表达影响根部生长素转运导致侧根生长发育受抑制。由此推测, 大豆GmPIN2家族蛋白不仅调控了根的向重力性, 并参与调控侧根生长和发育。
3 讨论
植物根系不仅起到固定植株的基本作用, 更是植物吸收水分和营养的重要器官[27-29]。发达的根系对植株完成正常生命活动具有重要意义。根系的生长发育受多种因素调控, 而生长素是根系发育调控的重要因素之一[30-31]。在根系发育过程中, PIN蛋白介导的生长素极性运输在主根, 侧根及根毛发育和根向地性反应过程中扮演了重要角色[32-35]。
本研究中通过进化树分析, 从大豆23个GmPIN蛋白中确定了与AtPIN2高度同源的2个GmPIN2蛋白: GmPIN2a和GmPIN2b。进一步序列分析表明, 大豆GmPIN2蛋白序列与结构与拟南芥、苜蓿、百脉根等双子叶植物PIN2蛋白序列及结构保持高度一致性, 而与单子叶植物水稻、玉米PIN2蛋白序列存在局部差异。尽管单子叶与双子叶植物中PIN2蛋白具有局部差异, 但PIN2蛋白在拟南芥、苜蓿、水稻和玉米中均主要表达于根冠表皮细胞并调控植物根系向重力性反应过程中发挥重要功能, 表明了PIN2蛋白介导的生长素极性运输功能在根向重力反应过程的功能保守性。单子叶与双子叶植物PIN2蛋白基因家族启动子区域含有大量与激素相关调控元件, 还包括与生长发育、光信号相关调控元件, 这表明PIN2基因家族生物学功能广泛, 并且涉及多种激素协调或拮抗调控, 共同调节植物生长发育。本研究中定位分析表明,与其他物种表达部位高度保守, 均在根部根冠表皮细胞中高表达。通过构建双突变体发现, 其不同株系双突变均表现出根部向重力性反应缺失表型, 表明大豆在介导植物根部向重力性反应过程中仍然功能保守。另外,在豆科特异器官根瘤组织顶端表皮细胞中高表达, 推测与根瘤发育过程中顶端生长素转运调控相关。在苜蓿中, 缺失并未导致根瘤数目发生显著变化[35]。大豆中是否在调控根瘤形成过程中发挥重要调控作用仍需进一步研究。
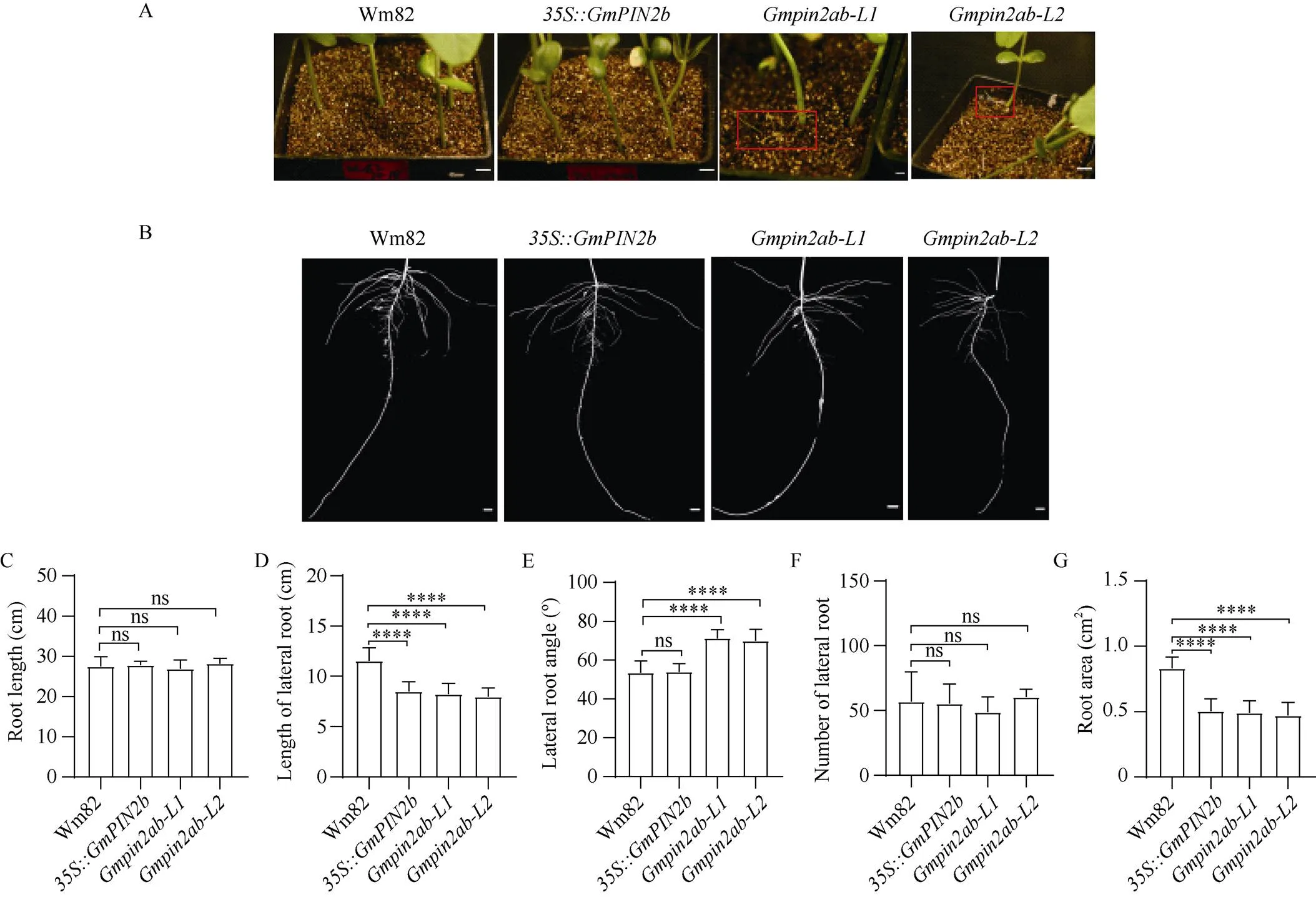
图5 野生型、Gmpin2ab-L1、Gmpin2ab-L2和35S::GmPIN2b植株表型
A:和向重力性缺失表型。红色框表示突变体向地上部分生长的根。B: 生长7 d的野生型、、和的扫描图, 比例尺为1 cm。值由假设方差相等的双尾检验确定, ****表示< 0.0001水平差异显著。ns: 没有显著差异, 样本数为7。
A:andare defective in root gravitropism. The red box indicates that the mutant is growing toward the ground. B: the scanning pattern of seven-day wild-type,,, and, Bars: 1 cm.-values are determined by a two-tailed Student’s-test assuming equal variances, **** indicates< 0.0001. ns: not significant.= 7.
此外, 在多种植物中均报道了PIN2蛋白在根系生长发育的调控作用, 在蒺藜苜蓿中,突变体根部生长素向基运输增加, 侧根数目减少。这说明蒺藜苜蓿在侧根发育过程中起重要作用[36]。在水稻中,基因的突变使根卷曲, 侧根起始发生变化, 过表达显著减少不定根数量和总根长[37-38]。本研究中发现, 缺失和导致根面积和侧根长度显著降低, 侧根夹角显著上升。而过表达则导致根面积和侧根长度显著降低, 而侧根夹角不变。表明缺失或过量表达可能均导致根部生长素向基转运紊乱, 从而均对侧根生长产生抑制作用, 进一步表明介导的生长素极性运输在大豆侧根发育过程中起到重要调控作用。近期拟南芥中报道响应土壤中磷含量, 以及不同N源调控主根和侧根发育[39-42]。而本研究中发现, 缺失和过表达对主根根长均为发生显著变化。是否介导不同N源及磷含量土壤中根系发育仍待深入研究。
4 结论
本研究构建拟南芥和大豆PIN蛋白家族系统进化树发现, 与拟南芥AtPIN2蛋白同一分支的大豆GmPIN2蛋白家族有2个成员: GmPIN2a和GmPIN2b。通过生物信息学分析发现, 双子叶植物和单子叶植物PIN2蛋白可能出现功能分化。进一步研究表明,和在地下部分根和根瘤组织中高表达, 这提示GmPIN2蛋白家族可能参与大豆根及根瘤发育过程生长素转运调控。通过CRISPR/Cas9基因编辑技术创制了大豆突变体:和, 发现突变体根部具有显著的重力缺失表型。与野生型相比,突变体和过表达的根面积和侧根长度显著降低, 但前者侧根夹角显著上升, 后者侧根夹角不变。本研究为深入探究大豆PIN蛋白介导生长素极性运输在根形态建成的作用机制奠定了研究基础。
[1] Peer W A, Blakeslee J J, Yang H, Murphy A S. Seven things we think we know about auxin transport., 2011, 4: 487–504.
[2] Friml J, Palme K. Polar auxin transport—old questions and new concepts?, 2002, 49: 273–284.
[3] Ljung K, Ostin A, Lioussanne L, Sandberg G. Developmental regulation of indole-3-acetic acid turnover in Scots pine seedlings., 2001, 125: 464–475.
[4] Balzan S, Johal G S, Carraro N. The role of auxin transporters in monocots development., 2014, 5: 393.
[5] Krecek P, Skupa P, Libus J, Naramoto S, Tejos R, Friml J, Zazímalová E. The PIN-FORMED (PIN) protein family of auxin transporters., 2009, 10: 249.
[6] Bohn-Courseau I. Auxin: a major regulator of organogenesis., 2010, 333: 290–296.
[7] Saini S, Sharma I, Kaur N, Pati P K. Auxin: a master regulator in plant root development., 2013, 32: 741–757.
[8] Overvoorde P, Fukaki H, Beeckman T. Auxin control of root development., 2010, 2: a001537.
[9] Olatunji D, Geelen D, Verstraeten I. Control of endogenous auxin levels in plant root development., 2017, 18: 2587.
[10] Blilou I, Xu J, Wildwater M, Willemsen V, Paponov I, Friml J, Heidstra R, Aida M, Palme K, Scheres B. The PIN auxin efflux facilitator network controls growth and patterning inroots., 2005, 433: 39–44.
[11] Benková E, Michniewicz M, Sauer M, Teichmann T, Seifertová D, Jürgens G, Friml J. Local, efflux-dependent auxin gradients as a common module for plant organ formation., 2003, 115: 591–602.
[12] Wang H Z, Yang K Z, Zou J J, Zhu L L, Xie Z D, Morita M T, Tasaka M, Friml J, Grotewold E, Beeckman T, Vanneste S, Sack F, Le J. Transcriptional regulation ofgenes by FOUR LIPS and MYB88 duringroot gravitropism., 2015, 6: 8822.
[13] Gao Z, Chen Z, Cui Y, Ke M, Xu H, Xu Q, Chen J, Li Y, Huang L, Zhao H, Huang D, Mai S, Xu T, Liu X, Li S, Guan Y, Yang W, Friml J, Petrášek J, Zhang J, Chen X.-dependent polar auxin transport is involved in soybean nodule development., 2021, 33: 2981–3003.
[14] Sańko-Sawczenko I, Łotocka B, Czarnocka W. Expression analysis ofgenes in root tips and nodules of., 2016, 17: 1197.
[15] Kohlen W, Ng J L P, Deinum E E, Mathesius U. Auxin transport, metabolism, and signaling during nodule initiation: indeterminate and determinate nodules., 2018, 69: 229–244.
[16] Plet J, Wasson A, Ariel F, Le Signor C, Baker D, Mathesius U, Crespi M, Frugier F.-dependent cytokinin signaling integrates bacterial and plant cues to coordinate symbiotic nodule organogenesis in., 2011, 65: 622–633.
[17] Huo X, Schnabel E, Hughes K, Frugoli J. RNAi phenotypes and the localization of a protein::GUS fusion imply a role forPIN genes in nodulation., 2006, 25: 156–165.
[18] Sańko-Sawczenko I, Dmitruk D, Łotocka B, Różańska E, Czarnocka W. Expression analysis ofgenes in root tips and nodules of., 2019, 20: 235.
[19] Xu M, Zhu L, Shou H, Wu P. Afamily gene,, involved in auxin-dependent adventitious root emergence and tillering in rice., 2005, 46: 1674–1681.
[20] Li Y, Zhu J, Wu L, Shao Y, Wu Y, Mao C. Functional divergence ofparalogous genes in rice., 2019, 60: 2720–2732.
[21] Li Z, Zhang X, Zhao Y, Li Y, Zhang G, Peng Z, Zhang J. Enhancing auxin accumulation in maize root tips improves root growth and dwarfs plant height., 2018, 16: 86–99.
[22] 郭安源, 朱其慧, 陈新, 罗静初. GSDS: 基因结构显示系统. 遗传, 2007, 29: 1023–1026.
Guo A Y, Zhu Q H, Chen X, Luo J C. GSDS: a gene structure display server., 2007, 29: 1023–1026 (in Chinese with English abstract).
[23] Chen C, Chen H, Zhang Y, Thomas H R, Frank M H, He Y, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020, 13: 1194–1202.
[24] Xie X, Ma X, Zhu Q, Zeng D, Li G, Liu Y G. CRISPR-GE: a convenient software toolkit for CRISPR-based genome editing., 2017, 10: 1246–1249.
[25] Bai M, Yuan J, Kuang H, Gong P, Li S, Zhang Z, Liu B, Sun J, Yang M, Yang L, Wang D, Song S, Guan Y. Generation of a multiplex mutagenesis populationpooled CRISPR-Cas9 in soya bean., 2020, 18: 721–731.
[26] Luschnig C, Gaxiola R A, Grisafi P, Fink G R., a root-specific protein involved in auxin transport, is required for gravitropism in., 1998, 12: 2175–2187.
[27] Motte H, Vanneste S, Beeckman T. Molecular and environmental regulation of root development., 2019, 70: 465–488.
[28] Raven J A, Edwards D. Roots: evolutionary origins and biogeochemical significance., 2001, 52: 381–401.
[29] Pires N D, Dolan L. Morphological evolution in land plants: new designs with old genes., 2012, 367: 508–518.
[30] Dubrovsky J G, Sauer M, Napsucialy-Mendivil S, Ivanchenko M G, Friml J, Shishkova S, Celenza J, Benková E. Auxin acts as a local morphogenetic trigger to specify lateral root founder cells., 2008, 105: 8790–8794.
[31] Krupinski P, Jönsson H. Modeling auxin-regulated development., 2010, 2: a001560.
[32] Qin H, He L, Huang R. The coordination of ethylene and other hormones in primary root development., 2019, 10: 874.
[33] Du Y, Scheres B. Lateral root formation and the multiple roles of auxin., 2018, 69: 155–167.
[34] Retzer K, Weckwerth W. The TOR-auxin connection upstream of root hair growth., 2021, 10: 150.
[35] Konstantinova N, Korbei B, Luschnig C. Auxin and root gravitropism: addressing basic cellular processes by exploiting a defined growth response., 2021, 22: 2749.
[36] Ng J L P, Welvaert A, Wen J, Chen R, Mathesius U. Theauxin transporter mediates basipetal auxin transport but is not necessary for nodulation., 2020, 71: 1562–1573.
[37] Inahashi H, Shelley I J, Yamauchi T, Nishiuchi S, Takahashi- Nosaka M, Matsunami M, Ogawa A, Noda Y, Inukai Y., which encodes a member of the auxin efflux carrier proteins, is involved in root elongation growth and lateral root formation patternsthe regulation of auxin distribution in rice., 2018, 164: 216–225.
[38] Chen Y, Fan X, Song W, Zhang Y, Xu G. Over-expression ofleads to increased tiller numbers, angle and shorter plant height through suppression of., 2012, 10: 139–149.
[39] Lin D L, Yao H Y, Jia L H, Tan J F, Xu Z H, Zheng W M, Xue H W. Phospholipase D-derived phosphatidic acid promotes root hair development under phosphorus deficiency by suppressing vacuolar degradation of PIN-FORMED2., 2020, 226: 142–155.
[40] Vega A, Fredes I, O’Brien J, Shen Z, Ötvös K, Abualia R, Benkova E, Briggs S P, Gutiérrez R A. Nitrate triggered phosphoproteome changes and aphosphosite modulating root system architecture., 2021, 22: e51813.
[41] Chai S, Li E, Zhang Y, Li S. NRT1.1-Mediated nitrate suppression of root coiling relies on PIN2- and AUX1-mediated auxin transport., 2020, 11: 671.
[42] Zou N, Li B, Chen H, Su Y, Kronzucker H J, Xiong L, Baluška F, Shi W.protects root gravitropism inunder ammonium stress., 2013, 200: 97–111.
Function of GmPIN2 family gene in regulating root development in soybean
LIANG Zheng1,3, KE Mei-Yu1,3, CHEN Zhi-Wei2, CHEN Xu3,*,and GAO Zhen3,*
1College of Life Science, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;2College of Resources and Environment, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;3Haixia Institute of Science and Technology, Horticultural Plant Biology and Metabolomics Center, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China
Auxin is one of the important plant hormones. Auxin transport is involved in the regulation of plant tissue and organ development. In, auxin transport is mainly regulated by PIN family proteins, among which AtPIN2 controls auxin basipetal transport and regulates root gravitation in. However, GmPIN2 family proteins and their functions are still largely unknown in soybean. In this study, we identified twohomologous genes ofandthrough phylogenetic and protein domain analysis. The relative expression level showed thatandwere highly expressed in root and nodule. Both genes were highly expressed in root epidermis and cortex, and the vascular bundles of the root nodule. Onlyexpressed in the epidermis and outer cortex at the tip of the root nodule. Furthermore,mutants were generated by CRISPR/Cas9 (Clustered Regularly Interspersed Short Palindromic Repeats/CRISPR Associated 9) gene editing technology.mutants had a significant loss of root gravitropism. Moreover, the loss ofresulted in a significant decrease of root area and lateral root length, while the lateral root angle ofsignificantly increased. Overexpression ofalso resulted in a decrease in root area and lateral root length, but the lateral root angle unchanged. In conclusion,andplay important roles in regulation of soybean root development via mediating auxin basipetal transport. This study laid a foundation for further analysis of the function and mechanism of GmPIN proteins in soybean root morphogenesis.
(soybean); auxin; PIN protein; root
10.3724/SP.J.1006.2023.14233
本研究由福建农林大学校杰青项目(XJQ201921)资助。
This study was supported by the Distinguished Young Scholar Program of Fujian Agriculture and Forestry University (XJQ201921).
通信作者(Corresponding authors):陈栩, E-mail: chenxu@inbox.com; 高震, E-mail: gaozhen0695@fafu.edu.cn
E-mail: 1879949813@qq.com
2021-12-08;
2022-05-05;
2022-05-13.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220512.1114.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
