多靶点全景数字病理:从原理到应用
2023-01-07张新华李才伟黄胜男吴俊楠任仕杰刘珂含高彤璐
张新华,李才伟,张 瑜,黄胜男,石 晗,吴俊楠,任仕杰,刘珂含,高彤璐,史 冰
(1. 海南省生物医学工程重点实验室, 海南大学 生物医学工程学院, 海南 海口 570100;2. 海南大学 计算机科学与技术学院, 海南 海口 570100)
1 引 言
肿瘤是危害人类生命健康的重大疾病,在世界各国均备受关注。根据肿瘤细胞的不同分子表达以及细胞形态,肿瘤又分为良性与恶性,其中,良性肿瘤并不致病,恶性肿瘤常被称为癌症。恶性肿瘤诊断的金标准即为病理诊断,其为重大疾病的诊断、预后和疗效评估提供客观依据。传统的病理诊断依赖病理医生对病理切片上的细胞和组织学病变进行阅读和分析,从而确定肿瘤类型[1]。然而,目前病理诊断行业面临着病理医生数量少且分布不均、阅片效率低、病理诊断时间长、信息传递困难等问题[2]。
数字病理(Digital Pathology)将计算机、网络会诊和病理诊断相结合,采用技术手段来加快和优化病理实验室的工作流程[3]。1980年,数字病理第一次被应用到临床实践中,但是早期数字病理技术分辨率低,无法准确观察细胞及特征[4]。1997年,Furness[5]等人将万维网(World Wide Web,WWW)和数字病理相结合,实现了病理数据之间的快速传输。2010年,Krupinski[6]等人利用数字面板(Digital Dashboard)技术控制病理检测系统,改善了检测流程,提高了病理诊断效率[7]。近十年,随着人工智能(Artificial Intelligence, AI)技术的复兴,基于AI技术的数字病理逐步实现了肿瘤图像的分割、识别等。2018年,Niazi[8]等人基于迁移学习的Inception v3算法实现了对乳腺癌组织中肿瘤细胞阴性与阳性的自动识别。该方法具有很高的灵敏度与特异性。数字病理从人工阅片转化为计算机阅片[9],该突破有助于计算机辅助诊断(Computer Aided Diagnostic, CAD)和远程会诊的开展,不仅节省了病理学家人工诊断的时间,且避免了主观误诊、漏诊的发生[10]。同时,数字病理解决了时间和地域原因产生的会诊困难问题,提高了病理诊断的准确性和高效性,有助于医疗资源更好地合理化分配[11]。
然而,局部病理图像无法展示病理组织的全貌。全景数字病理(Panorama Digital Pathology)技术通过对整张病理切片进行边界重叠的多次扫描采集,并利用快速精准的图像拼接技术实现了数字病理的全景成像,即全切片图像(Whole Slide Imaging, WSI)技术[12]。早期的WSI技术可以在相对较短的时间内产生较高分辨率的数字图像,同时在多倍率及多焦面上进行全玻片扫描。但由于成像及图像处理技术存在限制,导致全景数字病理图像质量较差[12-13]。因此,许多研究人员一直致力于全景成像的图像处理工作。例如,2007年,Ma[14]等人利用Autostitch软件实现了小鼠淋巴结组织的全景成像;2015年,Legesse[15]等人提出基于乘法校正算法,有效解决了全景成像过程中亮度不均匀问题,实现了头颈部鳞状细胞癌组织的全成像;2022年,Deshpande[16]等人利用SAFRON网络模型有效解决了全景图像处理过程出现的拼缝和伪影问题,实现了结直肠癌组织的全景成像。全景数字病理虽然实现了全玻片上的整张组织切片(单靶点)成像,但是组织病理的形态特征和结构分布复杂,疾病的诊断需要同时分析多个靶点[17-18]。
多靶点全景数字病理技术(Multi-Target Panorama Digital Pathology)可以在单张组织切片上原位检测出多种生物标记物的表达水平,借以识别组织的细胞表型、功能状态及其相互关系,并给出具有统计学意义的细胞组学数据[19]。目前,在多靶点全景数字病理系统开发方面,公司的研发进度展现了一定的优势。Akoya Biosciences、Olympus、Hamamatsu等公司的仪器已经投入到基础、医学研究中。2021年,约翰霍普金斯大学Janis M. Taube团 队 利 用Akoya Bioscience公 司的Vectra 3成像系统构建多光谱图像分析平台Astro Path,对接受PD-1抑制剂的黑色素瘤病人进行了全景多色数字病理分析,实现了黑色素瘤的免疫检查点抑制剂7种标志物的发现和患者生存预测[20]。
多靶点全景病理图像中包含细胞群体间的“空间信息”,分析这些“空间信息”对于理解疾病的病理、发展和预后至关重要。本文主要介绍多靶点全景数字病理的技术原理、技术特点及其在生物医学方面的应用。首先介绍了传统病理到多靶点全景数字病理的发展过程。接下来重点介绍了多靶点全景数字病理图像相关技术,涉及组织样本的准备、光学成像以及图像处理3个方面的相关技术。简单介绍了多靶点全景数字病理在肿瘤微环境以及肿瘤分子分型中的相关应用案例。最后,对多靶点全景数字病理的发展进行了总结与展望。
2 多靶点全景数字病理相关技术
2.1 样本准备
2.1.1 靶点筛选
靶点[21](生物标志物),即在血液、其他体液或组织中发现的生物分子,可以作为正常(或异常)过程(或状况)、疾病的标志(如癌症)。生物标记物可以用来区分肿瘤的性质、确定已诊断癌症患者的预后,预测疾病发展等,且已经在临床上广泛使用。然而,不同疾病或同一疾病不同阶段的靶点也不相同(表1)。合适的靶点对样本标记、定量分析至关重要,在选择时要考虑靶点的功能和表达部位等。
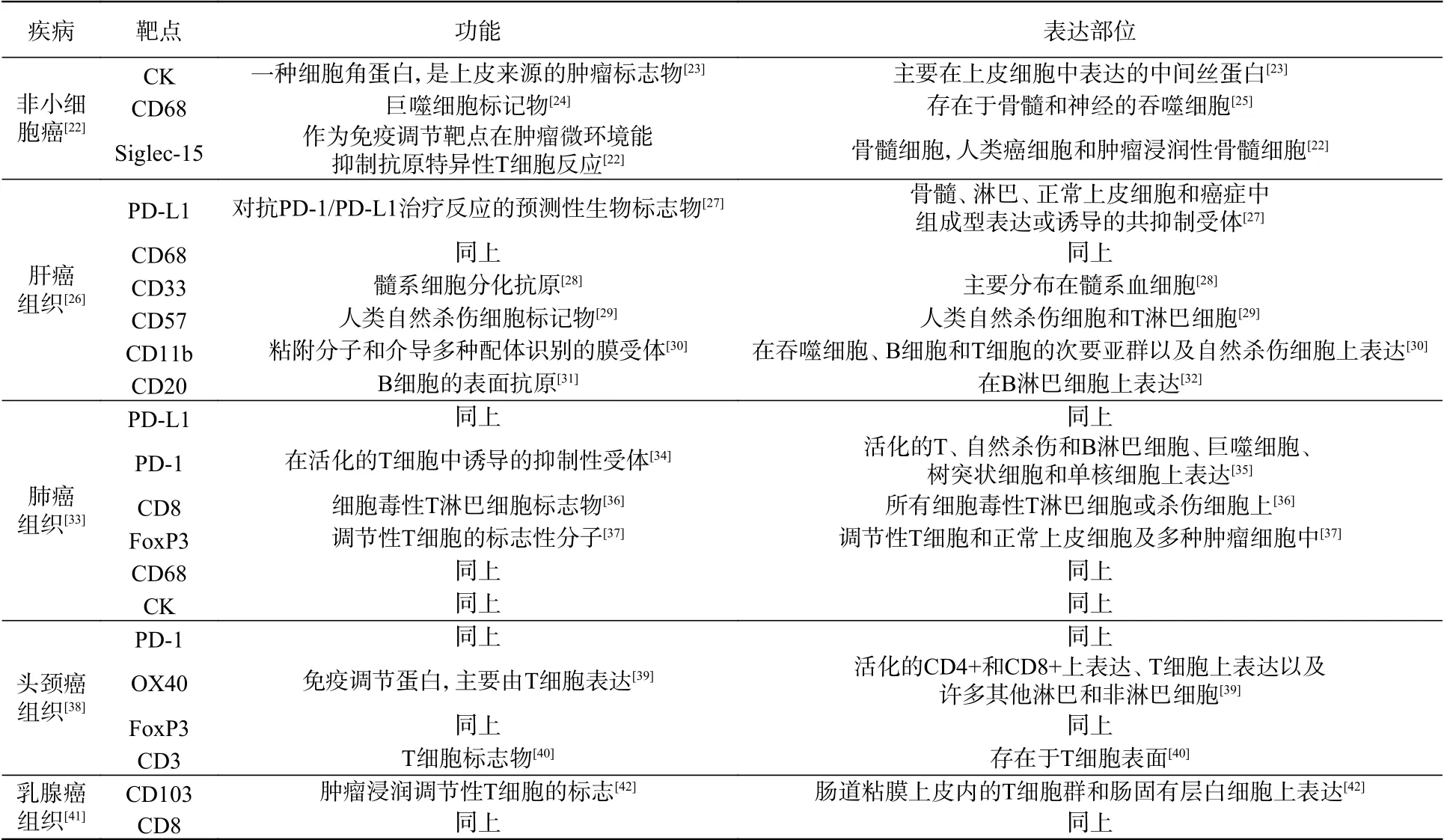
表1 靶点选择原则Tab. 1 Target selection principles
2.1.2 组织处理
病理组织切片是组织学、发育学和病理研究的主要实验方法和重要手段,在科研和临床诊断实践中发挥着重要作用。组织切片机的发明和应用允许精确切割具有一致厚度的切片且引起的损伤最小,生产的组织切片可用于多种分析[43]。首先,经验丰富的技术人员在手术病理检查过程中进行仔细检查,选取病变组织进行切除。切除下病变组织后立刻送到冰冻切片机中,加入包埋剂后冰冻,制成冰冻切片进行观察保存。检查后剩余的冰冻病理组织也可再制成石蜡切片,经固定等步骤后观察保存[44]。
病理组织切片主要包括两种,石蜡切片和冰冻切片。石蜡切片[45]需将提前确定好的新鲜组织用固定液处理,使组织块硬化并维持细胞形态。接着用乙醇或丙酮等脱水剂去除组织内的水分,进行浸蜡和包埋,使组织变硬便于切成薄片,最后用石蜡切片机处理,并置于载玻片中烘干保存[46]。冰冻切片则无需固定材料,直接取材速冻并加包埋剂,观察到组织出现白色冰体后使用冰冻切片机切割即可。制片观察后需保存于-80 °C冰箱内[47]。
2.1.3 组织标记
(1)免疫组织化学/免疫荧光
实验研究中,为了能够清楚地观察到组织或细胞样本中的结构特征和成分分布,需要借用荧光基团或呈色反应定位的染色方式,使目标区域直观呈现可视化效果。病理诊断研究中使用最广泛的方法是免疫组织化学(Immunohistochemistry,IHC)[48]。该方法将酶或荧光基团与抗体偶联作为标记物,能够在单张病理切片中进行某一种蛋白质的原位识别,并定位其分布情况。其中使用的荧光基团与抗体偶联方法又称为免疫荧光(Immunofluorescence, IF)[49]。IHC/IF可以表征肿瘤免疫微环境内特定的细胞种类、密度和空间分布特征,实现半定量分析。当需要检测多种蛋白质时,通常使用多张组织切片,对每张切片中的每种蛋白质单一标记,较为依赖病理学家的主观判断[50]。
常规的IHC/IF普遍采用间接标记方法,其流程如图1所示。首先,对样本上的抗原进行预处理并充分暴露后,选用封闭液将非特异性结合位点封闭。在基于辣根过氧化物酶的检测系统中,还应对内源性过氧化物酶进行阻断,降低非特异性反应可能。其次,加入特异性识别抗原表位的鼠源或兔源的单克隆一抗试剂进行孵育。最后,使用抗对应一抗物种来源的多克隆二抗试剂进行孵育,使二抗与一抗结合[51]。该方法能够有效放大反应信号,并且基于二抗制备的易得性和通用性可大大降低实验成本。标记完成后,要进行复染和封片操作用于成像观察和样本保存。

图1 常规免疫组织化学实验流程Fig. 1 Conventional immunohistochemistry staining procedures
(2)多重免疫组织化学/免疫荧光
多重免疫组织化学/免疫荧光(multiplex Immunohistochemistry/Immunofluorescence, mIHC/IF)[52]
技术的出现使得免疫细胞的亚群、丰度、功能状态以及它们在肿瘤微环境中的空间分布特征等信息更易被量化。该技术主要是对一张病理切片上的多种抗原靶点进行不同的抗体标记。除了酶和荧光基团,抗体还能与量子点[53]、DNA条形码[54]等物质进行偶联。
在mIHC/IF中,单片多重免疫组化连续染色(Multiplexed Immunohistochemical Consecutive Staining on Single Slide, MICSSS)[55]方法最早被用于组织的多重染色。其单一抗原染色原理和普通IHC一致,区别在于每次染色后均需要对组织进行化学洗脱,需要进行几种抗原的标记。该方法的问题在于,在逐轮的化学洗脱过程中,不同抗原信号可能在洗脱后减弱或者去除,因此还需要对标记流程进行优化。为了避免信号的大量损失,以及对低丰度靶标的检测过程进行研究,研究人员提出了基于酪胺信号放大(Tyramide Signal Amplification, TSA)[56]的间接标记法。该方法基于与二抗偶联的辣根过氧化物酶检测系统,在酶催化H2O2过程中,大量酪胺被激活形成共价结合位点,酪胺上连接着荧光基团、显色染料、生物素等标记物,然后与抗原周围的氨基酸残基形成牢固的共价键,实现检测信号数倍甚至几何级倍数的放大。加热可以除去抗体并保留共价结合的酪胺沉积。其流程如图2所示。重复染色操作可以得到多标记的病理切片,实现同时多色成像。TSA还可以与MCISSS相结合实现倍增的多靶点标记[57]。

图2 基于TSA的多重荧光免疫组化流程Fig. 2 Multiplex fluorescent immunohistochemistry based on TSA
(3)染料选择
根据实验需求,选择染料时应考虑理化性质、细胞毒性、色原或荧光强度、稳定性等因素。常用的染料有两种:一种是针对细胞器和细胞区室的特异性染料,如与DNA强力结合的荧光染料DAPI[58];另一种是基于抗原-抗体特异性反应的免疫染色法所需染料,将能够在成像系统中显色的物质与抗体偶联,并通过间接标记的手段来放大信号[59]。
明场成像和荧光成像时的染料选择方法也各有侧重。选择明场下可见的染料时,不需要特定可视化工具和软件便可评估,但是进行同时多色成像时,需要考虑肉眼是否能够准确区分这些颜色。选择荧光染料时,应考虑以下因素:(i)每种荧光染料具有不同的激发和发射光谱,需要根据实验室拥有的激光器进行选择,并考虑组织自发荧光带来的影响;(ii)同时多色成像时,尽量避免所选染料发射光谱重叠,防止出现光谱串扰;(iii)尽量避免选择易光漂白的荧光基团或采用抗淬灭剂,光稳定性较好的染料包括Alexa Fluor系列[60]、DyLight系列[61]、ATTO系列[62]等。此外,偶联抗体的染料在多靶点成像时需要考虑抗体之间的交叉反应。多次复染时的循环洗脱步骤中,需要考虑不同抗原的敏感性和损失程度,从而制定合适的实验方案。
2.2 光学成像技术与设备
在显微成像技术基础上发展的多靶点全景成像技术,主要包括基于分时或分光形式的多色成像技术、基于快速扫描的全景成像技术以及高通量成像技术几类。目前,国内外发展了许多成熟且适合用于多靶点全景成像的光学成像设备。下文将主要介绍用于多靶点全景成像技术的光学成像技术与设备。
2.2.1 关键技术
(1)多色成像技术
多色成像主要是利用探针信号的发射荧光波长差异,来实现对不同探针的荧光信号进行目标区分。目前常用的多色成像技术主要有分时多色、分光多色以及光谱成像几类。
分时多色成像技术[63]是通过依次采集发射波长不同的荧光探针信号,以获取时间序列相关的原始图像帧。该方法原理简单,但由于不同颜色通道图像的采集时间存在间隔,难以避免样品漂移,这需要后期图像处理的配准。相比较而言,分光多色成像技术[64]是采用滤色片组将被同时激发的不同探针荧光信号反射或透射到不同探测器或同一探测器的不同区域以实现多色成像。该方法虽然解决了分时成像技术中的样本漂移问题,但由于信号发射光谱分布可能存在频域上的重叠,使得荧光分子在密度较高、强度差异较大的时候存在严重的信号串扰问题。为了避免荧光信号串扰问题,这两种多色成像方法通常需要选择发射光谱中心间隔远的探针进行成像,因此,往往只能区分2 ~ 4种颜色[65]。
然而,数字病理需要观察多种细胞的形态、功能状态并研究他们之间的相互关系,这需要对组织进行多靶标观察,而2 ~ 4种颜色的多色成像技术很难满足数字病理的多色成像需求。为此,研究人员常采用光谱成像方法来实现对多靶标的成像。就图像获取方式而言,常用的光谱成像方法有扫描式和非扫描式两类;就图像光谱获取方式而言,光谱成像又可以分为滤光片光谱解析、干涉仪光谱解析以及棱镜等色散元件的光谱解析方法[66-68]。例如,Hess研究组[69]利用光学棱镜进行光谱解析,如图3所示。在该成像系统中,通过分光镜将荧光分为两路,其中一路图像记录荧光分子的位置信息,另一路荧光信号被棱镜或光谱相机进行光谱拆分以获取对应信号的全光谱信息,最终实现荧光分子的位置信息和光谱信息的同时检测,并利用光谱信息实现探针荧光信号的多重成像[70]。该方法能够识别荧光光谱重叠较严重的荧光团,能够实现对组织样本上多靶标信号的探测,很好地解决了传统分时和分光多色成像方法中颜色识别能力不足的问题。近年来,随着半导体技术的不断发展,各类新型快速可调滤光片器件相继问世,如液晶可调滤光片(Liquid Crystal Tunable Filter, LCTF)[71]、声光可调滤光片(Acousto-Optic Tunable Filter, AOTF)[72]。这些新型滤光片器件正推动着光谱成像技术向图像获取速度更快、光谱解析更精细的方向发展。另外,随着光电探测器制造相关技术的发展,探测器量子效率不断被提高,像素阵列不断扩大,这也让光谱成像技术的空间分辨率以及成像速度得到极大提升[73]。

图3 光谱成像示意图[68](F:滤光片;BS:分光镜;M1/M2/M3:反射镜;P:色散棱镜)Fig. 3 Schematic diagram of spectral imaging[68] (F: filter;BS: beam splitter; M1/M2/M3: mirror; P: dispersive prism)
利用光谱成像技术,Alex等人完成了对6种不同膜结合细胞器的相互作用研究[74];Janis M.Taube团队实现了肿瘤免疫疗法中6个标记物的多靶标全景成像[20]。
(2)全景成像技术
受限于成像设备的视场大小,一张全景病理图像的获取,需要对切片上众多单视场区域进行扫描成像,再根据各个视场之间的重叠部分进行多视场图像的融合[75]。其中,全景数字病理成像技术中的扫描方式主要有3种(图4)[76]。

图4 面扫描和线扫描[77]Fig. 4 Area scanning and line scan[77]
一是基于走停模式的面阵扫描:在成像过程中,载物台带着切片进行走停等待的移动操作,移动操作中相机采集每个区域的荧光图像。采用该方法成像时,每切换一个成像视场都需要重新对焦。但是,该方案中每次对焦都需要花费数秒时间,而一张大小为15 mm×15 mm的切片一般需要扫描上千个视场,因此,若采用基于走停模式的面阵扫描至少需要数十分钟才能完成一张切片的扫描成像[78]。为解决对焦时间长的问题,研究人员提出了许多改进方案[79],如焦面建模方案。该方案中,研究人员会预先选取切片上的部分区域进行焦面探测并建立模型,在成像时根据建模结果直接对焦。此方法只需花费约150秒就可以实现一张15 mm×15 mm切片扫描成像[77]。
二是基于线阵扫描的成像方法:将切片划分成若干相同大小、具有部分重叠的条带区域,成像系统根据焦面建模方案进行对焦;成像过程中,做匀速运动的载物台带动病理切片在线照明条件下与线阵传感器同步曝光,以实现对条带区域扫描成像;扫描完当前条带区域后移动至下一个条带,直至全切片扫描完成。在线扫描成像过程中不需要每个视场切换的走停等待,且图像拼接过程所需的计算量随着拼接视场区域减少而降低,因此成像速度较快[77]。例如滨松公司的Nano Zoomer采用的时间延迟积分线阵传感器能够增强图像信号,使得等效的曝光时间更短,同步扫描速度也更快,在40倍放大倍率下对15 mm×15 mm的病理切片进行成像只需45秒[80]。
三是基于线扫描成像的面阵扫描成像:该方法结合了以上两种方法的优点,将切片划分为若干区域,在每个区域分别采用线扫描进行图像采集。该方法能够在每个子区域进行更精确的对焦操作,同时解决了走停模式间歇时间长的问题,加快了扫描速度。目前,采用此设计的扫描成像仪主要有3D histech公司的Pannoramic 250 Flash III系统,该系统在40×放大倍率下对15 mm×15 mm的切片进行成像的耗时少于60秒[81]。
实现多靶点全景成像的必要前提是将上述多色成像技术和全景成像技术相结合。在上述3种多色成像技术中,分时多色方式目前在多色数字病理检查设备中使用较少。利用滤光片组进行分光全景多靶点成像技术不仅需要考虑设备与染料的组合,通过降低不同荧光波段的串扰,以获得高质量图像;而且需要通过优化滤光片组滤除无用荧光和不匹配的信号干扰,降低颜色串扰。基于光谱成像的全景多靶点成像技术有多种方法可以实现分光。例如,基于分段式元件或可调谐滤光片器件的方法,每次成像只能检测单个波段的信息,采集时间会随着检测波段的增加而相应增加。目前常用方法是滤光片阵列,即将多波段滤光片组集成到探测器的传感单元上,同时探测多个波段信息,但由于滤光片尺寸限制,目前最多只能检测4个成像波段。以上方法都需要考虑以下问题:(i)将多色技术和全景技术结合时,要考虑复杂光学系统的搭建;(ii)在成像过程中需要高精度高速率的控制系统。由于近年来光学元器件和现代控制技术的进步,全景多靶点成像技术通过对组织样品的快速成像,获得了组织样本病理信息特征。
(3)高通量成像技术
实现病理切片的高通量成像能力,提升病理设备的应用能力,是数字病理技术应用推广的关键[77]。为此,许多研究人员针对提升成像通量的方法进行了深入的研究与探索。得益于虚拟显微镜技术[82]和高速自动化系统的发展,研究人员目前已经开发出了一系列高通量全景数字病理成像扫描仪,在实现高通量成像方面取得了长足的进步[12]。数字病理切片扫描仪是一种集合光学、计算机科学、控制学、机械等诸多学科知识技术的产品,主要包含虚拟显微镜软件系统、光学成像系统、自动化玻片载物台系统、图像探测系统、光源照明系统等组件[77]。其中自动化玻片载物台是高速自动化成像系统的重要组成部分,高精度载物台对保证成像质量与提高扫描成像速度至关重要[83]。
总体而言,提高成像通量的关键是提高成像系统的扫描速度。在这方面,利用高精度位移台进行线扫描或连续面扫描成像的方式能够极大地提高成像速度。若配合快速对焦方法,系统能在数十秒的时间内实现对整个切片的全景成像。其次,病理切片成像结束后减少新切片的装载时间也是提高组织全景成像通量的重要环节。现今的数字病理扫描仪可自动快速重新装载切片样本,有效地避免了人工操作引入的失误并减少了人工操作时间。还有部分设备可进行多批次处理,能够实现单次装载就可实现数十至数百张切片的自动扫描成像[75],极大地提高了病理切片的扫描成像通量。例如,Olympus公司开发的研究级全玻片扫描系统VS 200(图5)采用多托盘加载器方案,能够实现单次装载210片,在明场20倍放大倍率下单张切片成像所需时间约为80秒[84]。

图5 Olympus VS200 研究级全玻片扫描系统[84]Fig. 5 Olympus VS200 research-grade slide scanner[84]
2.2.2 全景病理商品化设备
全景病理成像技术目前已经较为成熟,许多厂商生产的全景数字病理成像设备已经被广泛用于生物学、病理学、组织形态学等相关的科学研究及医疗诊断过程中。这里,将介绍几种商品化的全景病理成像设备,并对其性能参数进行对比,见表2。
表2中列出了目前科学研究和临床医疗中使用的大部分全景数字病理扫描成像设备。Zeiss公司的Axio Scan.Z1全自动数字玻片扫描系统可实现样本的明场及荧光成像。明场模式下,利用20倍放大倍率成像单张切片(15 mm×15 mm)所需时间为240秒。该系统的荧光成像模式最高可实现9色成像,且一次最高可装载100张玻片,采用滤光片式的分光多色方法的数字病理设备有Olympus、Zeiss等品牌。Zeiss公司的Axio Scan.Z1全自动数字玻片扫描系统拥有明场、荧光以及偏振光3种成像方式。该系统的荧光成像模式采用高灵敏sCOMS探测器,配合多个滤光轮可高速切换通道,最高可实现9色成像,且一次最高可装载100张玻片,能够快速获取高通量数据[85]。Olympus公司推出的全玻片扫描系统VS200[84]的成像模式多达5种,并可进行不同成像模式的组合观察。该系统使用电动滤光片转轮配合不同染色方案实现多靶点全景成像,并能够支持不同规格的载玻片。不同成像模式的组合能够更好地满足研究人员不同成像需要。采用光谱成像方式的光谱型成像设备有Akoya公司的Vectra系列和Polaris系列等。Akoya Biosciences公司的Vectra3自动定量病理成像系统采用多光谱成像方式,多路复用能力最多可分离7种颜色,结合inFORM®软件分析系统,能够检测肿瘤微环境中多个弱表达的生物标志物[20]。

表2 全景数字病理设备产品主要参数Tab. 2 The main parameters of panoramic digital pathology equipments
2.3 图像处理技术
基于多靶点全景高通量成像系统,可以获得多幅局部多色病理图像。在同一幅多色病理图像中包含多个靶点的病理信息,因此,首先要对多色病理图像进行颜色解析,然后利用多色拼接算法对多个子区域图像进行拼接构成全景数字病理图像并可视化,最后对多靶点全景数字病理图像进行定量分析,找出多靶点病理之间的关联。
2.3.1 图像重建
在当前常用的光谱成像方法中,多光谱成像显微镜系统先捕获多光谱图像,再由多个图像平面组成多层图像“立方体”,这些平面通过光谱来对应于液晶可调谐滤波器所选择的波长。然后,使用逆最小二乘拟合将多重染色样本的图像解混。解混分离每个荧光团的自荧光和重叠发射信号,从而去除自荧光背景,产生各个信号特定的“成分”平面[20],完成多色病理图像多色解析(靶点解析)。
靶点解析后的图像是小视场多色荧光图像,利用图像拼接技术提取相邻小视场多色图像重叠部分的特征点,通过匹配相邻图像的重叠区域的特征点,将多张小视场多色图像拼接成一张全景多色图像。图像拼接流程如图6所示。

图6 全景数字病理图像拼接流程Fig. 6 Mosaic process of panoramic digital pathological images
图像配准和图像融合是直接影响图像拼接性能的两个步骤。空间域中图像配准算法一般分为基于区域的图像配准和基于特征的图像配准。对于基于图像区域实现图像配准的方法,Plattard[86]等提出互信息法,通过重叠区域的联合熵来评判两张图像的信息相互包含程度,根据联合熵便可以实现图像的配准。基于图像特征的图像配准,提取具有特殊性质的特征点集作为匹配依据,常用的特征检测器有Harris、FAST(Features from Accelerated Segment Test)、SURF(Speeded-Up Robust Features)、SIFT(Scale-Invariant Feature Transform)。由于Harris会产生大量密集角点,FAST难以区分边缘点和噪声点,特征点配准方法中SIFT和其改进算法SURF[87]应用最为广泛。SIFT通过尺度空间筛选尺度和旋转不变兴趣点,根据图像局部区域的梯度方向对进一步筛选的关键点赋予方向,然后用一组向量对特征点及其邻域像素点信息进行描述。基于不同图像融合方法,拼接算法分为平滑过渡和最佳拼缝,基于平滑过渡拼接的方法可进一步分为基于羽化、金字塔和基于梯度的拼接[88]。
2.3.2 数据存储及可视化
医院扫描产生的图像数据量很大,拼接后一张全景图像大小就为800 M ~ 1 G。以南方医科大学南方医院采购医用设备招标项目为例,要求数字化病理扫描系统指标如下:放大倍率分别为20×和40×,持续产出量达到7小时400片[89]。在医院中每年将产生数百万张病理医学图像,年存储增量可达TB级。因此,在多靶点全景数字病理研究中要重点考虑病理图像数据存储及可视化方式。
病理数据存储策略需要考虑持续增长的容量需求,如兰乌特勒支大学医学中心病理系通过建立标准的数字病理存储系统实现全面数字病理[90]。其存储方式主要采用分层策略:第一层为低延迟、高带宽存储系统,其将为虚拟存储集群(操作系统、数据库)和数字切片提供本地短期存储。第二层是存储于网络连接(Network Attached Storage, NAS)上的全院系统,具备高度可扩展的存储容量(最高达PB级),数字图像在归档后将存储在该层。
全景图像可视化视野范围大,存储信息多,一次可实现整张全景图像可视化,但处理时间久,通常不能进行实时可视化。在查看大视场图像时,由于计算机屏幕的限制,不能一次性加载最高分辨图像。目前的可视化采用多分辨率层次金字塔模型,主要是根据原始图像构建图像序列,序列中的每个图像称为一个层,每一层都是原始图像不同分辨率下的图像。利用该方法每次可视化只需按需加载部分图像数据,不仅可以减少内存加载,而且可以解决文件存储和带宽之间的矛盾,实现快速实时可视化,方便医生浏览整张全景图像。
2.3.3 定量分析
(1)预处理
图像质量对后期定量分析很重要,处理不当会导致病理图像定量分析准确性下降。降低图像质量的原因可能有疾病异质性、切片制备、图像采集过程条件不一致等。采用预处理方法可以在一定程度上减少误差,如组织伪影检测、图像去噪、染色标准化、增强图像对比度等[91]。其中,组织伪影检测与染色标准化是常用的预处理方法。正确检测组织学图像中的组织和伪影是保证图像质量的前提。常用组织和伪影检测方法有滤波器与阈值化相结合[92]、语义分割算法[93]、特征提取分类器[94]等。染色标准化是图像处理的基础,Massimo Salv等人总结了三种染色标准化策略,一是全局颜色归一化:首先从模板图像中提取全局信息(例如RGB直方图、亮度),然后映射到源图像。二是染色分离后的颜色归一化:根据模板图像的颜色分布,分离单个染料的权重以改变原始图像。三是利用深度网络的颜色转移:采用风格转移方法将源图像的染色风格更改为模板风格[95]。接下来将从组织形态识别、细胞表型检测、空间距离分析方面定量分析多靶点全景病理图像。
(2)组织形态识别
在组织病理分析过程中,病理学家通常通过评估细胞核的多形性和细胞结构的空间排列来区分正常组织、非恶性和恶性病变[96]。针对某些疾病的病理诊断需求,以淋巴细胞识别、有丝分裂数目检测、细胞核大小识别为例,从整张病理切片中识别并分离出上述细胞或组织结构以进行数字病理分析。
传统方法主要依赖于数字图像处理技术,采用基于各种特征描述符的手工特征提取方法,例如根据颜色、纹理、形状和组合描述符等实现细胞或组织的识别和分类。2013年,Ciresan[97]等人首次使用滑动窗在数据集中截取大量小样本,然后采用卷积神经网络(Convolutional Neural Network, CNN)分类模型对样本做有无有丝分裂的二分类, 以检测有丝分裂数目。Chen[98]等人使用基于分离色彩通道的卷积神经网络生成细胞概率响应图,并在响应图中自动检测免疫细胞。回归/分类框架将对象识别视为一个回归或分类问题,采用统一框架直接获得最终结果(类别和位置)。
(3)细胞表型检测
细胞表型是多个基因和蛋白表达的细胞过程的集合体,决定了细胞特定的形态和功能。了解细胞表型对于了解癌症进展和免疫治疗反应的机制至关重要。将细胞内成分检测技术与多色免疫荧光分析方法相结合,可检测不同细胞亚群合成的细胞因子,如对淋巴细胞亚群和白血病免疫表型分析[99]。
目前有些学者构建了专业的数字图像分析软件,结合机器学习算法完成细胞表型检测。如Koelzer[100]等人使用多重IHC和数字图像分析软件对PDL1表达进行识别和计算定量,然后利用随机森林算法实现免疫细胞的分类和定量分析。Sun团队开发了新型计算工具Scissor,可以在单细胞数据中识别出与给定表型相关的细胞亚群[101]。Scissor首先通过量化每个单细胞和每个大量样品之间的相似性,整合与表型相关的大量表达数据和单细胞数据,然后优化与样本表型相关的矩阵回归模型,以识别相关亚群。基于深度学习(Deep Learning, DL)的图像分析也被广泛用于多靶点图像定量分析中的细胞表型检测。Harder等人使用高精度多分辨率配准方法和基于网络架构GooglieNet的卷积神经网络来区分黑色素和IHC标记的阳性免疫细胞[102]。
(4)空间距离分析
在组织细胞表型的基础上计算不同细胞之间的空间关系,进而对细胞之间的相互作用做出推测,也可以计算出组织交界面两侧的细胞密度,对细胞的浸润程度进行分析。如观察免疫T细胞与肿瘤细胞的聚集倾向和接触范围、判断不同样本组之间的免疫效果差异等。
将多染色面板和开源图像分析软件相结合用以实现免疫细胞的定量分析。Oguejiofor K等人通过Vectra自动多光谱成像系统[103]、Nuance FX多光谱成像系统软件和inFORM®高级图像分析软件实现了对肿瘤浸润淋巴细胞(Tumor Infiltrating Lymphocytes, TILs)与人乳头瘤病毒肿瘤状态和患者生存率之间的关系分析[104]。Failmezge[105]等人提出了拓扑肿瘤图(Topological Tumor Graphs,TTG),其基于自动图像分析提供的细胞空间映射,将每个细胞视为一个节点,如果它们在空间上接近35 μm,则绘制细胞之间的边缘,由此可得出细胞类型之间的空间相互作用。Barua等利用Gcross空间距离分布方法实现了每种细胞类型丰度和空间位置以及它们彼此之间接近度的分析,该方法可以在微米级半径范围内找到至少一个任意给定类型的免疫细胞,验证了肺癌细胞和特定免疫细胞在肿瘤微环境中的空间邻近性预后意义[106]。
3 多靶点全景数字病理应用
多靶点全景数字病理技术可以标记检测多种细胞结构和环境,通过分析位置分布和监测病理特征,有助于进一步了解细胞组织间的相互作用及功能。多靶点全景数字病理利用免疫荧光成像技术对细胞内部蛋白质分子等的分布状态进行精确观测,解决了传统检测手段面临的检测时间长、成像视野小、分辨率不足等问题,推动了医疗及生物应用的后续研究和发展。多靶点全景数字病理在生物医学领域的应用主要包括肿瘤微环境、肿瘤分子分型等。
3.1 肿瘤微环境
肿瘤微环境由癌细胞、恶性上皮细胞和多种基质细胞等构成,癌细胞的增殖、侵袭和转移等生物学行为受到宿主基质细胞特性的影响[107],因此,微环境的相互作用与癌细胞致病过程密切相关。在发掘生物标志物和潜在药物靶点上,肿瘤微环境利用其机制特性,为后续研究提供关键帮助[108],肿瘤微环境如图7(彩图见期刊电子版)所示[109]。

图7 肿瘤微环境[109]Fig. 7 Tumor micro environment[109]
利用肿瘤微环境中细胞结构及组织组成之间层次调节的特征,调控细胞对疾病的反应。但是一些蛋白标记物独特地分布于亚细胞体积中肿瘤块的不同部分,可以对这些蛋白标记物进行定向检测。非小细胞肺癌研究利用Akoya多色免疫荧光技术,对新辅助治疗前后的肿瘤免疫微环境进行7色多靶标标记,对肿瘤免疫微环境的免疫细胞浸润进行量化分析,验证了治疗前后肿瘤免疫微环境的动态变化[110]。目前已有的研究在如何构建肿瘤微环境上仍然有许多无法克服的问题,如大视场成像、分辨率等,而多靶点全景数字病理可以通过对肿瘤微环境标记出多个靶点,进行多方位的组织成像,成功定位细胞、产物的位置分布,有利于研究肿瘤微环境多个层次的异质性。Obradovic等人利用多靶点定量免疫荧光验证了异质性疾病肾透明细胞癌(Clear Cell Renal Cell Carcinoma, ccRCC)微环境中特异性的TREM2/APOE/C1Q上调巨噬细胞亚群,这一关键发现可作为预测肾透明细胞癌复发的潜在生物标志物[111]。
3.2 肿瘤分子分型
1999年美国癌症研究所提出了肿瘤分子分型的概念,即从形态学领域转向分子水平对肿瘤进行系统划分,其主要是通过分子水平表达差异(如抗体表达、免疫标志物)进行分型[112]。21世纪以来,各个团队也一直致力于研究肿瘤分型。2006年,Hu等利用微阵列数据针对乳腺癌分子建立了一个新的亚型预测因子,基于此,提出了一种新型的乳腺癌分子型,为肿瘤前瞻性提供了一种客观方法[113]。在此基础上,利用多靶点方法对肿瘤区域不同分子表达进行标记,进一步使用全景数字病理对肿瘤分子分型进行精确划分。Helmink等利用多色免疫荧光技术对三级淋巴结构进行多靶点标记(如图8(彩图见期刊电子版)所示),针对细胞内的生物标志物进行开发从而对其中的免疫细胞进行精确分型[114]。
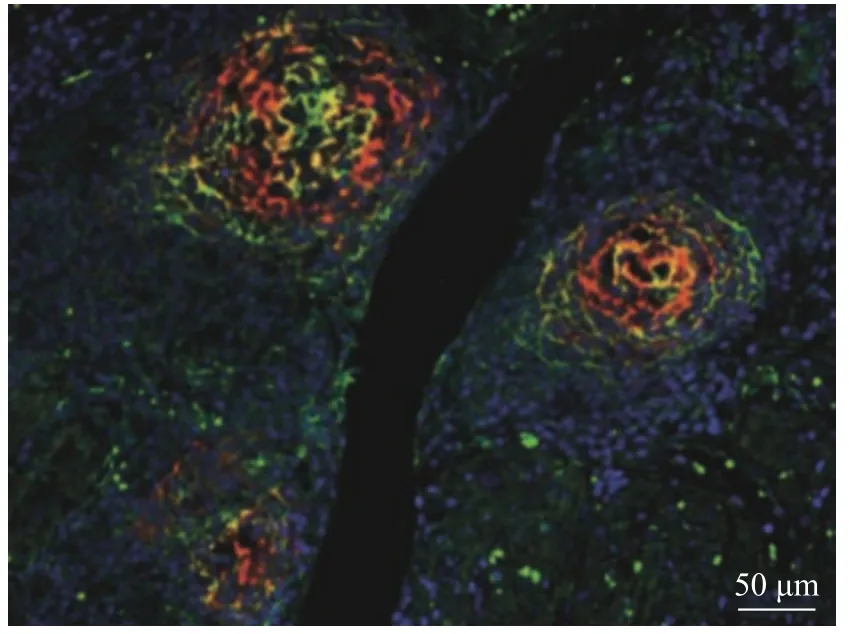
图8 三级淋巴结构免疫标记荧光成像[114]Fig. 8 Immunolabeling fluorescence imaging of tertiary lymphoid structures[114]
使用全景成像技术将玻片上的样本进行扫描采集成像生成图像数据集,并对这些数据集图像进行拼接,在大视场和高分辨率条件下观察多色免疫荧光染色情况[20],进而依据免疫标志物的差异从分子水平进行肿瘤分子分型。除此之外,多靶点全景数字病理技术可以根据肿瘤组织原位来测定不同免疫细胞亚群(如B细胞、T细胞)表型、状态及相互关系[115],为肿瘤分子分型提供分析数据和依据,进而为临床上个性化治疗制定策略和方案[111]。
4 结束语
多靶点全景数字病理是病理学和形态学研究的重要手段,其揭示了细胞组织之间相互作用以及功能变化。本文从基础技术和生物医学应用两方面介绍了多靶点全景数字病理。在基础技术中介绍了多靶点全景数字病理的生物样本处理、光学成像和图像处理3方面相关技术:(1)生物样本准备,使读者了解如何选择靶点、处理组织样本和样本标记,从而获得多靶点标记的组织样本;(2)光学成像,讲述了光谱解析的多色成像原理,介绍了面扫描与线扫描的全景成像技术,以及虚拟显微镜技术和高速自动化系统的结合实现了大视场高通量光学成像技术;(3)图像处理,介绍了如何通过颜色解析和图像拼接实现全景多靶点图像并定量分析多靶点之间的关联表达。在应用方面简单介绍了多靶点全景数字病理在肿瘤微环境和肿瘤分子分型方面的研究。这些研究有助于揭示细胞组织间的相互作用及功能变化,有助于推动多靶点全景病理图像在生物医学领域的研究。
虽然多靶点全景数字病理技术已被应用于生物医学领域,但是它的发展还不成熟,仍然有许多待完善的地方。例如在选择靶点方面,可以结合多靶点专家知识库,从而获得针对某种疾病更合适的靶点;在实现全景扫描成像方面,可以通过优化复杂的光学成像系统进而实现更高性能的“多色、全景、高通量”光学成像;在图像处理方面,可以结合深度学习来实现颜色解析和图像拼接,进而为定量分析提供高质量的图像数据,通过优化算法减少数据处理时间,实现数据实时可视化,方便病理医生浏览整张全景图像完成病理诊断等。
然而,通过调研该领域目前的研究现状以及分析实现多靶点全景数字病理的主要影响因素,多靶点全景数字病理的发展将面临的一些关键技术挑战如下:样本多标记免疫荧光染色时如何解决抗体冲突问题[116];光谱成像时重复信号串色干扰问题[117];定量分析时,如何解决定量分析标准化等问题[116]。这些关键问题的解决,需要进一步的深入研究。
